脑肿瘤的发病率越来越高, 严重危害人类的健康生活. 核磁共振成像(MRI)作为一种典型的非侵入式成像技术, 为脑肿瘤的诊断和治疗做出了突出的贡献. 卷积神经网络(CNN)被广泛地应用于医学图像中主要是因为固有的感应偏差, 可以实现特征的自动学习, 获取局部感受野等优点[1]. 在语义分割中实现模型精度跨度比较大的是FCN[2], 它全部由卷积组成. 基于FCN, Ronneberger 等人[3]提出了一种新颖的模型架构, 成为后来很多优秀模型的基准模型, 即U-Net. 它是由编码器和解码器, 以及编码器和解码器之间的跳跃连接组成, 跳跃连接实现了不同层次特征的融合, 促进上采样特征图细节特征的恢复. ResNet[4]中的残差结构, 将输出特征与原始特征相结合, 解决了深层网络降级的问题. 研究者们致力于找到效率高, 计算量和参数量小的分割方法. 群卷积、逐点卷积和深度可分离卷积在轻量级模型中起到主要的作用. 为了在有限的计算资源上实现高效的计算, ShuffleNet[5]利用逐点卷积和通道混洗的操作大大减少了计算量, 这对小规模网络精度的提升具有重大意义. ResNeXt[6]是一个多分支的结构, 通过简化分组卷积来降低模型的计算量. MobileNet[7]将普通的卷积替换为深度可分离卷积, 在精度不变的情况下, 使得模型的参数量降低.
因此, 训练一个参数少、计算量低并且简单高效的卷积神经网络是主流的发展方向.
在医学图像领域利用Transformer[8]来处理图像数据, 已经取得了巨大的成功, 这主要归功于Transformer对数据建模的远程依赖性, 能够实现特征的可扩展性, 高效性等. 从Vision Transformer (ViT)[9]和Swin Transformer[10]提出以来, 在医学图像中应用比较多的CNN和Transformer的混合模型[11–14], 将Transformer的变体与CNN相结合. 但是这些混合网络相对于CNN, 计算复杂度要高得多, 并且需要将图像转化为图像块的序列, 破坏了图像的空间结构, 为了获得感应偏差, 需要大量的数据进行训练, 这些缺点对于医学图像数据稀少的特点, 以及医学图像密集分割的性质来说是不利的.
为了在利用Transformer结构进行改进的同时保持卷积固有的感应偏差, ConvNeXt[15]重新考虑卷积神经网络在图像方面的作用, 用卷积神经网络来模拟ViT和Swin Transformer 的结构特点, 利用大的感受野和特征缩放行为来改进卷积神经网络, 并且在更简明的操作方法下, 实现和Transformer最先进的模型相当甚至更高的水平. 但是ConvNeXt只是应用在自然图像这些2D图像中. 显然, 自然图像与脑肿瘤图像无论是数据维度还是数据量方面都存在很大的不同. MedNeXt[1]将ConvNeXt块扩展到上采样和下采样的过程中, 构建了第1个完全ConvNeXt的U型结构. 在本文中, 我们设计了残差ConvNeXt模块, 将Transformer的远程表征学习与CNN的感应偏差相结合. 并且改变了传统的U型结构, 提出了一个非对称3D U型卷积神经网络用于MRI脑肿瘤分割.
1 方法本节详细介绍所提出的非对称U型卷积神经网络ASUNet. 首先, 介绍总体的架构组成. 其次, 介绍用于下采样的残差ConvNeXt模块. 最后介绍解码器架构.
1.1 提出的ASUNetASUNet是一个具有编码器解码器结构的轻量级模型. 如图1所示, 不同颜色的长方体表示不同的功能模块, 每个长方体上面的信息表示特征图的尺寸和通道数. ASUNet是由收缩路径, 跳跃连接和扩展路径组成. 从图中可以看出, 首先, ASUNet是一个不对称的U型结构, 编码器的数量要多于解码器的数量. 为了防止在下采样的过程中特征图尺寸缩小的幅度过大导致特征细节信息严重丢失, 保持最后一个下采样特征图的尺寸不变. 此外, 将最后两个编码器的输出在通道级别上进行连接, 通道数量的增加有助于特征更详细的表示, 这种连接方式可以保存上一阶段网络的一部分信息从而保证信息的有效性. 其次, 与传统的U型网络在下采样过程中使用步长为2的残差连接进行特征提取不同的是, 我们在下采样部分采用残差ConvNeXt模块进行特征提取, 对特征图在通道和感受野两个方面进行缩放和扩展, 减少特征信息在下采样过程中不必要的丢失问题. 然后, 在上采样之后, 采用
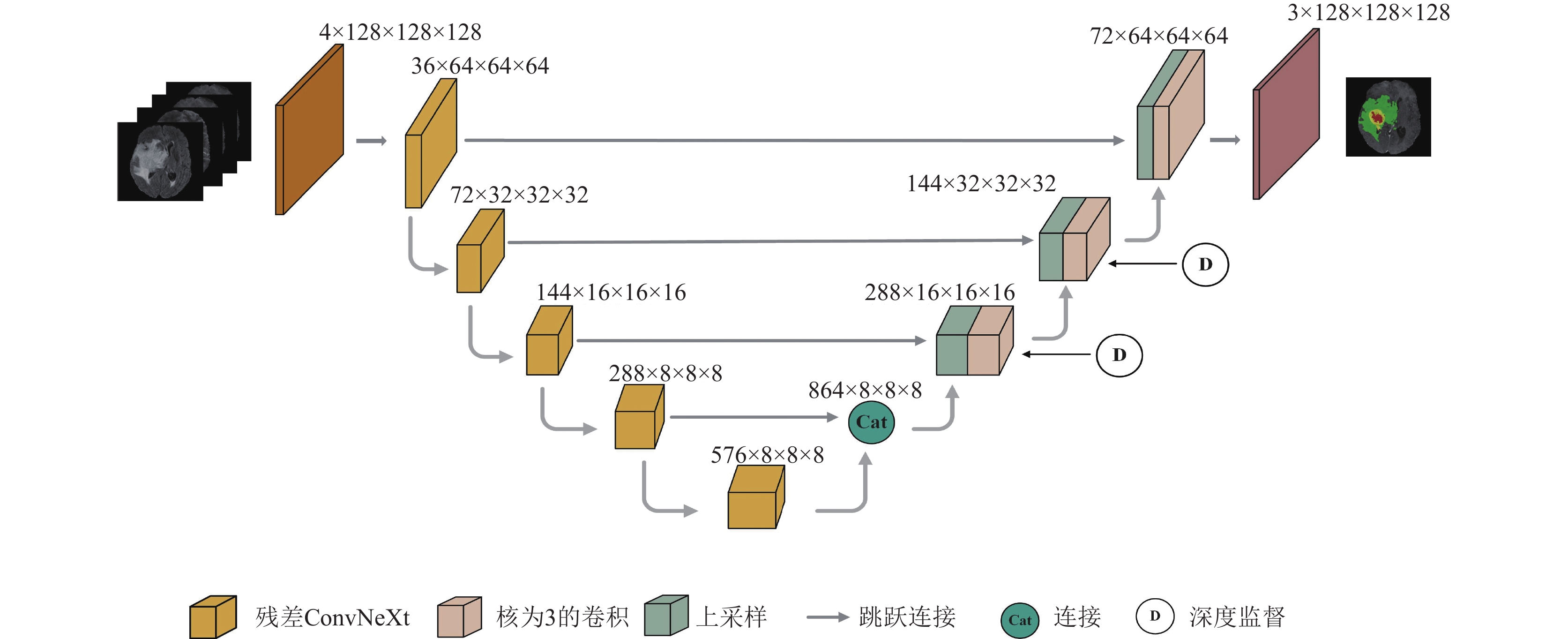
|
图 1 提出的ASUNet架构 |
1.2 残差ConvNeXt 模块
在下采样的过程中我们设计了残差ConvNeXt模块, 丰富了下采样过程中的语义特征信息, 有利于密集语义分割任务. 我们对传统的瓶颈结构进行改进, 传统的瓶颈结构组成如图2(b)所示. 除此之外, 如图2(c), MedNeXt在下采样的过程中也采用了具有残差结构的MedNeXt模块, 但是采用将主路径与捷径连接直接相加的方式, 并且在捷径连接中采用卷积核大小为1. 我们所提出的残差ConvNeXt根据ResNet的设计特点, 将主路径与捷径连接相加, 然后送到激活层. 为了将不同感受野所提取的特征进行融合, 在捷径连接中采用了卷积核大小为3的卷积. 通过第2.6.1节的实验, 发现ResNet的残差连接方法性能更好, 更适合于医学图像分割. 改进后的残差ConvNeXt模块如图2(d)所示, 主要分为3部分: 深度卷积层、扩张层、收缩层. 从图2中可以看出, 第1层深度卷积层是由核大小为K的深度卷积(DW)组成的, 采用深度卷积层, 有效地降低了参数的数量. 如图2(a)所示, 在Swin Transformer的多头自注意力(MSA)中, 所采用的滑动窗口大小至少为7, 并且在ConvNeXt模块中采用核大小为7的卷积. 通过第2.6.1节的实验表明, 当
总之, 采用深度卷积是在空间维度上混合信息,
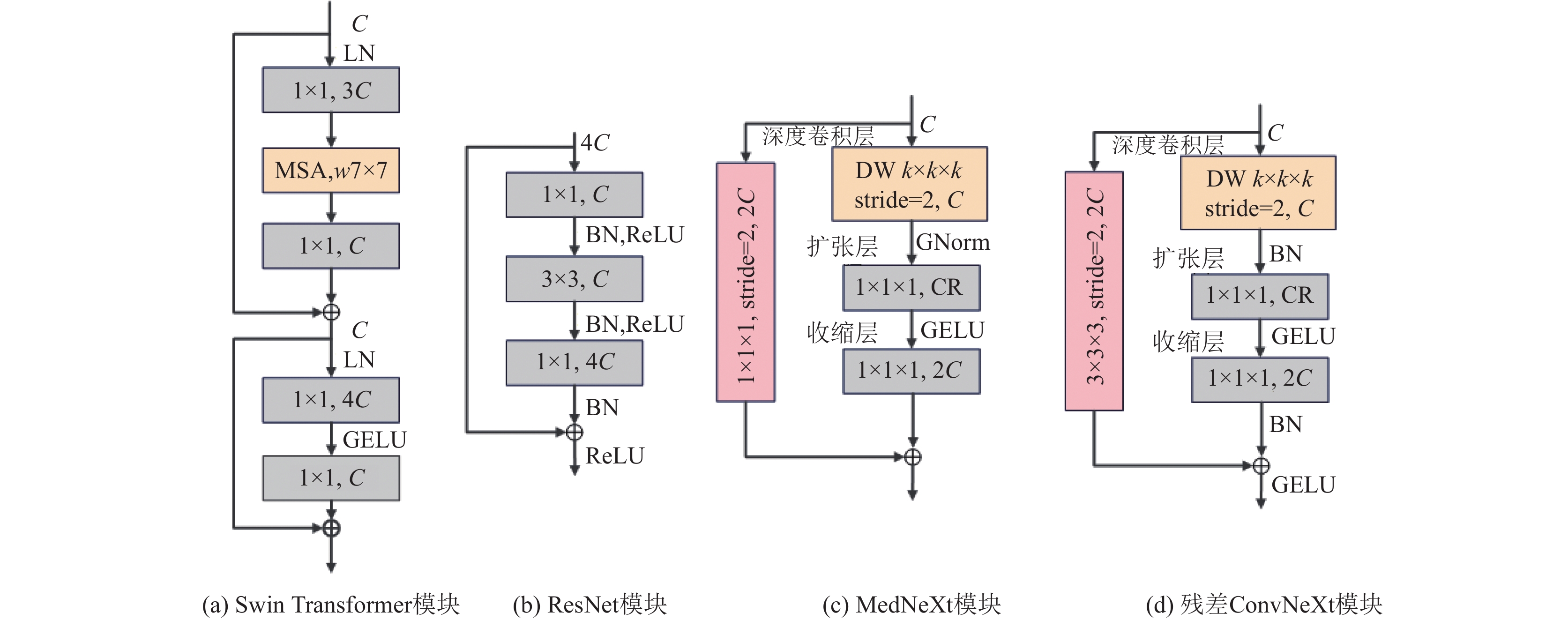
|
图 2 4种模块详细结构 |
1.3 解码器结构
解码器中的上采样模块主要包括转置卷积、批量归一化(batch norm, BN)和ReLU激活函数, 在上采样的过程中, 首先通过转置卷积逐渐恢复特征图的分辨率, 转置卷积减少了网络扩展情况下的特征损失, 同时实现了降维, 并且逐步降低特征图的通道数. 在转置卷积之后, 采用BN和ReLU激活函数, 加快模型的收敛. 在上采样模块之后, 采用
在本实验中, 使用BraTS 2020和FeTS 2021数据集进行实验, 这两个数据集都是由MICCAI脑肿瘤分割挑战赛提供的完全注释的公开数据集, 从不同患者中利用不同的MRI扫描仪所获得的, 这些MRI图像都是严格对齐的, 重采样至1×1×1 mm的各向同性分辨率, 并剥离颅骨[16]. MRI数据集通常包括4个序列: T1加权序列(T1)、T2加权序列(T2)、T1加权增强序列(T1ce)和FLAIR序列(FLAIR). 脑肿瘤组织在不同序列中表现不同, 每一个序列的作用都是为了突出观察脑肿瘤内某一个组织的位置, 大小和形状, 并提供脑肿瘤的互补信息, 便于分割. BraTS 2020由训练集和验证集组成, 其中训练集是由369个病例组成, 验证集包括125个病例. FeTS 2021训练集包括341个病例, 验证集有111个病例. 每一个病例的MRI图像都是由4个模态组成. 训练集提供事实标签, 主要包含4类: 背景(标签0)、坏死(标签1)、瘤周水肿(标签2)和增强的肿瘤(标签4). 而我们的评估指标是针对这4类标签组合而成的3个子区域进行评估, 分别是: 标签4组成的增强肿瘤区域(ET)、由标签1和4组成的肿瘤核心区(TC)、除了背景区域外的整个肿瘤区域(WT). 验证集并没有提供真实标签, 我们将训练好的模型对验证集进行分割, 然后将分割结果上传至CBICA在线评估平台, 通过这个官方平台来获得预测分割图的准确程度.
2.2 实现细节本文的模型使用PyTorch实现, 在3台NVIDIA RTX A5000上训练了
为了避免由于脑肿瘤的数据量比较稀少而导致的过拟合问题, 首先本次实验将MRI脑肿瘤图像进行了随机裁剪, 将图像尺寸由
由于脑肿瘤在整个MRI图像中占比非常小, 在预测的过程中出现了很多评分为0的数值, 这是假阳性导致的结果, 所以在预测的过程中, 使用测试时间增强的技术(TTA), 将验证集输入图像进行水平, 垂直, 对角等多个角度进行7次翻转, 然后分别送入训练好的模型进行测试, 测试完成的7次分割结果再分别进行相对应的角度翻转, 翻转回原来的角度. 我们将翻转后的7次分割结果与原始图像的分割结果求取平均值得到最终的分割图. 通过使用TTA技术, 可以对验证数据集进行数量以及特征的增强, 很大程度上解决了假阳性的问题, 并且模型精度得到了提升.
2.2.2 损失函数损失函数是为了测量真实分割与预测分割之间的偏差, 在实验中使用软骰子损失函数直接优化重叠度量[17], 产生了比通常使用的加权损失函数更好的精度, 这个值在
| L(G,Y)=1−2JJ∑j=1∑Ii=1Gi,jYi,j∑Ii=1G2i,j+∑Ii=1Y2i,j | (1) |
其中,
我们采用骰子相似系数(Dice)和豪斯多夫距离 (Hausdorff)两个评价指标来测试模型的分割精度, 这些指标是针对WT, TC和ET来计算的, 所以我们不根据提供的事实标签
| Dice=2TPFN+FP+2TP | (2) |
豪斯多夫距离目的是测量预测的分割结果和真实分割图之间的距离差距, 这个数值越小说明预测结果图越接近真实分割图. 豪斯多夫距离被定义为:
| Hausdorff95=max(maxp∈P(95%)maxt∈T‖p−t‖,maxt∈T(95%)maxp∈P‖t−p‖) | (3) |
其中,
本节将展示ASUNet在BraTS 2020验证集上与其他先进的模型在骰子分数和豪斯多夫距离上的比较. 在BraTS 2020验证集上, ASUNet通过实验在ET、WT和TC上分别取得了77.08%、90.83%、83.41%和24.13 mm、4.92 mm、12.28 mm的Dice分数和豪斯多夫距离. 表1中的数据对比表明, ASUNet在WT和TC上表现出了较大的优势. 此外, 为了验证ASUNet和真实分割图(GT)直观上的差异, 我们将训练集随机划分为4:1, 将1/5的训练集抽出GT作为测试集, 剩余的数据集用于训练, 将训练好的模型来预测测试集的数据. 由于测试集是训练集的一部分, 所以GT是可以获得的, 进而比较预测分割结果图与GT之间视觉分割的差异. 可视化结果如图3所示, 其中前4列表示MRI脑肿瘤的4个模态, GT表示脑肿瘤的真实分割图, 最后一列是所提出模型的预测分割图. 从图3可以看出, ASUNet的分割预测结果与GT是相近的, 进一步证明ASUNet模型的有效性.
2.4.1 与基于Transformer和CNN的混合模型进行比较在基于Transformer和CNN的混合模型中, TransBTS[12]结合CNN来提取局部特征信息, 然后将提取的局部特征信息送入Transformer中实现全局信息建模, 相比于TransBTS, ASUNet在ET的分割上低了1.65%, 这主要是由于TransBTS同时在深度维度和空间维度上有效的建模局部和全局特征, 促进特征信息的充分表示. 但TransBTS的参数量高达32.99M, 比ASUNet的参数高出2.7倍. UNETR[18]方法采用ViT作为编解码器. ASUNet是一个完全基于卷积的网络, 在捕获多尺度上下文信息方面存在不足, 导致对具有不同形状和尺度的脑结构的次优分割. 在TC的分割上, 豪斯多夫距离相比于UNETR差了1.65. SwinBTS[19]将Swin Transformer作为编解码器并使用CNN进行上下采样来获得最终的分割结果. Peiris等人[20]利用自我注意和交叉注意设计了一个新颖的Transformer架构.
| 表 1 在BraTS 2020数据集上将所提出的模型与先进的BraTS方法进行比较 |
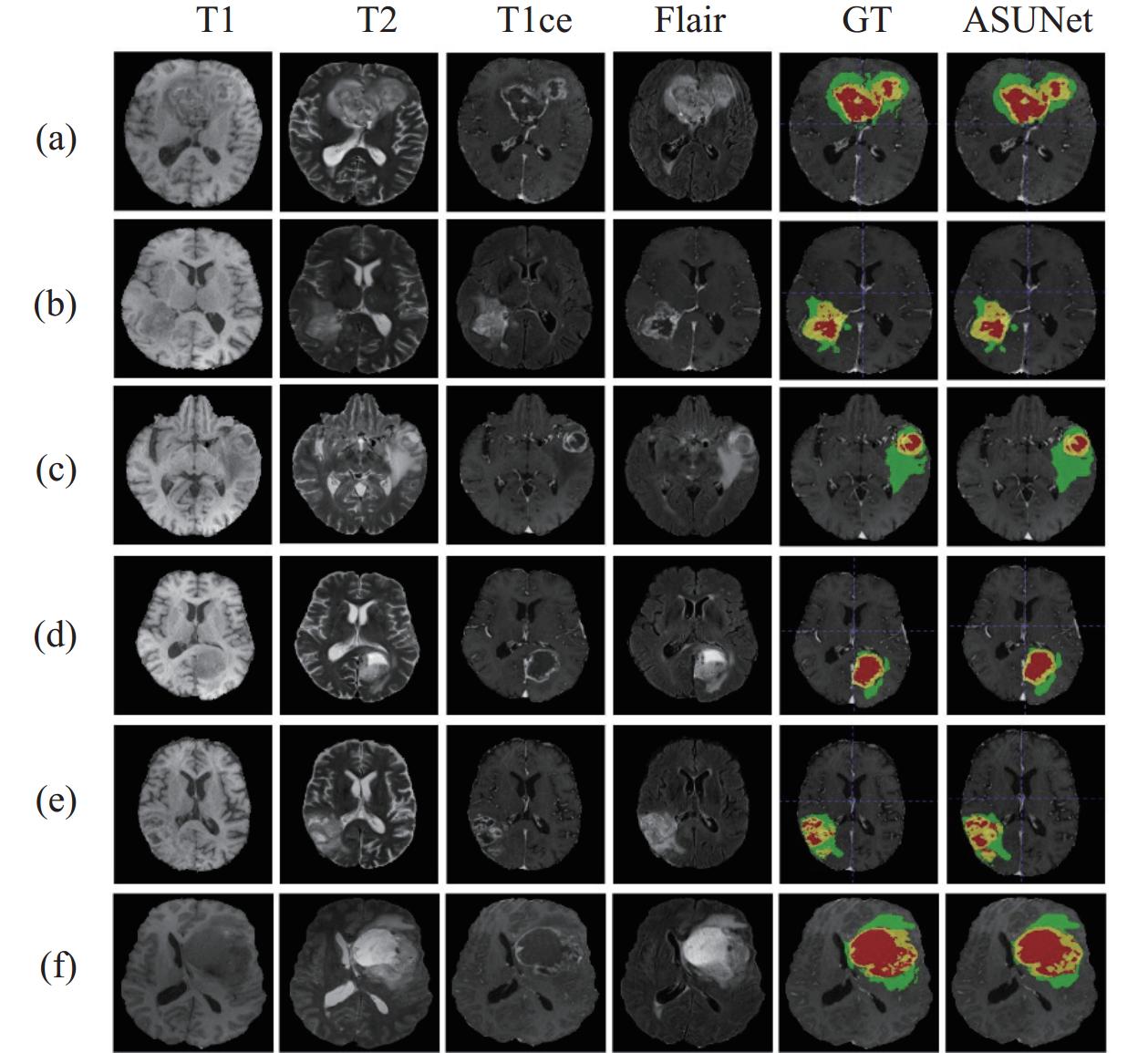
|
图 3 在BraTS 2020数据集上的视觉分割结果 |
2.4.2 与基于CNN的模型进行比较
在基于CNN的方法中, CH-UNet[21]提出了角注意模块分别在3D MRI脑肿瘤的3个轴方向上提取信息. Point-UNet[22]设计了一种高效的上下文感知点采样机制; Point-UNet在ET分割上的Dice得分, 相比于ASUNet高出了1.9%. 但是在WT和TC上分别比ASUNet低了1.12%和0.66%, 并且模型复杂度要高于ASUNet. Akbar等人[23]提出将atrous卷积和注意力进行有效的结合. DR-Unet104[24]使用104层的卷积层和瓶颈残差单元, 但是使用的2D卷积, 对于3D脑肿瘤图像的结构细节信息并未完全获取, 导致在ET和TC的较小区域分割上效果较差, 仅为67.56%和67.21%. Attention-UNet[26]将注意力与U-Net进行结合. 该注意力模块产生的门控信号有效地抑制了输入图像的不相关信息同时突出特定局部区域的相关特征. ASUNet通过跳跃连接直接将低级语义信息和高级语义信息相连接的方式, 并不能充分发挥低级语义信息在上采样的作用, 这就导致了ASUNet模型在ET小区域图像的分割性能较差, 在ET的Dice分数上低了4.99%. 但是ASUNet对远程依赖关系进行有效的建模, 并且结合卷积固有的感应偏差, 在WT和TC上获得了比较好的分割效果.
2.5 在FeTS 2021上的实验结果展示本节在FeTS 2021数据集上进行实验来验证该网络的泛化性能. 如表2所示, 展示了ASUNet与其他先进模型在骰子分数和豪斯多夫距离上的比较. 其中, Khan等人[27]提出两种权重聚合方法, 在FeTS 2021挑战赛中获得了第2名. 观察表2, 可以发现该方法在ET上获得了最佳的豪斯多夫距离, 但它的骰子分数在3个脑肿瘤子区域中是最低的. Hu等人[28]提出了一种基于集成学习的方法, 但它可能导致模型过拟合. 从表2可以看出, 该方法在ET上的骰子得分很差, 比ASUNet低了4.1%. Pawar等人[29]介绍了一种两阶段的方法用于脑肿瘤精确分割, 细化了脑肿瘤区域的边界位置信息. 与其他最先进的模型相比, 实现了最佳性能, 但需要训练2次, 这就导致2倍的参数量和计算量, 而且相比ASUNet, 在ET、WT和TC上分别低0.5%、1.2%和0.7%. 观察表2, 可以明显发现, ASUNet模型在所有脑肿瘤子区域上的Dice分数明显优于其他最先进的方法. 虽然ET的豪斯多夫距离不是最好的, 但WT和TC的豪斯多夫距离明显优于其他最先进的方法. 综上所述, 在BraTS 2020和FeTS 2021数据集上的实验结果表明, ASUNet表现出较强的泛化能力.
| 表 2 在FeTS 2020数据集上将所提出的模型与先进的BraTS方法进行比较 |
为了直观地说明ASUNet和GT预测分割结果之间的差异, 使用与BraTS 2020相同的数据集划分方法来预测ASUNet的分割结果. 图4显示了ASUNet预测分割图与真实分割图之间的视觉比较. 从图4可以发现, ASUNet的分割结果与GT非常相似, 同时进一步表明了其对FeTS 2021数据集的适用性.
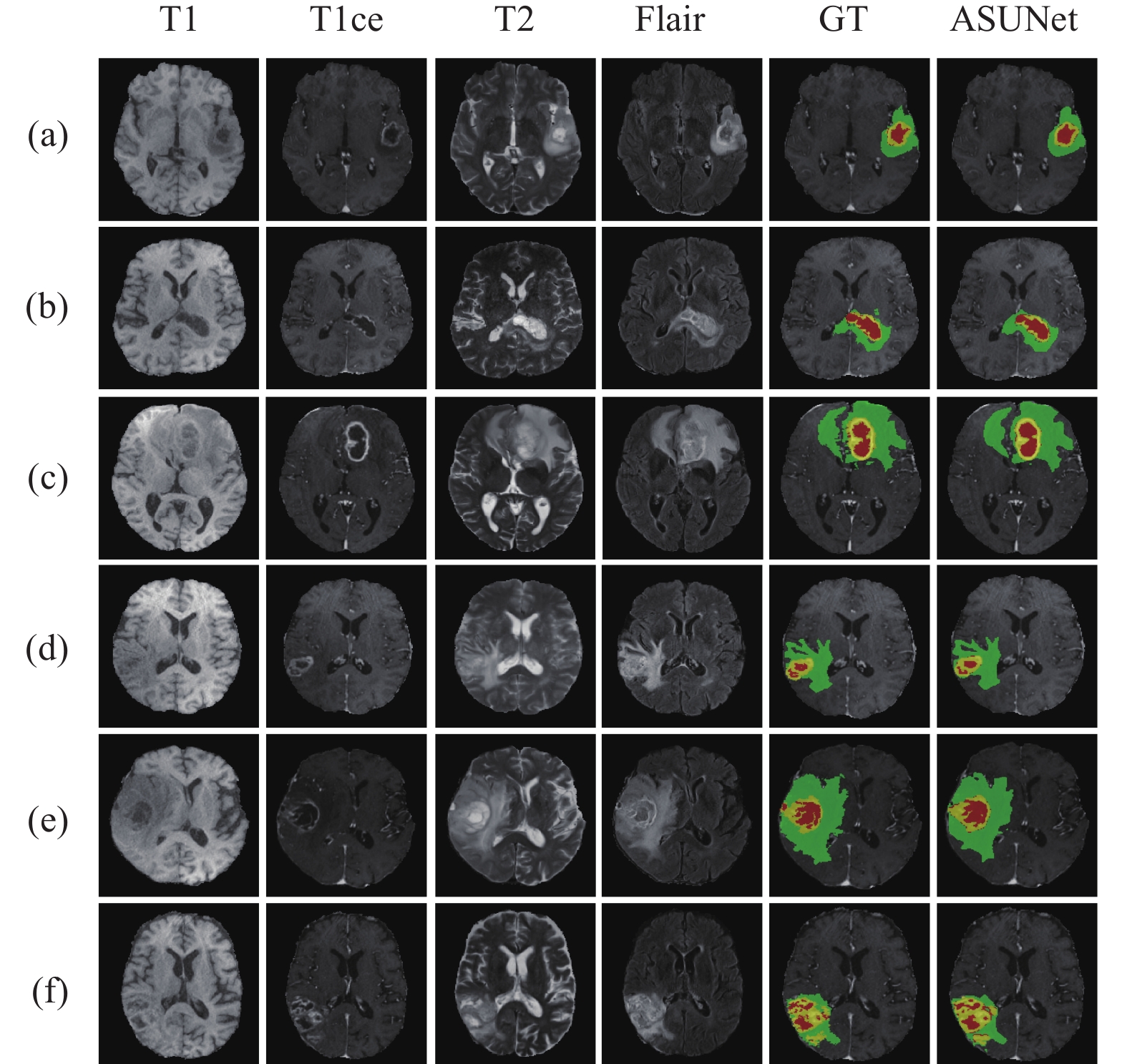
|
图 4 在FeTS 2021数据集上的视觉分割结果 |
2.6 消融实验
为了更好地说明ASUNet中的每一个模块发挥的重要作用, 本节将利用BraTS 2020数据集进行消融实验来验证. 我们分别评估使用残差ConvNeXt模块进行下采样的有效性, 对比U型结构不同设计对实验结果的影响, 以及有无后处理、有无深度监督对实验结果的影响.
2.6.1 不同下采样策略对模型的影响本节通过采用不同的下采样策略来验证残差ConvNeXt模块的有效性, 所对比的下采样方法是步长为2的残差结构, 配置如图5所示, 其中C表示通道数, Stride表示步长. 在下采样中, 采用步长为2的残差结构, 以一种常规卷积的方式降低特征图的尺寸, 增加特征图的通道数, 但是忽略了下采样过程中特征细节丢失的问题.

|
图 5 用于下采样步长为2的残差结构 |
从表3中的实验数据可以明显发现, 当采用步长为2的残差连接时, ET和TC的Dice得分非常低, 分别为71.41%、81.00%. 并且从参数的数量可以发现, 残差ConvNeXt模块极大地降低了参数的数量, 大约是传统残差结构的1/2. 残差ConvNeXt模块利用特征尺寸和通道的缩放和扩展, 不仅保持了下采样过程中语义信息丰富性, 而且以一种计算代价小的方式建立了远程依赖关系, 保持了卷积固有的感应偏差.
| 表 3 在BraTS 2020验证集上对不同的下采样策略进行Dice值的对比 |
表3也显示, 使用残差ConvNeXt进行下采样在ET、WT和TC的Dice分数上提升了5.67%、0.61%和2.41%. 在残差ConvNeXt模块的深度卷积层, 不同的K值产生了不同的分割结果, 从表3还可以看出, 虽然残差ConvNeXt模块采用K=5时, 在ET上获得了最高的Dice分数, 但是当采用K=7时, 在WT和TC上的分割效果是最好的, 因此在深度卷积层, 我们将K设置为7.
2.6.2 U型结构的不同设计对模型的影响为了在编码过程中提取详细的语义特征信息, 我们采用非对称的U型网络架构, 在非对称的U型结构中, 底层采用了残差连接的方式, 扩充了U型结构底层的通道数, 促进特征图更详细的表示. 在不改变网络的其他功能模块的前提下, 我们将特征图按照对称U型结构进行实验, 在下采样的过程中将特征图的尺寸减少到
| 表 4 在BraTS 2020验证集上进行不对称U型结构和对称U型结构的骰子分数Dice值对比 (%) |
2.6.3 后处理对模型的影响
本文应用TTA技术来提高模型的稳健性, 由于脑肿瘤MRI图像的类别不均衡性, 其中有一些MRI图像的标签为0, 这样就会导致假阳性的出现, 为了避免出现这种情况, 通过使用TTA技术来扩充验证集的数量. 实验结果如表5所示, 从中可以发现, 通过使用TTA技术, ASUNet在ET, TC和WT上的Dice分数值分别增长了0.23%, 0.06%和0.57%, 此外TTA作为后处理技术同样也适用于其他类型的医学图像分割以及用于其他模型的精度提升.
| 表 5 在BraTS 2020验证集上使用TTA与不使用TTA的Dice值对比 (%) |
2.6.4 深度监督对模型的影响
本节中对U型结构有无深度监督进行了比较, 在上采样过程中加入深度监督, 通过使用不同的权重, 对每一个上采样特征图根据不同的权重求和计算损失, 在没有深度监督的U型结构中, 保持其他结构不变. 如表6所示, 我们可以发现, 加入深度监督在ET、WT和TC上分别提升了4.59%、0.41%和0.26%.
| 表 6 在BraTS 2020验证集上使用深度监督与不使用深度监督的Dice值对比 (%) |
3 结论与展望
本文提出了一个新型的非对称U型卷积神经网络ASUNet, 用于MRI脑肿瘤图像的精确分割. 首先, 为了减少特征语义信息丢失, 我们设计了一个非对称的U型框架结构, 在U型结构的底层建立残差连接, 保存了之前阶段的特征信息, 解决了网络训练过程中的梯度问题. 其次, 在下采样中使用残差ConvNeXt模块, 结合深度可分离卷积和逐点卷积, 避免了在下采样过程中丢失过多的细节信息. 最后, 我们在上采样的过程中采用了深度监督算法, 通过对每一层的上采样特征图进行监督, 有助于最终的预测分割图更接近于真实分割图. 在BraTS 2020数据集的实验中, 与多个先进的模型进行对比, 本文的方法取得了较好的结果. 此外, 本文还对不同的下采样方法、不同U型结构的设计以及TTA和深度监督对模型效果的影响进行了消融实验, 实验数据表明网络在设计和实现上也有独特的思路.
通过一系列实验证明, ASUNet具有良好的分割性能. 但也有一定的局限性: ASUNet只在多模式公共数据集上进行实验. 目前, 我们正在与医院谈判合作, 以获得更多临床数据. 在未来的工作中, 我们将使用临床数据, 以便真正将其应用于医学临床研究.
| [1] |
Roy S, Koehler G, Ulrich C, et al. MedNeXt: Transformer-driven scaling of convNets for medical image segmentation. Proceedings of the 26th International Conference on Medical Image Computing and Computer-assisted Intervention. Vancouver: Springer, 2023. 405–415.
|
| [2] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015. 3431–3440.
|
| [3] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [4] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [5] |
Zhang XY, Zhou XY, Lin MX, et al. ShuffleNet: An extremely efficient convolutional neural network for mobile devices. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake: IEEE, 2018. 6848–6856.
|
| [6] |
Xie SN, Girshick R, Dollár P, et al. Aggregated residual transformations for deep neural networks. Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017. 5987–5995.
|
| [7] |
Howard AG, Zhu ML, Chen B, et al. MobileNets: Efficient convolutional neural networks for mobile vision applications. arXiv:1704.04861, 2017.
|
| [8] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017 6000–6010.
|
| [9] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale. Proceedings of the 9th International Conference on Learning Representations. OpenReview.net, 2020.
|
| [10] |
Liu Z, Lin YT, Cao Y, et al. Swin Transformer: Hierarchical vision Transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 9992–10002.
|
| [11] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [12] |
Wang WX, Chen C, Ding M, et al. TransBTS: Multimodal brain tumor segmentation using Transformer. Proceedings of the 24th International Conference on Medical Image Computing and Computer-assisted Intervention. Strasbourg: Springer, 2021. 109–119.
|
| [13] |
Li JY, Wang WX, Chen C, et al. TransBTSV2: Towards better and more efficient volumetric segmentation of medical images. arXiv:2201.12785, 2022.
|
| [14] |
Xie YT, Zhang JP, Shen CH, et al. CoTr: Efficiently bridging CNN and Transformer for 3D medical image segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-Assisted Intervention. Strasbourg: Springer, 2021. 171–180.
|
| [15] |
Liu Z, Mao HZ, Wu CY, et al. A convNet for the 2020s. Proceedings of the 2022 IEEE/CVF Conference on Computer Vision and Pattern Recognition. New Orleans: IEEE, 2022. 11966–11976.
|
| [16] |
Hatamizadeh A, Nath V, Tang YC, et al. Swin UNETR: Swin Transformers for semantic segmentation of brain tumors in MRI images. Proceedings of the 7th International MICCAI Brainlesion Workshop. Springer, 2021. 272–284.
|
| [17] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [18] |
Hatamizadeh A, Tang YC, Nath V, et al. UNETR: Transformers for 3D medical image segmentation. Proceedings of the 2022 IEEE/CVF Winter Conference on Applications of Computer Vision. New Orleans: IEEE, 2022. 1748–1758.
|
| [19] |
Jiang Y, Zhang Y, Lin X, et al. SwinBTS: A method for 3D multimodal brain tumor segmentation using Swin Transformer. Brain Sciences, 2022, 12(6): 797. DOI:10.3390/brainsci12060797 |
| [20] |
Peiris H, Hayat M, Chen ZL, et al. A robust volumetric Transformer for accurate 3D tumor segmentation. Proceedings of the 25th International Conference on Medical Image Computing and Computer-assisted Intervention. Singapore: Springer, 2022. 162–172.
|
| [21] |
Xu WJ, Yang HH, Zhang MY, et al. Brain tumor segmentation with corner attention and high-dimensional perceptual loss. Biomedical Signal Processing and Control, 2022, 73: 103438. DOI:10.1016/j.bspc.2021.103438 |
| [22] |
Ho NV, Nguyen T, Diep GH, et al. Point-UNet: A context-aware point-based neural network for volumetric segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-assisted Intervention. Strasbourg: Springer, 2021. 644–655.
|
| [23] |
Akbar AS, Fatichah C, Suciati N. Single level UNet3D with multipath residual attention block for brain tumor segmentation. Journal of King Saud University—Computer and Information Sciences, 2022, 34(6): 3247-3258. DOI:10.1016/j.jksuci.2022.03.022 |
| [24] |
Colman J, Zhang L, Duan WT, et al. DR-Unet104 for Multimodal MRI brain tumor segmentation. Proceedings of the 6th International MICCAI Brainlesion Workshop. Lima: Springer, 2021. 410–419.
|
| [25] |
Çiçek Ö, Abdulkadir A, Lienkamp SS, et al. 3D U-Net: Learning dense volumetric segmentation from sparse annotation. Proceedings of the 19th International Conference on Medical Image Computing and Computer-assisted Intervention. Athens: Springer, 2016. 424–432.
|
| [26] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [27] |
Khan MI, Jafaritadi M, Alhoniemi E, et al. Adaptive weight aggregation in federated learning for brain tumor segmentation. Proceedings of the 7th International MICCAI Brainlesion Workshop. Springer, 2021. 455–469.
|
| [28] |
Hu JY, Gu XJ, Wang ZQ, et al. Mixture of calibrated networks for domain generalization in brain tumor segmentation. Knowledge-based Systems, 2023, 270: 110520. DOI:10.1016/j.knosys.2023.110520 |
| [29] |
Pawar K, Zhong SJ, Chen ZL, et al. Brain tumor segmentation using two-stage convolutional neural network for federated evaluation. Proceedings of the 2021 International MICCAI Brainlesion Workshop. Springer, 2021. 494–505.
|
| [30] |
Nalawade S, Ganesh C, Wagner B, et al. Federated learning for brain tumor segmentation using MRI and Transformers. Proceedings of the 2021 International MICCAI Brainlesion Workshop. Springer, 2021. 444–454.
|
 2024, Vol. 33
2024, Vol. 33



