2. 福建医科大学附属第一医院 放疗科, 福州 350001;
3. 福建医科大学肿瘤临床医学院 & 福建省肿瘤医院 放射诊断科, 福州 350014;
4. 福建师范大学 数字福建环境监测物联网实验室, 福州 350117
2. Radiotherapy Department, the First Affiliated Hospital of Fujian Medical University, Fuzhou 350001, China;
3. Department of Radiodiagnosis, Fujian Cancer Hospital & Clinical Oncology School of Fujian Medical University, Fuzhou 350014, China;
4. Digital Fujian Environmental Monitoring Internet of Things Laboratory, Fujian Normal University, Fuzhou 350117, China
脑胶质瘤为颅内原发恶性肿瘤, 在我国年发病率为每10万人5–8例, 目前该疾病的发病机制可能与高剂量电离辐射暴露、相关基因遗传突变等有关, 其临床表现主要包括颅内压增高、神经功能及认知功能障碍和癫痫发作3大类[1]. 2021年版 WHO 中枢神经系统肿瘤分类将脑胶质瘤分为1–4级, 其中1、2级为低级别脑胶质瘤, 3、4 级为高级别脑胶质瘤[2], 2022年美国国立综合癌症网络(NCCN)指南还将胶质瘤进一步细分为5类[3].
影像学检查作为该疾病的重要诊断与鉴别诊断方法之一, 其能够定位, 即肿瘤位置大小等形态, 并能初步定性, 即初步判断肿瘤的恶性程度, 有助于胶质瘤分级, 故对于胶质瘤的诊断、治疗、甚至预后判断均有十分重要的作用. 作为软组织病变, 相比于电子计算机体层扫描(CT), 因核磁共振(MR)检查利用氢原子成像, 故对于脑组织更具有优势, 对于脑胶质瘤疾病全程管理, 均起到重要的指导作用.
脑胶质瘤的MR扫描通常需要取得T1加权图像、T2加权图像、液体衰减反转恢复图像(FLAIR)序列的图像, 以及进行加入对比剂的MR增强扫描, 即实现多模态的图像获取. 在胶质瘤的MR影像上, 通常呈现长T1、长T2的信号影, FLAIR俗称压水象, 其设计之初即为主要应用于颅脑, 通过使正常脑脊液信号衰减, 从而肿瘤水肿区域征象更为明显. 增强扫描则征象各异, 根据不同性质的肿瘤会有不同的结果. 通常边缘轮廓清晰平整光滑、周围水肿不明显是低级别胶质瘤的表现; 而边缘轮廓不整、周围大片水肿并伴坏死则常常提示高级别胶质瘤, 同时还可能具脑中线结构移位、脑池、脑沟受压等征象, 并且会有更加明显的不同类型的异常强化影. 如图1所示为典型脑胶质瘤MR影像学表现, 其中, 图1(a)为FLAIR图像, 图1(b)为T1加权图像, 图1(c)为T1加权增强图像, 图1(d)为T2加权图像. 可见额颞叶占位, T1等信号, 边缘见略高信号, T2高信号, FLAIR仍呈高信号表现, 周围见水肿影, 增强扫描见边缘环形强化, 右侧脑室受压变形; 中线结构左侧偏移.
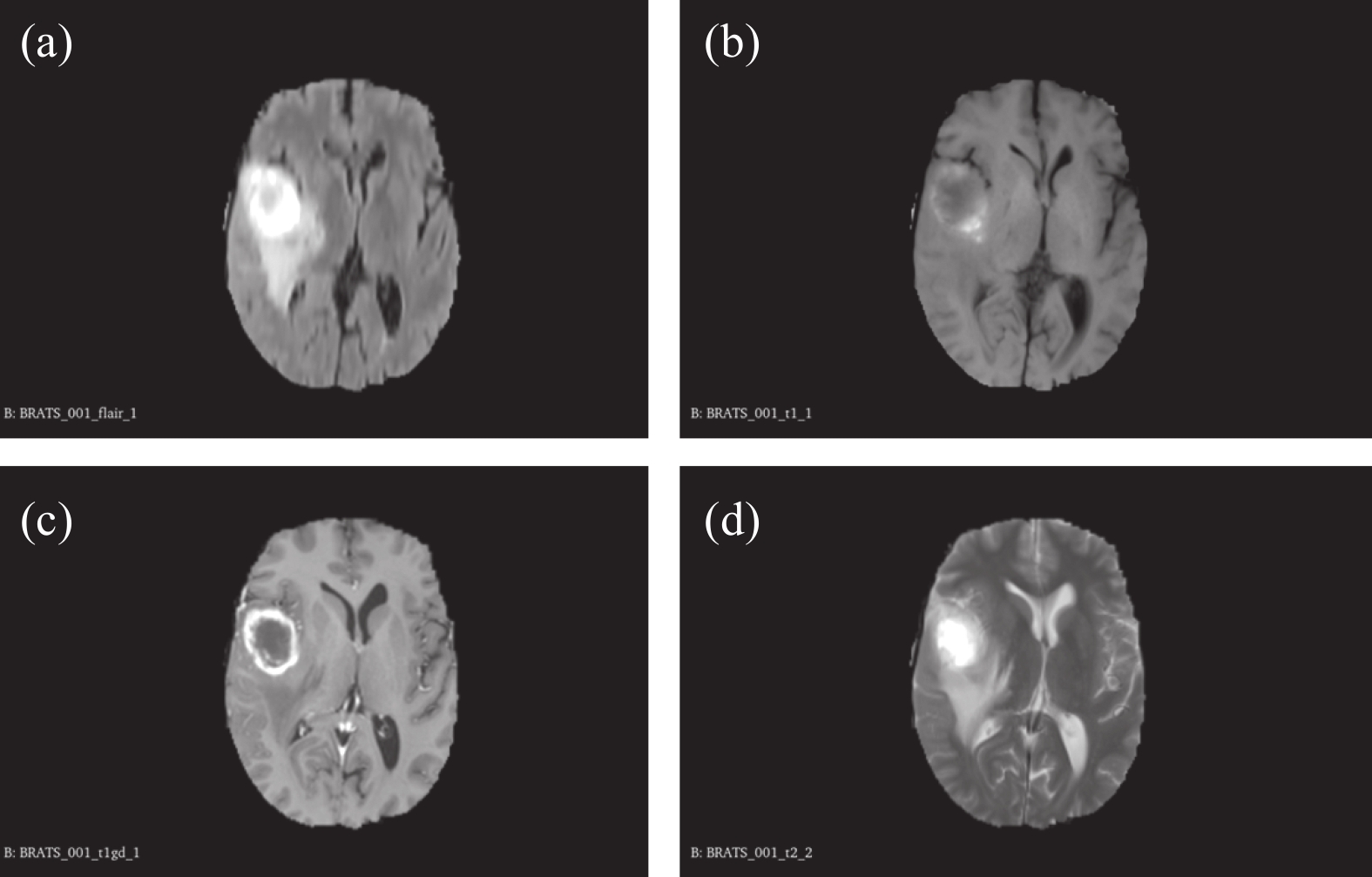
|
图 1 脑胶质瘤MR影像学表现 |
图像分割是实现计算机辅助诊断的重要步骤, 近年来, 较多方法被应用于脑胶质瘤的核磁共振图像分割, 包括传统基于图像处理的方法, 如使用基于阈值的分割方法, 如全局阈值、局部阈值等[4,5]; 基于区域的分割方法, 如区域生长、活动轮廓、分水岭[6–8]等, 目前已广泛应用于脑胶质瘤核磁共振图像分割, 这些方法操作简单、速度快, 但会出现过度分割问题. 基于传统机器学习的图像分割方法, 是一种通过对已知的数据或者某种规律性特点学习来预测推理出未知的样本类别. 根据算法是否依赖于标记的训练集, 机器学习方法能够分成监督、无监督和半监督3种方法. 监督方法借助标签指导, 通过手工提取脑胶质瘤图像部分的特征, 例如图像像素、纹理、梯度等, 将其提供给分类器如决策树、随机森林以实现分割, 手工提取的大量图像特征未必能完全表示脑胶质瘤图像信息, 会导致一般化能力下降, 而小样本数据或过于低维度的数据分类器分类效果会出现下降的问题. 聚类(clustering)是一种无监督机器学习方法, 如K均值聚类方法已被用于脑胶质瘤核磁共振图像分割[9], 这种方法实现速度快, 然而受初始状态影响较大, 取得分割结果较不稳定. 近年来, 基于深度学习的方法也广泛应用于脑胶质瘤核磁共振图像分割[10], 这种方法可以通过精致设计的神经网络来更好地提取特征[11], 从而实现更加精准的分割结果, 其中基于典型的基于全卷积网络[12]的U-Net方法被设计用于医学图像的分割[13], 采用跳跃连接的方法, 能够将图像的深层信息和浅层信息相结合, 实现对医学图像的分割任务.
而过于深层的神经网络, 其感受野不断增大, 虽捕获更多全局特征, 但会使得模型丢失了局部的特征信息, 图卷积网络(GCN)应运而生[14], 其用来对图数据结构进行处理, 每个节点的参数和权值在全程共享, 通过传递节点的信息从而更新节点的状态, 其结果是该结构能够实现对局部信息的更多关注. 由于脑胶质瘤本身高度异质性, 其大小形态不一, 可生长于颅脑中任何部位, 其常见于大脑及脑干, 绝大部分为浸润性生长, 使得影像学表现不规则, 边缘部分模糊, 部分病例常见的水肿征象及增强时的不均匀强化甚至经过治疗后可能出现的难以区分坏死和复发征象等进一步加大对图像该部分精确分割的难度; 同时MR影像的众多模态增加分割需要的众多必要信息的同时引入不必要的信息, 如T1增强图像中肿瘤部分由于血脑屏障破坏对比剂渗入导致其呈现高信号, 而正常硬脑膜中因有丰富的毛细血管网, 亦可呈现高信号, 故可能干扰分割结果. 作为图像中局部信息, 利用GCN结构可以对U-Net生成的特征图进行进一步细致处理, 结合邻接关系, 从而获得更加准确分割结果, 对上述问题具有一定帮助. 图数据结构是一种不同于传统的欧氏结构的数据, 而常见的CNN通常只能处理欧氏结构的数据. 图具有节点和边的特点, GCN能够借助图结构, 获得深层次的特征, 并且随着图节点保存信息, 而不会随着网络的加深丢失局部的特征信息.
综上, 本文将CNN和GCN结合, 利用 GCN 将图像分割问题转化为图节点分类问题来实现对脑胶质瘤核磁共振图像的分割. 本文将分为3部分叙述: 首先介绍GCN的基本原理, 阐述方法的相关步骤; 其次介绍实验所使用的数据集、实验环境、评价指标以及实验结果; 最后对本文方法进行总结并给出展望.
1 本文方法本文方法分为两个阶段进行, 如图2所示, 第1阶段将多模态即T1加权、T2加权、T1增强、FLAIR序列整合为4通道的脑核磁共振图像输入U-Net, 通过上采样、下采样及跳跃连接的方式, 提取多尺度特征; 并将浅层特征和深层特征相结合、多模态特征进行融合; 第2阶段, 利用U-Net的输出中的64维向量进行输入, 将 U-Net 获得的数据转化为 GCN 所需的图结构数据, 重新构造边的结构, 输入GCN, 随即图像分割问题转化为图节点分类问题, 关注图的节点之间关系问题, 利用 GCN 对图结构数据进行分类.
1.1 基于U-Net的特征提取如图2所示, 本文特征提取部分所采用的U-Net网络是一个编码器-解码器结构, 两部分内容相互对称. 图2主要为U-Net网络的结果示意, 左半部分即编码器, 亦被称为收缩部分, 右半部分为解码器, 亦被称为扩展部分. 收缩部分用于获取上下文信息, 由堆叠的卷积、池化操作实现下采样, 扩展部分用于精确的定位, 由转置的卷积(反卷积)操作实现上采样.
在收缩部分包含多个收缩块; 每个块包括两个3×3卷积层, 然后是一个2×2池化层, 它使用校正线性单元(ReLU)激活函数[15,16]. 随着网络层次递进, 特征图的数量逐渐增加, 从而能够有效地学习复杂结构, 将特征图数量增加到
扩展部分中的每个块由两个3×3卷积层和一个2×2上采样层组成. 为了保持对称性, 从每个块到下一块, 特征图的数量减少一半. U-Net中使用跳过连接(copy and crop)将相应收缩层的特征图附加到每个扩展部分的末尾, 使用拼接(concatenate)的方法实现特征融合, 这些连接保证了收缩部分特征将用于图像重建. 最后一层是1×1卷积层, 用于生成分割结果.
池化层实现了网络对图像的多尺度特征识别, 共有5个池化层结构, 实现5尺度特征识别. 图2中位于前层的卷积层将输出低级别特征图, 识别出肿瘤的大致位置及轮廓, 位于深层则输出高级别特征图, 得到更细致的肿瘤边缘细节. 如图2右侧部分, 本文将截取U-Net网络输出中间层部分的特征图, 它是通过下采样、上采样、在不同连接层提取的多尺度、多级别特征, 作为后续GCN的输入内容.

|
图 2 U-Net网络结构 |
1.2 GCN结构设计
图卷积网络将传统数据的卷积操作推广至图结构数据中. 其总体思想为生成一个函数f, 通过聚合操作获得节点自身的特征以及邻接节点的特征.
图结构数据一般表示为, G = (V, E). V表示图的顶点(节点)集合, E表示图的边集合, 图在计算机中可使用邻接矩阵存储, 邻接矩阵表示图的各个顶点(节点)的关系, 其各个元素表示图各个顶点(节点)之间的相邻关系.
图卷积网络通常分为基于空间域以及基于谱域的图卷积网络, 本文使用基于谱域的图卷积网络. 基于谱的图卷积网络利用图的拉普拉斯矩阵的特征值及特征向量实现特征提取, 如将特征分解后的拉普拉斯矩阵L表示为:
| L=UΛUT | (1) |
则图的Fourier变换为:
| ˆf=UTf | (2) |
其中, 左侧为Fourier系数, f则被定义为图信号, 图的Fourier逆变换为:
| f=Uˆf | (3) |
由此, 根据卷积定理, 图数据结构的卷积是两者Fourier变换的乘积的逆变换, 表示为:
| (f∗g)G=U(UTf⋅UTg) | (4) |
图卷积网络的图卷积操作为[14]:
| H(l+1)=σ(ˆD−12ˆAˆD−12HlWl) | (5) |
其中,
通过多层卷积, 图卷积网络提取出各个节点的信息, 从而进行之后的分割任务. 图卷积操作先单独对每个节点特征进行变换, 而后通过聚合来实现获取邻接节点的特征信息, 多层卷积层则可以接收到来自更远的节点特征, 不同于CNN, 在其中每一层图卷积层的参数是共享的. 如图3, 本文将使用两层卷积层的图卷积网络, 将U-Net输出的特征图转换为图结构G, 此后进行膨胀邻域、稀疏化处理后, 输入GCN, 图3中V为输入GCN的节点信息结构, 其大小N与原始图像有关, 本文使用U-Net输出的64维特征图作为GCN的输入, 图3中E为经过边膨胀邻域、稀疏化处理的边结构, GCN卷积层使用谱域下的图卷积操作, 结果为产生若干个特征图, 使用ReLU单元激活, 最终对图节点进行分类.
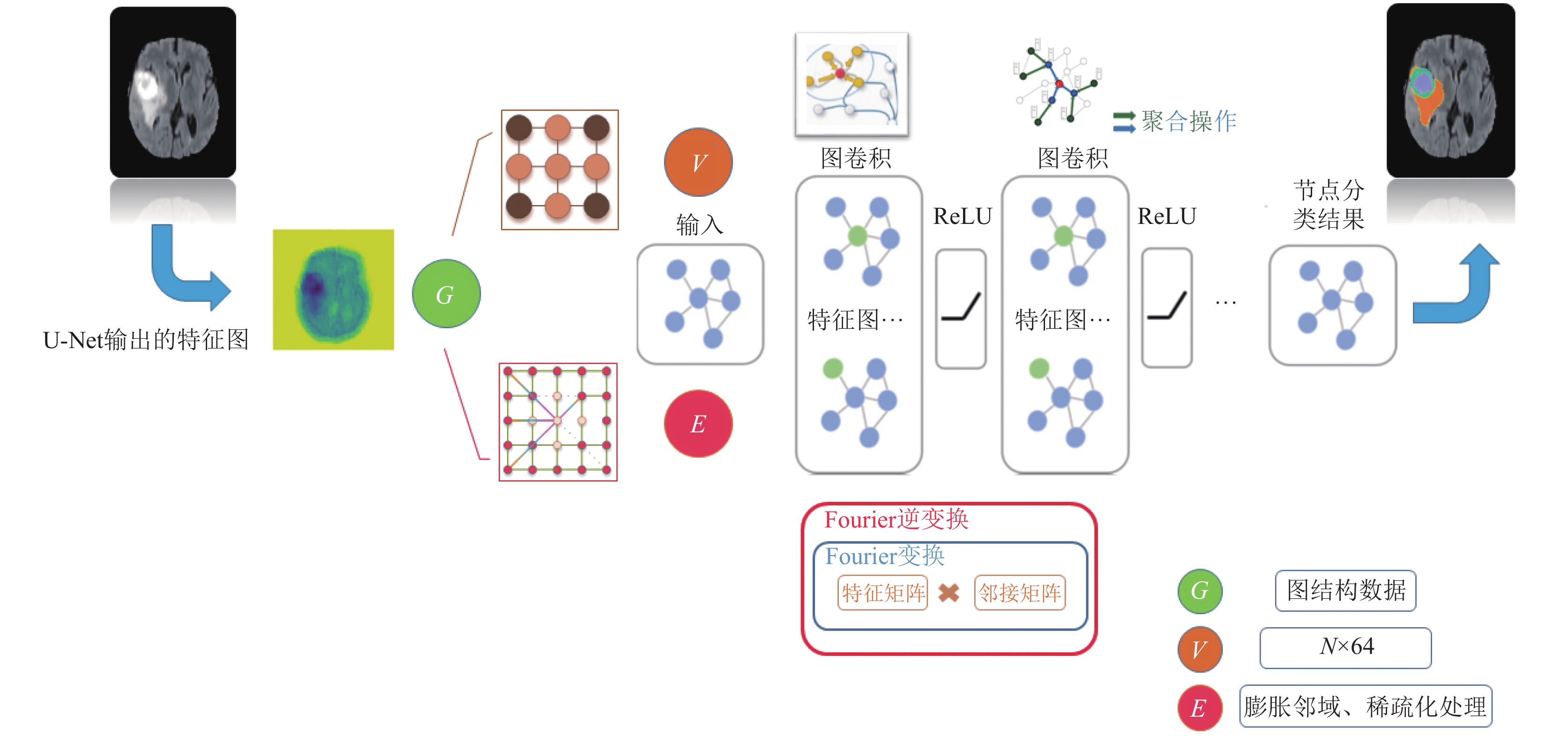
|
图 3 基于GCN的MR图像分割流程 |
1.3 图结构边的重构
本文方法中, GCN所输入训练的数据, 需要将U-Net训练获得到的中间特征转换为图数据结构. 在此使用余弦相似度考虑像素点至图结构节点的转换关系:
| cos(θ)=∑ni=1xi×yi√∑ni=1(xi)2×√∑ni=1(yi)2 | (6) |
余弦相似度用于表示两个节点之间的相似程度, 相似性越大则余弦相似度越大, 若两个点的相似度过于小(本文设置为小于0.75), 则舍弃该条边. 通过设置膨胀邻域的方法, 对边结构进行重塑, 不仅考虑原始像素的4邻域或8邻域的邻接节点信息, 亦通过改变不同的膨胀值, 增大邻接关系, 以增加深层特征的学习能力.
此外, 增加邻接关系同时可能也增加了实际无用的边, 本文运用稀疏化剪枝处理以再次去除低相似度的边[18], 通过计算每一个节点的权值[19], 按照权重升序排列插入将每个节点的权重及边加入队列, 将前e条边移出队列并放入集合中, 此后找到并删除集合中重复的边, 如图4为膨胀邻域和稀疏化处理过程, 其中输入为特征图像素间原始邻接关系结构, 经过建立新的邻接关系并删除相似度低的弱关系, 得到处理后的图结构. 若关注中心的点, 原始的图为4邻域, 经过膨胀邻域增加了4个邻接关系, 而后通过稀疏化处理, 假设图中的e为6, 即处理后的图中心节点最多保留6条边, 实线为通过计算后需要保留的边, 虚线为只保留一个节点的边, 最终得到处理后的图结构.
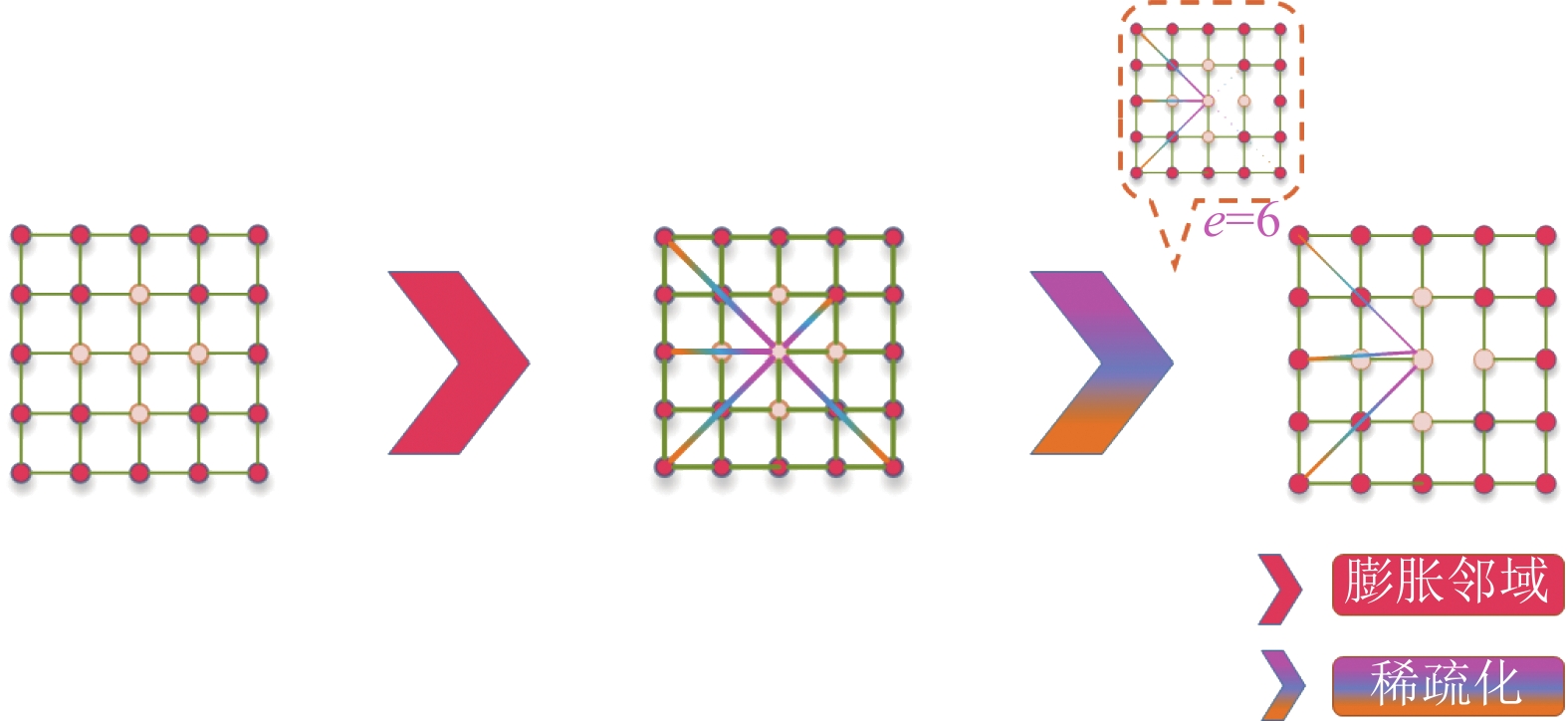
|
图 4 GCN中的膨胀邻域和稀疏化处理 |
利用边缘构建策略, 其倾向于关注附近节点和具有相似特征的节点, 这些节点不仅关注本地信息, 而且能够访问远程信息, 增加更多的邻接关系, 不同区域的脑肿瘤之间的关系通过同结构实现有效地学习. 与传统卷积神经网络的有限感受野相比, 图卷积模块具有更大的全局感受野, 这使得每个节点的信息在图学习的前向传播中完全整合其他节点的信息.
2 实验结果与分析在实际实验中, 本文使用的数据集为国际医学图像计算和计算机辅助干预协会(MICCAI)提供的BraTS 2018, 该数据集共有285名病例, 且已知其中210例为高级别胶质瘤, 75例为低级别胶质瘤. 每个病例均有T1加权图像(T1WI)、T2加权图像(T2WI)、FLAIR图像及T1WI引入钆类对比剂(GD-DTPA)的增强图像共4种模态的图像, 4种模态每个序列共有155个断层面, 图像尺寸均为240×240像素, 每个病例图像已整合4个模态形成四维数据, 本文保留每个病例、每个模态中第50–130张断层面, 以舍弃部分不含肿瘤的断层面, 其范围基本覆盖幕上及部分幕下范围. 该数据集已事先进行偏置场校准、颅骨分离、插值, 并按照相同解剖模板进行配准; 并提供已分割的标准标签, 根据数据集的定义, 给出的分割区域有整体肿瘤区域(whole tumor, WT)和增强肿瘤区域(enhance tumor, ET)以及肿瘤核心区域(tumor core, TC), 本文的实验测试结果也以该定义为准.
对于实验环境, 本文采用基于Python 3的PyTorch 1.7.1深度学习框架, 运行于Ubuntu 20.04.3操作系统上, 所使用的图形处理单元(graphics processing unit, GPU)为GeForce RTX 3090. 在参数设置上, 对于U-Net部分, 训练的batch size大小为32, 使用Adam作为优化算法, 学习率设置为10−3, 衰减权重为10−4. 为了加速训练, 假如损失函数的值超过20轮不再减小, 则提早停止训练. 对于GCN部分, 训练的batch size大小为32, dropout比率设置为0.5, Adam作为优化算法, 学习率设置为3×10−3, 衰减权重为10−4.
集真实分割标签为标准, 使用Dice损失函数[20], 公式如下:
| Dice(g,p)=1−2∑ni=1pigi∑ni=1(p2i+g2i) | (7) |
其中, g表示图像的真实标签, p表示网络的预测结果, n表示图像的像素点个数.
对于GCN部分, 使用交叉熵损失函数, 公式如下:
| L=−1m∑mi=1∑nj=1yijlog(x′ij) | (8) |
其中, m为数据集样本数量, n为类别数量,
实验先对数据集进行预处理, 设置训练集和测试集的比例为70%和30%, 将图像大小调整为160×160, 选择128张断层面的图像进入实验, 然后将数据集的4个模态即4个序列的图像连接成4×128×160×160作为输入数据. 然后进行归一化处理, 将图像中灰度值小于30的像素点设置为0, 接着将每个像素点减去非0部分的均值, 最后再除以非0部分的标准差. 并对训练集进行小于20°的随机旋转、随机平移等操作, 概率值为0.4.
2.1 消融实验及对比实验本文所提出的方法, 结合了U-Net和GCN, 因此实验对单独使用U-Net方法, 单独使用GCN方法, 以及结合U-Net和GCN的方法进行测试.
为验证本文方法的性能, 除U-Net外, 实验还选取其他常用于医学图像分割任务的先进方法进行比较, 分别为FCN[12]、HTTU-Net[21]. FCN是一种用于语义分割的方法, 适用于多种自然场景的通用图像. 它通过对基于CNN的分类网络进行改造, 使用卷积层替换全连接层从而实现对图像的分割. HTTU-Net是设计专用于脑胶质瘤核磁共振图像分割的方法, 其基于原始U-Net的编码器-解码器结构, 它将处理流程划分为具有不同大小的卷积核双通道[21], 之后通过卷积和Softmax操作得出分割结果.
如图5所示为编号001病例第95张切片结果, 从左至右依次为原始影像、单独使用U-Net方法的分割结果、结合使用U-Net和GCN的结果以及真实分割的结果; 真实分割结果为BraTS 2018数据集中带有的专家标注的标签的可视化展示, 作为金标准, 是实验各项性能的参考. 从上至下依次为FLAIR、T1WI、T1增强、T2WI影像. 其可见: 右侧脑室旁占位, 脑室受压变形, 中线结构偏移, 呈长T1长T2信号, 增强见边缘强化. 其中第2、3列的红色为真实分割的轮廓叠加表示. 从中可见, 相比于单独使用U-Net方法, 结合使用GCN以后, 其与真实分割的边缘重合度更高(红色边缘), FLAIR及T2WI上见不规则的水肿区域边缘与真实分割结果接近; 同时增强肿瘤区域对应于T1增强图像的边缘环状高信号, 说明对于增强肿瘤区域判断正确; 且3种区域的交界位置也得到了更加细致的结果, 更加接近真实分割效果.
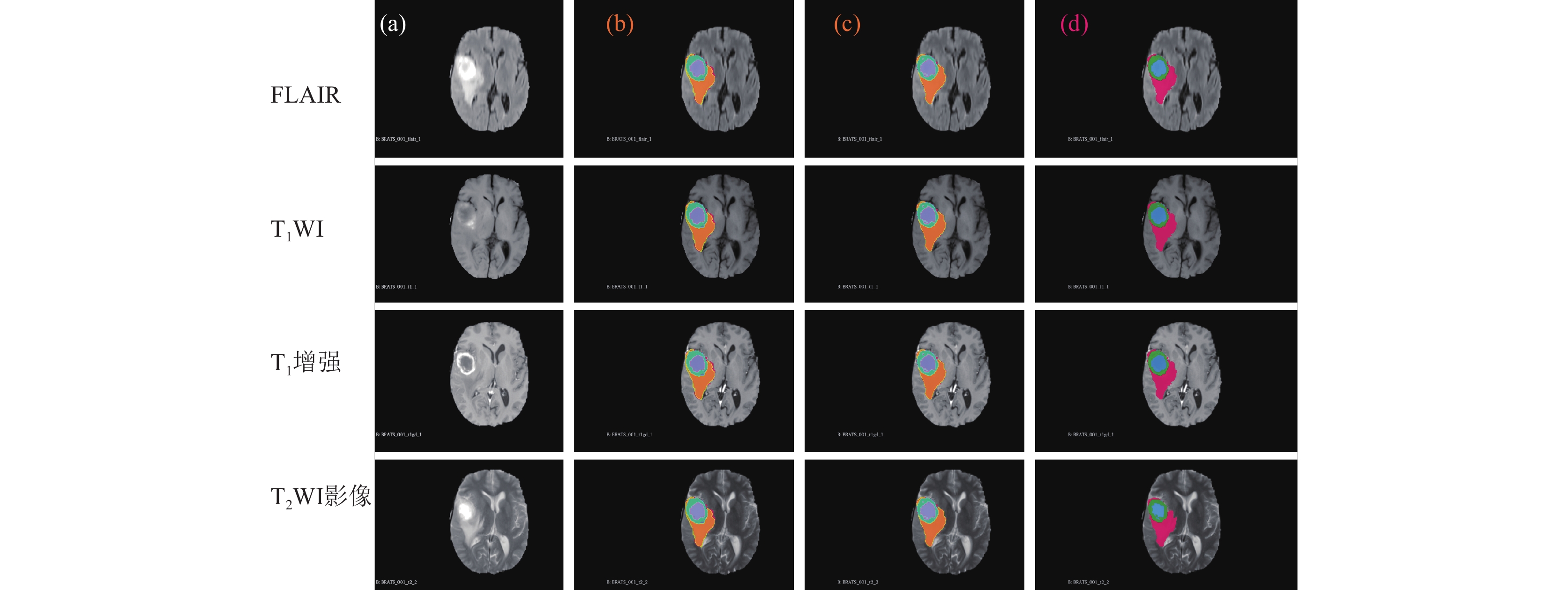
|
图 5 第75张切片分割结果多模态图例显示 |
为对实验进行定量分析, 使用准确度(accuracy, ACC)、灵敏度(sensitivity, SEN)、Dice来度量实验效果, 公式如下:
| ACC=TP+TNTP+FP+TN+FN | (9) |
| SEN=TPTP+FN | (10) |
| Dice=2TPTP+2FP+FN | (11) |
其中, TP为正确分类肿瘤区域的数量, TN表示正确分类背景区域的数量, FP表示背景区域被错误分类为肿瘤区域的数量, FN为肿瘤区域被错误分类为背景区域的数量.
表1为测试不同方法在BraTS 2018数据集中整体平均水平, 以数据集中自带的分割标签为标准, 计算利用不同方法预测的结果与标准分割的差异. 从表1实验结果可以看出, 定量测试本文方法在Dice指标、敏感度、准确度上各项数值均相比于单独使用U-Net的方法有了提升, 其中准确度已达到99.6%, 灵敏度达到89.2%, 说明结合使用U-Net和GCN方法, 从该定量结果上看, 对于分割脑胶质瘤核磁共振图像具有较好的效果.
| 表 1 消融实验测试结果 |
根据上文所提数据集分割区域的定义, 分别测试本文的方法对3个区域分割的性能, 为验证本方法进一步的一般化能力, 扩展至BraTS 2018数据集的所有序列图像进行综合计算后, 结果如表2所示, 同分割的图像结果相对应, 根据分区域的分割结果, 相比其余两个区域, 定量测试本文方法对WT即整体肿瘤区域分割效果较佳, 其中准确度、灵敏度、Dice均超过95%, 体现本文方法在该数据集上运行稳定, 具有一定的一般化能力, 该区域能够获得更好的效果, 因为WT区域实际上包括了增强肿瘤、肿瘤核心即ET、TC区域, 其作为最大的亚区域, 故提高了整体的性能值; 对于肿瘤核心区域TC, 本文方法在准确度、灵敏度、Dice均超过93%的结果; 对于增强肿瘤区域ET, 其指标略有下降, 准确度为92%, 考虑其原因应是增强肿瘤区域仅于T1增强图像显示较明显高信号, 呈现边缘环状强化影, 其余模态显示增强边缘部分不清, 区分度不高, 结合多模态图像计算指标后, 略降低了性能, 图5印证了此结果.
| 表 2 结合U-Net和GCN的肿瘤区域分割结果 |
表3为将本文方法分别与FCN、HTTU-Net进行对比的测试结果, 从中可看出通过结合3个区域的平均性能, 本文方法在准确度、灵敏度及Dice指标均不逊于上述两方法, 其中准确度、灵敏度具有更加明显的优势, 其验证了本文方法取得了较好的结果.
| 表 3 与其他方法对比测试结果 |
2.2 图像序列三维重建实验
为便于观察整体分割效果, 利用ITK-SNAP[22]和3D-slicer[23]中的三维重建模块, 对叠加分割结果FLAIR图像中第50–130张断层面进行三维重建, 图6(c)显示的是第95张断层面, 图6(d)为冠状位、图6(e)为矢状位的重建结果, 十字线中心指向肿瘤核心区域的大致位置; 上方为本文方法分割结果的三维视图, 图6(a)为叠加分割结果颅脑右侧观, 其可见肿瘤区域的体表投影; 图6(b)为单独肿瘤部分重建结果, 图中的RAS坐标按照解剖学方位定义: R (right)表示以患者为中心右侧, L (left)表示左侧, A (anterior)表示前方, S (superior)表示上方, I (inferior)表示下方. 其中矢状面重建可见肿瘤前后生长侵袭程度, 冠状面重建可见右侧脑室受压变形情况; 三维肿瘤视图可见肿瘤核心区域大部位于颞叶, 边缘另见大量体积水肿区域. 原断层面显示肿瘤部分边缘与脑组织界限不清, MR信号相近, 难以区分, 通过叠加分割肿瘤三维重建后的图像, 可从各个方向及任意角度观察肿瘤位置、形态结构, 可见其形状与运用本文方法分割结果相吻合, 体现本文方法分割该区域的有效性.

|
图 6 叠加分割的三维重建影像 |
3 结论与展望
脑胶质瘤作为一类严重的神经系统恶性肿瘤, 实现对该疾病的计算机辅助诊断, 对于该疾病的全程管理具有帮助, 作为影像学检查中的重要部分, 核磁共振对该疾病诊断及治疗具有十分的意义[24], 本文着重研究脑胶质瘤核磁共振图像分割问题.
本文使用一个结合了图卷积网络和卷积神经网络的方法, 来实现对脑胶质瘤核磁共振图像的分割. 第1步先通过U-Net网络训练输入的多模态胶质瘤核磁共振图像, 生成中间特征图, 在第2步将中间特征图转化为图数据结构后输入GCN网络, 对图的各个节点进行分类, 从而实现对图像的分割. 同传统的卷积神经网络相比, 使用图卷积网络的分割方法, 图的节点在非欧氏空间中工作, 大大增加了灵活度, 使其可以处理任意形状任意大小的图像信息, 并且能自动学习图像的局部特征, 提高了分割的准确度, 而适用于局部信息的图神经网络, 恰适合如本文的脑胶质瘤图像的应用, 本文使用基于谱域的两层图卷积网络, 即可实现对图节点的良好分类.
对公开数据集的实验结果可知, 本文的方法取得了较好的分割效果, 采用结合U-Net结构进行特征提取可以改善随着网络层数不断增加而丢失局部的信息的问题. 与此同时, 图卷积网络结构能够有效关注局部特征, 减小了同类像素内的差异, 增大了不同类间像素的差异, 适合本文的脑胶质瘤核磁共振图像分割; 除此之外, 其亦可适用于类似需要关注局部信息的图像分割等问题, 该方法将有可能拓展至其领域使用的可能. 后续的研究中通过改进图结构的构造方式, 可以进一步降低计算量, 提高分割效果.
| [1] |
国家卫生健康委员会医政医管局, 中国抗癌协会脑胶质瘤专业委员会, 中国医师协会脑胶质瘤专业委员会. 脑胶质瘤诊疗指南(2022版). 中华神经外科杂志, 2022, 38(8): 757-777. DOI:10.3760/cma.j.cn112050-20220510-00239 |
| [2] |
Louis DN, Perry A, Wesseling P, et al. The 2021 WHO classification of tumors of the central nervous system: A summary. Neuro-oncology, 2021, 23(8): 1231-1251. DOI:10.1093/neuonc/noab106 |
| [3] |
Horbinski C, Nabors LB, Portnow J, et al. NCCN guidelines® insights: Central nervous system cancers, version 2. Journal of the National Comprehensive Cancer Network, 2023, 21(1): 12-20. |
| [4] |
Sharma SR, Alshathri S, Singh B, et al. Hybrid multilevel thresholding image segmentation approach for brain MRI. Diagnostics, 2023, 13(5): 925. DOI:10.3390/diagnostics13050925 |
| [5] |
Ye ZW, Song ZL, Li PF, et al. A modified threshold score-based multilevel thresholding segmentation technique for brain magnetic resonance images using opposition-based learning hybrid rice optimization algorithm. International Journal of Imaging Systems and Technology, 2023, 33(2): 622-643. DOI:10.1002/ima.22830 |
| [6] |
贺江琳, 刘世伟, 王星月, 等. 基于混合水平集的脑胶质瘤分割方法. 中国医学物理学杂志, 2019, 36(4): 414-419. DOI:10.3969/j.issn.1005-202X.2019.04.009 |
| [7] |
UmaMaheswaran SK, Ahmad F, Hegde R, et al. Enhanced non-contrast computed tomography images for early acute stroke detection using machine learning approach. Expert Systems with Applications, 2024, 240: 122559. DOI:10.1016/j.eswa.2023.122559 |
| [8] |
Moussaoui H, El Akkad N, Benslimane M. A brain tumor segmentation and detection technique based on birch and marker watershed. SN Computer Science, 2023, 4(4): 339. DOI:10.1007/s42979-023-01802-4 |
| [9] |
Jamazi C, Manita G, Chhabra A, et al. Mutated Aquila optimizer for assisting brain tumor segmentation. Biomedical Signal Processing and Control, 2024, 88: 105089. DOI:10.1016/j.bspc.2023.105089 |
| [10] |
李锵, 白柯鑫, 赵柳, 等. MRI脑肿瘤图像分割研究进展及挑战. 中国图象图形学报, 2020, 25(3): 419-431. DOI:10.11834/jig.190524 |
| [11] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe: ACM, 2012. 1097–1105.
|
| [12] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [13] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [14] |
Kipf TN, Welling M. Semi-supervised classification with graph convolutional networks. Proceedings of the 5th International Conference on Learning Representations. Toulon: ICLR, 2017.
|
| [15] |
Nair V, Hinton GE. Rectified linear units improve restricted Boltzmann machines. Proceedings of the 27th International Conference on Machine Learning. Haifa: ICML, 2010. 807–814.
|
| [16] |
Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks. Proceedings of the 14th International Conference on Artificial Intelligence and Statistics. Fort Lauderdale: AISTATS, 2011. 315–323.
|
| [17] |
Hinton GE, Srivastava N, Krizhevsky A, et al. Improving neural networks by preventing co-adaptation of feature detectors. arXiv:1207.0580, 2012.
|
| [18] |
战宇, 潘海为, 韩启龙, 等. 一种运用图熵的医学图像聚类方法. 小型微型计算机系统, 2016, 37(7): 1594-1599. DOI:10.3969/j.issn.1000-1220.2016.07.044 |
| [19] |
Yang B, Pan HW, Yu JY, et al. Classification of medical images with synergic graph convolutional networks. Proceedings of the 35th IEEE International Conference on Data Engineering Workshops (ICDEW). Macao: IEEE, 2019. 253–258.
|
| [20] |
Sudre CH, Li WQ, Vercauteren T, et al. Generalised dice overlap as a deep learning loss function for highly unbalanced segmentations. Proceedings of the 3rd International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Québec City: Springer, 2017. 240–248.
|
| [21] |
Aboelenein NM, Songhao P, Koubaa A, et al. HTTU-Net: Hybrid two track U-Net for automatic brain tumor segmentation. IEEE Access, 2020, 8: 101406-101415. DOI:10.1109/ACCESS.2020.2998601 |
| [22] |
Yushkevich PA, Piven J, Hazlett HC, et al. User-guided 3D active contour segmentation of anatomical structures: Significantly improved efficiency and reliability. NeuroImage, 2006, 31(3): 1116-1128. DOI:10.1016/j.neuroimage.2006.01.015 |
| [23] |
Fedorov A, Beichel R, Kalpathy-Cramer J, et al. 3D slicer as an image computing platform for the quantitative imaging network. Magnetic Resonance Imaging, 2012, 30(9): 1323-1341. DOI:10.1016/j.mri.2012.05.001 |
| [24] |
祝自虹, 王惠, 尹勇. 低级别脑胶质瘤放射治疗进展. 中华肿瘤防治杂志, 2022, 29(6): 381-386. |
 2024, Vol. 33
2024, Vol. 33



