2. 沈阳工业大学 软件学院, 沈阳 110870
2. School of Software, Shenyang University of Technology, Shenyang 110870, China
急性淋巴细胞白血病(ALL)是一种严重的血液癌症, 开始于白细胞产生的地方, 如骨髓和淋巴系统的疾病[1]. 在这个情况下, 骨髓产生大量未成熟的淋巴细胞(又称“母细胞”)而不是健康的粒细胞或红细胞. 急性淋巴细胞白血病主要影响儿童和青少年, 同时也可出现在成年人中[2]. 目前在全世界范围内, 这种疾病的发病率以及疾病管理都有所不同. 根据全球权威服务机构卫生数据科学参考中心(global burden of disease study)的报告, 在全球范围内, 急性淋巴细胞白血病的发病人数在2017年达到了近10万例[3]. 并且全球各个地区的发病率存在着显著的差距. 北美和欧洲地区的确诊病例数量相对较多, 而亚洲、非洲和拉丁美洲等资源匮乏地区的病例因为检测设施和技术的限制[4], 而有所低估, 影响全球各地成千上万的人. 针对这种情况, 早期准确诊断对于成功治疗及预防复发至关重要. ALL可通过多种检查进行诊断, 如体检、血液检查、血细胞计数、骨髓造影、腰椎穿刺和骨髓活检[5], 与其他癌症诊断不同的是, 白血病不会产生可以通过医学成像检测到的肿瘤, 这是一个非常主观和耗时的任务[6]. 白细胞通过其深紫色样外观来检测, 但由于其形状和质地的可变性, 分析和进一步处理变得非常复杂, 当前的诊断方法依赖于在显微镜下手动观察细胞的形态变化, 以辨识出数量异常增多的淋巴细胞[7], 分析大量的血液显微镜样品对于医学检测人员来说是一个问题, 可能会导致诊断不够精确[8]. 此外, 一些自身免疫性疾病和感染性疾病(由血细胞或良性淋巴细胞增加引起)的体征类似于ALL, 而这种情况不需要复杂的治疗. 近年来, 伴随计算机视觉和医疗健康领域的进步, 为解决这一问题提供了新的可能性[9].
传统的图像分类任务通常涉及手动设计或选择特征提取方法. 这些方法包括色彩直方图、边缘检测、纹理分析等各种方式. 特征提取后, 使用某种分类器, 如Mohapatra等人[10]提出了通过使用具有形状、纹理和颜色特征的支持向量机(support vector machine, SVM)[11]进行白细胞分类, 提出了一种基于形状和颜色特征以及分类器集成的方法. Singhal等人[12]使用具有局部二进制模式(local binary patterns, LBP)和灰度共生矩阵(gray-level co-occurrence matrix, GLCM)的SVM分类器对ALL进行分类. 其中的挑战性主要在于, 找到捕获图像本质并具有区分度的特征是一个困难且需大量专业知识的过程. 这个过程也可能受到图像光照、角度、尺度等变化的影响, 导致分类效果降低.
相比于传统方法, 深度学习中的卷积神经网络(convolutional neural network, CNN)的优势在于它能自动学习和识别从基本到复杂的图像特征[13]. 给定足够的数据, 深度学习模型逐层抽象并学习高级特征, 消除了手动设计和选择特征的必要性. 如Ali等人[14]结合 EfficientNet和迁移学习进行多类皮肤癌分类, Dafni Rose等人[15]使用最优区域生长分割和MobileNet模型进行乳腺癌识别和分类. 王俊洁等人[16]采用基于Sobel算子的算法, 生成强调细胞边界的标签, 通过计算图像的梯度来提取边缘信息, 增强了深度学习模型对细胞边界的学习能力, 构建了多分支压缩和激励网络模型, 实现蕈样肉芽肿细胞分类. Hosseini等人[17]在设计B-ALL (B细胞急性淋巴细胞白血病)分类程序时, 提出先使用K-means[18]对图像进行聚类处理, 接下来通过添加掩模, 可以去除图像中不必要的部分, 结合CNN模型来实现图像分割, 从而在提高分割效果的同时降低计算资源的需求. 张芳等人[19]提出了深层多尺度聚合3D U-Net网络分割模型, 是一种常用于图像分割任务的神经网络模型, 提升了对小尺度肾肿瘤和肿瘤边缘的分割精度. U-Net[20]在医学图像分割领域特别受欢迎, 用了全卷积网络(FCN)的概念, 并引入了长距离跳跃连接来保证局部和全局特征同时被考虑, 能有效地解决浅层特征和深层语义特征的融合问题. 然而, 在急性淋巴性白血病(ALL)的分类中, 图像上细胞的大小、形态等非常相似, 借助于微小的局部差异才可以区分出种类, 不仅如此, 由于伦理和隐私等问题, 医学图像数据获取困难, 此外深度学习模型理解性、过拟合风险, 以及泛化能力等方面仍面临着重要问题需要解决[21].
针对这些挑战, 本文提出了一种融合注意力与残差网络的淋巴细胞白血病分类网络模型(a dilated pyramid and shuffle-attention integrated model, ADSA-Net). 主要工作如下: 1)采用ResNeXt[22]为主干网络, ResNeXt借鉴了VGG[23]和ResNet[24]的设计理念, 并引入了“Cardinality”概念(即群组卷积的数量), 这有助于增强网络的表达能力. 2)采用图像增强技术以减少背景噪声的影响, 关注感兴趣目标区域. 3)增加空洞金字塔特征提取以更有效地获取ALL细胞的关键信息. 4)添加注意力机制提高网络对重要特征的提取能力.
1 本文方法ADSA-Net的主干网络架构采用了ResNeXt50作为基础, 网络结构如图1所示. 首先, 经过数据增强, 包括对比度增强, 图像锐化, 减少背景的影响, 进入7×7卷积, 对比ResNeXt原模型, 删除了最大池化层, 减少了图像特征流失, 用4个Layer提取特征, 对应的残差块的数量分别为3, 4, 6, 3. 图像通过网络的第1个Layer进行特征提取, 获得包含丰富底层信息的特征映射, 这些特征信息对ALL细胞识别起着关键作用, 所以在得到的特征提取图后面添加ADSPP模块, 利用更加广阔感受野取得更多信息, 并将得到的特征图送入Layer3, 4中, 将处理得到的多尺度语义特征结合后面特征图到一起, 融合更多信息; 接着, 在Layer1, 2, 3后面添加SAE模块, 调节模型对不同特征的注意力, 帮助模型聚焦更加重要的区域. 最后, 将上述模块得到的特征映射信息输入平均池化层, 经过全连接层输出得到分类结果.
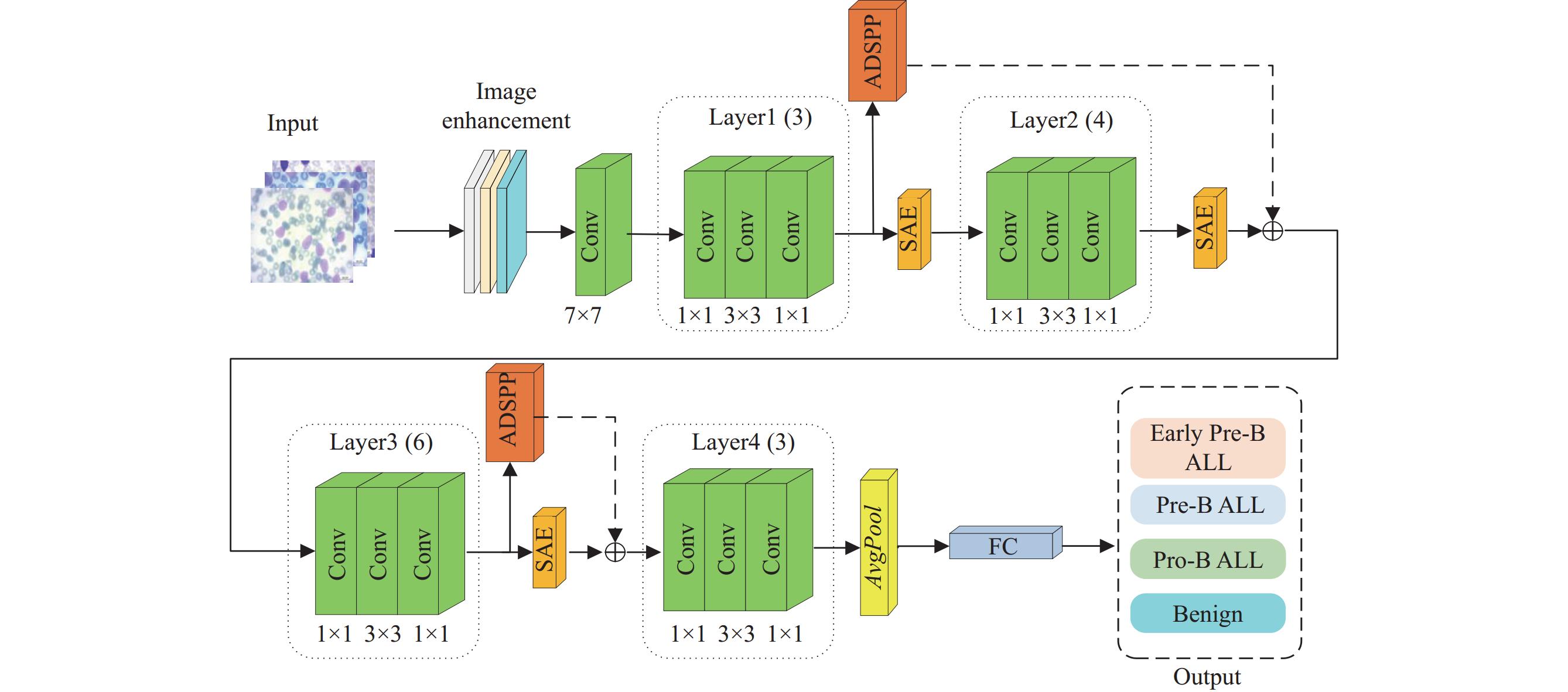
|
图 1 ADSA-Net网络结构图 |
1.1 数据增强
图像增强技术是在保持图像内容的前提下, 通过调整对比度, 图像锐化等方式, 使图像更具有视觉效果或更适合特定任务. 在对ALL细胞进行分类时, 需清晰地看到细胞的特征, 这就需进行对比度的调整. 有效的对比度增强能提高细胞图像质量, 从而帮助改善模型对细胞分类的性能. 自适应Gamma矫正(adaptive Gamma correction)[25]是传统的Gamma矫正方法的扩展. 在自适应Gamma 矫正中, Gamma值
| O=I11+m−ˉI | (1) |
其中,
锐化是一种增强图像细节的技术, 使得图像的边缘和转换变得更加清晰[26]. 在过程中, 需要应用卷积运算. 给定一个核(或者叫滤波器)
| (I⊗H)(x,y)=a∑i=−ab∑j=−bI(x−i,y−j)⊗H(i,j) | (2) |
其中,
空洞空间金字塔池化[27]是一种用于捕获多尺度信息的卷积策略, 基本上通过使用不同比例的空洞卷积(也称膨胀卷积)来实现[28], 模块结构图如图3所示. 相对于普通卷积, 空洞卷积除了卷积核的大小外, 还有一个空洞(膨胀)率, 用来表示展开的大小. 膨胀卷积与一般卷积的相似之处在于卷积核的大小相同, 即神经网络中的参数个数保持不变, 不同之处在于膨胀卷积具有更大的接受野, 减少图像信息丢失, 使网络在获得了更多的数据, 同时保证了图像信息的连续性. 相较标准卷积, 卷积层感受野计算公式为:
| rn=rn−1+(k−1)∏n−1i=1Si | (3) |
其中,

|
图 2 图像增强效果图 |

|
图 3 自适应空洞金字塔模块 |
自适应空洞金字塔池化(adaptive dilated spatial pyramid pooling, ADSPP)模块的主要思想是保留原始全卷积网络的空洞卷积, 使用不同的空洞率(dilation rate)来处理不同尺度的特征, 更好地捕捉不同尺度的特征. 通过改变积核中空洞的大小, 可以有效地扩大感受野, 从而捕获图像中更大范围的信息[29]. 对于空洞卷积可以表示为:
| [xout(i,j)=∑m,nxin(k⋅m,k⋅n)⋅w(m,n)] | (4) |
其中,
传统的注意力模块中, 采用不同大小的卷积核或者全连接层从输入特征图中获取全局的上下文信息, 对输入特征图进行权重调整[31]. 这类自我注意力机制对于某些需要精细化理解的任务确实非常有效, 但是对于一些类似于图片分类的任务, 这样可能会带来过多的参数和复杂度.
本文根据SA (shuffle attention)新型注意力[32]思想, 以更好地处理图像的各种扭曲和平移等空间变化, 增强模型对输入内容的理解能力, 有助于增加模型的注意力表达能力, 并提高模型的预测性能. 模块结构如图4所示.

|
图 4 SAE注意力机制 |
首先, 输入特征图
| x=Reshape(x,(b∗self.groups,h,w)) | (5) |
其中, *表示元素相乘操作.
将其平均分割成两部分
| xn=x0∗Sigmoid(cweight⋅AvgPool(x0)+cbias) | (6) |
| xs=x1∗Sigmoid(sweight⋅GroupNorm(x1)+sbias) | (7) |
使用一维卷积将以上生成的
| xsconv=Conv1d([xn;xs]) | (8) |
将得到的特征图送入预定义的空间变换器网络[35](spatial Transformer network, STN)中, 主要包括3个步骤: 本地化网络(localization network)、网格生成器(grid generator)和采样器(sampler).
本地化网络: 此网络接收输入特征图并预测出透视变换矩阵
| θ=floc(U) | (9) |
网格生成器(grid generator): 网格生成器用于根据已经计算好的线性变换参数
| [x′iy′i]=[θ11θ12θ13θ21θ22θ23][xiyi1] | (10) |
采样器: 采样器根据生成的采样网格来从输入特征图中抽取值并双线性插值.
| Vi=H∑nW∑mUnmmax(0,1−|x′i−m|)max(0,1−|y′i−n|) | (11) |
这里
| xsconv_stn=STN(xsconv) | (12) |
最后再把
| out=Concatenate[xs;xsconv_stn] | (13) |
通过封装好的shuffle (打乱)操作对输出进行通道排列使得相互关联的通道在一起, 最后输出的就是最终结果.
2 实验结果及分析 2.1 实验环境及参数设置本文实验环境为Ubuntu 20.04操作系统, 硬件配置为Intel(R) Xeon(R) CPU E5-2686 v4、64 GB内存和Nvidia GeForce GTX RTX 3090显卡. 采用Python语言, 深度学习模型基于PyTorch框架, 集成开发环境为PyCharm. 为了保证模型处于最优状态, 在多次实验验证后设置参数如下: 将网络训练时期的迭代次数设置为100次, 批量大小设置为32次, 并采用Adam优化器(其初始学习率为0.0001).
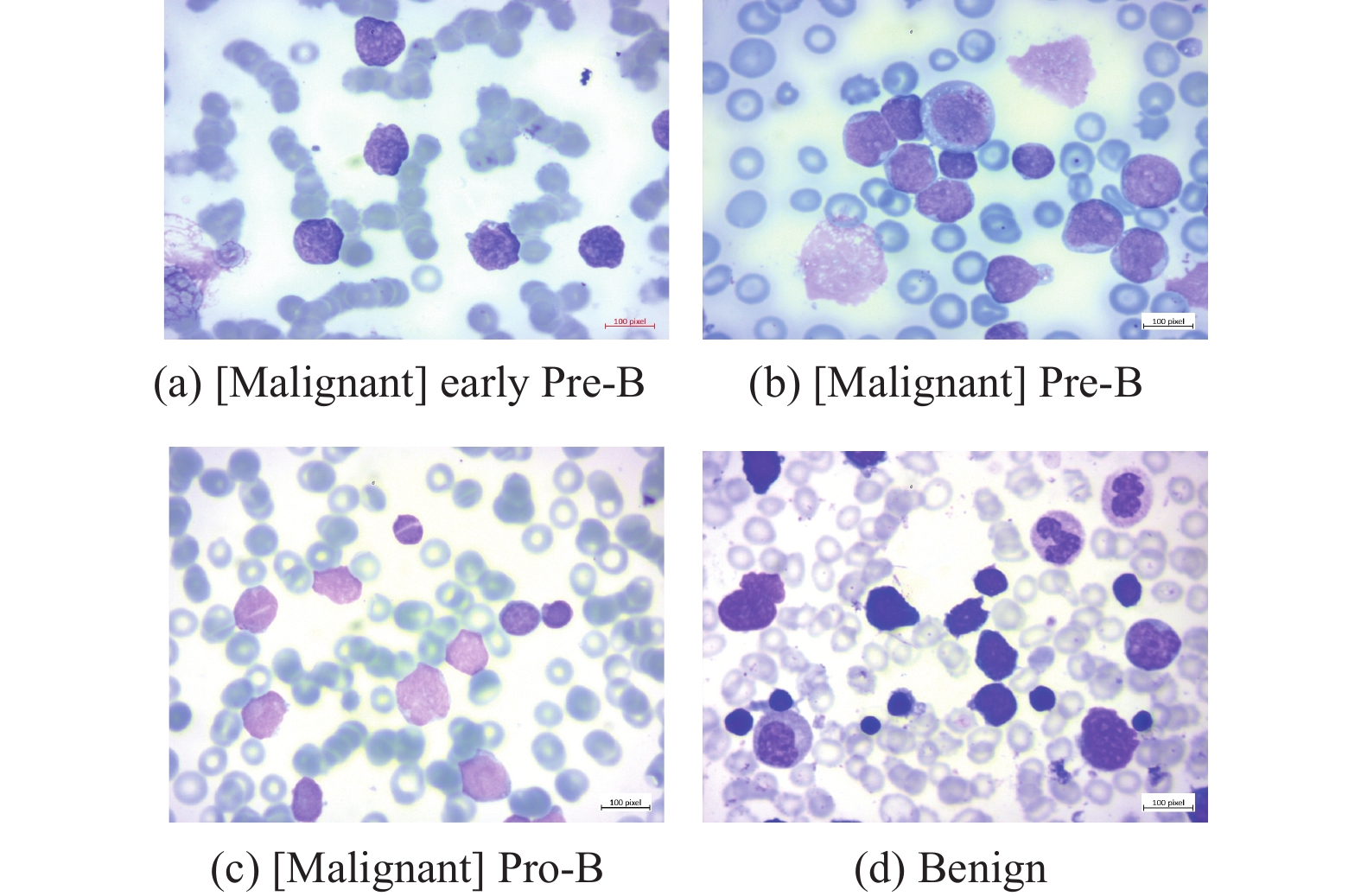
2.2 数据集本文在blood cells cancer (ALL)大型血细胞数据集图像数据集上进行实验, 该数据集的图像是在 Taleqani 医院(伊朗德黑兰)的骨髓实验室准备. 包含89名疑似 ALL 患者的3242张PBS图像, 其血样由熟练的实验室工作人员制备和染色. 该数据集分为良性和恶性两类. 第1类(良性)含有与B-ALL病例非常相似的造血原, 但这种造血前体是良性的, 不需要化疗, 通常会自行消失在血液中. 后者是具有3种恶性淋巴细胞亚型的ALL组: 早期Pre-B、Pre-B和Pro-B ALL. 所有图像均使用蔡司相机在显微镜下以 100倍放大倍数拍摄并保存为JPG文件. 数据集具体名称和数量如表1所示, 各类图像如图5所示.
| 表 1 数据集及其子类 |
|
图 5 数据集中4类图像 |
2.3 数据集预处理
将每类细胞图像样本按75%:15%:15% 划分为训练集、验证集和测试集. 对于深度卷积神经网络的优异分类性能需要经过大量的数据训练才能得到体现. 由此. 本文采用随机旋转. 添加噪声和水平翻转等. 对训练集样本进行数据增强. 将训练集扩充到11340张. 并将数据集图像转换成 224×224×3的统一尺寸.
2.4 评价指标本文中通过构建混淆矩阵来可视化分类模型在已知真实值的测试数据集上的性能, 采用临床相关的统计量评估提出的分类模型. 召回率、准确率和准确率计算这些评价指标需要以下参数: 真阳性(TP):该方法对阳性病例的正确预测次数; 真阴性(TN): 阴性病例的正确预测数; 假阳性(FP): 阳性病例的错误预测数; 假阴性(FN): 通过该方法对阴性情况的错误预测的数量.
Accuracy是一个用于分类模型的指标, 指预测正确数量所占总量的比例, 公式如下:
| Accuracy=TP+TNTP+FP+TN+FN×100% | (14) |
Precision指评估模型预测正确的类别数量占预测该类别总数量的比值, 公式如下:
| Precision=TPTP+FP×100% | (15) |
Recall指在评估模型预测正确的类别数量占该类别实际数量的比值, 公式如下:
| Recall=TPTP+FN×100% | (16) |
F1-score指Precision和Recall的加权调和平均值, 公式如下:
| F1-score=2×Precision×RecallPrecision+Recall | (17) |
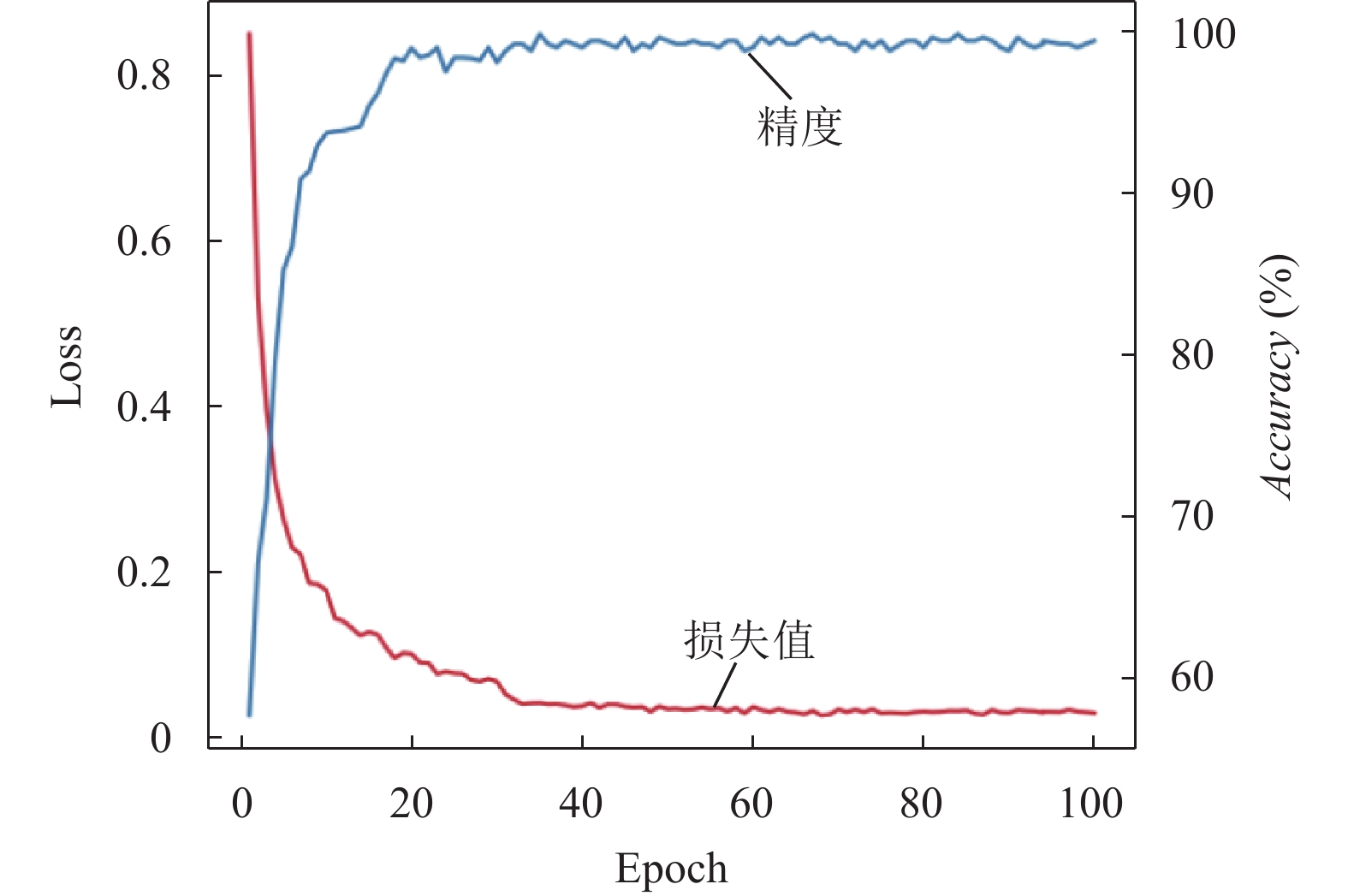
图6可以看出, 随着训练轮次的增加, 训练集的分类精度逐渐提高, 损失值逐渐减少并趋于稳定, 说明模型分类结果和实际结果的误差逐渐降低, 此外, 也说明了模型体现出了较好的泛化能力.
为了进一步验证本文网络模型的有效性, 将其与经典的图像分类网络VGG19, ResNet34等以及近几年流行的EfficientNet, Swin-Transformer等进行了对比. 这些模型在过去被广泛应用于各类图像分类任务, 并显示出良好的性能. 从表2可以看出, 相较于其他本文提出的网络模型表现出更高的准确性, 可以更准确地识别出正向类别, 误报率远低于其他模型, 在召回率方面也有显著的提高, 对于那些易于被漏掉的类别, 识别更加完整, 考虑到精确度和召回率的综合性能, F1-score比其他模型也有提高.
|
图 6 训练过程的损失和精度 |
| 表 2 不同网络模型性能对比(%) |
PR曲线(precision-recall curve)主要用于评估分类模型在各种阈值下的性能. 以召回率作为横坐标, 准确率作为纵坐标. 图7为模型进行分类后, 各类细胞的PR图.
混淆矩阵是一种常用的多分类结果的可视化工具, 可用于评估分类模型的性能. 混淆矩阵的行代表真实类别, 列代表预测类别. 对于每个类别, 混淆矩阵记录了模型的预测结果中的正确分类和错误分类. 对角线上的元素表示每个类别被正确预测的数量. 这些值越大, 表示模型对于这些类别的分类结果可靠性越高.
图8为网络验证时生成的混淆矩阵, 把模型预测的结果和实际标签进行对比, 然后按照真阳性(TP)、假阳性(FP)、真阴性(TN)、假阴性(FN)这4类情况进行统计, 可以看出模型性能较优, 但是还存在少量错分样本, 由于ALL细胞图像特征相似、临床特征也非常相似, 导致很难区分.
2.6 消融实验为了更好地展示出加入ADSPP和SAE注意力模块的重要性, 进行了消融实验, 验证各个模块对ALL分类结果的影响, 在其每个实验中, 都设置相同的超参数. 将ResNeXt50为基准(base), 完成此次消融实验, 结果为表3所示.

|
图 7 PR图 |

|
图 8 混淆矩阵 |
| 表 3 消融实验结果对比(%) |
在表3中可见, 将ADSPP和SAE注意力机制分别引入模型时, 网络对ALL分类的性能均有了提高. 说明ADSPP能够帮助模型更好地理解图像中的空间结构信息, 并有效地增强其对 ALL 的识别能力, SAE注意力机制使得模型能更专注于图像中与分类最相关的区域. 同时引入ADSPP和注意力机制, 我们观察到了比单独使用这些策略时更优的性能. 这相当于综合利用了ADSPP的多尺度信息以及注意力机制的区域聚焦, 使得模型在解析和理解输入图像时更为高效和精准.
3 结论与展望本文在处理ALL分类问题的研究过程中, 我们采用了以ResNeXt50网络为基础的分类网络模型. 首先, 应用数据增强技术对输入图像进行预处理, 有效降低背景噪声, 并将关注焦点集中在主要的特征上. 接着, 通过自适应空洞金字塔特征提取方法, 一定程度地改进了模型对诸如尺度和上下文信息等重要细节的感知能力, 从而优化了特征挖掘过程. 此外, 经过引入改进后SA注意力机制, 显著增强了模型对关键区域的关注度, 为模型提供了更明确、更有针对性的学习方向. 尽管目前方案在一定程度上改善了ALL分类的性能, 但仍需要进一步完善和调整以适应不同类型数据或更复杂情形的挑战.
| [1] |
Das PK, Meher S. An efficient deep convolutional neural network based detection and classification of acute lymphoblastic leukemia. Expert Systems with Applications, 2021, 183: 115311. DOI:10.1016/j.eswa.2021.115311 |
| [2] |
Brady SW, Roberts KG, Gu ZH, et al. The genomic landscape of pediatric acute lymphoblastic leukemia. Nature Genetics, 2022, 54(9): 1376-1389. DOI:10.1038/s41588-022-01159-z |
| [3] |
Force LM, Abdollahpour I, Advani SM, et al. The global burden of childhood and adolescent cancer in 2017: An analysis of the global burden of disease study 2017. The Lancet Oncology, 2019, 20(9): 1211-1225. DOI:10.1016/S1470-2045(19)30339-0 |
| [4] |
Hu YF, Zhang XP, Zhang AJ, et al. Global burden and attributable risk factors of acute lymphoblastic leukemia in 204 countries and territories in 1990–2019: Estimation based on global burden of disease study 2019. Hematological Oncology, 2022, 40(1): 93-105. DOI:10.1002/hon.2936 |
| [5] |
Rodrigues LF, Backes AR, Travençolo BAN, et al. Optimizing a deep residual neural network with genetic algorithm for acute lymphoblastic leukemia classification. Journal of Digital Imaging, 2022, 35(3): 623-637. DOI:10.1007/s10278-022-00600-3 |
| [6] |
Ghaderzadeh M, Asadi F, Hosseini A, et al. Machine learning in detection and classification of leukemia using smear blood images: A systematic review. Hindawi Limited, 2021, 2021: 9933481. DOI:10.1155/2021/9933481 |
| [7] |
Dasariraju S, Huo M, Mccalla S. Detection and classification of immature leukocytes for diagnosis of acute myeloid leukemia using random forest algorithm. Bioengineering, 2020, 7(4): 120. DOI:10.3390/bioengineering7040120 |
| [8] |
Ghaderzadeh M, Aria M, Hosseini A, et al. A fast and efficient CNN model for B-ALL diagnosis and its subtypes classification using peripheral blood smear images. International Journal of Intelligent Systems, 2022, 37(8): 5113-5133. DOI:10.1002/int.22753 |
| [9] |
张雪怡, 孙博, 陈癸霖, 等. 计算机视觉在医学图像中的应用及其对罕见肌肉骨骼疾病的影响. 罕见病研究, 2023, 2(4): 589-595. |
| [10] |
Mohapatra S, Samanta SS, Patra D, et al. Fuzzy based blood image segmentation for automated leukemia detection. Proceedings of the 2011 International Conference on Devices and Communications. Mesra: IEEE, 2011. 1–5. [doi: 10.1109/ICDECOM.2011.5738491]
|
| [11] |
Chandra MA, Bedi SS. Survey on SVM and their application in image classification. International Journal of Information Technology, 2021, 13(5): 1-11. DOI:10.1007/s41870-017-0080-1 |
| [12] |
Singhal V, Singh P. Local binary pattern for automatic detection of acute lymphoblastic leukemia. Proceedings of the 20th National Conference on Communications (NCC). Kanpur: IEEE, 2014. 1–5. [doi: 10.1109/NCC.2014.6811261]
|
| [13] |
Li ZW, Liu F, Yang WJ, et al. A survey of convolutional neural networks: Analysis, applications, and prospects. IEEE Transactions on Neural Networks and Learning Systems, 2022, 33(12): 6999-7019. DOI:10.1109/TNNLS.2021.3084827 |
| [14] |
Ali K, Shaikh ZA, Khan AA, et al. Multiclass skin cancer classification using EfficientNets—A first step towards preventing skin cancer. Neuroscience Informatics, 2022, 2(4): 100034. DOI:10.1016/j.neuri.2021.100034 |
| [15] |
Dafni Rose J, VijayaKumar K, Singh L, et al. Computer-aided diagnosis for breast cancer detection and classification using optimal region growing segmentation with MobileNet model. Concurrent Engineering, 2022, 30(2): 181-189. DOI:10.1177/1063293X221080518 |
| [16] |
王俊洁, 徐聪聪, 赵增瑞, 等. 多分支压缩和激励网络的蕈样肉芽肿细胞分类. 北京邮电大学学报, 2022, 45(4): 31-36. |
| [17] |
Hosseini A, Eshraghi MA, Taami T, et al. A mobile application based on efficient lightweight CNN model for classification of B-ALL cancer from non-cancerous cells: A design and implementation study. Informatics in Medicine Unlocked, 2023, 39: 101244. DOI:10.1016/j.imu.2023.101244 |
| [18] |
Ikotun AM, Ezugwu AE, Abualigah L, et al. K-means clustering algorithms: A comprehensive review, variants analysis, and advances in the era of big data. Information Sciences, 2023, 622: 178-210. DOI:10.1016/j.ins.2022.11.139 |
| [19] |
张芳, 郝思敏, 耿磊. 基于深层多尺度聚合3D U-Net的肾脏与肾肿瘤分割方法. 天津工业大学学报, 2023, 42(6): 84-90. |
| [20] |
Jena B, Jain S, Nayak GK, et al. Analysis of depth variation of U-NET architecture for brain tumor segmentation. Multimedia Tools and Applications, 2023, 82(7): 10723-10743. DOI:10.1007/s11042-022-13730-1 |
| [21] |
马梓博, 米悦, 张波, 等. 面向异质性医学图像处理的深度学习算法综述. 软件学报, 2023, 34(10): 4870-4915. DOI:10.13328/j.cnki.jos.006680 |
| [22] |
Xie SN, Girshick R, Dollár P, et al. Aggregated residual transformations for deep neural networks. Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017. 5987–5995.
|
| [23] |
Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition. Proceedings of the 3rd International Conference on Learning Representations. San Diego: ICLR, 2015.
|
| [24] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [25] |
侯利霞, 聂丰英, 万里勇. 多尺度自适应Gamma矫正的低照图像增强. 云南大学学报(自然科学版), 2023, 45(1): 57-66. |
| [26] |
路琨婷, 费蓉蓉, 张选德. 融合卷积神经网络的遥感图像全色锐化. 计算机应用, 2023, 43(9): 2963-2969. |
| [27] |
尹稳, 周冬明, 范腾, 等. 基于密集空洞空间金字塔池化和注意力机制的皮肤病灶图像分割方法. 生物医学工程学杂志, 2022, 39(6): 1108-1116. |
| [28] |
Chen LC, Papandreou G, Kokkinos I, et al. DeepLab: Semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4): 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [29] |
刘糠继, 时培成, 齐恒, 等. 基于多分支空洞卷积与自适应特征融合的FCOS目标检测算法. 井冈山大学学报(自然科学版), 2023, 44(6): 75-83. |
| [30] |
董婷, 魏珑, 叶晓丹, 等. 基于空洞空间卷积池化金字塔结构和注意力机制的全卷积残差网络磨玻璃肺结节分割方法. 生物医学工程学杂志, 2022, 39(3): 441-451. |
| [31] |
肖斌, 郭经伟, 张兴鹏, 等. 基于融合池化和注意力增强的细粒度视觉分类网络. 模式识别与人工智能, 2023, 36(7): 661-670. DOI:10.16451/j.cnki.issn1003-6059.202307007 |
| [32] |
Zhang QL, Yang YB. SA-Net: Shuffle attention for deep convolutional neural networks. Proceedings of the 2021 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). Toronto: IEEE, 2021. 2235–2239. [doi: 10.1109/ICASSP39728.2021.9414568]
|
| [33] |
汪亚航, 宋晓宁, 吴小俊. 结合混合池化的双流人脸活体检测网络. 中国图象图形学报, 2020, 25(7): 1408-1420. |
| [34] |
虞欣, 郑肇葆, 孟令奎, 等. 遥感影像场景识别的贝叶斯共轭批次归一化方法. 武汉大学学报(信息科学版), 1–17. (2023-12-12). [doi: 10.13203/j.whugis20220632]
|
| [35] |
Zhong ZL, Li Y, Ma LF, et al. Spectral-spatial Transformer network for hyperspectral image classification: A factorized architecture search framework. IEEE Transactions on Geoscience and Remote Sensing, 2022, 60: 5514715. |
 2024, Vol. 33
2024, Vol. 33



