2. 重庆师范大学 重庆国家应用数学中心, 重庆 401331
2. National Center for Applied Mathematics in Chongqing, Chongqing Normal University, Chongqing 401331, China
多器官医学图像的自动分割在计算机辅助诊断中有着重要作用[1]. 它通过提取器官区域的信息, 辅助医生做出适当的诊断. 自21世纪来, 随着医学成像领域的不断发展, 由此产生了大量的医学图像, 从中手动分割器官会耗费大量时间, 且分割同一器官时, 不同医生也会产生判断偏差[2,3]. 因此, 在医学图像中, 多器官分割仍然是一项具有挑战性的任务.
CNN已被广泛应用于医学图像分割领域[4]. Ronneberger等人[5]提出的U-Net采用端到端的网络形式, 编码器采用卷积层和池化层, 分层提取图像的局部特征. 解码器采用上采样操作, 将特征图恢复到原始图像的尺寸. 为提升医学图像分割任务的精度, U-Net通过跳跃连接, 将编码器的特征图与解码器的特征图相融合, 从而保留更多的细节信息. U-Net中的这种跳跃连接, 大幅度减少带标签的训练集. 后续的U-Net变种模型, 如Zhou等人[6]提出的U-Net++, 通过加入密集连接方式重新设计编码器-解码器结构. Huang等人[7]提出的UNet 3+, 通过全尺度跳跃连接, 将来自不同尺度特征图的低级细节与高级语义相结合的一种编码器-解码器结构. 这些模型通过捕获目标特征以及位置信息对目标区域进行分割, 在医学图像分割领域取得了一定的效果, 但由于感受野限制和平移不变性所带来固有的局限性, 导致CNN在全局特征能力的建模和位置信息的识别是存在缺陷的. 在一些复杂的场景中, 受限的感受野不足以捕获全局特征信息, 平移不变性会使得模型对物体位置信息的提取能力有所下降, 从而导致分割性能下降[8,9].
Dosovitskiy等人[10]提出的视觉Transformer (vision Transformer, ViT) 克服了CNN的缺点. ViT借助独特的自注意力机制 (self-attention, SA), 可以对输入位置与其他位置相关性进行建模, 使Transformer能够轻松捕捉不同位置之间的长距离依赖关系, 有助于全局特征信息的提取[11,12]. Wang等人[13]在Transformer中引入CNN中金字塔结构, 以处理不同尺度的特征. 与传统的ViT不同, 金字塔结构允许PVT在不同尺度上执行特征提取和处理, 从而提高多尺度目标分割的性能. 虽然ViT缓解了CNN中感受野限制和平移不变性的局限性, 但ViT在捕获局部特征表示上表现不佳, 将Transformer应用于特定情况下的医学图像分割会面临新的挑战[14,15]. 为达到与CNN相似性能, 需要大量数据进行训练. 此外, 由于SA需要将每个位置与其他位置计算相似度分数, 所带来的计算复杂度是二次方[16]. 在医学图像分割任务中, 面对高维的医学图像数据时, 会降低模型的处理速度.
Liu等人[17]提出的Swin Transformer降低了ViT的二次方计算复杂度. 作者采用一种移动窗口划分方法, 将SA限制在一个局部的窗口中, 捕获局部特征的同时降低自注意力的计算复杂度. 尽管Swin Transformer有能力在对全局上下文表示进行建模的同时, 提高计算效率, 但对局部特征能力的提取仍然受到限制. Chen等人[18]提出的TransUNet缓解Transformer在医学图像中的弱局部表示. TransUNet是首个将Transformer应用于医学图像分割领域的U型网络, 编码器采用CNN和Transformer提取局部和全局特征, 解码器通过上采样与高分辨率的局部特征相融合, 以达到精准定位的效果. 然而, TransUNet的缺点是参数量大和计算效率低.
针对CNN和Transformer的上述缺点, 设计多级特征交互融合Transformer模型. 该模型首先通过CNN提取分级局部特征, 首级局部特征经Swin Transformer输出多级全局特征, 随后将同级的全局和局部特征进行加性融合, 融合后的特征再经过全局和局部特征增强 (global and local feature enhancement, GLFE) 模块, 在通道上做深度卷积降低运算参数量, 其次在X方向和Y方向进行聚合增强特征表示, 再采用逐点卷积对特征进行升维. 多个GLFE模块的输出采用Shen等人[19]提出的高效注意力 (efficient attention, EA) 和Lin等人[20]提出的交叉注意力 (cross attention, CA) 的双注意力机制组成的多级融合 (multi-level feature fusion, MLFF) 模块进行特征交叉融合. 最后, 通过上采样操作将交叉融合后的特征映射成原始图像尺寸, 通过分割头输出分割掩码.
● 设计GLFE模块, 使模型可以同时捕获跨通道信息以及长距离位置信息. 仿真实验表明, 加入GLFE模块在平均DSC和平均HD95系数上提升0.58%和降低9.01 mm, GLFE模块可以提升模型的定位能力.
● 设计MLFF模块, 使模型在降低计算复杂度的同时计算多级特征向量之间的注意力权重. 仿真实验表明, 添加MLFF模块的模型在平均DSC和平均HD95系数上提升2.67%和降低8.53 mm, MLFF模块可以提升模型识别目标的能力和定位能力.
● 提出一种多级特征交互Transformer的多器官医学图像分割模型. 实验结果表明所提模型在Synapse和ACDC数据集上与代表性模型相比, 在平均DSC系数和平均HD95系数上具有竞争性优势.
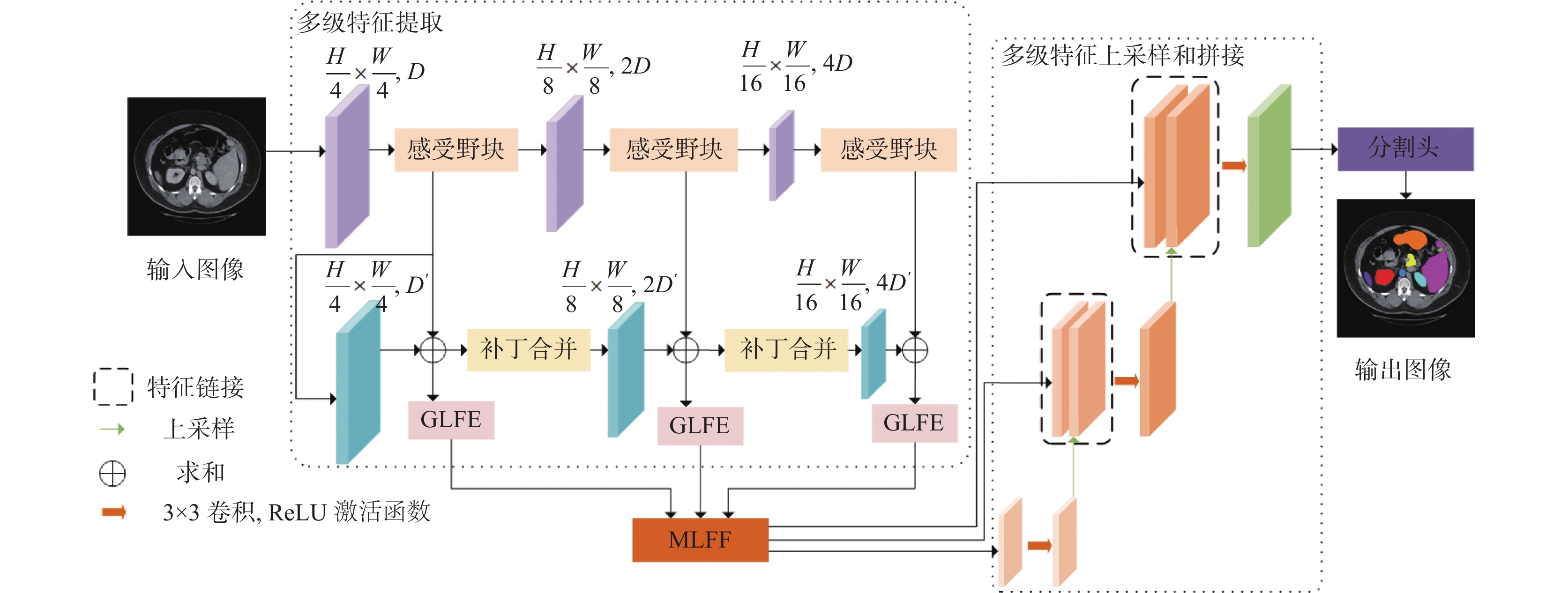
2 所提模型结构所提模型的网络结构如图1所示, 其中浅紫色和深蓝色块分别表示CNN和Swin Transformer层级. 首先编码器采用多级特征提取结构提取局部和全局特征信息, 通过GLFE模块将上述同级特征交互后再增强特征表示. 其次使用MLFF模块计算不同级特征之间的注意力权重, 捕获上下文语义信息. 最后, 将MLFF的特征映射经过级联上采样器和分割头, 输出分割掩码.
2.1 多级特征提取结构如图1所示, 多级特征提取结构由CNN和Swin Transformer组成. CNN和Swin Transformer分别包括3个不同的层级, 通过增大感受野的方式对图像进行分级特征提取.
|
图 1 整体网络框架图 |
首先使用不同分辨率的CNN作为多级特征提取器, 以提取不同层级的特征图. 给定一个具有空间维度
在首层局部特征的基础上使用Swin Transformer[17]输出多级全局特征. ViT[10]主要由L层多头自注意力 (multi-head self-attention, MSA) 和多层感知机 (multi-layer perceptron, MLP) 组成, 在MSA和MLP之前采用层归一化 (LayerNorm, LN) 操作. 与ViT不同的是, Swin Tranformer模块采用滑动窗口机制构建窗口多头自注意力 (window based MSA, W-MSA) 和滑动窗口多头自注意力 (shifted window based MSA, SW-MSA). 在W-MSA中, 在
| ˆzl=W-MSA(LN(zl−1))+zl−1 | (1) |
| zl=MLP(LN(ˆzl))+ˆzl | (2) |
| ˆzl+1=SW-MSA(LN(zl))+zl | (3) |
| zl+1=MLP(LN(ˆzl+1))+ˆzl+1 | (4) |
其中,
常见的特征融合操作有拼接, 相加, 相乘操作, 但在某些条件下会带来一系列问题. 例如, 相加操作是将两个相同维度的特征进行叠加, 通过增加描述图像的特征信息量, 提高模型的表示能力. 同时会使得不正确的特征信息被加入融合后的特征中, 导致一些重要特征的丢失, 从而降低模型的分割性能.
GLFE模块如图2所示, 实现降低卷积计算的参数量的同时整合空间位置信息. 先用深度卷积对特征进行降维, 降维后的特征在X方向和Y方向通过全局平均池化压缩成一维向量, 最后使用逐点卷积对特征进行升维. 压缩成一维向量的计算公式如下:
| zhc(h)=1W∑0⩽i⩽Wxc(h,i) | (5) |
| zwc(w)=1H∑0⩽j⩽Hxc(j,w) | (6) |
其中,

|
图 2 全局和局部特征增强模块 |
2.3 多级融合模块
不同级别的特征对应不同的语义信息, 如何将这些语义信息进行关联, 是模型捕获目标区域信息的关键. 针对GLFE输出的多级特征信息, 设计多级融合模块, 使模型同时捕获输入特征映射的空间重要性和关联不同特征映射之间的语义内容.
图3所示为MLFF模块. 增强后的多级特征作为输入, 采用EA和CA的双注意力结构捕获输入特征映的空间重要性的同时计算注意力权重. 标准的SA的计算复杂度是
| EA(Q,K,V)=ρq(Q)(ρk(K)TV) | (7) |
其中,
| CLSl=GAP(LN(Pl)) | (8) |
其中,
| zl=[fl(CLSl),Pl+1] | (9) |
| pl=fl(CLSl)+EA(LN(zl)) | (10) |
| pl′=fl′(pl)+CA(LN(pl)) | (11) |
| Zl=[Pl,gl′(pl′)] | (12) |
其中,
MLFF模块的输出为3个特征映射

|
图 3 多级特征融合模块 |
3 实验 3.1 数据集和评价指标
Synapse: Synapse数据集由30例病人共3779张腹部轴向临床CT图像构成. CT体积由85–198张切片组成. 为保证实验的一致性, 与TransUNet[18]的数据集划分设置相同, 18个病例用于模型的训练, 12个病例用于模型的测试.
ACDC: ACDC数据集由100个心脏磁共振图像序列构成. 每个序列包含收缩末期和舒张末期, 包括左心室, 右心室和心肌等心脏结构. 为保证实验的一致性, 与TransUNet[18]的数据集划分设置相同, 70样本用于训练, 10个样本用于验证和20个样本用于测试.
为了分析所提模型在Synapse和ACDC数据集上的实验结果. 研究中使用平均骰子相似系数 (Dice similarity coefficient, DSC) 和平均95%的豪斯多夫距离 (Hausdorff distance, HD95) 作为评估指标.
DSC: Dice相似系数是一种用于比较两个样本相似度的指标. 在医学图像分割领域, 它的取值范围为0–1, 其中1表示两个样本完全一致, 0表示两个样本完全不同. 在医学图像分割中, Dice相似系数用于评估预测结果与真实结果之间的重叠程度, 即两者之间的交集与并集的比值. 较高的Dice相似系数通常意味着较好的分割效果. 计算公式如下:
| Dice(P,T)=2|P∩T||P|+|T| | (13) |
其中,
HD95: HD是一种用于比较两个集合之间相似度的度量方法, 其度量单位是mm. 在医学图像分割领域, HD被广泛用于评估预测分割结果与真实分割结果之间的相似度, 特别是用于评估分割结果的边界或轮廓的准确性. 计算过程如式(14)所示. HD95是通过HD结果值乘以95%, 目的是消除离群值的一个非常小的子集的影响.
| H(X,Y)=max{h(X,Y),h(Y,X)} | (14) |
其中,
所有实验均在Linux操作系统上进行, CPU配置为i7-12600KF, 显卡配置为RTX 3060. 深度学习框架为PyTorch 1.10和Python 3.7, CUDA版本为11.2. 训练分割模型时, 通过翻转和旋转数据集中的样本, 改善数据多样性; 从ImageNet上获得CNN和Swin Transformer模块的预训练权重, 使用该权重对所提模型进行初始化; 模型输入图像大小为224×224, 训练期间的批量大小和学习率分别为8和0.01; 所提模型使用SGD算法进行优化, 其中动量设置为0.9, 权重衰减设置为0.000 1.
3.3 对比实验为了验证所提模型在多器官医学图像分割任务上的有效性. 与近两年较新的医学图像分割网络 (如MT-Unet, LGNet和RFE-UNet) 进行比较. 所有实验都在统一的基准和评价指标上进行, 不同的模型在两个数据集上的实验结果如表1和表2所示.
| 表 1 所提模型在Synapse数据集上的实验结果 |
| 表 2 所提模型在ACDC数据集上的实验结果 (%) |
表1为所提模型在Synapse数据集上实验结果, 加粗字体表示最佳结果. 所提模型在平均DSC系数上到达最高的80.17%, 在平均HD95系数上降低为19.20 mm. 所提模型相比于基于CNN的U-Net, 在平均DSC系数上提升3.32%. 在平均HD95系数上降低20.50 mm. 相比于基于Transformer的Swin-Unet, 在平均DSC系数上提升1.04%, 在平均HD95系数上降低2.35 mm. 相比于基于CNN-Transformer的TransUNet, 平均DSC系数上分别提升2.69%, 在平均HD95系数上降低12.49 mm. 实验结果表明, 所提模型在这两个评估指标上都表现出优秀的性能, 特别是在脾脏分割上优于其他模型, 而在胰腺和胃的分割上也优于大多数模型.
表2为所提模型在ACDC数据集上的实验结果, 表中加粗字体表示最佳结果. 所提模型在平均DSC系数上取得最高的值为91.07%. 对比基于Transformer的Swin-Unet, 在平均DSC系数提升3%, 对比基于CNN-Transformer混合所提模型的TransUNet和HiFormer, 平均DSC系数分别提升1.36%和1.87%. 实验结果表明, 所提模型对心脏数据集的分割精度更高.
3.4 可视化结果及分析图4为部分多器官分割实例的定性结果, 图中红色矩形框中的分割效果差异更大. 首先, 与基于混合架构的模型相比, 基于Transformer的模型在捕捉局部细节方面较弱. 在病例2中, Swin-Unet在分割胃和胰腺方面表现不佳, 主要因为基于Transformer的模型在建模过程中强调上下文信息, 而忽略局部细节的重要性. 因此, 基于Transformer的模型会丢失局部信息, 进而影响分割性能. 其次, 基于混合架构的模型直接将局部特征与全局特征相结合, 这会引入信息冗余或信息丢失, 导致模型分割效果不好, 如TransUNet对胃和胰腺的分割是残缺的. 最后, 考虑到器官的复杂性和多样性, 局部特征和全局特征的融合对于优化分割性能至关重要. 与其他模型相比, 所提模型显示出优秀的分割结果. 首先, 通过GLFE模块增强特征表示. 其次, MLFF模块在允许不同分辨率的局部和全局特征融合. 特别是, 该模型在精确分割复杂结构方面表现出色, 从而产生更精确的分割结果. 在4个案例中, 所提模型在胃、脾脏和胰腺分割上具有优势, 与表1中的结果相符合.
图5为部分心脏分割实例的定性结果, 图中红色矩形框中的分割效果差异更大. 首先基于混合架构的模型, 在有效融合局部和全局特征方面面临挑战. 例如, 在病例1中, TransUNet对于收缩末期的右心室分割是不足的. HiFormer缓解了这个问题, 它通过DLF模块巧妙地融合局部和全局特性. 其次基于Transformer的模型, 由于局部特征相关性建模的不足, 往往会出现分割错误. 在案例2中, DAE-Former对于收缩末期的右心室分割是残缺的. Swin-Unet和MT-Unet对收缩末期的右心室分割不够准确. 最后所提模型在分割舒张末期和收缩末期的心肌方面表现优秀, 与表2中的结果一致.

|
图 4 Synapse数据集上可视化结果比较 |

|
图 5 ACDC数据集上的可视化结果比较 |
3.5 消融实验
为了验证所设计的MLFF模块和GLFE模块的有效性, 在Synapse和ACDC数据集上对所提模型中的MLFF模块和GLFE模块进行消融实验.
GLFE模块对模型的分割是有贡献的. 在模型训练相同条件下, 实验结果如表3和表4所示. 表3中, 在使用GLFE模块比不用GLFE模块导致平均DSC系数增加1.71%, 平均HD95系数下降9.01 mm. 表4中, 添加GLFE模块比不添加GLFE模块使左心室, 心肌和右心室在DSC系数上分别提升1.57%, 0.39%和0.01%, 总体提升0.66%. 融合后的局部特征和全局特征, 通过GLFE模块, 帮助网络在局部特征和全局特征关注更为重要的信息, 从而避免信息的冗余和丢失. 实验结果表明, GLFE模块在平均DSC和HD95系数上均有提升, 可以提高模型对目标边缘区域的分割性能, 验证了GLFE模块对模型是有贡献的.
| 表 3 GLFE模块在Synapse数据集上的影响 |
| 表 4 GLFE模块在ACDC数据集上的影响 (%) |
MLFF模块对模型的分割也是有贡献的. 在模型训练相同条件下, 实验结果如表5和表6所示. 表5中, MLFF模块在平均DSC系数上提升2.67%, 在HD95系数上降低8.53 mm. 表6中, 添加MLFF模块相较于不添加MLFF模块在平均DSC系数上, 使左心室提升2.37%, 心肌提升2.81%, 右心室提升1.32%, 总体提升2.17%. MLFF模块通过将3个不同层次的特征表示进行关联, 从而提高模型的分割性能. 实验结果表明, 所设计的MLFF模块在两个数据集上对平均DSC和HD95系数均有提升, 验证了MLFF模块对模型是有贡献的.
| 表 5 MLFF模块在Synapse数据集上的影响 |
| 表 6 MLFF模块在ACDC数据集上的影响 (%) |
3.6 计算复杂度分析
所提模型与不同医学图像分割模型参数量的比较如表7所示. 提出的模型在平均DSC系数上优于其他模型, 在参数量上优于大部分模型. 实验结果表明, 与基于CNN和基于Transformer的模型相比, 所提模型不论是在评估指标或模型计算力和参数量上, 具有一定的竞争优势.
| 表 7 模型计算力和参数比较 |
4 结论与展望
针对多器官医学图像分割任务, 提出一种多级特征交互融合Transformer模型. 所提模型通过CNN和Swin Transformer分别提取局部特征和全局特征; 通过GLFE模块在降低卷积计算的参数量的同时捕捉融合后局部特征和全局特征的长期依赖关系和精确的位置信息; 通过MLFF模块捕获输入特征映射的空间重要性和关联不同级特征映射之间的语义内容, 从而得到精细化融合后的特征. 实验结果表明, 所提模型在Synapse和ACDC数据集上的分割性能优于基于Transformer和基于CNN的模型. 所提模型有助于提高医生的工作效率, 但模型的泛化能力仍待提高.
| [1] |
Bao H, Zhu YQ, Li Q. Hybrid-scale contextual fusion network for medical image segmentation. Computers in Biology and Medicine, 2023, 152: 106439. DOI:10.1016/j.compbiomed.2022.106439 |
| [2] |
Dhamija T, Gupta A, Gupta S, et al. Semantic segmentation in medical images through transfused convolution and Transformer networks. Applied Intelligence, 2023, 53(1): 1132-1148. DOI:10.1007/s10489-022-03642-w |
| [3] |
Zhang YC, Jiao RS, Liao QC, et al. Uncertainty-guided mutual consistency learning for semi-supervised medical image segmentation. Artificial Intelligence in Medicine, 2023, 138: 102476. DOI:10.1016/j.artmed.2022.102476 |
| [4] |
徐光宪, 冯春, 马飞. 基于UNet的医学图像分割综述. 计算机科学与探索, 2023, 17(8): 1776-1792. DOI:10.3778/j.issn.1673-9418.2301044 |
| [5] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [6] |
Zhou ZW, Rahman Siddiquee MM, Tajbakhsh N, et al. UNet++: A nested U-Net architecture for medical image segmentation. Proceedings of the 4th International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Granada: Springer, 2018. 3–11. [doi: 10.1007/978-3-030-00889-5_1]
|
| [7] |
Huang HM, Lin LF, Tong RF, et al. UNet 3+: A full-scale connected UNet for medical image segmentation. Proceedings of the 2020 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). Barcelona: IEEE, 2020. 1055–1059.
|
| [8] |
Ranjbarzadeh R, Tataei Sarshar N, Jafarzadeh Ghoushchi S, et al. MRFE-CNN: Multi-route feature extraction model for breast tumor segmentation in Mammograms using a convolutional neural network. Annals of Operations Research, 2023, 328(1): 1021-1042. DOI:10.1007/s10479-022-04755-8 |
| [9] |
Nirthika R, Manivannan S, Ramanan A, et al. Pooling in convolutional neural networks for medical image analysis: A survey and an empirical study. Neural Computing and Applications, 2022, 34(7): 5321-5347. DOI:10.1007/s00521-022-06953-8 |
| [10] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale. Proceedings of the 9th International Conference on Learning Representations. OpenReview.net, 2021.
|
| [11] |
Jiang Y, Liang J, Cheng TT, et al. MTPA_Unet: Multi-scale Transformer-position attention retinal vessel segmentation network joint Transformer and CNN. Sensors, 2022, 22(12): 4592. DOI:10.3390/s22124592 |
| [12] |
Wu YL, Wang GL, Wang ZY, et al. DI-Unet: Dimensional interaction self-attention for medical image segmentation. Biomedical Signal Processing and Control, 2022, 78: 103896. DOI:10.1016/j.bspc.2022.103896 |
| [13] |
Wang WH, Xie EZ, Li X, et al. Pyramid vision Transformer: A versatile backbone for dense prediction without convolutions. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 548–558.
|
| [14] |
石磊, 籍庆余, 陈清威, 等. 视觉Transformer在医学图像分析中的应用研究综述. 计算机工程与应用, 2023, 59(8): 41-55. DOI:10.3778/j.issn.1002-8331.2206-0022 |
| [15] |
傅励瑶, 尹梦晓, 杨锋. 基于Transformer的U型医学图像分割网络综述. 计算机应用, 2023, 43(5): 1584-1595. |
| [16] |
Azad R, Arimond R, Aghdam EK, et al. Dae-former: Dual attention-guided efficient Transformer for medical image segmentation. Proceedings of the 6th International Workshop on Predictive Intelligence in Medicine. Vancouver: Springer, 2023. 83–95.
|
| [17] |
Liu Z, Lin YT, Cao Y, et al. Swin Transformer: Hierarchical vision Transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 9992–10002.
|
| [18] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [19] |
Shen ZR, Zhang MY, Zhao HY, et al. Efficient attention: Attention with linear complexities. Proceedings of the 2021 IEEE Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2021. 3530–3538.
|
| [20] |
Lin HZ, Cheng X, Wu XY, et al. CAT: Cross attention in vision Transformer. Proceedings of the 2022 IEEE International Conference on Multimedia and Expo (ICME). Taipei: IEEE, 2022. 1–6.
|
| [21] |
Liu ST, Huang D. Receptive field block net for accurate and fast object detection. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 404–419.
|
| [22] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [23] |
Fu SH, Lu YY, Wang Y, et al. Domain adaptive relational reasoning for 3D multi-organ segmentation. Proceedings of the 23rd International Conference on Medical Image Computing and Computer Assisted Intervention. Lima: Springer, 2020. 656–666.
|
| [24] |
Schlemper J, Oktay O, Schaap M, et al. Attention gated networks: Learning to leverage salient regions in medical images. Medical Image Analysis, 2019, 53: 197-207. DOI:10.1016/j.media.2019.01.012 |
| [25] |
Cao H, Wang YY, Chen J, et al. Swin-Unet: Unet-like pure Transformer for medical image segmentation. Proceedings of the 2023 European Conference on Computer Vision. Tel Aviv: Springer, 2023. 205–218.
|
| [26] |
Yao C, Hu MH, Li QL, et al. TransClaw U-Net: Claw U-Net with Transformers for medical image segmentation. Proceedings of the 5th International Conference on Information Communication and Signal Processing (ICICSP). Shenzhen: IEEE, 2022. 280–284.
|
| [27] |
Wang HY, Xie SA, Lin LF, et al. Mixed Transformer U-Net for medical image segmentation. Proceedings of the 2022 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). Singapore: IEEE, 2022. 2390–2394.
|
| [28] |
Yuan FN, Zhang ZX, Fang ZJ. An effective CNN and Transformer complementary network for medical image segmentation. Pattern Recognition, 2023, 136: 109228. DOI:10.1016/j.patcog.2022.109228 |
| [29] |
Azad R, Heidari M, Shariatnia M, et al. TransDeepLab: Convolution-free Transformer-based DeepLab v3+ for medical image segmentation. Proceedings of the 5th International Workshop on Predictive Intelligence in Medicine. Singapore: Springer, 2022. 91–102.
|
| [30] |
Zhong XX, Xu LH, Li CQ, et al. RFE-UNet: Remote feature exploration with local learning for medical image segmentation. Sensors, 2023, 23(13): 6228. DOI:10.3390/s23136228 |
| [31] |
Xu GP, Zhang X, Liao WT, et al. LGNet: Local and global representation learning for fast biomedical image segmentation. Journal of Innovative Optical Health Sciences, 2023, 16(4): 2243001. DOI:10.1142/S1793545822430015 |
| [32] |
Heidari M, Kazerouni A, Soltany M, et al. HiFormer: Hierarchical multi-scale representations using Transformers for medical image segmentation. Proceedings of the 2023 IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2023. 6191–6201.
|
| [33] |
Liu YH, Wang H, Chen ZG, et al. TransUNet+: Redesigning the skip connection to enhance features in medical image segmentation. Knowledge-based Systems, 2022, 256: 109859. DOI:10.1016/j.knosys.2022.109859 |
 2024, Vol. 33
2024, Vol. 33



