2. 武汉科技大学 智能信息处理与实时工业系统湖北省重点实验室, 武汉 430081
2. Hubei Province Key Laboratory of Intelligent Information Processing and Real-time Industrial System, Wuhan University of Science and Technology, Wuhan 430081, China
结直肠癌[1]是消化道常见的恶性肿瘤之一, 多数情况下起源于腺瘤性息肉. 早期阶段的息肉通常是良性的, 但若未得到及时治疗, 随着时间推移, 存在演变为恶性的风险. 因此, 及早发现对于提高结直肠癌患者的存活率至关重要. 目前, 结肠镜检查被认为是最有效的筛查和诊断方法. 它能协助医生确定结肠息肉的具体形态和位置. 然而, 该方法在一定程度上依赖医生的专业经验. 结肠息肉本身具有不同大小、形状、颜色、纹理, 且其与周围粘膜组织的边界可能模糊不清. 在临床研究中, 利用计算机辅助诊断系统[2,3]来帮助临床医生对结肠息肉进行准确的定位和分割. 因此, 精准的结肠息肉分割算法显得尤为重要.
目前, 国内外杰出的研究者提出了许多结肠息肉分割模型, 这些模型展现了巨大的潜力. 大致上, 这些模型可以分为两类: 传统的无监督息肉分割算法和基于深度学习的有监督息肉分割算法. 传统的无监督分割算法通常利用图像自身的特征, 如阈值、边缘和区域合并等方法[4]进行分割. 基于阈值和边缘的算法具有较高的运算效率, 但容易受到背景噪声和人工标注结果的影响, 因此难以实现对结肠息肉区域的精准分割. 而基于区域合并的算法则容易导致结肠息肉区域的过度分割现象, 从而影响分割的准确性.
基于深度学习的息肉分割算法, 是在利用大量已人工标注好的数据来训练算法从而实现对结肠息肉的高精度分割, 并且由于卷积神经网络在提取图像特征方面具有突出表现, 因此将其广泛地应用于结肠息肉分割领域. 其中, U-Net模型[5]的出现使得息肉分割技术有了突破性的进展, U-Net本身是利用编解码的结构来提取图像中不同尺度的特征, 从而实现息肉的精准分割. Cao等[6]利用具有移位窗口的分层Swin Transformer[7]作为编码器来提取上下文特征以及具有补丁扩展层的基于对称 Swin Transformer 的解码器来执行上采样操作以恢复特征图的空间分辨率. Mahmud等[8]提出PolypSegNet (polyp segmentation network), 通过在编码器和解码器的每个尺度中利用深度扩张卷积聚合来自不同区域的特征, 同时将来自所有编码器单元层的不同尺度的上下文信息与各个解码器层进行互连, 从而提高了信息传递的效率. Wu等[9]提出了一种基于Swin Transformer的息肉分割网络MSRAformer (multiscale spatial reverse attention network), 通过金字塔结构的编码器来提取不同阶段的特征, 并利用多尺度通道注意模块来提取多尺度特征信息. Yeung等[10]采用双注意力焦点门机制, 将空间和通道的注意力融合到单个的注意力门[11]模块中, 以促进对特征的有选择性学习. Xia等[12]在U-Net的基础上引入了亮度先验融合模块将亮度信息融合到高级语义特征中, 以引导网络定位可能的息肉区域, 并且使用全局反向注意模块将亮度先验模块的输出和初始预测图结合, 实现长距离依赖以及细化预测结果. Yue等[13]提出了注意力引导的金字塔上下文网络APCNet (attention-guided pyramid context network), 采用注意力引导的多层聚合策略, 利用不同层的互补信息细化各层的上下文特征. 虽然基于深度学习的方法在结肠息肉分割中取得了显著进步, 其准确性和泛化性相较传统方法有了明显提高, 但仍然面临一些挑战. 其中包括图像特征信息利用不够充分、病灶区域与其背景之间的对比度较低从而导致分割结果不准确等问题. 针对以上问题, 本文基于U-Net网络提出了一种融合多尺度门控卷积和窗口注意力的结肠息肉分割模型MGW-Net (colon polyp seg-mentation fusing multi-scale gate convolution and window attention), 相较于传统的结肠息肉分割算法, 本文贡献如下.
(1)利用多尺度门控卷积块(multi-scale gate convolution module, MGCM)取代传统的卷积块, 其通过多尺度特征提取使模型拥有较大的感受野, 能够适应不同大小和形状的病灶区域. 同时结合空间与通道注意力以及改进的注意力门机制重新分配空间与通道特征权重, 强化病灶特征的响应, 以区分病灶与周围背景区域, 从而更精确地定位和分割息肉区域.
(2)为了充分利用U型网络丰富的图像特征信息, 在跳跃连接处引入了多信息融合增强模块(multi-information fusion enhancement module, MFEM), 通过改进的密集空洞卷积充分提取U型网络底部的丰富语义信息, 并构建混合增强的残差窗口注意力[7]捕获U型网络当前层的局部和全局特征.
(3)通过与其他分割网络在公开的结肠息肉数据集上进行比较, 验证了该方法在息肉分割方面具有较高的分割精度以及较好的泛化性能.
1 网络构建 1.1 网络总体架构针对传统的结肠息肉分割模型在对息肉的分割过程出现的分割精度不足和易受到病灶区域[14]影响等问题, 本文提出了一种融合多尺度门控卷积和窗口注意力的结肠息肉分割模型MGW-Net. 网络模型整体架构如图1所示. MGW-Net是在U-Net的基础上进行的改进, 使用多尺度门控卷积块MGCM取代传统的卷积块来充分提取息肉的特征信息, 并将输入图像的通道数改为32; 其次, 利用最大池化进行下采样操作, 为了减少模型在跳跃连接处的信息损失和利用模型底部数据蕴含的丰富图像语义信息, 在跳跃连接处引入了多信息融合增强模块MFEM来对U型网络当前层的特征信息进行挖掘以及引入模型底部数据来进行特征增强处理; 最后, 将最后一层解码层的结果输入带有Sigmoid激活函数的1×1的卷积中, 来获取最终预测的结肠息肉分割图像, 从而实现对结肠息肉的精准分割.
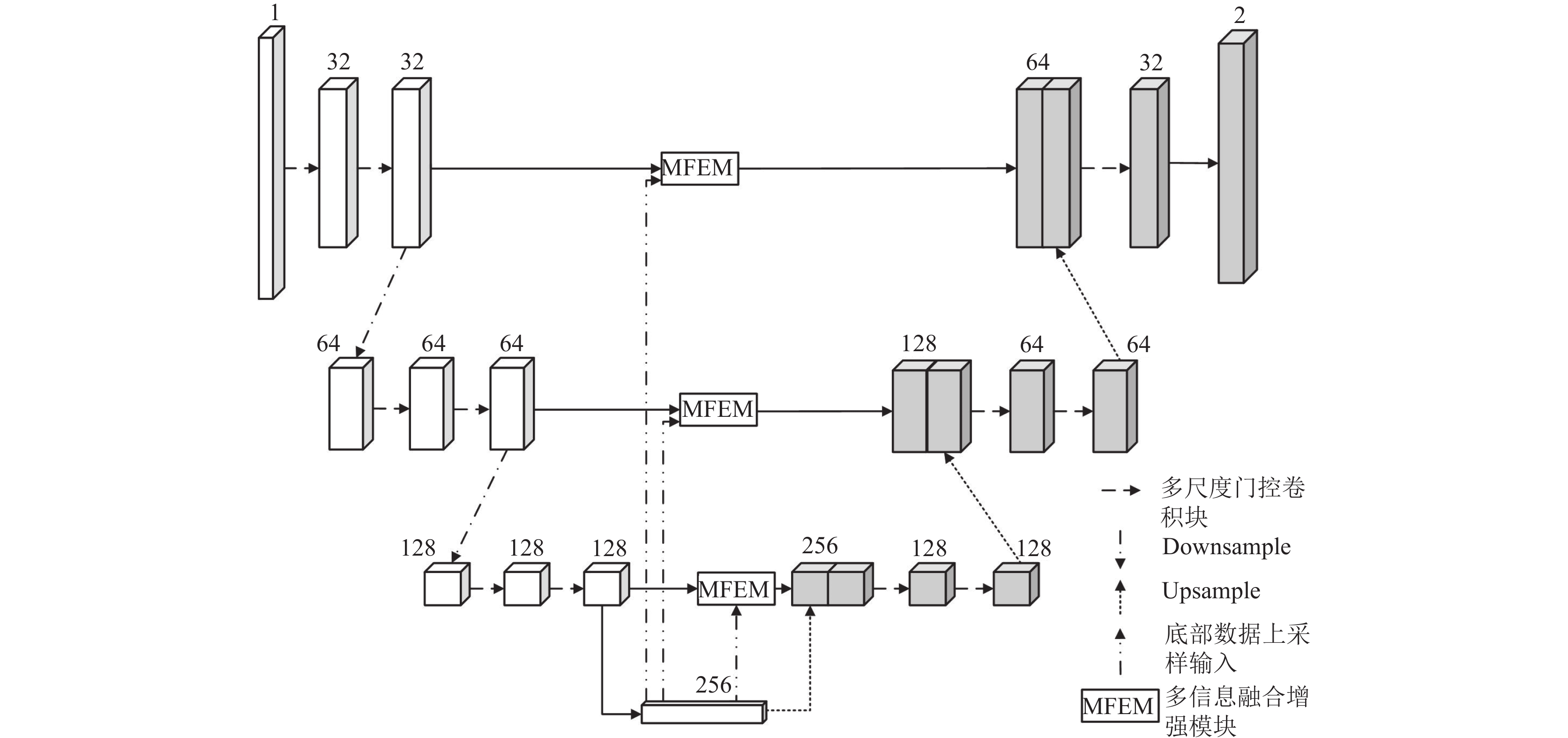
|
图 1 MGW-Net总体架构 |
1.2 多尺度门控卷积块
传统的卷积块在训练过程中难以针对性地对结肠息肉像素进行训练, 从而可能导致在适应不同大小、形状以及对比度较低的息肉时存在挑战. 受文献[10,15,16]启发, 本文设计了多尺度门控卷积块MGCM来弥补这些缺陷, 其主要利用多尺度门控压缩激励模块(multi-scale gate squeeze-excitation module, MGSM) 对不同尺寸和形状的息肉进行更准确的识别和建模、同时采用双重残差注意力模块(double residual attention module, DRAM)和双重注意力门控模块(double attention gate module, DAGM) 来强化息肉特征的响应, 进一步区分息肉与周围背景区域. MGCM结构如图2所示.
1.2.1 多尺度门控压缩激励模块MGSM与传统网络利用单一分支捕获单一尺度特征相比, 采用了4个包含不同大小的卷积块的并行分支设计, 同时在4个分支的最后阶段参考压缩激励块[17]以及注意力门机制, 设计了一个门控压缩激励模块(gate squeeze-excitation block, Gate-SE Block). MGSM结构如图2所示. 多尺度可以在增加感受野的同时, 能够捕获不同尺度的特征信息, 更好地识别并覆盖特征图病灶可能存在的各种形状、大小和位置. 压缩激励块通过学习通道间的相关依赖关系来动态地调整特征图的权重, 使得更重要的特征得到更大的权重, 有助于提高特征表示的表达能力. 而注意力门则可以自动学习并集中注意力在相关的部分, 提高对于重要信息的关注度. 因此, 本文将压缩激励块与注意力门进行结合可以使网络不再简单地整合所有通道信息, 而是能够灵活地强调在当前任务中最为显著和重要的特征, 抑制不重要的特征. Gate-SE Block结构如图3所示. 同时在多尺度中添加Gate-SE Block可以选择性地增强或减弱特定尺度下的特征, 网络更有可能学习到结肠息肉图像的普遍特征, 以及适应不同大小和形状的病灶区域, 避免过拟合的现象发生.
在MGSM模块中, 假设输入特征图A
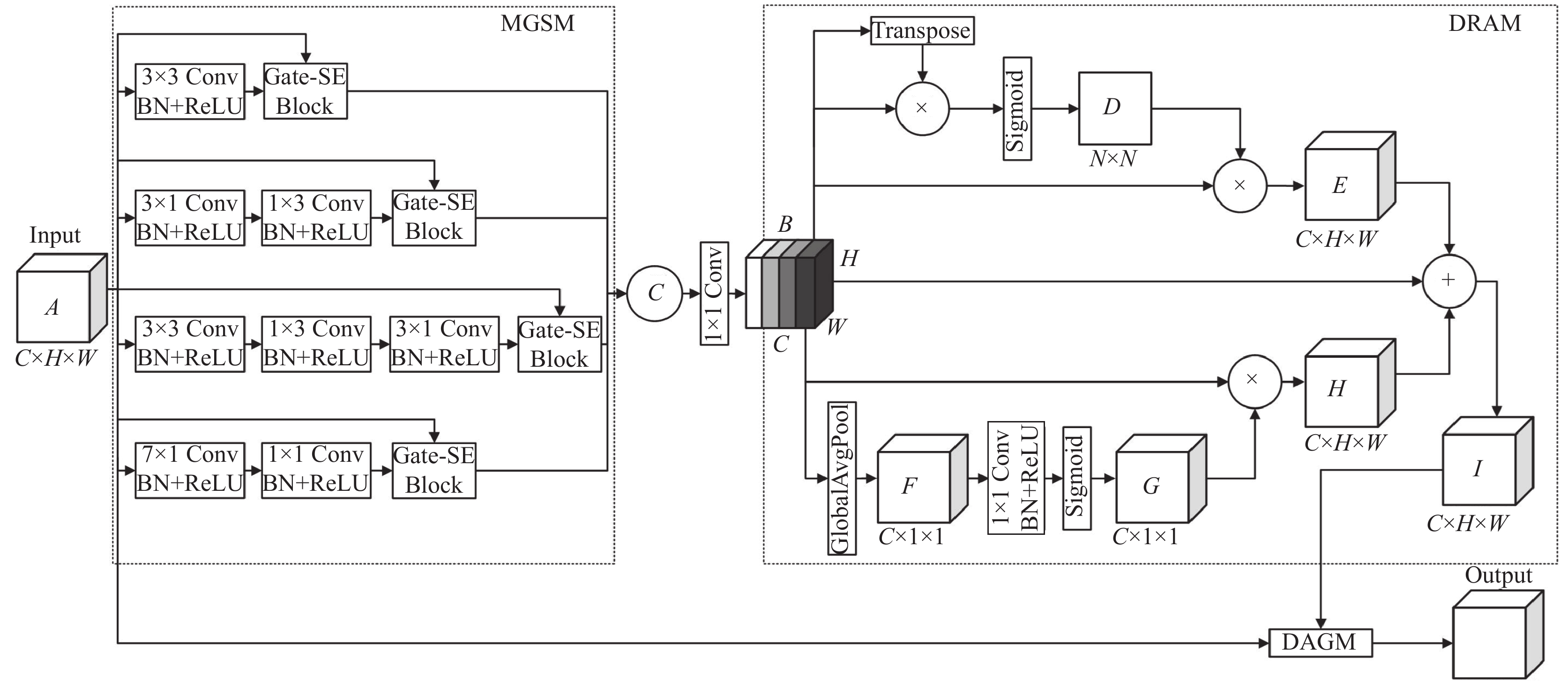
|
图 2 多尺度门控卷积块 |
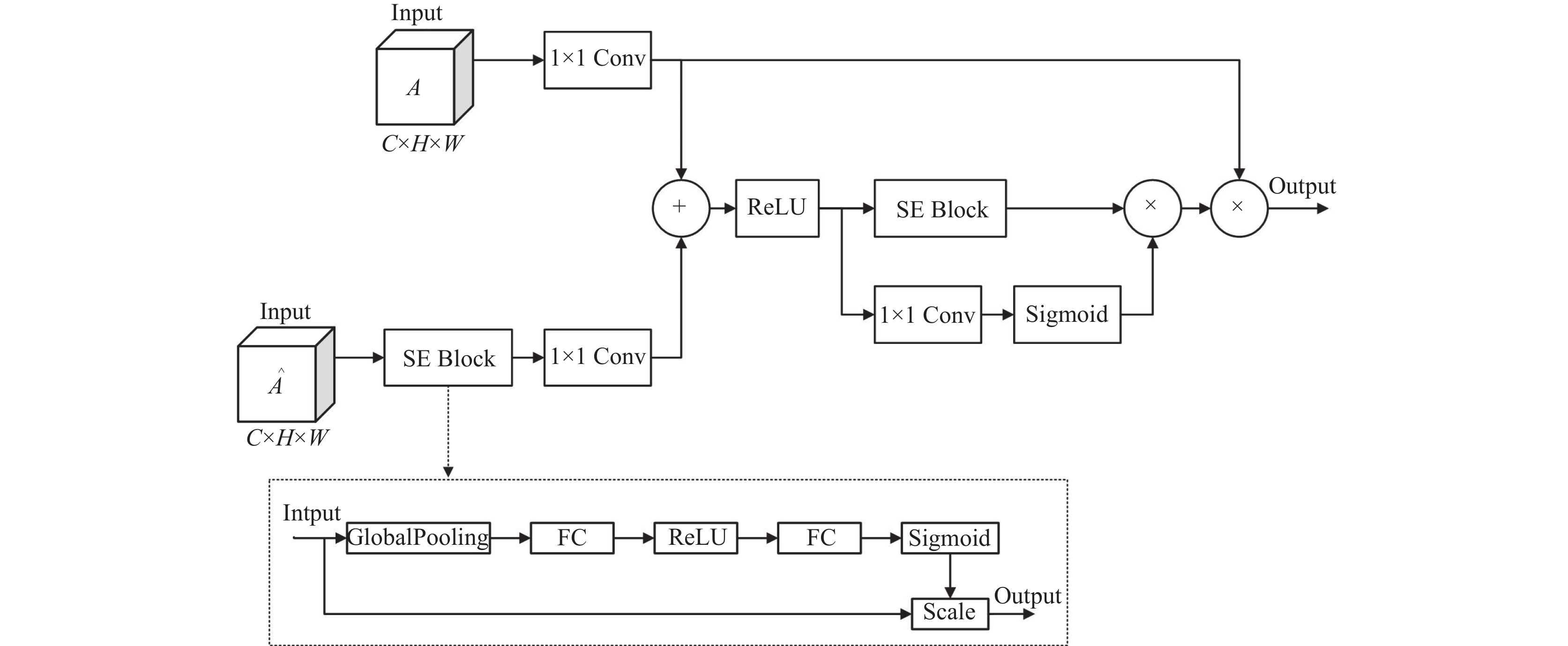
|
图 3 Gate-SE Block |
1.2.2 双重残差注意力模块
为了更全面地捕获息肉图像内部特征的空间和通道相关性, 并且解决权重更新为零以及难以进行更新的问题, 通过引入残差连接, 设计了空间与通道融合的双重残差注意力模块DRAM, 能够更准确地关注息肉区域, 进一步提高特征表征的准确性和多样性. 如图2所示, 首先为了获取空间特征, 将特征图B和B的转置进行逐元素相乘, 然后通过Softmax层来计算出空间上的注意力权重D
结合焦点门[10]和注意力门的概念, 本文设计了一种改进的双重注意力门控模块DAGM, 将双重残差注意力模块DRAM的空间注意力和通道注意力融合于其中, 其模块结构如图4所示. DAGM借助注意力门机制, 让卷积块更加精准地聚焦于输入数据中的空间和通道信息部分, 并抑制其他不相关的信息. 这提升了对关键空间和通道信息的提取能力, 有效地学习和优化特征数据之间的联系与差异. 从而强化了对结肠息肉病灶的响应, 从而有助于区分病灶区域与周围背景区域. 在图4中, 首先对输入特征图I和A分别进行1×1卷积处理, 随后进行矩阵加法, 并结合非线性激活函数(ReLU)来构建注意力系数. 接着, 利用空间和通道注意力模块分别处理注意力系数之后将它们连接在一起, 以进一步细化相关特征. 最后再将注意力系数进行1×1卷积和Sigmoid 操作, 并将其输出结果与经过空间和通道注意力细化后的输出以及A进行连接起来.
1.3 多信息融合增强模块尽管使用MGCM取代传统的卷积块可以缓解梯度消失和充分挖掘息肉特征信息. 但在编解码过程中, 直接的跳跃连接仍会不可避免地带来一定程度的噪声干扰, 从而造成漏分现象. 本文考虑到U-Net模型当前层的局部特征和全局特征信息未得到有效利用, 以及底部是在空间上的集成并蕴含了较多的图像语义信息, 为了减少漏分现象, 因此本文参考密集空洞卷积块[18]以及Swin Transformer[7], 设计了多信息融合增强模块MFEM, 其利用底部信息增强模块(bottom information enhancement module, BEM)来挖掘U型网络底部丰富的语义信息, 以及通过混合增强的残差窗口注意力模块(hybrid enhanced residual window attention module, HERWM)来捕获U型网络当前层的局部和全局特征. MFEM结构如图5所示.
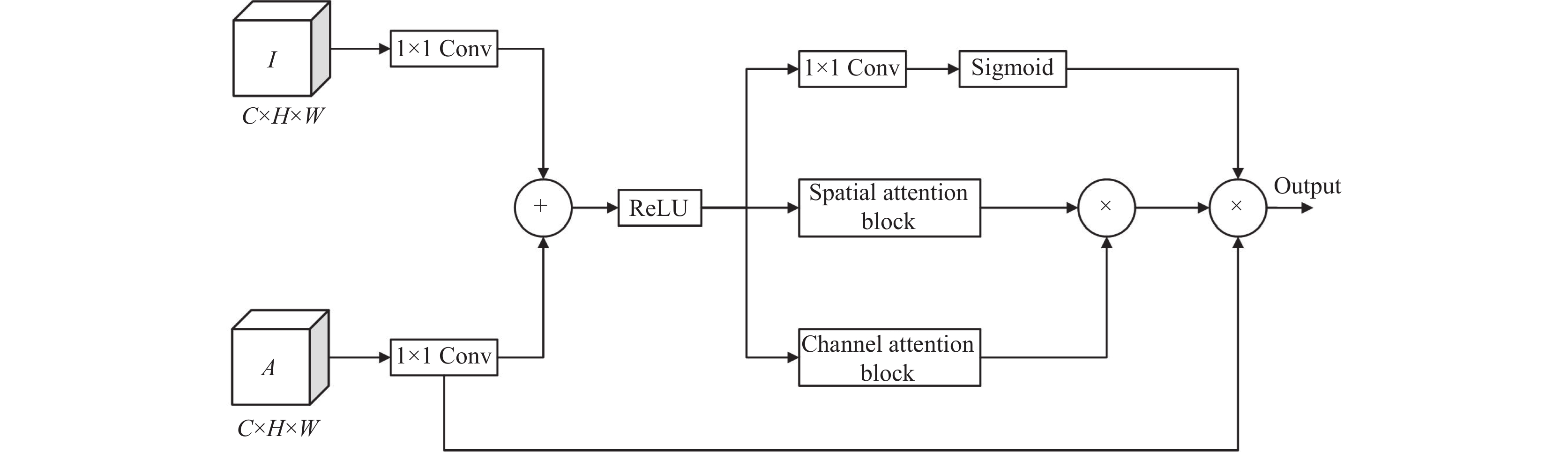
|
图 4 双重注意力门控模块 |
1.3.1 底部信息增强模块
在BEM中, 通过引入U型网络底部数据来对跳跃连接部分进行信息增强处理, 假设当前层的输入数据为J
| {b1=upsample(upsample(upsample(B)))b2=upsample(upsample(B))b3=upsample(B)) | (1) |
其中, upsample表示利用转置卷积进行上采样操作; B表示模型底部数据; b1、b2 和b3分别代表不同层进行上采样操作的输出结果. 为了方便处理与模型运算, 提前将模型底部数据进行上采样操作使之与当前跳跃连接层输入图像的数据维度保持一致.
其次, 为了充分挖掘底部数据所蕴含的信息, 将底部数据进行密集空洞卷积操作. 空洞卷积通过在卷积核中引入空洞, 扩大卷积核的感受野, 使得网络能够捕获更广阔范围内的信息. 本文通过将空洞卷积以串联方式进行堆叠, 串联堆叠空洞卷积可以逐步扩大感受野, 让网络更好地理解输入数据的全局信息, 有助于捕捉更大范围的特征. 其次, 每个分支的输出不仅作为下一分支的输入, 同时与下一个分支的空洞卷积结果直接进行相加操作, 可以引入更多不同尺度的特征表示, 进一步增强特征表达能力. 如图5所示, 将K依次经过感受野为3、5、9、19的串联分支, 使用1×1的卷积进行线性激活, 并将操作结果进行相加输出特征映射L

|
图 5 多信息融合增强模块 |
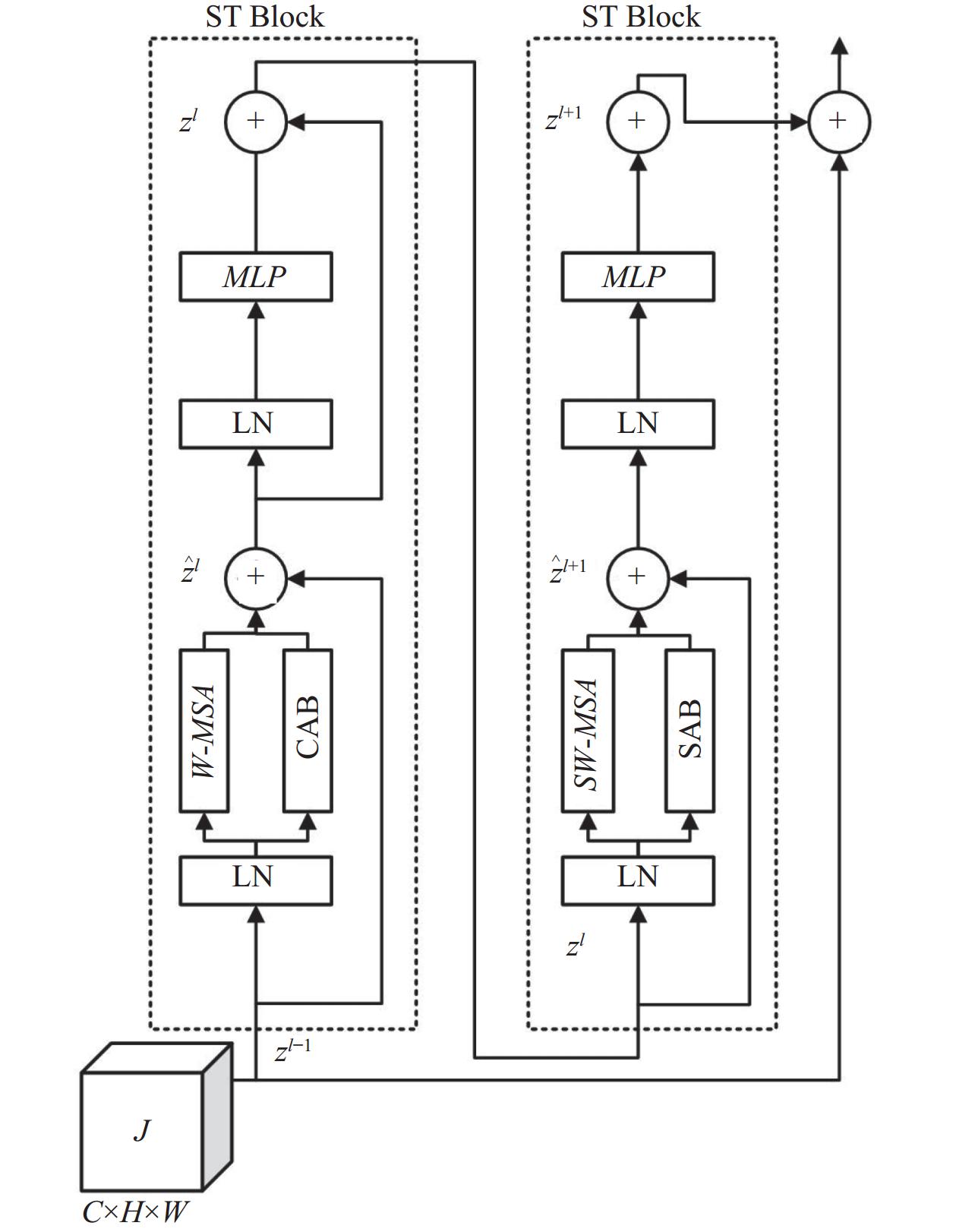
1.3.2 混合增强的残差窗口注意力模块
为了有效利用U型网络当前层的局部和全局特征, 通过引入Swin Transformer Block (ST Block)并结合DRAM模块的通道注意力(CAB)和空间注意力(SAB)来实现更好的特征提取. HERWM模块结构如图6所示. 首先, 利用ST Block常规窗口多头自注意力(W-MSA)对输入特征进行局部窗口划分, 并计算每一个小窗口内的自注意力, 再通过CAB计算输入特征的通道注意力权重, 将二者进行相加, 强化每一个小窗口的重要特征并抑制非重要的特征, 从而捕获局部区域的特征信息. 但由于窗口的划分, 导致各个窗口无法进行信息交互, 所以利用移位窗口多头自注意力(SW-MSA)来实现让特征信息在相邻的窗口中进行传递, 同时加入SAB来保留原始空间上的特征信息, 进一步提升关键区域的特征表达; 最后, 以残差连接的方式与当前层输入特征进行连接, 从而更好地捕捉图像中的全局结构和局部细节. 其数学定义如下:
| {ˆzl=W-MSA(LN(zl−1))+CAB(zl−1)+zl−1zl=MLP(LN(ˆzl))+ˆzlˆzl+1=SW-MSA(LN(zl))+SAB(zl)+zlzl+1=MLP(LN(ˆzl+1))+ˆzl+1xi=zl+1+zl−1 | (2) |
其中, ẑl和zl分别表示(S)W-MSA模块和MLP模块的输出特征; W-MSA和 SW-MSA分别表示使用常规和移位窗口多头自注意力; LN表示进行层归一化操作; CAB和SAB分别表示通道注意力和空间注意力; zl–1表示跳跃连接处的输入图像; xi表示经过HERWM模块的输出结果.
最后, 为了使底部数据的丰富语义信息与当前层的特征信息进行融合. 如图5所示, 将L经过1×1的卷积、平均池化和Softmax激活函数生成空间权重图, 将空间权重图与HERWM输出结果进行相乘, 最后与J进行相加操作输出特征图M
|
图 6 混合增强的残差窗口注意力模块 |
2 实验与分析 2.1 实验环境与参数设置
本文运行实验的环境为Windows 10操作系统、CPU Intel Core i9-12900 5.1 GHz、16 GB RAM以及NVIDIA GTX 3090 图形处理器. 使用深度学习框架PyTorch 1.7.1来进行训练MGW-Net. 实验的批处理大小(batch_size)为8, 初始学习率为0.000 1, 迭代次数为200, 每训练20次将学习率设置为原本的1/10, 各卷积层的权值使用Kaiming进行初始化, 使用Adam优化器来更新模型的参数.
在实验数据集方面, 使用CVC-ClinicDB[19]和Kvasir-SEG[20]公开结肠息肉分割数据集来验证本文模型的有效性, 并使用CVC-ColonDB[21]、CVC-300[22]和ETIS[23]公开结肠息肉分割数据集来验证本文模型算法的泛化性能. CVC-ClinicDB数据集是由612张分辨率为384×288像素大小的彩色结肠息肉图片组成. Kvasir-SEG数据集共有1000张分辨率从332×487到1920×1070像素大小的息肉图片. 由于Kvasir-SEG数据集并未划分训练集、验证集和测试集. 为了保证实验的可靠性, 从CVC-ClinicDB和Kvasir-SEG划分80%用于训练集, 10%用于验证集, 10%用于测试集.
2.2 评价指标和损失函数结肠息肉分割本质是将每个像素分为背景部分和息肉部分, 是一种二分类问题. 本文使用了交并比系数(intersection over union, IoU)、Dice相似系数(Dice similarity coefficient, DSC)、平均绝对值误差(mean absolute error, MAE)、
IoU作为语义分割常用的评价标准, 其代表着预测结果的真实性, 越接近于1, 越表示分割结果越接近真实标签. 本文对所有测试结果的交并比系数总和取平均值, 记作mIoU. IoU的计算公式如下:
| IoU = \frac{{TP}}{{FP + TP + FN}} | (3) |
Dice系数表示预测结果与真实标签的交并集之比, 越接近于1, 越表示预测结果于真实标签的相似度高. 本文对所有测试结果的Dice相似系数总和取平均值, 记作mDice. Dice的计算公式如下:
| Dice = \frac{{2TP}}{{FP + 2TP + FN}} | (4) |
其中, TP (true positive)、FP (false positive)和FN (false negative)分别表示真阳性、假阳性和假阴性.
MAE用于比较预测值ŷ与实际值y 之间的逐像素绝对值差异. MAE计算公式如下:
| MAE(y, \hat y) = \frac{1}{n}\sum\limits_{i = 0}^{n - 1} {|{y_i}} - {\hat y_i}| | (5) |
| E_\phi ^{\max } = \frac{1}{{W \times H}}\sum\limits_{i = 1}^W {\sum\limits_{j = 1}^H {\mathop \phi \nolimits_{FM} } } \left( {i, j} \right) | (6) |
其中, W、H分别表示图像的宽度和高度; ϕFM表示增强的对齐矩阵.
| F_\beta ^w = \left( {1 + {\beta ^2}} \right)\frac{{Precision \times Recall}}{{{\beta ^2} \times Precision + Recall}} | (7) |
其中, β设置为1; Precision为加权精度值; Recall为加权召回值.
Sα表示衡量预测结果和真实标签之间的结构相似性. Sα计算公式如下:
| {S_\alpha } = \alpha \times {S_o} + (1 - \alpha ) \times {S_r} | (8) |
其中, α是一个权衡参数, 默认设置为0.5; So表示预测结果与真实标签之间的重叠程度; Sr表示预测结果与真实标签之间的结构相似性.
由于背景的像素占比较大, 难以在息肉边界处准确定义像素类别, 因此本文采用二值交叉熵(binary cross entropy, BCE)损失函数和交并比(IoU)损失函数相结合, 其定义为:
| \left\{\begin{gathered} {L_{{\text{BCE}}}} = - \displaystyle\sum\limits_{i \in I} {{y_i}\log ({{\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\frown}$}}{y} }_i}) + (1 - {y_i})} \log (1 - {{\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\frown}$}}{y} }_i}) \\ {L_{{{IoU}}}} = 1 - \frac{{\displaystyle\sum\limits_{i \in I} {{y_i}{{\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\frown}$}}{y} }_i}} }}{{\displaystyle\sum\limits_{i \in I} {{y_i} + {{\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\frown}$}}{y} }_i} - {y_i}{{\overset{\lower0.5em\hbox{$\smash{\scriptscriptstyle\frown}$}}{y} }_i}} }} \\ {L_{{\text{loss}}}} = {L_{{\text{BCE}}}} + {L_{{{IoU}}}} \\ \end{gathered} \right. | (9) |
为了验证本文所提的MGW-Net模型对U-Net的有效改进, 将其与不同的先进模型进行对比实验. 本文选取U-Net、PraNet[27]、SANet[28]、UACANet(S)[29]、UACANet(L)[29]和CFA-Net[30]这几个模型, 其中, S和L分别表示网络中采用的卷积通道数大小. 与MGW-Net分别在CVC-ClinicDB和Kvasir-SEG数据集上进行对比实验, 采用相同的实验条件和调试策略, 实验可视化结果如图7所示.

|
图 7 不同模型的分割结果 |
从图7中可以发现, U-Net作为结肠息肉分割的经典算法, 在分割出息肉的大体位置和分布上的性能较为不错, 但是在分割息肉的时候容易受到背景噪声像素的影响, 从而会导致息肉分割结果的断裂与缺失现象. PraNet采用并行部分解码器(PPD)聚合高层特征, 同时使用反向注意(RA)模块来挖掘结肠息肉边缘信息, 但在息肉分割方面仍会出现息肉分割不完全以及误分割的现象. SANet通过颜色交换操作来降低因下采样样本颜色不一致问题带来的过拟合问题, 同时提出浅层注意力模块来改进小息肉分割, 并且引入概率校正策略(PCS)来缓解小息肉像素不平衡问题, 通过实验可以发现该模型在对息肉的分割方面已经表现出较为不错的性能可以分割出较为连续的分割结果, 但会出现误分割现象. UACANet模型是在U-Net的基础上在解码部分计算显著映射, 来对息肉像素进行增强处理, 从而实现对息肉的分割, 从实验结果中, 可以发现UACANet在息肉分割方面已经可以在做到对息肉区域较为精准地分割并且卷积通道数的增加对于息肉分割结果具有一定的增强效果, 但在息肉的边缘位置分割上存在不精准问题. CFA-Net采用了跨级特征聚合网络的策略, 利用边界预测网络生成边界感知特征, 然后通过分层策略将这些特征融合到分割网络中, 同时引入了跨级别特征融合模块, 能够整合不同级别的相邻特征, 处理息肉尺度变化并包含多尺度信息, 进而生成更精细的息肉分割图, 从实验结果来看其拥有比较高的准确性, 但是对小息肉分割精度较低. 本文所提出的算法MGW-Net, 不仅在分割结果上与专家手动标注的结果保持一致, 并且相较于其他息肉分割算法, 在对息肉边缘位置的分割方面不会出现明显的误分割和漏分割现象, 可以保障结肠息肉分割结果的整体性和连贯性, 降低背景噪声的影响. 从而验证MGW-Net在对结肠息肉分割方面具有一定的抗干扰性, 可以实现对结肠息肉的高精度分割. 在CVC-ClinicDB和Kvasir-SEG数据集的实验结果分别如表1和表2所示.
| 表 1 不同模型在CVC-ClinicDB数据集上的评估结果 |
| 表 2 不同模型在Kvasir-SEG数据集上的评估结果 |
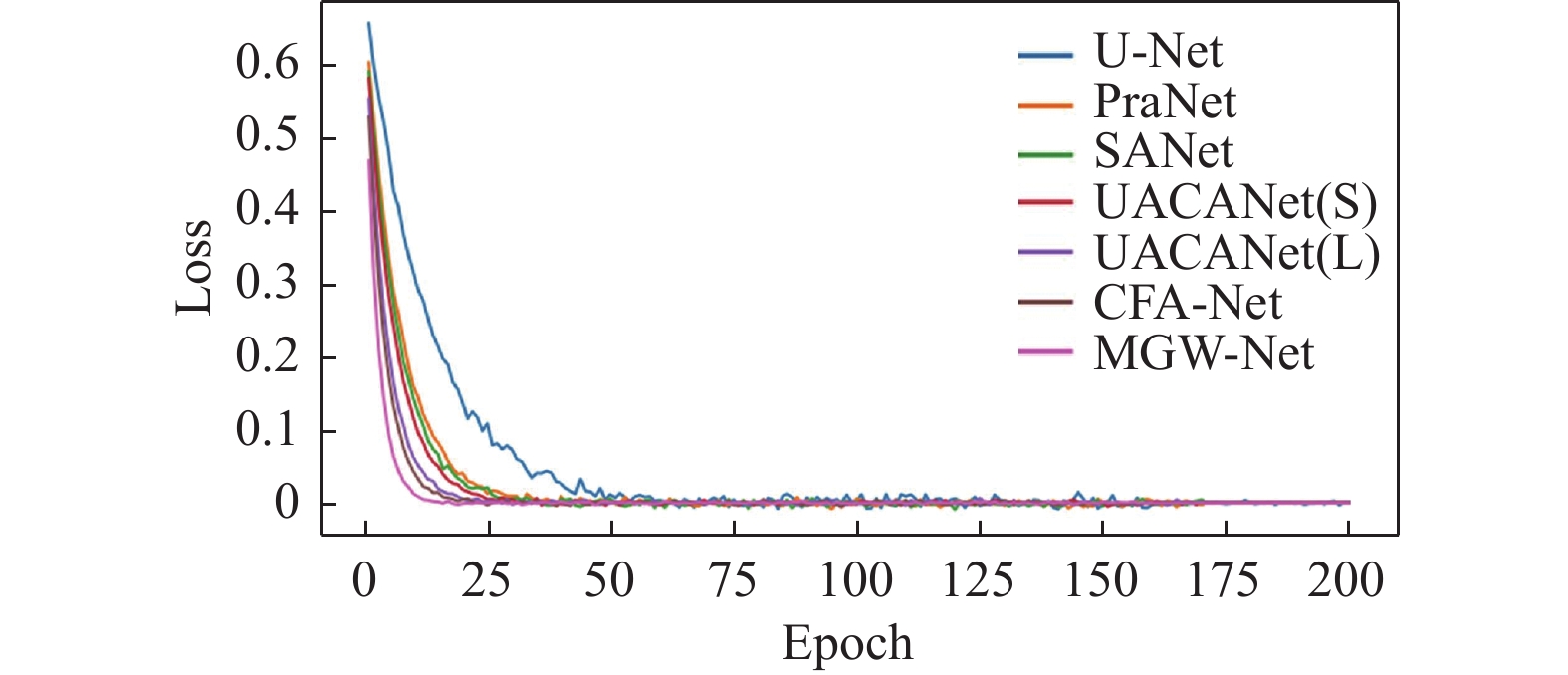
从表1可知, 在CVC-ClinicDB数据集上, 分割结果较为领先的CFA-Net的mDice为0.933, mIoU为0.883; 而本文所提出的MGW-Net的mDice为0.938, mIoU为0.894, 与CFA-Net相比, 本文所提出的模型在mDice提高了0.5%, mIoU上提高了1.1%, 其他几个指标也达到了前列水平. 同时, 从图8可知, 在CVC-ClinicDB数据集上进行单独训练, 本文提出的MGW-Net在收敛速度方面优于其他对比模型.
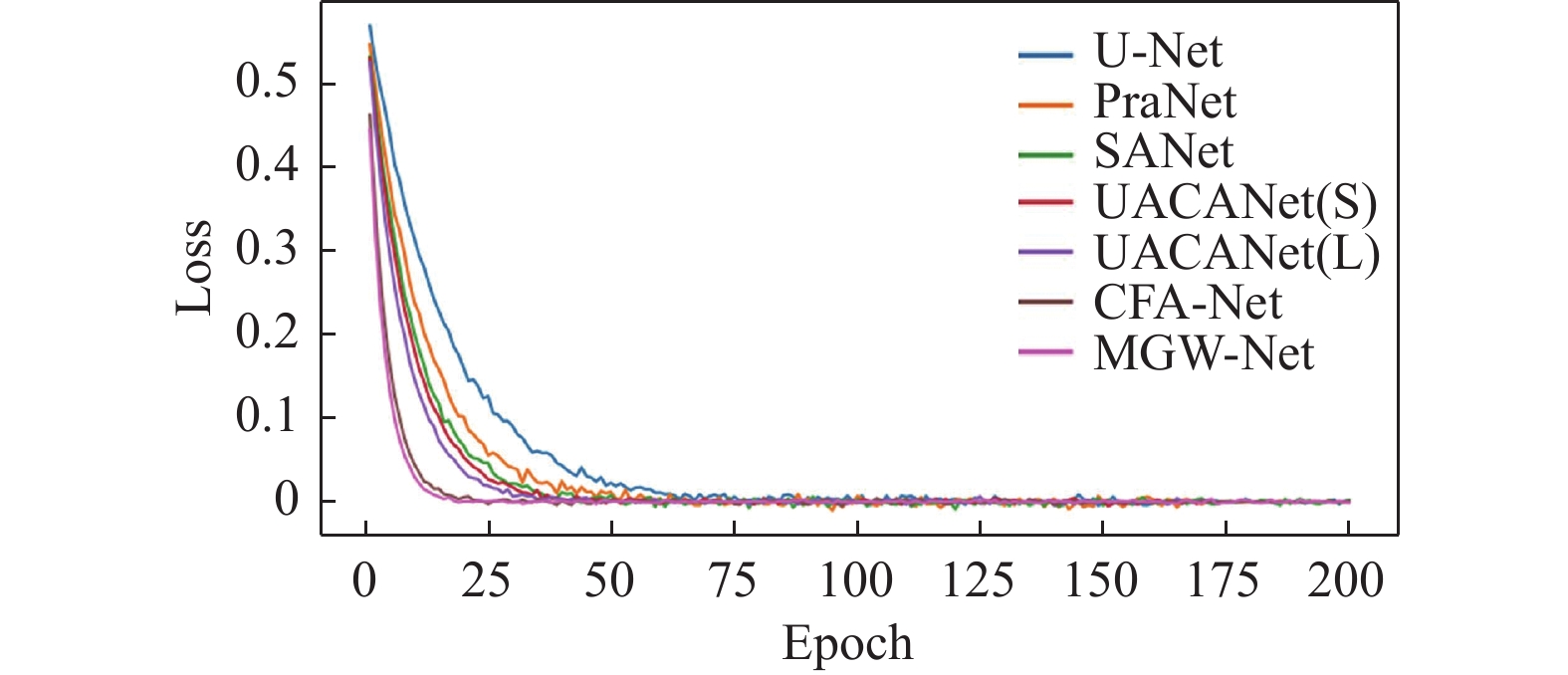
从表2可知, 在Kvasir-SEG数据集中, 性能表现较好的CFA-Net的mDice为0.915, mIoU为0.861; 而本文所提出的MGW-Net的mDice为0.927, mIoU为0.879, 与CFA-Net相比, 本文所提出的模型在mDice上提高了1.2%, 以及在mIoU上提高了1.8%. 并且其他几个指标在对比模型中均达到最优. 同时, 从图9可知, 在Kvasir-SEG数据集上进行单独训练, 本文所提出的MGW-Net的收敛速度与CFA-Net相当, 并优于其他对比模型.
|
图 8 不同模型在CVC-ClinicDB数据集上的loss变化 |
|
图 9 不同模型在Kvasir-SEG数据集上的loss变化 |
2.4 消融实验
为了进一步地证实本文中各个模块的有效性, 本文在CVC-ClinicDB和Kvasir-SEG这两个数据集上分别进行了消融实验, 实验结果如表3所示. 其中, M1表示基准的U-Net网络模型, M2表示在U-Net的基础上加入MGCM模块, M3表示在M1的基础上加入MFEM模块, M4表示在M2的基础上加入MFEM模块, 也即是本文所提出的模型.
| 表 3 消融实验结果 |
由表3可知, M1仅采用传统的U-Net模型在对息肉方面已经有了不错的精度, 但分割精度仍需一定程度的提升; M2和M3是分别在U-Net基准模型的基础上新加入了本文所设计的模块, 并且在各项指标相较于M1均有不同程度的提升, 表明MGCM模块的确可以充分提取出息肉的细微特征信息避免过拟合现象, MFEM模块可以有效地利用模型底部语义信息以及模型当前层的局部特征和全局特征, 充分提高编解码的传输效率并抑制噪声干扰. M4相较于M2和M3在各个指标上均有一定程度的提高, 表明本文所提出的MGW-Net模型的确具有一定的合理性和有效性.
2.5 泛化性能由于本文采用Kvasir-SEG和CVC-ClinicDB数据集作为训练集, 为了验证本文模型算法的泛化性, 因此采用CVC-ColonDB、CVC-300和ETIS数据集作为测试集进行验证. 实验结果如表4所示.
| 表 4 泛化实验结果 |
由表4可知, 本文所提出的MGW-Net与对比模型相比, 在CVC-300数据集上mDice、mIoU分别为91.2%、85.0%, 在ETIS数据集上mDice、mIoU分别为79.0%、73.1%, 均达到了最优水平, 其他指标也处于前列. 同时在CVC-ColonDB数据集上MGW-Net的各项指标也处于前列水平. 综合比较结果, 本文所提出的MGW-Net模型算法在未知数据集上具有较强的泛化能力.
3 结束语针对传统的结肠息肉分割模型在对息肉分割方面存在的图像特征信息利用不充分、分割精度不足问题, 本文提出了一种融合多尺度门控卷积和窗口注意力的结肠息肉分割模型(MGW-Net), 通过使用多尺度门控卷积块替代传统的卷积块, 实现了对不同大小、形状以及对比度较低的息肉的精准定位与分割; 同时在跳跃连接处构建了多信息融合增强模块, 通过有效利用当前层的局部特征和全局特征信息以及融入模型底部数据所蕴含的丰富语义信息来减少编解码过程带来的信息损失. 通过MGW-Net在CVC-ClinicDB和Kvasir-SEG数据集上的实验结果表明, 该模型相较于其他先进模型, 可以比较完整的分割不同大小的息肉区域. 同时, 在CVC-ColonDB、CVC-300和ETIS数据集上的实验结果表明该模型具有较强的泛化性能. 但MGW-Net在对小型息肉边缘分割方面仍会出现精度较低的现象, 并且模型在物理设备的性能不足的时候推理速度仍有待进一步提高, 今后将进一步地提高模型的分割性能以满足临床需要.
| [1] |
Sinicrope FA. Increasing incidence of early-onset colorectal cancer. New England Journal of Medicine, 2022, 386(16): 1547-1558. DOI:10.1056/NEJMra2200869 |
| [2] |
Ibrahim AU, Kibarer AG, Al-Turjman F. Computer-aided detection of tuberculosis from microbiological and radiographic images. Data Intelligence, 2023, 5(4): 1008-1032. DOI:10.1162/dint_a_00198 |
| [3] |
Peng SG. Application of medical image detection technology based on deep learning in pneumoconiosis diagnosis. Data Intelligence, 2023, 5(4): 1033-1047. DOI:10.1162/dint_a_00228 |
| [4] |
Lingwal S, Bhatia KK, Singh M. Semantic segmentation of landcover for cropland mapping and area estimation using Machine Learning techniques. Data Intelligence, 2023, 5(2): 370-387. DOI:10.1162/dint_a_00145 |
| [5] |
Yin XX, Sun L, Fu YH, et al. U-Net-based medical image segmentation. Journal of Healthcare Engineering, 2022, 2022: 4189781. |
| [6] |
Cao H, Wang YY, Chen J, et al. Swin-Unet: Unet-like pure Transformer for medical image segmentation. Proceedings of the 2023 European Conference on Computer Vision. Tel Aviv: Springer, 2023. 205–218.
|
| [7] |
Liu Z, Lin YT, Cao Y, et al. Swin Transformer: Hierarchical vision Transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 9992–10002.
|
| [8] |
Mahmud T, Paul B, Fattah SA. PolypSegNet: A modified encoder-decoder architecture for automated polyp segmentation from colonoscopy images. Computers in Biology and Medicine, 2021, 128: 104119. DOI:10.1016/j.compbiomed.2020.104119 |
| [9] |
Wu C, Long C, Li SJ, et al. MSRAformer: Multiscale spatial reverse attention network for polyp segmentation. Computers in Biology and Medicine, 2022, 151: 106274. DOI:10.1016/j.compbiomed.2022.106274 |
| [10] |
Yeung M, Sala E, Schönlieb CB, et al. Focus U-Net: A novel dual attention-gated CNN for polyp segmentation during colonoscopy. Computers in Biology and Medicine, 2021, 137: 104815. DOI:10.1016/j.compbiomed.2021.104815 |
| [11] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [12] |
Xia HY, Qin YL, Tan YM, et al. BA-Net: Brightness prior guided attention network for colonic polyp segmentation. Biocybernetics and Biomedical Engineering, 2023, 43(3): 603-615. DOI:10.1016/j.bbe.2023.08.001 |
| [13] |
Yue GH, Li SY, Cong RM, et al. Attention-guided pyramid context network for polyp segmentation in colonoscopy images. IEEE Transactions on Instrumentation and Measurement, 2023, 72: 5008213. |
| [14] |
Zhang RF, Lai PW, Wan X, et al. Lesion-aware dynamic kernel for polyp segmentation. Proceedings of the 25th International Conference on Medical Image Computing and Computer-assisted Intervention. Singapore: Springer, 2022. 99–109.
|
| [15] |
Yu F, Koltun V. Multi-scale context aggregation by dilated convolutions. Proceedings of the 4th International Conference on Learning Representations. San Juan, 2016.
|
| [16] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 3–19.
|
| [17] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [18] |
Chen LC, Papandreou G, Schroff F, et al. Rethinking atrous convolution for semantic image segmentation. arXiv:1706.05587, 2017.
|
| [19] |
Bernal J, Sánchez FJ, Fernández-Esparrach G, et al. WM-DOVA maps for accurate polyp highlighting in colonoscopy: Validation vs. saliency maps from physicians. Computerized Medical Imaging and Graphics, 2015, 43: 99-111. DOI:10.1016/j.compmedimag.2015.02.007 |
| [20] |
Jha D, Smedsrud PH, Riegler MA, et al. Kvasir-SEG: A segmented polyp dataset. Proceedings of the 26th International Conference on Multimedia Modeling. Daejeon: Springer, 2020. 451–462.
|
| [21] |
Tajbakhsh N, Gurudu SR, Liang JM. Automated polyp detection in colonoscopy videos using shape and context information. IEEE Transactions on Medical Imaging, 2016, 35(2): 630-644. DOI:10.1109/TMI.2015.2487997 |
| [22] |
Vázquez D, Bernal J, Sánchez FJ, et al. A benchmark for endoluminal scene segmentation of colonoscopy images. Journal of Healthcare Engineering, 2017, 2017: 4037190. |
| [23] |
Silva J, Histace A, Romain O, et al. Toward embedded detection of polyps in WCE images for early diagnosis of colorectal cancer. International Journal of Computer Assisted Radiology and Surgery, 2014, 9(2): 283-293. DOI:10.1007/s11548-013-0926-3 |
| [24] |
Fan DP, Gong C, Cao Y, et al. Enhanced-alignment measure for binary foreground map evaluation. Proceedings of the 27th International Joint Conference on Artificial Intelligence. Stockholm: IJCAI.org, 2018. 698–704.
|
| [25] |
Margolin R, Zelnik-Manor L, Tal A. How to evaluate foreground maps? Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition. Columbus: IEEE, 2014. 248–255.
|
| [26] |
Fan DP, Cheng MM, Liu Y, et al. Structure-measure: A new way to evaluate foreground maps. Proceedings of the 2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017. 4558–4567.
|
| [27] |
Fan DP, Ji GP, Zhou T, et al. PraNet: Parallel reverse attention network for polyp segmentation. Proceedings of the 23rd International Conference on Medical Image Computing and Computer-assisted Intervention. Lima: Springer, 2020. 263–273.
|
| [28] |
Wei J, Hu YW, Zhang RM, et al. Shallow attention network for polyp segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention. Strasbourg: Springer, 2021. 699–708.
|
| [29] |
Kim T, Lee H, Kim D. UACANet: Uncertainty augmented context attention for polyp segmentation. Proceedings of the 29th ACM International Conference on Multimedia. Chengdu: ACM, 2021. 2167–2175.
|
| [30] |
Zhou T, Zhou Y, He KL, et al. Cross-level feature aggregation network for polyp segmentation. Pattern Recognition, 2023, 140: 109555. DOI:10.1016/j.patcog.2023.109555 |
 2024, Vol. 33
2024, Vol. 33



