随着医学影像设备的快速发展和普及、成像技术已广泛应用于临床实践, 成为疾病诊断、手术规划和预后评估等过程中不可或缺的辅助工具. 医学影像分割是医学影像分析的重要领域, 也是计算机辅助诊断、监测、干预和治疗的必要组成部分, 其关键任务之一就是分割医学图像中的目标对象(如器官或病变)[1,2], 从而为下游任务提供如详细分析[3]、合理诊断、预测和预防疾病等方面的有价值信息. 但目前医学影像分割仍存在数据掩码少[4]、组织边界信息弱[5]以及分割精度低等问题. 为了探索这些问题, Ben-Cohen等人[6]首次尝试将全卷积网络(FCN)应用于计算机断层扫描(CT)检查中的肝脏分割和肝转移检测任务, 对一个相对较小的数据集上进行研究, 结果表明FCN优于基于补丁的卷积神经网络(CNN)和基于稀疏性的分类方案. U-Net由Ronneberger等人[7]首次提出, 该架构由一个用于捕捉上下文的收缩路径和一个用于精确定位的对称扩展路径组成, 因此这种网络可以通过几张图像进行端到端[8]训练, 这使得U-Net成为医学图像分割任务的首选网络. Zhou等人[9]发现了一种编码器和解码器子网络通过一系列嵌套的密集跳跃路径连接的深度监督编码器-解码器网络, 该网络中重新设计的跳跃路径的主要目的就是缩小编码器和解码器子网络特征图之间的语义差距[7]. NnU-Net是由Isensee等人提出的[10], 它是一种基于二维和三维vanilla U-Net的鲁棒性自适应框架, 在Isensee等人在研究过程中摒弃了拟议网络设计中多余的花哨设计, 重点关注在影响方法性能和通用性的关键方面.
近年来, 随着网络的更新壮大, 神经网络架构的轻量化得到了改善, 但提高医学图像分割的准确性仍然是一个巨大的挑战. Valanarasu等人[11]就提出了一种门控轴向注意模型, 通过在自我注意中引入额外的控制机制以扩展现有的架构. 基于多层感知机(MLP)的网络, 如MLP-Mixer (完全基于MLP的架构)[12]、gMLP[13]也被发现是可以较好地完成计算机视觉下游任务的重要手段. UNeXt[14]是一种将CNN和MLP相结合的网络, 其特点是模型的参数量较小、推理速度较快. 因此在医学图像分割中的应用得到研究者越来越广泛的关注. 但UNeXt仍存在忽略网络体系结构设计(例如跳跃连接)[15], 导致其在分割准确度方面还有待提升.
基于此, 本文提出了一个可以有效增强特征间融合的多级多层感知机模块的LM-UNet (lateral MLP augmented U-Net for medical image segmentation)模型, 该模型网络底部使用混合MLP块的对称结构. LM-UNet模型改进了U-Net[7]的跳跃连接, 在编码器端的每个层级上的一组卷积和降采样后得到的特征图进行卷积-维度卷积操作并特定标记化, 然后分别在通道信息和空间信息对特征信息进行全连接操作, 最后将增强后的特征融合到解码器中. LM-UNet模型包含3个阶段: 卷积阶段、多层感知机阶段和横向多层感知机增强阶段. 其中卷积阶段用于提取图像的局部特征, 多层感知机阶段用于提取全局特征和增强边界信息, 横向多层感知机增强阶段用于加强特征信息的融合[16]以及增强局部特征信息和全局信息之间的联系.
2 方法图1表示的是LM-UNet模型的总体架构. LM-UNet模型是一个分层的编码器-解码器架构, 包括横向MLP块、混合MLP块和卷积模块, 其中编码器执行的初始操作由卷积和降采样、卷积和维度卷积组成. 每个横向MLP块由一个令牌化特征模块和多个混合MLP块组成, 横向MLP块的输入大小为

|
图 1 LM-UNet 的总体架构 |
2.1 卷积块
输入图像为
| $ x'_i = MaxPool(BN(Conv({x_i}))),\; i \in \left\{ {1, 2, 3} \right\} $ | (1) |
其中, xi表示第i输入特征图,
在多层级令牌化特征图的输入端, 本文采用了双卷积块(卷积-维度卷积)操作. 如图2所示, 首先, 通过卷积操作将特征图的通道数做升维操作, 然后经过维度卷积块(本质是卷积层), 其目的是降低在低层中的特征图维度, 因为参数过大的特征融合到混合MLP模块中容易造成过拟合现象. 因此本文添加了维度卷积块, 在令牌化特征图的输入端尽量缩减通道维度并且通道数是有规律的依次进行升维和降维.

|
图 2 多层级令牌化特征图的输入 |
2.2 混合MLP块
混合器将输入作为S个非重叠图像片段序列, 并将每个片段序列投射到所需的隐藏维度C上, 从而得到一个二维实值输入
| $ u'_i= x'_i + {W_2}\sigma ({W_1}LN(x'_i)), \;\;i = 1, \cdots, C $ | (2) |
| $ v'_i = u'_i + {W_4}\sigma ({W_3}LN(u'_i)),\;\; j = 1, \cdots, S $ | (3) |
其中, W1、W2、W3、W4为权重. 原始输入图像的分辨率为H×W, 每个补丁的分辨率为P×P, 补丁的数量为S=H×W/P2个. LN()表示归一化层. 这里的

|
图 3 混合 MLP 模块核心思想 |
2.3 横向MLP块
根据卷积块操作得到的特征输出, Fi是每个输出特征图的维度, 让
| $ x'_i = Tok(x'_i) $ | (4) |
| $ {\mathit{FM}_i} = Resi{\textit{z}}e\left( {LN\left( {x'_i} \right)} \right) $ | (5) |
| $ {\mathit{FM}}'_i = MixerLayer\left( {F{M_i}} \right) $ | (6) |
| $ Ou{t_i} = {\mathit{FM}}'_i + y'_i$ | (7) |
其中,
交叉熵损失(cross entropy loss)和骰子损失(Dice loss)都被广泛用于一般的分割任务中. 由于交叉熵损失在类别分布均匀的情况下表现较好, 而骰子损失则更适用于大尺寸的目标对象, 因此本文使用二元交叉熵(binary cross entropy)和骰子损失的线性组合来训练网络. 损失函数如式(8)所示.
| $ L = aBCE\left( {Y, y} \right) + bDice\left( {Y, y} \right) $ | (8) |
其中, L表示预测值Y和目标值y之间的损失函数, 参数a和b为可调的超参数.
2.5 数据规格和数据处理为了评估LM-UNet模型, 在ISIC国际皮肤成像数据集[19]、OAI-ZIB软骨数据集[20]和BUSI乳腺超声数据集[21]这3个不同的数据集上进行实验. ISIC 2018数据集由2594幅图像组成, 其中既有相机获取的皮肤病图像, 也有相应的皮损区域分割图, 所有图像的分辨率被调整为512×512. OAI-ZIB数据集最初由507个带分割的三维
表1展示的是U-Net、U-Net++、ResUNet、TransUNet、UNeXt和LM-UNet这6种模型在ISIC 2018数据集上分割性能指标. 由表1中的指标可以看出, 本文提出的模型分割性能最佳, ISIC 2018数据集上的平均IoU系数最大值对应模型为LM-UNet, 其平均IoU系数为0.8652, 较UNeXt提高3.2%. 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet的分割性能也有不同程度的提高, 平均IoU系数分别比前四者高10.8%、9.8%、5.3%和2.8%.
| 表 1 ISIC 2018数据集不同模型的实验分割性能 |
GFLOPs是用来衡量神经网络模型计算速度和能力的指标, 从表1–表3中看出, LM-UNet模型展现出了较高的计算速度和推理能力. 相比较TransUNet模型, 对于医学图像数据而言, 基于Transformer的模型耗时长, 这里从数据中看出, LM-UNet模型比基于Transformer的模型能够更快地处理数据, 因此在训练和推理任务中更高效.
| 表 2 OAI-ZIB 2D数据集不同模型的实验分割性能 |
| 表 3 BUSI数据集不同模型的实验分割性能 |
LM-UNet在跳跃连接部分添加了横向MLP块并二次加强了特征信息间的融合, 使模型具有多层级强化特征融合和边界信息处理的特点, 这也是LM-UNet在ISIC数据集上获得更高的分割性能的根本原因.
U-Net、U-Net++、ResUNet、TransUNet、UNeXt和LM-UNet这6种模型在OAI-ZIB 2D数据集上分割性能指标如表2所示. 分析表2不难看出OAI-ZIB 2D数据集上分割效果最好的模型为LM-UNet, 平均IoU系数0.8470, 两个指标均为6种模型中的最大值. LM-UNet具有最大平均IoU系数0.8470, 比UNeXt高4.2%. 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet在OAI-ZIB 2D数据集上分割性能也均较前四者有不同程度的提高, 平均IoU系数提高2.8%–9.1%. LM-UNet在OAI-ZIB 2D数据集上的高分割性能再次印证了模型的合理性.
表3是BUSI数据集上6种模型的分割性能指标. 从表3中分析可得, LM-UNet模型在BUSI数据集上同样达到了最优分割性能, 平均IoU系数为0.8470, 为6种模型中的最优值. LM-UNet模型比UNeXt模型的平均IoU系数提高1.5%. 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet的分割指标提高率如下: 平均IoU系数提高1.5%–6.8%. BUSI数据集上的实验结果表明, 在2D医学图像分割任务中, 横向MLP块具有优化性能和数据泛化的潜力, 以及更高的捕捉高水平语义信息能力.
图4表示的是6个模型在3个不同数据集上得到的分割性能指标平均Dice系数的可视化结果. 其平均Dice系数0.9258, 为6种模型中的最高值. 相比之下, 模型UNeXt的平均Dice系数仅为0.9051, LM-UNet模型较UNeXt模型提高2%. 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet的分割性能也有不同程度的提高, 平均Dice系数较前四者依次提高7.4%、7.2%、3.8%和1.9%; OAI-ZIB 2D数据集上分割效果最好的模型为LM-UNet, 平均Dice系数为0.9170, 并且, LM-UNet模型比UNeXt模型的平均Dice系数提高2.5%. 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet在OAI-ZIB 2D数据集上分割性能也均较前四者有不同程度的提高, 平均Dice系数提高1.7%–5.7%. 在BUSI数据集上, 平均Dice系数为0.9170, 为6种模型中的最优值. LM-UNet模型比UNeXt模型的平均Dice系数提高0.8%, 与U-Net++、ResUNet、TransUNet和U-Net模型相比, LM-UNet的分割指标提高率如下: 平均Dice系数提高0.8%–4.8%.
相比于其他模型, LM-UNet模型在2D医学图像分割应用中具备更优的数据泛化性和鲁棒性. 不同特性的医学图像数据集展现出了各自独特的语义信息和分割挑战. 而LM-UNet模型在3种数据集上均具备较高的分割性能, 这说明该模型可以更好地在训练过程中学习到更多不同类别图像特性和丰富的语义信息, 并能够通过横向MLP块对多层级特征的加强融合以及对多层级特征边界信息的处理, 最终在多个数据集上获得最优分割性能和最接近真实分割值的分割结果.
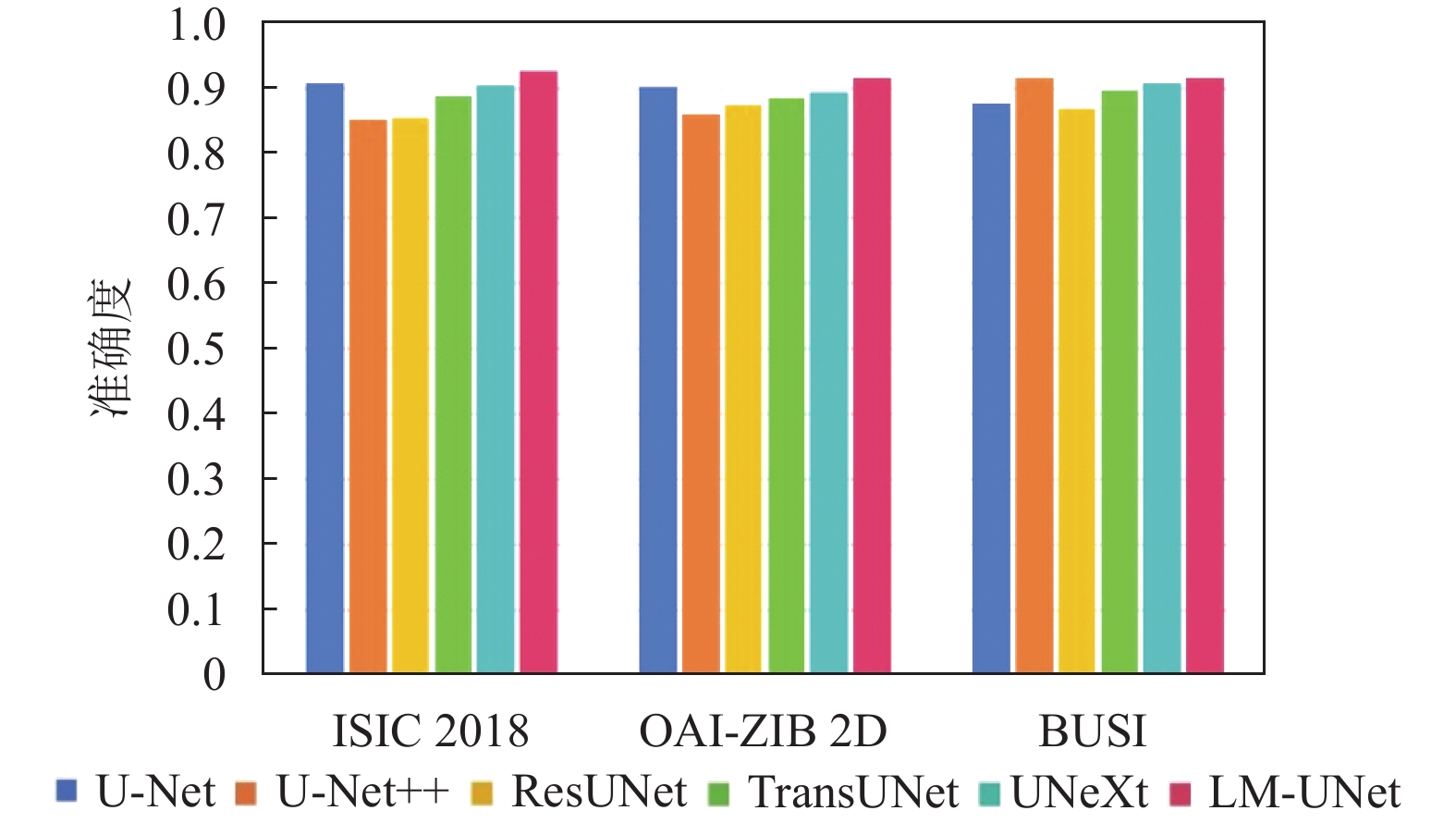
|
图 4 不同模型在3种数据集上的平均 Dice 系数对比 |
3.2 消融实验
选取ISIC 2018数据集和OAI-ZIB 2D数据集进行消融实验, 消融实验结果如表4、表5所示.
| 表 4 ISIC 2018数据集上的消融实验 |
| 表 5 OAI-ZIB 2D数据集上的消融实验 |
将混合MLP块中的令牌混合维度(Td)初始参数设置为32, 通道混合维度(Cd)初始为256. 现有的MLP网络模型通常在宽和高两个方向上进行改进或在模型的瓶颈处进行创新. FF Only[22]在分类任务中的结果表明, 当模型扩大时, 前馈式会有很好的表现. 相比于一个轻量级的模型架构UNeXt, LM-UNet模型的参数量在相对合理的范围内增加, 其本质是因为MLP的全连接特性, 横向MLP块能够较好地建立模型低层级的局部特征间的联系, 并且还能够加强不同层级的特征之间的融合. 横向MLP块对特征图的令牌化分块处理后, 将每块的空间信息和通道信息均使用了全连接操作来进行充分的融合, 可以更好地对特征信息进行学习以及对分割图像的边界进行了加强处理. 因此, 本研究建立了由低层级的横向MLP块、4个对称的混合MLP块和U-Net组成的混合模型. 本文提出的LM-UNet模型适当地扩大了模型参数量并优化了跳跃连接(SC). 但是, 对于这项分割任务来说, 参数量不宜过大, 因为过大的参数量可能会导致基于MLP的模型出现过拟合. 对不同的参数进行实验, 发现参数越大, 效果可能越差. 实验结果表明, 在一般的跳跃连接中, 模型的平均Dice系数为89.54%, 参数为7.44M.
由表4中可以看出, 在前3层(FTL)添加横向MLP块(LMB)操作时, 当参数增加到14.14M, 平均Dice系数增加了3%. 若只在低层(LL)或者只在高层(HL)添加横向MLP块, 并不能够提高分割精度, 如表4所示, 尝试只在低层添加横向MLP块, 发现实验结果参数数量相对较多, 而且无法对后续的特征图进行特征融合, 最终导致对图像语义信息的获取不够充分, 空间信息和信道信息的特征间的联系也没有得到增强. 为了验证LM-UNet模型的稳定性和通用性, 在几个具有不同特征的医学图像数据集上分别进行测试, 结果均显示取得了良好的分割效果. 这说明LM-UNet模型的确提高了医学图像分割的精确度.
表5显示, 如果Td和Cd值过大, 模型很容易出现过拟合. 通过对比实验结果, 发现当(Td, Cd)的组合为(32, 256)时, 分割效果最好, 在OAI-ZIB 2D数据集上的平均Dice系数达到91.70%, 平均IoU系数达到84.70%.
3.3 混合U-Net架构和MLP的优势多阶段设计是CNN模型的标准设计, 但目前在基于Transformer或基于MLP的模型设计中, 多阶段设计的重要性却在很大程度上被忽视了. 研究发现, 无论单独选择CNN、Transformer或MLP这3种网络结构中的哪一种作为单级框架, 其性能都远不及多级框架[23,24]的性能.
通过轻量级深度卷积, 卷积模型在执行计算机视觉下游任务过程中具备较高性能. 当模型规模较小时, MLP具有很强的性能, 但当模型规模增大时, MLP就会出现严重的过拟合问题, 过拟合是MLP实现SOTA性能的主要障碍[25], 因此, 控制模型的参数量以避免过拟合问题是决定模型优劣的关键. U-Net已被广泛应用于医疗分割, 因此沿用这一架构, 使用CNN获取特征的局部关系, 而全局关系则利用MLP获取, 通过MLP融合空间信息和信道信息可以有效避免过多计算量和全局信息替换. 在CNN结合MLP基础上获得的LM-UNet模型保留了跳跃连接, 并且能够通过横向MLP块进一步优化多层级特征信息融合及边缘信息提取, 增强编码器和解码器之间的特征信息传递.
3.4 实验结果6种模型在3个不同数据集上的分割效果如图5所示. 与其他5种模型相比, 由于横向MLP块加强了通道特征信息和空间特征信息之间的关系, LM-UNet模型让图像的边界信息分割的更加明确. 与基准模型U-Net一样, 图像分割的模糊混浊边界得到了改善, 分割出的轮廓更加平滑、轮廓更加清晰. 不同数据上的结果显示LM-UNet模型可以针对不同类型的数据集进行分割, 并获取更接近标签值(ground truth, GT)的分割效果.
皮肤数据集(A)上分割效果的可视化结果显示, LM-UNet模型在边缘的平滑度、边界轮廓的完整性和分割的准确性方面都取得了最佳结果. 对于医学影像分割来说, 从不同尺度的特征图中获得的信息是不同的, 横向MLP块在建立空间和通道信息联系方面发挥着关键作用, 不同的权重矩阵有助于提高LM-UNet模型的分割性能.

|
图 5 ISIC 2018 数据集(A)、OAI-ZIB 2D 数据集(B)和 BUSI 数据集(C)上不同模型的分割效果 |
从软骨图像(B)的分割图中可以看出, LM-UNet模型不论是对于上软骨组织还是下软骨组织都可以清晰分割出来并最接近标签真实值.
乳腺超声图像(C)的特点是背景和待分割区域不太明显, 但该模型仍能捕捉局部信息范围之间的联系和差异, 在合理的参数范围内获得更完整的语义信息, 分割结果也更加与标签真实值接近.
3.5 讨论通过图5的对比, 可以更加直观地看到, 在横向MLP块的作用下, LM-UNet模型实现的图像分割结果拥有更清晰、平滑的边界和轮廓, 其分割准确率显著高于其他模型. 横向MLP块通过混合空间信息和通道信息, 有效改善了边界特征的分割并提升了边界分割精度. 此外, 横向MLP块在模型的前3层进行二次特征提取, 增强了多层级特征中局部与全局特征间的联系, 从而使LM-UNet模型获得更精确的分割结果, 得到的分割图更加平滑、清晰, “云状”轮廓的结构更少.
4 结论与展望本文提出了基于卷积和MLP混合的深度学习模型LM-UNet, 该模型通过增加横向MLP块有效改进了一般的跳跃连接. 在ISIC 2018、OAI-ZIB和BUSI这3个不同的数据集上进行实验, 对比实验结果表明, LM-UNet模型相比于U-Net、U-Net++、ResUNet、TransUNet和UNeXt模型有着更接近于标签值的分割结果, 且LM-UNet模型在ISIC 2018、OAI-ZIB和BUSI这3个数据集上的平均Dice系数相比于UNeXt模型分别提高0.8%、2.0%、2.5%, 平均IoU系数分别提高1.5%、2.9%、4.2%. 消融实验的结果也再次印证LM-UNet模型具有最佳的分割性能. 虽然本研究中LM-UNet模型具有较高的医学图像分割效果, 但MLP本身存在的过拟合问题仍未完全解决, 因此, 在后续的研究中应该重点关注如何精简MLP的全连接操作.
| [1] |
Spanhol FA, Oliveira LS, Petitjean C, et al. A dataset for breast cancer histopathological image classification. IEEE Transactions on Biomedical Engineering, 2016, 63(7): 1455-1462. DOI:10.1109/TBME.2015.2496264 |
| [2] |
Shi C, Xu CH, He JF, et al. Graph-based convolution feature aggregation for retinal vessel segmentation. Simulation Modelling Practice and Theory, 2022, 121: 102653. DOI:10.1016/j.simpat.2022.102653 |
| [3] |
Guan H, Liu MX. Domain adaptation for medical image analysis: A survey. IEEE Transactions on Biomedical Engineering, 2022, 69(3): 1173-1185. DOI:10.1109/TBME.2021.3117407 |
| [4] |
Despotović I, Goossens B, Philips W. MRI segmentation of the human brain: Challenges, methods, and applications. Computational and Mathematical Methods in Medicine, 2015, 2015: 450341. |
| [5] |
Razzak MI, Naz S, Zaib A. Deep learning for medical image processing: Overview, challenges and the future. In: Dey N, Ashour AS, Borra S, eds. Classification in BioApps. Cham: Springer, 2018. 323–350.
|
| [6] |
Ben-Cohen A, Diamant I, Klang E, et al. Fully convolutional network for liver segmentation and lesions detection. Proceedings of the 1st International Workshop on Deep Learning in Medical Image Analysis. Athens: Springer, 2016. 77–85.
|
| [7] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [8] |
Xu CH, Shi C, Chen YN. End-to-end dilated convolution network for document image semantic segmentation. Journal of Central South University, 2021, 28(6): 1765-1774. DOI:10.1007/s11771-021-4731-9 |
| [9] |
Zhou ZW, Rahman Siddiquee M, Tajbakhsh N, et al. UNet++: A nested U-Net architecture for medical image segmentation. Proceedings of the 4th International Workshop on Deep Learning in Medical Image Analysis. Granada: Springer, 2018. 3–11.
|
| [10] |
Isensee F, Petersen J, Klein A, et al. Abstract: NnU-Net: Self-adapting framework for U-Net-based medical image segmentation. In: Handels H, Deserno TM, Maier A, et al., eds. Bildverarbeitung für die Medizin 2019. Wiesbaden: Springer, 2019. 22.
|
| [11] |
Valanarasu JMJ, Oza P, Hacihaliloglu I, et al. Medical Transformer: Gated axial-attention for medical image segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-assisted Intervention. Strasbourg: Springer, 2021. 36–46.
|
| [12] |
Tolstikhin I, Houlsby N, Kolesnikov A, et al. Supplementary materials for: MLP-Mixer: An all-MLP architecture for vision. Proceedings of the 35th Conference on Neural Information Processing Systems. 2021. 24261–24272.
|
| [13] |
Liu HX, Dai ZH, So DR, et al. Pay attention to MLPs. Proceedings of the 35th Conference on Neural Information Processing Systems. 2021. 9204–9215.
|
| [14] |
Valanarasu JMJ, Patel VM. UNeXt: MLP-based rapid medical image segmentation network. Proceedings of the 25th International Conference on Medical Image Computing and Computer-assisted Intervention. Singapore: Springer, 2022. 23–33.
|
| [15] |
Li H, Xu Z, Taylor G, et al. Visualizing the loss landscape of neural nets. Proceedings of the 32nd International Conference on Neural Information Processing Systems. Montréal: Curran Associates Inc., 2018. 6391–6401.
|
| [16] |
陈珊, 郑琴, 秦绪佳. 基于二维经验模式分解的医学图像融合方法. 计算机系统应用, 2014, 23(6): 105-110. |
| [17] |
Hendrycks D, Gimpel K. Gaussian error linear units (GELUs). arXiv:1606.08415, 2016.
|
| [18] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale. arXiv:2010.11929, 2020.
|
| [19] |
Codella N, Rotemberg V, Tschandl P, et al. Skin lesion analysis toward melanoma detection 2018: A challenge hosted by the international skin imaging collaboration (ISIC). arXiv:1902.03368, 2019.
|
| [20] |
Liu Q, Xu ZL, Jiao YN, et al. iSegFormer: Interactive segmentation via Transformers with application to 3D knee MR images. Proceedings of the 25th International Conference on Medical Image Computing and Computer-assisted Intervention. Singapore: Springer, 2022. 464–474.
|
| [21] |
Al-Dhabyani W, Gomaa M, Khaled H, et al. Dataset of breast ultrasound images. Data in Brief, 2020, 28: 104863. DOI:10.1016/j.dib.2019.104863 |
| [22] |
Melas-Kyriazi L. Do you even need attention? A stack of feed-forward layers does surprisingly well on ImageNet. arXiv:2105.02723, 2021.
|
| [23] |
王金祥, 付立军, 尹鹏滨, 等. 基于CNN与Transformer的医学图像分割. 计算机系统应用, 2023, 32(4): 141-148. DOI:10.15888/j.cnki.csa.009010 |
| [24] |
张潮, 宋亚林, 袁明阳. 基于MultiResUNet-SMIS的皮肤黑色素瘤图像分割. 计算机系统应用, 2023, 32(6): 221-230. DOI:10.15888/j.cnki.csa.009113 |
| [25] |
Zhao YC, Wang GT, Tang CX, et al. A battle of network structures: An empirical study of CNN, Transformer, and MLP. arXiv:2108.13002, 2021.
|
 2024, Vol. 33
2024, Vol. 33


