骨关节疾病自古以来是人类最高发的疾病类别之一, 常见的骨关节疾病包括关节炎、颈椎炎、类风湿关节炎等. 20世纪以来, 由于人口老龄化的不断加快, 这类疾病日趋广泛, 给关节外科医师带来了巨大的挑战. 而医学影像是疾病诊断、筛选、评估和确定治疗方案的重要工具, 图像分割技术在临床诊断和治疗中有着重要作用, 能够显著提高诊断效率和准确性. 早期的医学图像分割方法往往依赖于边缘检测、模板匹配技术、统计形状模型等. 尽管这些方法取得了一定的成效, 但特征提取算法的复杂性、局限性、稳定性等因素限制了图像分割技术的进一步发展[1]. 近年来, 在医学图像分割研究中, 深度学习的快速发展使得图像分割算法成为一种有效的工具, 它能够用来提取并分割医学图像中的骨关节结构, 帮助外科医生更有效地进行临床诊断和治疗.
卷积神经网络(convolutional neural network, CNN)是深度学习领域的重要成果, 自卷积神经网络在手写字体识别领域获得巨大突破后[2], 逐渐开始应用在图像处理领域. CNN在医疗影像分析中发挥着重要作用, 用于特征学习的CNN对图像噪声、模糊、对比度等不敏感, 因此在医学图像处理中提供了很好的分割结果. 它可以用于肿瘤检测、疾病诊断、器官分割等任务, 帮助医生更准确地进行诊断和治疗. Long等人[3]2015年提出了全卷积网络(fully convolutional network, FCN)结构. FCN可以对图像进行像素级的分类, 从而解决了语义级别的图像分割问题.
2015年, Ronneberger等人[4]提出了U-Net网络结构, U-Net是一种基于FCN的语义分割网络, 适用于医学图像分割. 但是U-Net网络只能关注到局部性, 不能很好地建立显式的长距离依赖, 也没有完全捕获全局上下文关系. Transformer结构开始用于视觉领域之后, 基于U-Net和Transformer结合的相关研究逐渐兴起. 由于CNN的平移不变性和捕捉长期依赖能力的不足, 限制了U-Net模型的性能, 而Transformer擅长捕捉序列之间的长期依赖关系. 于是, 研究者们尝试将Transformer结构融入到以CNN为主体的U-Net中, 提出了TransUNet网络[5]. TransUNet是第1个将Transformer结构应用于医学图像分割的研究, 它大大地提高了U-Net的分割性能. 但是TransUNet仍然采用CNN和Transformer混合编码的结构, 解码器也是基于CNN的上采样, 这种混合编码的结构没有完全发挥出Transformer的优势. 此前, MSRA提出了Swin Transformer[6]作为新的视觉Transformer backbone, 更适合用于医学图像分割, 基于Swin Transformer的Swin-Unet应运而生[7]. Swin-Unet采用纯Transformer结构的U-shaped网络, 大量实验表明, Swin-Unet在多个器官和心脏数据集上具有良好的分割精度和鲁棒泛化能力.
注意力机制(attention mechanism)最初在机器翻译领域[8]取得成功, 随后被广泛应用于图像处理[9]、语音识别、自然语言处理等方面. 注意力机制可以从众多信息中选择出对当前任务目标更关键的信息, 同时抑制那些不重要的信息. 目前, 注意力机制广泛应用于在图像处理中, 在文献[10]中, Hu等人提出了SENet, 他们在通道维度上对特征层进行特征提取, 从而提高了通道特征信息的表达能力. Fu等人[11]提出双重注意网络, 他们通过自注意机制自适应地融合局部语义特征, 更有效地捕获空间和通道维度上的全局依赖关系. 大量实验研究表明, 注意力机制能够促进图像分割模型更准确地关注关键信息, 提高分割结果的准确性和有效性, 提升模型的效率和可解释性.
目前图像分割也广泛应用于软骨、半月板等人体关节的临床研究, 基于卷积神经网络的图像分割方法可以通过学习关节区域的特征来实现准确的分割. 刘云鹏等人[12]提出一种基于图像块和全卷积神经网络相结合的自动分割算法, 用于对肩关节MRI图像进行分割. Liao等人[13]提出了一种CNN+Bi-RNN方法来确定椎骨的位置, 改善了对特征信息的获取和处理, 提高了分割结果的准确性. Zeng等人[14]提出了一种深度监督的3D U-Net, 用于近端股骨的图像分割. Nie等人[15]使用3D FCN对颅颌面进行分割. 宋平等人[16]构建了PointRend_Unet神经网络, 该方法用于对膝关节CT图像进行三维重建与分割, 有效提高了算法的准确性和分割速度, 提高了人工全膝关节置换术前规划的工作效率.
但是上述这些方法都没有关注编码器和解码器中间的连接通路, 也没有对信息接收提供有效的跳跃连接. 因此, 在先前工作的基础上, 我们基于Swin-Unet模型, 提出了一种新的图像分割算法——DH-Swin Unet, 我们在跳跃连接中引入密集连接的Swin Transformer块, 从而可以更好地使用不同尺度下的移位窗口方案, 增强网络的多尺度特征表示能力并改善长距离相关性. 另外我们在跳跃路径中引入混合注意力机制, 来增强特征描述. 我们构建了一个关节术后感染数据集, 其中包括髋骨数据和膝盖数据. 通过在我们的数据集上进行多任务分割实验, 结果表明, 所提出的方法在不同数据集上取得了更好的分割结果.
1 研究方法 1.1 网络架构图1展示了我们提出的医学图像分割模型的结构. 我们基于Swin-Unet, 提出了一种由编码器、解码器和跳跃连接组成的U型对称网络模型——DH-Swin Unet, 跳跃连接采用由密集Swin Transformer块(DSTB)和混合注意力模块(hybrid attention block)组成的跳跃连接路径, 从而加强了编码器和解码器之间的信息传递有效性. 来自编码器的特征信息在进入解码器之前先经过密集连接的Swin Tansformer块, 再经过混合注意力模块, 最后输出到解码层.
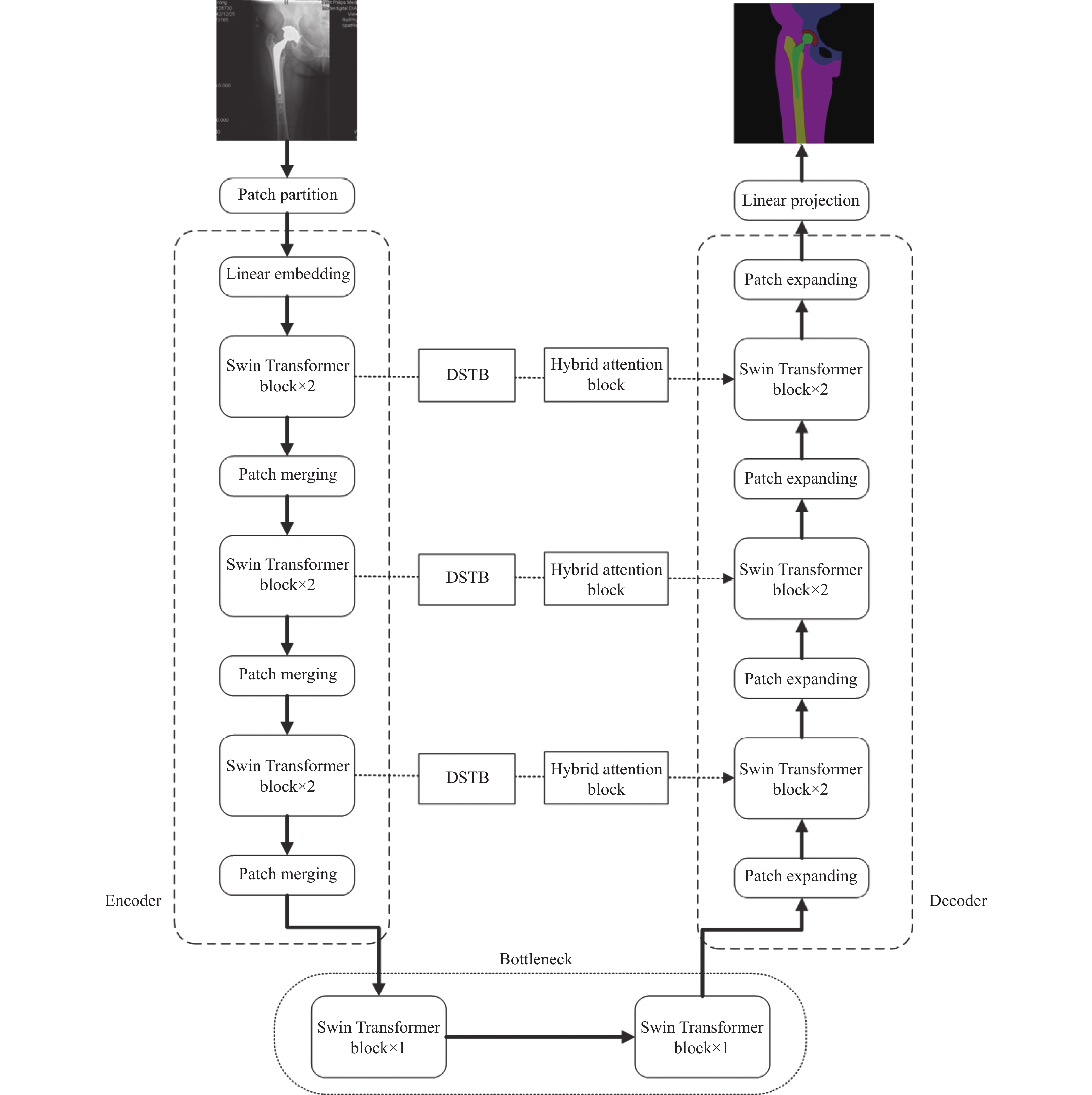
|
图 1 DH-Swin Unet模型结构 |
1.2 跳跃路径
U-Net的大多数变体都使用了简单的跳跃连接, 但是这种跳跃连接不利于长距离的依赖, 会带来语义差距大、信息损失、细粒度细节丢失等问题[17]. U-Net[4]中, 来自编码器的特征与解码器直接相连, 这就要求它们具有相同的比例. 然而, Zhou等人[18]提出即使这些特征图处于相同的比例, 它们在语义上并不相似, 且缺少理论支持这种融合是最优策略. 因此, 他们将注意力放在重新设置编码器和解码器之间的跳跃连接上, 采用密集的卷积块替换了这些简单的连接, 使得编码器特征的语义信息更丰富, 并在合并之前使其语义级别更接近等待的解码器. 这些密集跳跃连接也在文献[19]的Mask-RCNN分割中被证明了有效性. 同样, 密集U-Net++[20]使用半密集U-Net[21], 包含密集跳跃连接和跳跃路径, 每个图层上的密集块使用之前所有图层的聚合要素, 得到更优的结果.
基于以上研究的启发, 我们采用由密集Swin Transformer块和混合注意力模块组成的跳跃连接路径, 来增强模型的显式远程依赖, 促进特征信息在编码器和解码器之间的有效传递.
1.3 密集Swin Transformer模块受密集连接的启发, 我们在跳跃连接路径中采用了密集连接Swin Transformer块, 每个DSTB由多个Swin Transformer组成, 具体结构如图2和图3所示. 每个Transformer将特征信息传播给下一个Swin Transformer. Swin Transformer块是基于平移窗口(shifted windows)构造的, 每个Swin Transformer块由LayerNorm (LN)层、多头自注意模块、剩余连接和具有GELU非线性的两层MLP组成. 在两个连续的Transformer模块中分别采用了基于窗口的多头自注意(W-MSA)模块和位移的基于窗口的多头自注意(SW-MSA)模块[6]. 这个机制表示为:
| ˆzl=W-MSA(LN(zl−1))+zl−1 | (1) |
| zl=MLP(LN(ˆzl))+ˆzl | (2) |
| ˆzl+1=SW-MSA(LN(ˆzl))+ˆzl | (3) |
| zl+1=MLP(LN(ˆzl+1))+ˆzl+1 | (4) |
其中,
| Attention(Q,K,V)=Softmax(QKT√d+B)V | (5) |
其中,
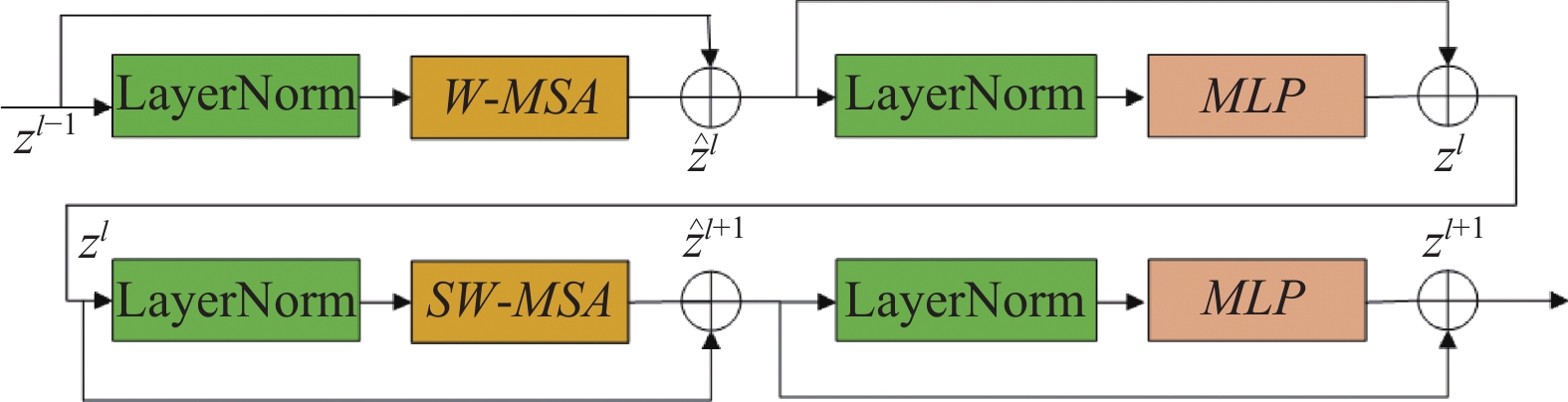
|
图 2 Swin Transformer结构 |
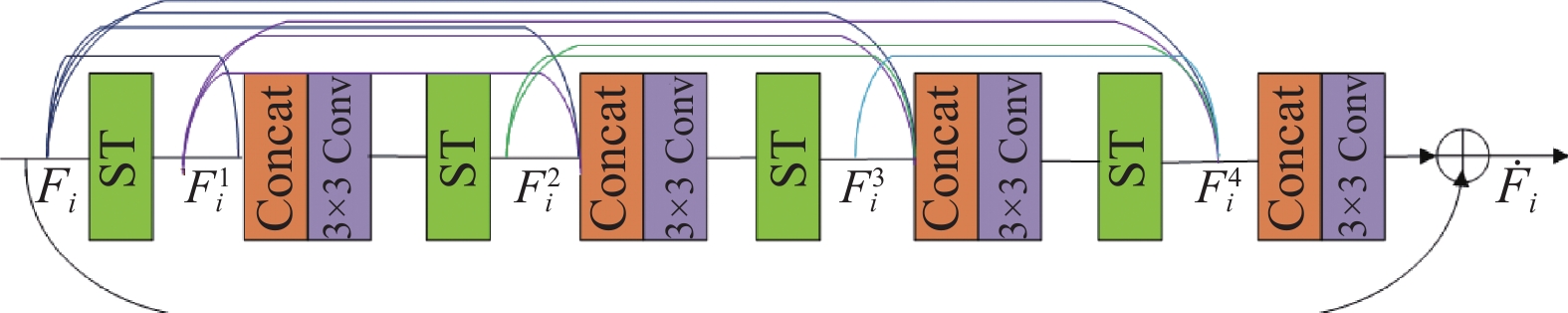
|
图 3 Dense Swin Transformer block结构 |
图3中
| Fti=PST(PFF(Fi,F1i,⋯,Ft−1i)) | (6) |
其中,
在本文中, 我们采用混合注意力机制, 顺序捕获跨多尺度编码器特征的通道和空间依赖性来减小编码器和解码器特征之间的语义差距.
1.4.1 通道注意力模块通道注意力(CA)模块关注图像的关键特征, 如图4所示, 输入特征图的通道信息是一个
| Mchannel(FC)=σ(MLP(Avg(FC))+MLP(Max(FC)))=σ(W1(W0(FAvgC))+W1(W0(FMaxC))) | (7) |
其中,
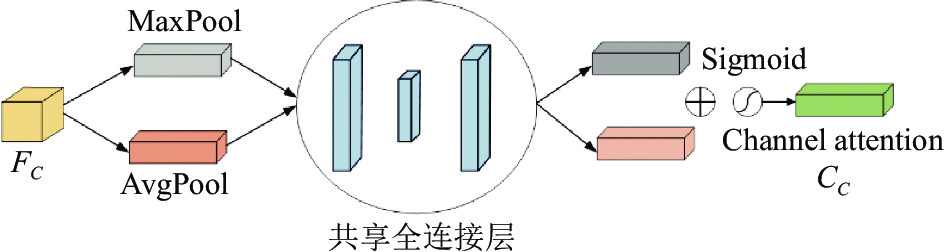
|
图 4 通道注意力模块 |
1.4.2 空间注意力模块
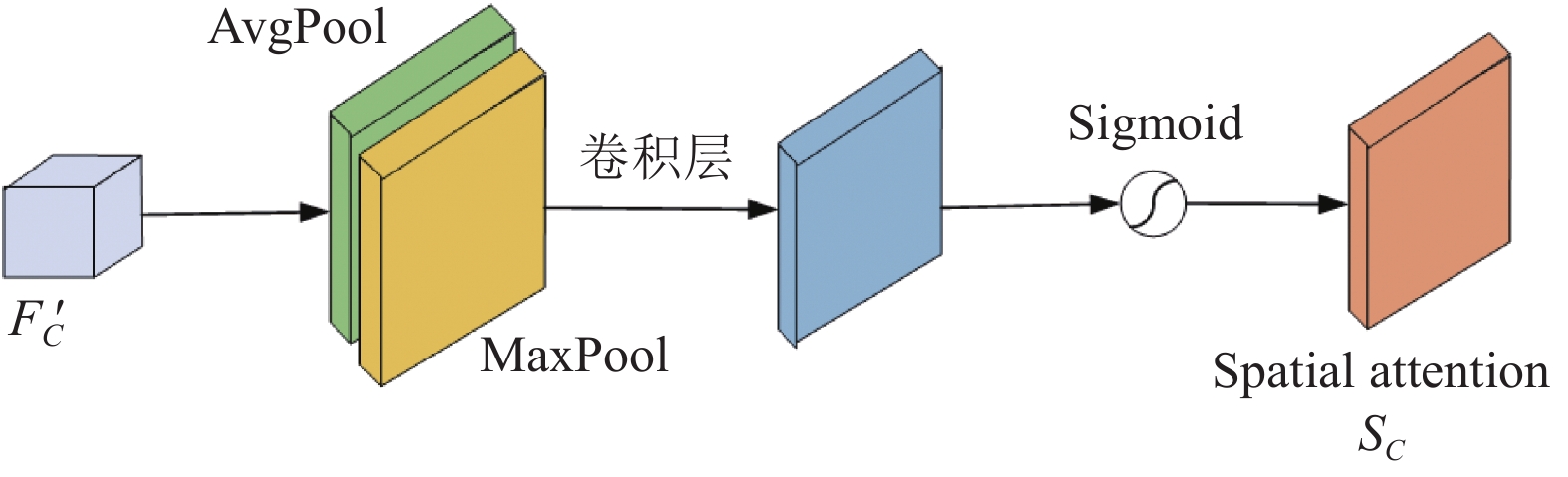
空间注意力(SA)模块的目的是找到特征图中的关键位置关系. 如图5所示, 与通道注意力相似, 给定一个
| Mspatial(F′C)=σ(conv3×3([AvgPool(F′C),MaxPool(F′C)]))=σ(conv3×3([F′AvgC,F′MaxC])) | (8) |
其中,
|
图 5 空间注意力模块 |
1.4.3 混合注意力模块
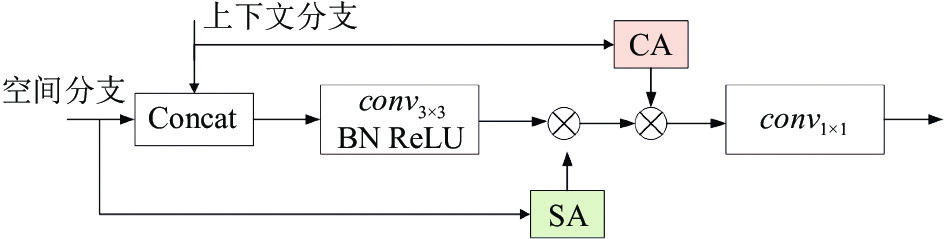
通过在跳跃路径中引入混合注意力模块, 来增强模型的特征描述. 如图6所示, 混合注意力模块首先对空间分支和上下文分支的输出特征进行连接, 接着对融合的特征进行3×3卷积、批量归一化和ReLU单元处理, 再将空间分支的输出通过空间注意力模块进行处理, 得到的结果与之前融合的特征相乘. 再将上下文分支的输出应用于通道注意力模块, 得到的注意力图乘以之前的融合特征, 结果添加到融合特征, 最后对获得的特征进行卷积和上采样操作得到最后的特征图.
|
图 6 混合注意力模块 |
2 实验 2.1 数据集和评价指标
本文的实验数据来自某三甲医院的临床医学图像数据, 其中包括髋骨图像数据757例, 膝盖图像数据241例, 分别实现了多分类医学图像分割任务. 为避免样本过少可能出现的过拟合问题, 我们通过随机旋转、缩放、几何平移等方法对数据集进行了预处理, 最终得到4500张髋骨图像, 1700张膝盖图像作为数据集. 我们用各类别的平均Dice相似系数(DSC)和平均Hausdorff距离(HD)来评估我们的方法. DSC用来度量两个集合的相似度, 侧重于度量分割的内部填充的准确性. HD主要用来度量边界的分割准确度. 它们的定义如下:
| DSC=2|A∩B||A|+|B| | (9) |
| HD(A,B)=max(h(A,B),h(B,A)) | (10) |
| h(A,B)=maxa∈A{minb∈B∥a−b∥} | (11) |
| h(B,A)=maxb∈B{mina∈A∥b−a∥} | (12) |
其中, A代表医学影像的标签图, B代表算法分割的预测图. 式(10)为双向HD, 式(11)和式(12)分别为从A到B和从B到A的单向HD.
2.2 实验细节基于Python 3.7, PyTorch 1.6.0, Nvidia 1080Ti GPU完成实验. 训练时超参数使用如下相同设置: batch_size为4, 初始学习率为0.01, 优化器为随机梯度下降(SGD)混合使用交叉熵损失函数CrossEntropyLoss()和Dice Loss()两个损失函数.
在相同的实验条件下, 将我们的模型分割结果与经典的分割网络U-Net, U-Net++, TransUNet, Swin-Unet等进行对比分析, 实验结果如表1, 表2所示.
| 表 1 不同方法在髋骨数据集上的分割结果 |
从表1中可以看出, 在髋骨数据集上, 我们的模型得到的DSC和HD参数分别为86.79%和32.05 mm, 取得了较为准确的分割结果. 与经典的医学图像分割网络U-Net, U-Net++相比, 我们的模型分割结果在DSC和HD上得到了较大的提升. 与TransUNet和Swin-Unet相比, 我们的DSC分别提高了0.56%, 2.63%, HD分别下降了32.63 mm, 2.51 mm. 与其他关节图像分割方法CNN+Bi-RNN、PointRend_Unet相比, 我们的分割结果得到的DSC分别提高了2.79%和2.09%, 说明我们的模型加强了编码器和解码器之间的信息传递, 减少了信息损失, 提高了算法的分割精度. 我们还在膝盖数据集上评估了我们的方法. 所有实验都是在相同的设置下进行的, 以便进行公平的比较. 表2总结了实验结果, 在膝盖数据集上, 得到的DSC和HD参数分别为88.17%和29.57 mm, 也取得了较好的分割结果. 这表明了我们提出的模型具有通用性和鲁棒性.
| 表 2 不同方法在膝盖数据集上的分割结果 |
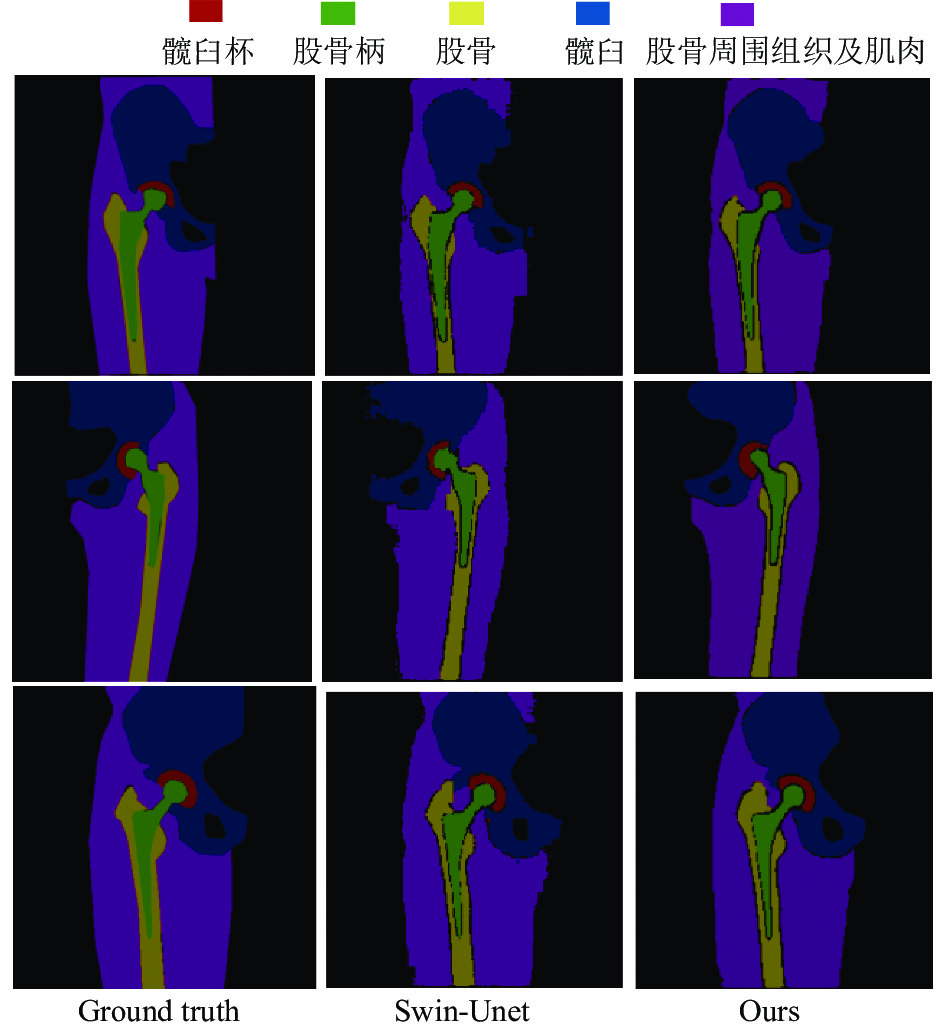
通过对我们提出的模型与Swin-Unet网络的分割结果进行可视化对比分析, 结果如图7. 可以看出我们的模型得到的分割结果边界更加清晰和光滑, 而且对不同类别的分割结果更加准确. 说明本文提出的算法提高了信息传递的有效性.
2.3 消融实验通过分别移除跳跃路径上的密集Swin Transformer模块和混合注意力模块, 来验证它们的有效性, 并在我们的数据集上完成了消融实验, 结果如表3、表4所示. 我们将Swin-Unet作为基准实验模型, 实验表明, 当算法中仅有DSTB模块时, 取得了DSC为86.59%, HD为32.32 mm的结果, 较好地提升了分割的准确性; 仅有混合注意力模块时, DSC为84.41%, 小幅度地提高了分割结果的准确性. 当同时加入DSTB模块和混合注意力模块时, 在髋骨数据集上取得了DSC为86.79%、HD为32.05 mm的分割结果, 在膝盖数据集上取得了DSC为88.17%、HD为29.57 mm的分割结果, 均有显著提升. 因此说明二者对于算法性能的提升都不可缺少, 也证明了我们提出的算法的有效性.
|
图 7 不同方法的分割结果图 |
| 表 3 在髋骨数据集上的消融实验 |
| 表 4 在膝盖数据集上的消融实验 |
3 结束语
在本文中, 我们提出了一种基于Swin-Unet的图像分割方法——DH-Swin Unet, 用于关节图像分割. 我们基于Swin-Unet网络, 重新设计改进其跳跃连接路径, 我们在跳跃连接中引入密集连接的Swin Transformer块和混合注意力模块, 来增强特征描述和信息传递. 通过在多种医学图像数据上进行实验, 结果表明我们所提出的模型具有更好的密集连接效率, 更稳定的信息传递效果. 其中在髋骨数据集上得到了DSC为86.79%、HD为32.05 mm的分割结果, 说明本文所提出的算法的有效性.
| [1] |
Wang RS, Lei T, Cui RX, et al. Medical image segmentation using deep learning: A survey. IET Image Processing, 2022, 16(5): 1243-1267. DOI:10.1049/ipr2.12419 |
| [2] |
LeCun Y, Bottou L, Bengio Y, et al. Gradient-based learning applied to document recognition. Proceedings of the IEEE, 1998, 86(11): 2278-2324. DOI:10.1109/5.726791 |
| [3] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015. 3431–3440.
|
| [4] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [5] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [6] |
Liu Z, Lin YT, Cao Y, et al. Swin Transformer: Hierarchical vision Transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 10012–10022.
|
| [7] |
Cao H, Wang YY, Chen J, et al. Swin-Unet: Unet-like pure Transformer for medical image segmentation. Proceedings of the 2022 European Conference on Computer Vision. Tel Aviv: Springer, 2022. 205–218.
|
| [8] |
Bahdanau D, Cho K, Bengio Y. Neural machine translation by jointly learning to align and translate. Proceedings of the 3rd International Conference on Learning Representations. San Diego, 2015.
|
| [9] |
Hou RB, Chang H, Ma BP, et al. Cross attention network for few-shot classification. arXiv:1910.07677v1, 2019.
|
| [10] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [11] |
Fu J, Liu J, Tian HJ, et al. Dual attention network for scene segmentation. Proceedings of the 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019. 3146–3154.
|
| [12] |
刘云鹏, 蔡文立, 洪国斌, 等. 应用图像块和全卷积神经网络的肩关节MRI自动分割. 中国图象图形学报, 2018, 23(10): 1558-1570. DOI:10.11834/jig.180044 |
| [13] |
Liao HF, Mesfin A, Luo JB. Joint vertebrae identification and localization in spinal CT images by combining short- and long-range contextual information. IEEE Transactions on Medical Imaging, 2018, 37(5): 1266-1275. DOI:10.1109/TMI.2018.2798293 |
| [14] |
Zeng GD, Yang X, Li J, et al. 3D U-Net with multi-level deep supervision: Fully automatic segmentation of proximal femur in 3D MR images. Proceedings of the 8th International Workshop on Machine Learning in Medical Imaging. Quebec City: Springer, 2017. 274–282.
|
| [15] |
Nie D, Wang L, Trullo R, et al. Segmentation of craniomaxillofacial bony structures from MRI with a 3D deep-learning based cascade framework. Proceedings of the 8th International Workshop on Machine Learning in Medical Imaging. Quebec City: Springer, 2017. 266–273.
|
| [16] |
宋平, 范哲奇, 智信, 等. 基于深度学习的膝关节CT图像自动分割准确性验证研究. 中国修复重建外科杂志, 2022, 36(5): 534-539. |
| [17] |
Chaurasia A, Culurciello E. LinkNet: Exploiting encoder representations for efficient semantic segmentation. Proceedings of the 2017 IEEE Visual Communications and Image Processing. St. Petersburg: IEEE, 2017. 1–4.
|
| [18] |
Zhou ZW, Siddiquee MR, Tajbakhsh N, et al. UNet++: Redesigning skip connections to exploit multiscale features in image segmentation. IEEE Transactions on Medical Imaging, 2020, 39(6): 1856-1867. DOI:10.1109/TMI.2019.2959609 |
| [19] |
Zhao HS, Qi XJ, Shen XY, et al. ICNet for real-time semantic segmentation on high-resolution images. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 405–420.
|
| [20] |
Chen FJ, Ding Y, Wu ZX, et al. An improved framework called Du++ applied to brain tumor segmentation. Proceedings of the 15th International Computer Conference on Wavelet Active Media Technology and Information Processing. Chengdu: IEEE, 2018. 85–88.
|
| [21] |
Wu ZX, Chen FJ, Wu DY. A novel framework called HDU for segmentation of brain tumor. Proceedings of the 15th International Computer Conference on Wavelet Active Media Technology and Information Processing. Chengdu: IEEE, 2018. 81–84.
|
| [22] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 3–19.
|
 2024, Vol. 33
2024, Vol. 33



