随着计算机断层扫描(CT)、核磁共振成像(MRI)等医学影像技术的广泛应用, 医生可以更准确地观察人体内部组织结构, 从而做出更精确的诊断. 在腹部CT图像中, 对器官进行精确分割具有关键意义, 对于进行器官形态与大小量化、疾病诊断、放射学分析、手术规划和导航等临床应用来说尤为重要[1]. 作为腹部相关疾病诊断的常规检查方法, 腹部CT扫描具备重要的临床应用价值. 然而, 在传统临床诊断中, 手工完成器官分割任务依赖经验丰富的专业人员, 耗时且繁琐. 为了解决这一问题, 计算机断层扫描(CT)的自动分割方法得到了广泛研究. 尽管取得了一定的进展, 但由于腹部CT图像的复杂背景和模糊的组织边缘, 多器官分割任务依然具有极高的挑战性.
CT图像自动分割方法主要包括传统方法和基于深度学习的方法. 其中, 传统的CT图像分割方法采用低层次的体素处理, 包括阈值分割方法[2]、边缘检测方法、区域生长[3]和聚类[4]等方法. 这些方法是通过强度或梯度的差异来实现的, 但需要专业知识和大量的人工设计的特征, 并且分割效果受到特征选择的影响[5]. 例如: 基于阈值的分割方法对阈值的选择极其敏感, 并在灰度值不均匀和边界模糊的多器官分割中鲁棒性较差. 尽管传统方法在某些方面具有优势和应用价值, 但仍需要更高效、更精准的分割工具来提高分割精度. 近年来, 深度学习方法在医学图像分割领域得到广泛应用. 这些方法利用卷积神经网络等复杂结构, 以自动学习多层次的图像特征. 其中, U-Net模型[6]是一种代表性的深度学习模型, 具备自动提取图像特征的能力. 然而, 目前这些方法针对单一器官分割问题, 基于深度学习的多器官分割方法仍处于起步阶段, 需要进一步的研究和完善以实现高精度的分割结果.
为实现自动高效的腹部多器官分割, 研究人员提出多种解决方案. 例如Chu等人[7]提出了一种通过使用患者特定的加权概率图谱进行3D腹部多器官分割. Gibson等人[8]提出了一个基于FCN网络的Dense-VNet用于腹部多器分割. Roth等人[9]通过使用密集的块级跳跃连接来级联两个相同的3DVnet来分割CT图像中多个器官, 以提高级联结构效率, 并且提高了准确率. 这些深度学习算法在多器官分割任务中表现出了较好的效果, 同时也存在着共同问题: 针对位置相对固定且体积较大的器官有较好的分割效果, 如: 肝脏、左右肾脏、脾脏等. 但针对主动脉、胰腺、胆囊等形变较大、体积较小且边缘模糊的器官分割效果相对较差.
为了解决以上困难, 本文提出了一种改进模型, 旨在进一步优化基于U-Net的多器官分割模型的性能. 本文提出了两个改进模块, 并将其引入U-Net模型. 首先, 我们引入注意力机制模块(A2B), 通过自适应调整特征的权重, 减少对背景特征的关注, 从而更好地区分前景和背景. 同时, 通过残差连接有助于信息传递和梯度流动. 其次, 我们引入多尺度空洞卷积模块(MDCB). 该模块利用空洞卷积和跳跃连接的机制, 能够捕获不同尺度上的细节信息, 适应不同大小的器官结构, 提高分割结果的细腻程度, 进而提高体积较小器官的分割性能和鲁棒性.
1 相关工作 1.1 U-Net分割网络目前, 将深度学习技术应用于医学图像分割以定位病灶并辅助诊断已成为流行趋势. 其中, U-Net是一种常见的方法, 其采用编码器和解码器的对称结构, 并通过级联操作来融合浅层和深层的信息. U-Net具备简单的网络结构和良好泛化能力, 已被广泛用作医学图像分割的基准方法. 研究人员对U-Net进行了多种改进, 并将其应用于不同的医学分割任务. 例如: Bi等人[10]提出一种改进的U-Net模型, 并将其应用于甲状腺超声分割, 该模型引入了BPSM、AMFFM和ASTM来提取更丰富的特征, 以获得更准确甲状腺结节分割结果. Yang等人[11]提出了一种名为ADS-UNet的全新阶段式加性训练算法, 该算法应用于组织病理学领域. 该算法降低了组成特征之间的相关性, 增强了资源利用效率. Wang等人[12]提出了一种多尺度大卷积核MLKCA-UNet模型, 应用于脊髓MRI图像. 该模型利用不同卷积核的大核卷积来处理特征映射, 显著减少了计算量. Sun等人[13]提出了一种改进的U-Net, 用于腺体分割. 通过引入双分支注意力模块和多尺度融合模块, 以引导细化特征并提高分割的准确性. 这些方法都没有考虑到腹部CT图像中器官分布密集、形状大小可变等特点, 因此在多器官分割任务上的性能还有待提升. 多器官分割对医学分割模型获取信息的能力提出了更高的要求.
1.2 注意力机制在计算机视觉任务中, 存在与研究任务相关的目标信息和与任务无关的背景噪声的问题. 为了解决这一问题, 注意力机制被引入, 模仿人类场景感知和观察的过程, 能够快速扫描场景, 并有选择性地聚焦于突出区域. 在相关领域, 研究者已经做出许多成功的实验与研究. Schlemper 等人[14]提出了attention gate (AG)并将其与U-Net结合应用于胎儿超声筛查. Roy等人[15]在全卷积神经网络上引入了3个挤压和激发(SE)模块, 以提高通道方向特征的利用率, 并将其用于多器官分割. Wang等人[16]提出了深度注意力特征(DAF)模块, 抑制CNN低层特征的背景噪声, 增加高层特征的目标细节信息, 提高网络性能, 并用于状态TRUS图像分割. Hou等人[17]提出了一种基于注意力机制的特征融合算法, 并将其应用于主动脉分割. Duan等人[18]提出了一种边缘注意力机制来引导网络关注目标区域的边界信息. 现有注意力机制缺乏灵活性, 通常是静态的, 即对于给定输入具有固定的权重分布. 然而, 在某些情况下, 图像特征的重要性可能会随着上下文的变化而变化. 因此, 我们需要更灵活的注意力机制来适应不同上下文并充分利用图像特征的动态变化.
2 基于改进的U-Net骨干网络为了提高多器官分割精度同时控制计算成本, 本文采用U-Net作为分割模型的主干网络, 主干网络的结构如图1所示. 主干网络主要由两个部分组成, 左侧是编码路径, 右侧是解码路径. 编码网络的每一层由两个卷积层组成, 包括3×3卷积和2×2的池化层. 每次池化操作后, 卷积核的数量会增加两倍. 通过逐步的卷积和池化操作, 从而提取医学图像的特征, 而池化操作可以确保感受野最大化, 但同时也会逐渐减小图像的分辨率. U-Net解码路径中普通卷积模块可能会导致特征信息提取不足和微小特征的丢失. 因此, 在解码路径中每一层都将普通卷积和空洞卷积融合, 每个空洞卷积层都具有不同的空洞率(dilation rate), 分别为1, 3, 5. 通过增加卷积核的感受野, 有助于理解CT图像的背景, 可以在保持计算效率的同时获得更大范围的上下文信息. 由于复杂背景的影响, 特征提取过程容易丢失目标信息. 因此在跳跃连接处加入了注意力机制, 能够让网络自适应聚焦在重要的图像特征上, 从而提高特征提取效果.
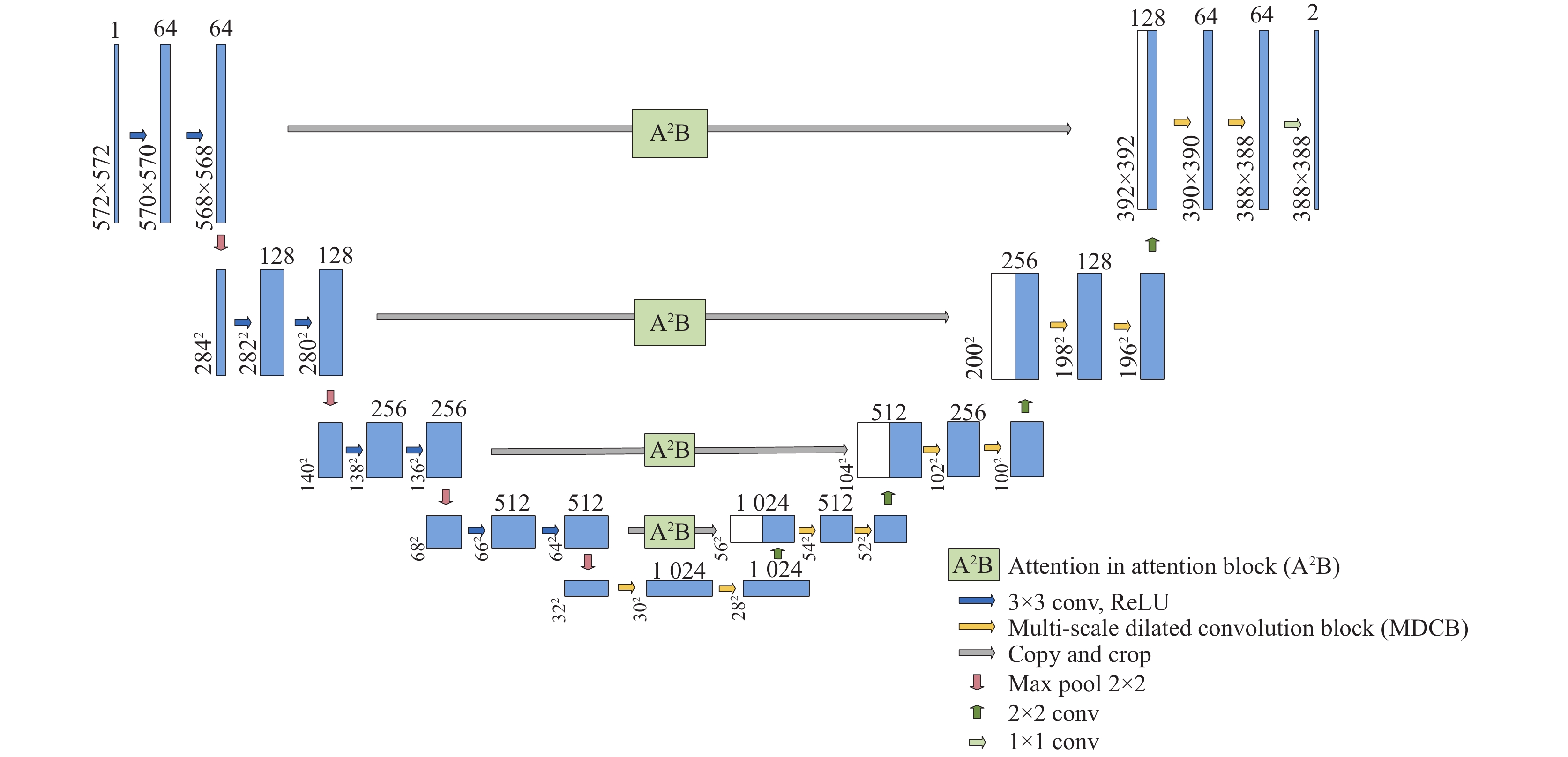
|
图 1 改进后的U-Net网络结构 |
2.1 注意力机制模块
虽然U-Net网络在轻量级方面具有优势, 但其计算能力有限. 为了将计算资源分配给相对重要的任务并减少复杂的计算参数, 本文引入了一种注意力机制 (attention in attention block, A2B) [19]. 该模块具有强大的特征选择和细化能力, 非常适用于图像分割任务. A2B是一种可学习的动态关注模块, 可以自动剔除一些不重要的关注特征, 以实现注意分支和非注意分支的平衡. 每个动态注意模块使用加权求和的方式来控制注意分支和非注意分支的动态加权贡献. 该模块的结构如图2所示. 动态注意力模块通过使用相同的输入特征作为两个独立分支来生成权重, 从而实现动态的关注和权衡公式如式(1)所示:
| xn=f1×1(πnan×xnan+πattnnxattnn) | (1) |
其中,
| πn=fda(xn−1) | (2) |
| πnan+πattnn=1 | (3) |
其中,

|
图 2 A2B的网络架构 |
2.2 多尺度空洞卷积模块
空洞卷积[20]主要应用于图像分割任务中, 可以有效地增加卷积模块的感受野, 图3中展示了本文提出的多尺度空洞卷积模块 (multi-scale dilated convolution block, MDCB)的整体结构. 对于空洞卷积来说, 选取不同的空洞率 (dilation rate)可以获取不同尺度的像素信息.
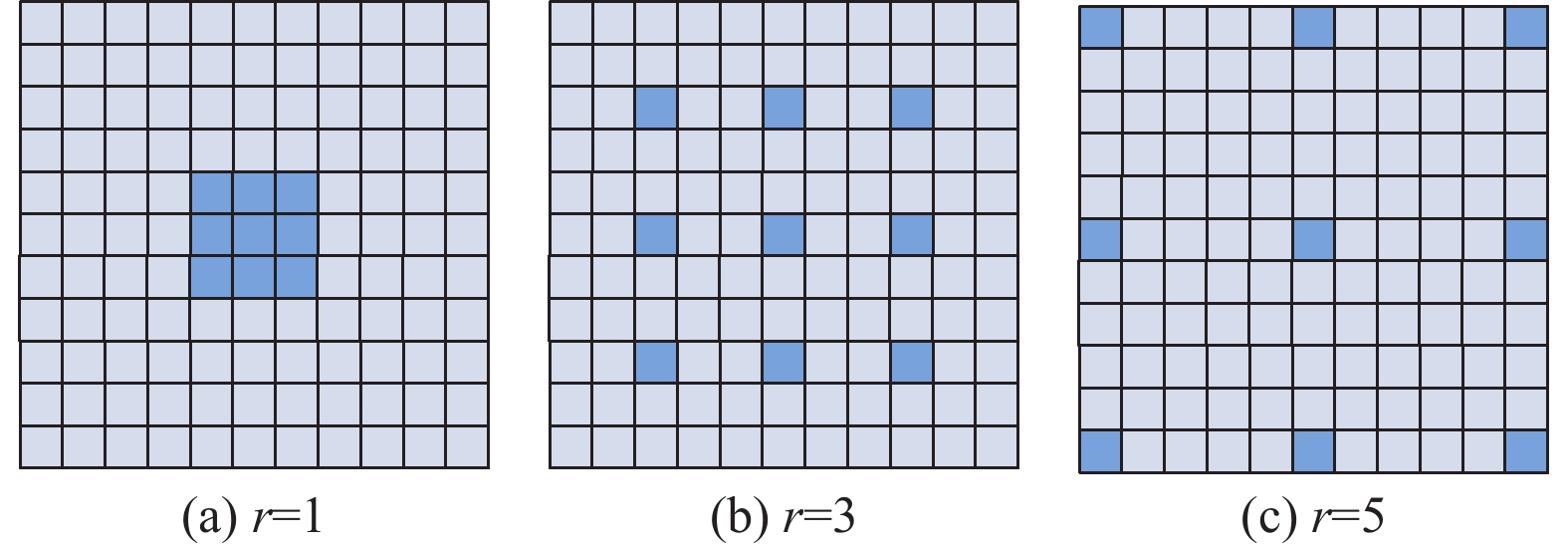
|
图 3 不同空洞率的空洞卷积示意图 |
为了在多器官图像中获取多尺度的空间信息, 采用3种空洞率对输入特征进行处理, 空洞率分别为1, 3, 5. 如图3所示, 首先将一个1×1卷积层和3×3卷积层的组合对输入特征图进行处理, 得到中间特征图
本文采用MICCAI 2015多图谱腹部标记挑战赛中的Synapse数据集进行多器官CT分割. 该数据集包含30名受试者的腹部CT扫描图像, 共计3779张轴向增强的临床CT图像, 涵盖了8个腹部器官. 每个CT图像由85–198个512×512像素的切片组成, 像素空间分辨率为[0.54–0.54]×[0.98–0.98]×[2.5–5.0] mm3. 实验使用了95% Hausdorff距离(HD)和Dice相似系数(DSC)对8个腹部器官(主动脉、胆囊、左肾、右肾、肝脏、胰腺、脾脏、胃)进行评估. 在Synapse数据集中选取18个病例(包含2212张切片)作为训练集, 12个病例(包含1474张切片)作为测试集.
3.2 实验训练本实验是在Ubuntu 20.04操作系统下进行, 编程环境为Python 3.8, 深度学习框架为PyTorch 1.11.0, CUDA 11.3, 显卡RTX3090 GPU, 显存24 GB. 训练次数为300 epoch, 初试阶段学习率设置为0.01, 批量大小为24, 输入图像大小为224. 此外, 采用Adam优化器对所有模型进行优化, 其中权重衰减设置为1×10−4.
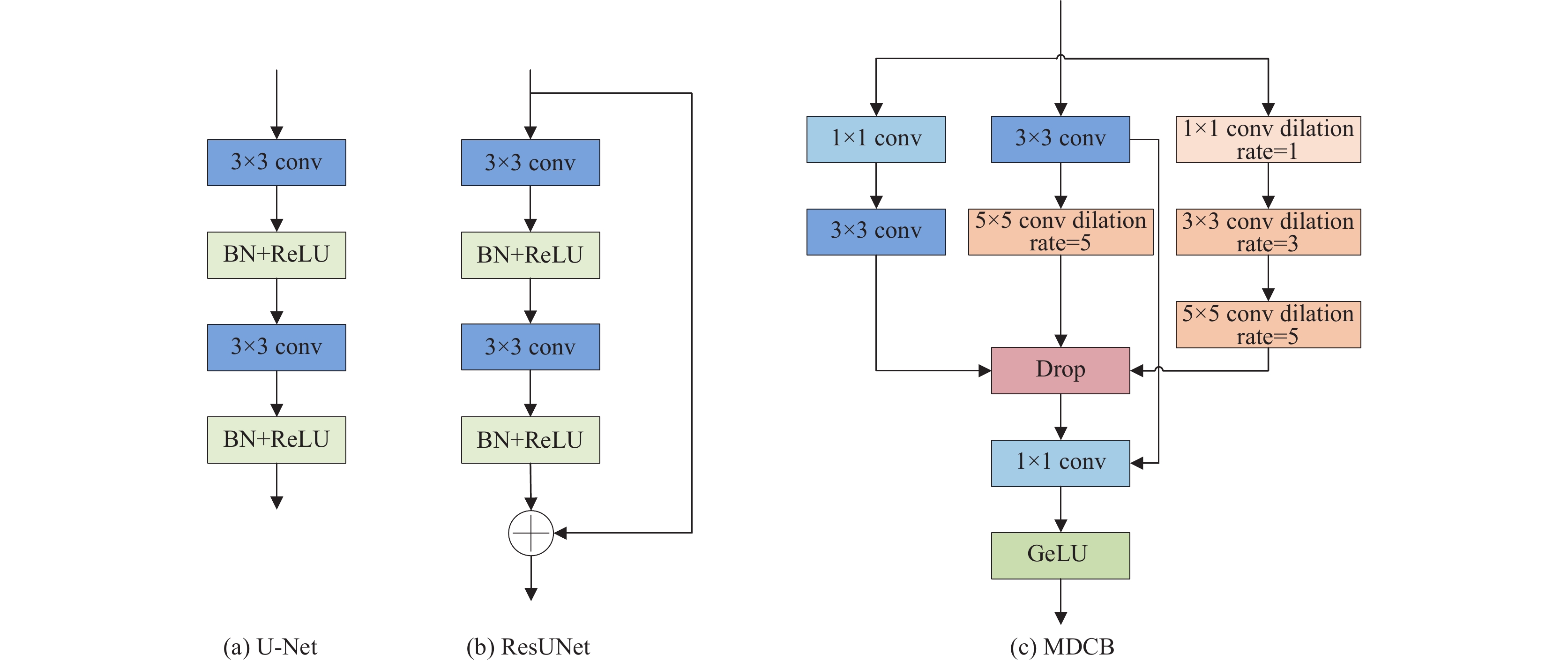
|
图 4 不同模型卷积块的比较 |
为了应对多器官分割中类别分布不平衡的挑战, 构建了Dice损失
| Ltotal=LCE+λLDice | (4) |
其中,
为了验证模型的分割性能, 我们选择了300个epoch进行验证损失的评估, 如图5所示. 从图5中可以明显看出, 未经改进的图像损失函数表现出较大的波动, 导致训练不够稳定, 损失值高于改进后的图像损失值, 进而导致预测结果的质量较差. 经过改进后的模型经过了100个epoch的训练, 损失函数的变化趋势变得相对平稳, 不再表现出明显的波动. 这表明改进后的模型训练更加稳定. 与未改进的模型相比, 改进后的模型表现出更好的性能, 损失值更低, 表明模型对数据的拟合更好, 能够更准确地进行分割任务.
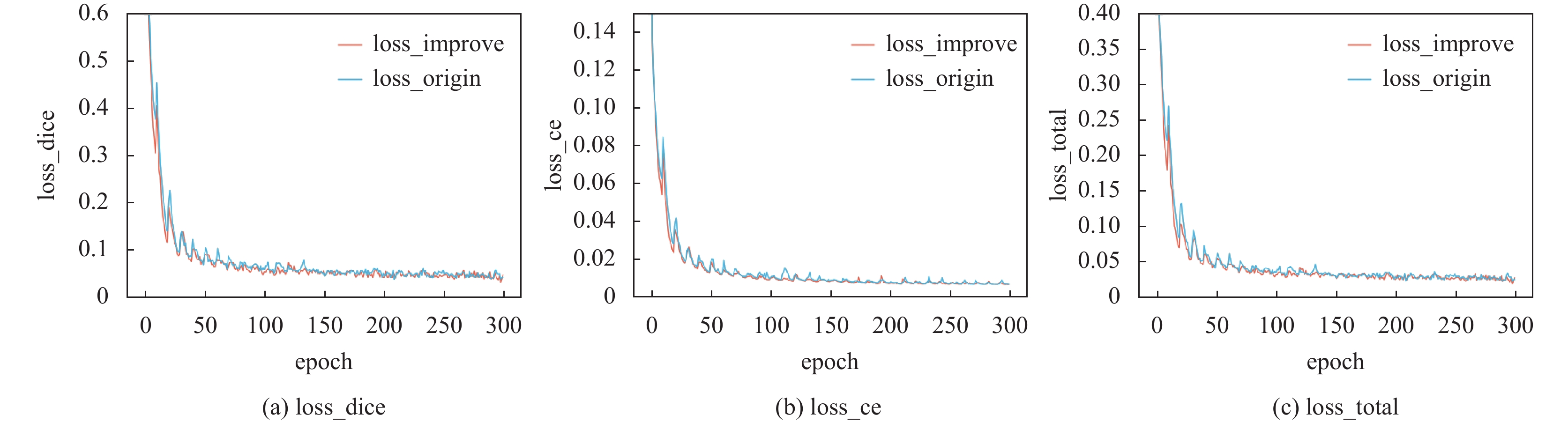
|
图 5 损失函数对比图 |
3.3 评价指标
为了进行分割结果的定量性能评估, 本文采用了Dice相似系数(Dice similarity coefficient, DSC)、95%豪斯多夫距离作为评价指标. Dice相似性系数用于衡量分割结果与地面真值之间的重叠程度, 其取值范围为
| DSC=2×|X∩Y||X|+|Y| | (5) |
其中,
HD是
| HD(X,Y)=max{maxx∈Xminy∈Yd(x,y),maxy∈Yminx∈Xd(x,y)} | (6) |
其中,
为了实现更具临床应用价值的多器官分割模型并克服背景复杂、边界模糊、大小形状不同带来的挑战, 研究提出了两种基于U-Net架构的改进模型, 分别是注意力机制模块(A2B)、多尺度空洞卷积模块(MDCB). 这些改进模型旨在提高分割模型的性能和准确性. 如表1所示.
| 表 1 Synapse数据集上不同模块的消融研究 |
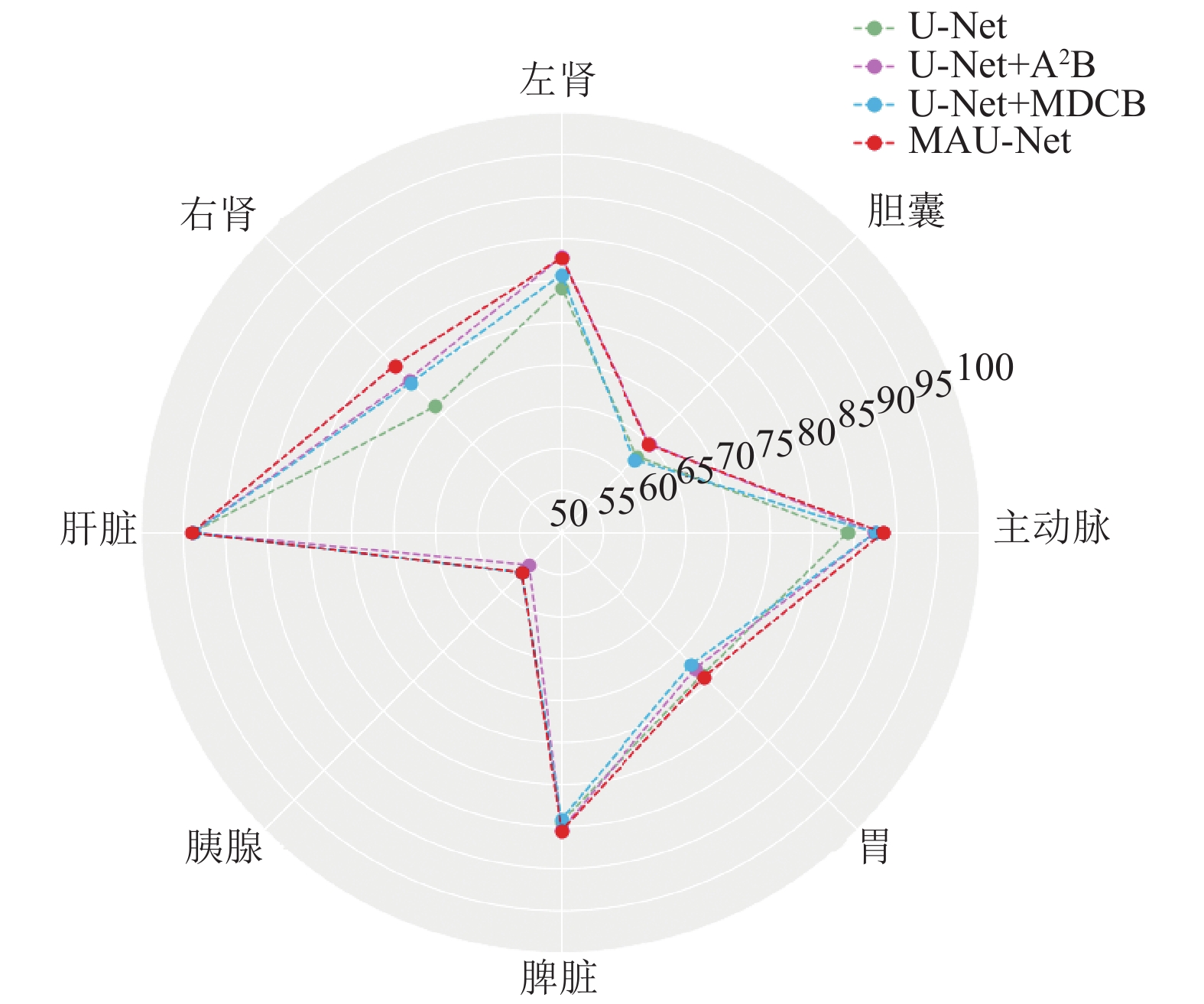
从消融研究可以发现, 相比于原始U-Net, 加入A²B注意力机制模块的网络在主动脉、胆囊、左肾、右肾、胰腺、脾脏上DSC分别提升了3.29%、2.11%、3.64%、4.49%、7.21%、0.96%. 此外, 平均HD也降低了2.47 mm. 同样对比原始U-Net, 加入多尺度空洞卷积模块的网络在主动脉、左肾、右肾、脾脏上Dice分别提升了3.32%、1.53%、4.05%、8.51%. 同时, HD也降低了4.95 mm. 综上所述, 加入注意力机制模块(A2B)和多尺度空洞卷积模块(MDCB)的U-Net网络在大多数器官中展现出了良好的性能提升. 图6为表1的视觉雷达图, 用于比较加入不同模块的Dice在不同器官上的性能. 雷达图清晰地展示了各个器官的数值, 并且突出显示了改进后模型Dice在所有器官上都取得了最佳值和最大的覆盖面积. 改进后的模型不仅在每个器官的分割数值上表现优异, 还能更全面地捕捉到器官的整体轮廓, 提高了分割结果的准确度和精度.
3.5 对比实验在相同的实验条件下, 所提出的模型与表2中挑选出的几个最先进的模型进行了比较研究. 所有结果递归比较, 比较表2中所示的方法在同一数据集上训练结果证明所提出的模型在分割精度和分割准确度上的优势.
|
图 6 不同模块在Synapse多器官CT数据集Dice系数比较 |
| 表 2 不同方法在Synapse多器官CT数据集上的实验结果比较 |
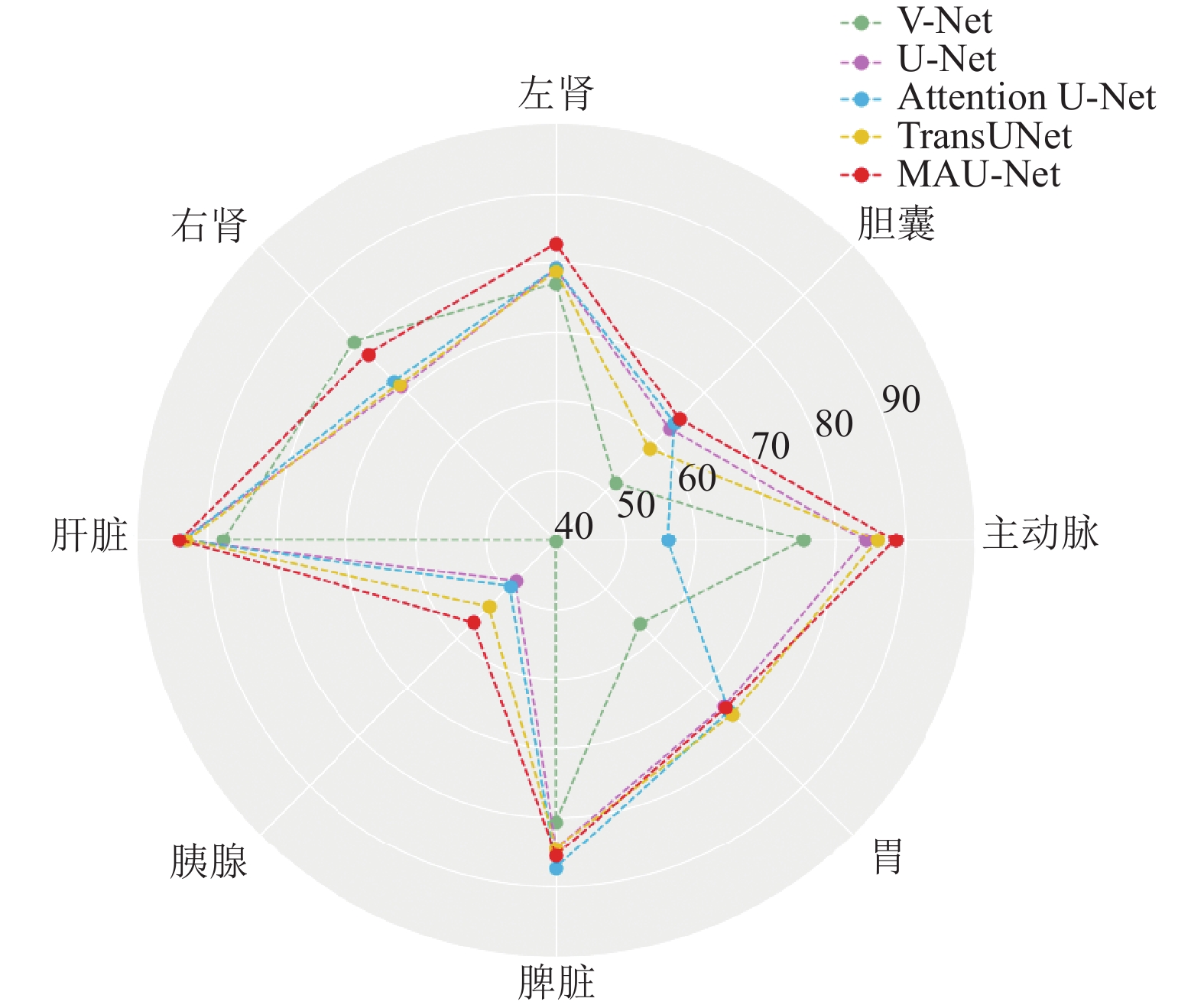
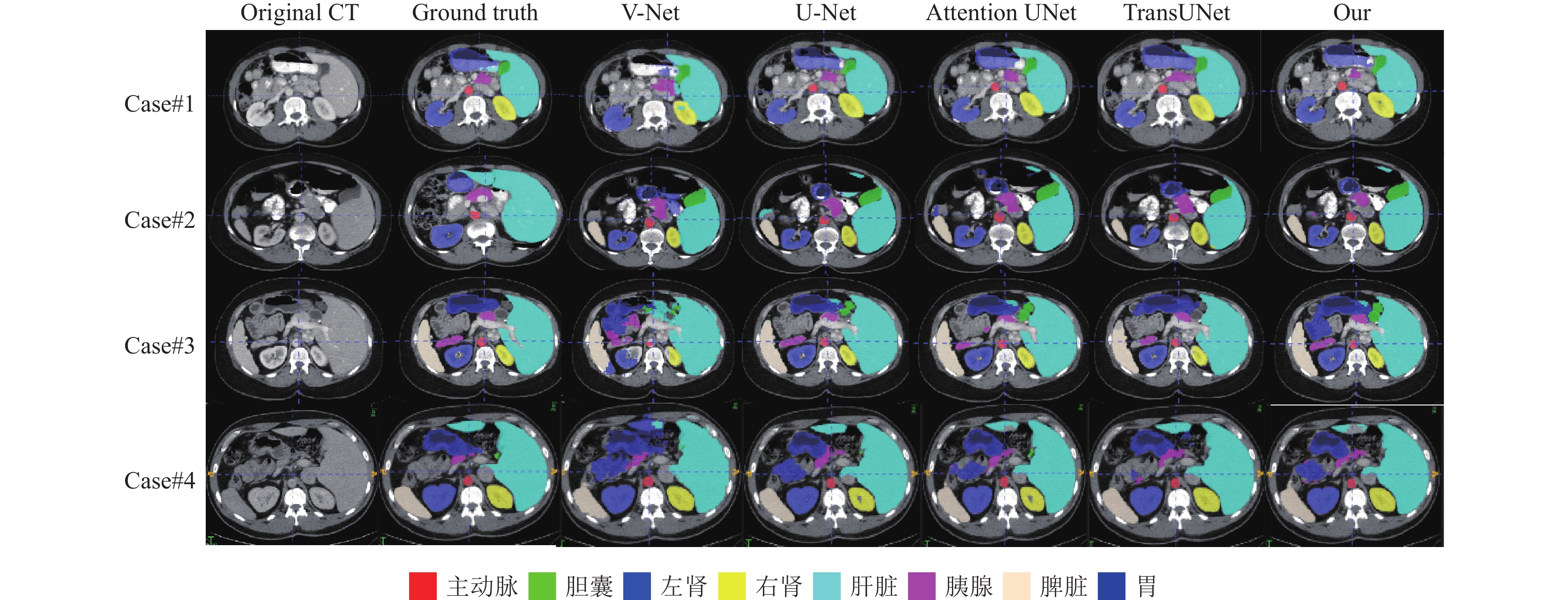
表2显示了经过改进后的网络与6个基线网络(V-Net[21]、U-Net[6]、Attention U-Net[22]、ViT[23]、R50+ViT[23]、TransUNet[24])在性能上进行比较. 结果表明, 具有多尺度空洞卷积模块和注意力模块的网络在大多数器官中表现出最佳性能. 例如: 在主动脉中, 改进后的网络的DSC为88.43%, 而其余基线网络的最佳DSC为85.97%. 然而, 对于一些数据量较小的器官(如右肾、胃)上的表现比其他网络差, 例如V-Net中右肾的DSC为80.75%, 而改进后的DSC仅为78.13%. 对于所有器官平均DSC中, 改进后网络的平均DSC为78.07%, 而其余基线网络最佳平均DSC为75.23%. 此外, 改进后的网络在所有这些网络中平均HD最低, 为32.03 mm. 为了更加直观地展示效果, 图7为表2的视觉雷达图, 用于比较不同方法的DSC在各个器官上的性能. 在图中可以观察到改进后的模型在大部分器官上都达到了最佳值和最大的覆盖面积. 这一结果综合体现了改进后方法的优越性. 此外, 图8展示分割结果的可视化, 可以看出改进后的模型分割边缘更加平滑, 没有明显的锯齿状或不连续性, 这进一步证明了改进后模型在提高分割结果质量方面的优势.
|
图 7 不同方法在Synapse多器官CT数据集上Dice系数比较 |
|
图 8 不同方法的多器官分割结果示例 |
4 结论与展望
为了获得更具临床应用潜力的腹部多器官分割, 解决背景复杂、边界模糊、大小形状不同带来的困难, 本研究提出了一种基于改进的U-Net网络, 加入注意力机制模块(A2B)和多尺度空洞卷积模块(MDCB)可以提高模型对关键特征的学习和利用能力, 增强对上下文信息和细节信息的感知能力, 进而提高模型的性能和泛化能力. 因此, 所提出的模型可以更好地处理各种器官的不同形状和大小. 通过实验证明, 该模型较对比方法而言在临床应用中更具优越性. 所提出的模型2D输入未能利用CT序列图像的空间信息. 因此未来可以利用一些低成本、高效的序列分割策略进一步改进所提出的模型.
| [1] |
Fu YB, Lei Y, Wang TH, et al. A review of deep learning based methods for medical image multi-organ segmentation. Physica Medica, 2021, 85: 107-122. DOI:10.1016/j.ejmp.2021.05.003 |
| [2] |
Moghadas-Dastjerdi H, Ahmadzadeh M, Samani A. Towards computer based lung disease diagnosis using accurate lung air segmentation of CT images in exhalation and inhalation phases. Expert Systems with Applications, 2017, 71: 396-403. DOI:10.1016/j.eswa.2016.11.013 |
| [3] |
Bozkurt F, Köse C, Sarı A. An inverse approach for automatic segmentation of carotid and vertebral arteries in CTA. Expert Systems with Applications, 2018, 93: 358-375. DOI:10.1016/j.eswa.2017.10.041 |
| [4] |
Kaur H, Kaur N, Neeru N. Evolution of multiorgan segmentation techniques from traditional to deep learning in abdominal CT images—A systematic review. Displays, 2022, 73: 102223. DOI:10.1016/j.displa.2022.102223 |
| [5] |
Shen NY, Wang ZY, Li J, et al. Multi-organ segmentation network for abdominal CT images based on spatial attention and deformable convolution. Expert Systems with Applications, 2023, 211: 118625. DOI:10.1016/j.eswa.2022.118625 |
| [6] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [7] |
Chu CW, Oda M, Kitasaka T, et al. Multi-organ segmentation from 3D abdominal CT images using patient-specific weighted-probabilistic atlas. Proceedings of the 2013 Medical Imaging Conference on Image Processing. Florida: SPIE, 2013. 1090–1096.
|
| [8] |
Gibson E, Giganti F, Hu YP, et al. Automatic multi-organ segmentation on abdominal CT with dense V-networks. IEEE Transactions on Medical Imaging, 2018, 37(8): 1822-1834. DOI:10.1109/TMI.2018.2806309 |
| [9] |
Roth HR, Oda H, Zhou XR, et al. An application of cascaded 3D fully convolutional networks for medical image segmentation. Computerized Medical Imaging and Graphics, 2018, 66: 90-99. DOI:10.1016/j.compmedimag.2018.03.001 |
| [10] |
Bi H, Cai CJ, Sun JW, et al. BPAT-UNet: Boundary preserving assembled Transformer UNet for ultrasound thyroid nodule segmentation. Computer Methods and Programs in Biomedicine, 2023, 238: 107614. DOI:10.1016/j.cmpb.2023.107614 |
| [11] |
Yang YL, Dasmahapatra S, Mahmoodi S. ADS_UNet: A nested UNet for histopathology image segmentation. Expert Systems with Applications, 2023, 226: 120128. DOI:10.1016/j.eswa.2023.120128 |
| [12] |
Wang B, Qin J, Lv LR, et al. MLKCA-UNet: Multiscale large-kernel convolution and attention in UNet for spine MRI segmentation. Optik, 2023, 272: 170277. DOI:10.1016/j.ijleo.2022.170277 |
| [13] |
Sun JM, Zhang X, Li XM, et al. DARMF-UNet: A dual-branch attention-guided refinement network with multi-scale features fusion U-Net for gland segmentation. Computers in Biology and Medicine, 2023, 163: 107218. DOI:10.1016/j.compbiomed.2023.107218 |
| [14] |
Schlemper J, Oktay O, Schaap M, et al. Attention gated networks: Learning to leverage salient regions in medical images. Medical Image Analysis, 2019, 53: 197-207. DOI:10.1016/j.media.2019.01.012 |
| [15] |
Roy AG, Navab N, Wachinger C. Concurrent spatial and channel ‘squeeze & excitation’ in fully convolutional networks. Proceedings of the 21st International Conference on Medical Image Computing and Computer-assisted Intervention. Granada: Springer, 2018. 421–429.
|
| [16] |
Wang Y, Deng ZJ, Hu XW, et al. Deep attentional features for prostate segmentation in ultrasound. Proceedings of the 21st International Conference on Medical Image Computing and Computer-assisted Intervention. Granada: Springer, 2018. 523–530.
|
| [17] |
Hou GM, Qin JH, Xiang XY, et al. AF-Net: A medical image segmentation network based on attention mechanism and feature fusion. Computers, Materials & Continua, 2021, 69(2): 1877–1891.
|
| [18] |
Duan XJ, Shi MC, Wang JM, et al. Segmentation of the aortic dissection from CT images based on spatial continuity prior model. Proceedings of the 8th International Conference on Information Technology in Medicine and Education. Fuzhou: IEEE, 2016. 275–280.
|
| [19] |
Chen HY, Gu JJ, Zhang Z. Attention in attention network for image super-resolution. arXiv:2104.09497, 2021.
|
| [20] |
Yu F, Koltun V. Multi-scale context aggregation by dilated convolutions. Proceedings of the 4th International Conference on Learning Representations. San Juan, 2016.
|
| [21] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision. Stanford: IEEE, 2016. 565–571.
|
| [22] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [23] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale. Proceedings of the 9th International Conference on Learning Representations. OpenReview.net, 2021.
|
| [24] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
 2024, Vol. 33
2024, Vol. 33



