2. 中国科学院大学, 北京 100049;
3. 中国医科大学附属第四医院, 沈阳 110032;
4. 东软集团股份有限公司 医疗解决方案事业本部, 沈阳 110003
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. The Fourth Affiliated Hospital of China Medical University, Shenyang 110032, China;
4. Medical Solutions Business Division, Neusoft Group Co. Ltd., Shenyang 110003, China
不同的患者通常存在异质性, 异质性解释了不同的潜在疾病机制. 为解决患者群异质性, 有针对地实现更好预后的首选策略就是对于临床特征更为相似的同质患者进行分离. 通过寻找疾病相似的发展途径, 可针对不同群体采用不同类型的治疗干预, 为不同类患者提供针对其健康状况的精准医疗, 患者分型有助于研究特定类型的复杂疾病[1]. 电子病历可以为了解人口健康管理提供重要的资源, 并帮助制定更好的医疗保健运营政策决策[2], 在电子病历中隐藏着被现有的临床描述所限制的多种子类型, 通过学习患者深层表示, 对其进行聚类来发掘被隐藏模式的巨大潜力, 对病情和治疗产生更深入了解. 从复杂的患者数据中分析异质患者获取其子类型极具挑战, 深度学习已被应用于获得更稳健的患者表征, 以挖掘疾病的亚型分类, Lopez-Martinez-Carrasco等人提出一种基于跟踪的聚类, 通过以前分区的重叠聚类评估来寻找患者表型[3]; Landi等人提出一种基于词嵌入、卷积神经网络和自编码器(即ConvAE)的表示学习模型, 将患者轨迹转换为低维潜在向量, 通过聚类实现患者分层[4]; Chaudhary等人提出了基于自编码器的多组学数据特征提取, 进行肝细胞癌的生存亚群聚类[5].
因医疗数据多为时序数据, 利用机器学习等技术可以从数据本身获取其疾病描述和其他临床概念, 且用于此方面的数据具有纵向时序特点, 所以捕捉各个记录间的时序关系就尤为重要, Lu等人提出了基于 LSTM临床预测模型以预测临床事件, 采用果蝇优化算法解决临床事件间的时间间隔不同问题, 提高了网络的训练效率和预测精度[6]; 赵奎等人使用将诊疗日向量和相邻两个诊疗日之间的时间间隔进行拼接, 提高了诊疗项目预测精度[7]; Bai等人提出了一种新的可解释深度学习模型, 称为时间轴, 学习每种医疗代码的时间衰减因子通过分析时间轴的注意力权重和疾病进展函数, 预测未来就诊风险随时间的变化[8]; Zhang等人提出了一种基于注意力的时间感知疾病进展模型ATTAIN, 将注意力机制产生的权重与时间间隔通过衰减函数转换为衰减权重结合起来, 得到一个可解释的、临床合理的预测模型, 有助于了解感染性休克的进展行为[9].
循环神经网络(RNN)是时间上的展开, 常用于处理以时间序列数据作为输入的预测问题, 但其在梯度反向传播过程中存在梯度消失问题. 为解决RNN存在的问题, Hochreiter等人提出了长短期神经网络(long short term memory, LSTM)[10], 引入了“门”机制调节信息流, 学习序列中信息的记忆遗忘与否, 能够更好地捕捉较长距离的依赖关系, 然而标准的LSTM网络仅能用于处理规则的时间间隔, 而我们现实世界的序列数据, 尤其是医疗领域中患者就诊的时序数据通常为不规律时间间隔数据, 记录之间的时间差异从一天到几年不等, 这些时间间隔可能预示着某些疾病的发生, 若一位患者在短期内频繁入院, 其自身可能存在严重的健康问题, 而若两次就诊间间隔较长时间, 则前一次的就诊记录往往不能对当次诊断提供有用信息, 此时使用LSTM处理数据则不能抓住不规律间隔元素之间的依赖性.
Baytas等人提出的时间感知长短期神经网络(time-aware long short term memory, T-LSTM)[11]是LSTM的一种变体, 在LSTM记忆单元中加入时间不规则性以提高LSTM性能, 通过使用衰减函数将时间间隔转换为权重, 将单元记忆分解为短期记忆和长期记忆, 使用由衰减函数转换的时间间隔权重折算, 改变上一单元记忆对于当前输出的影响. 而T-LSTM同LSTM一样, 仅能编码从前到后的信息, 这总使得后序列信息的重要程度大于前序列, 而在处理患者入院就诊的时序数据时, 过去和未来的观测值可能包含了不同的信息, 患者诊断结果则是由其上下文记录共同决定, 对于理解疾病的发展轨迹和特征的演变, 捕捉病情变化、病程发展等动态信息至关重要, 能够更准确地预测患者的疾病状态或诊断结果. 医疗数据包含了患者的长期观测记录, 而这些观测数据之间存在着复杂的时间关系和演变模式, 通过考虑时间的因素, 模型能够更好地捕捉到医疗数据的动态变化和趋势, 提高模型对医疗数据的建模能力, 自动编码器提供了一种从原始数据直接学习映射的无监督方式[12], 原始序列由编码器重建, 其所得到的重建表示为输入序列的摘要[13] , 在患者疾病分型研究中, 自动编码器可以通过学习患者就诊数据的高级特征表示, 发现其中潜在的亚型或模式, 从而实现患者疾病的分型. 因此本文提出一种患者疾病分型模型, BT-DST模型, 本模型使用双向时间感知长短期神经网络(bi-directional time-aware long short term memory, Bi-T-LSTM)构造的双向自动编码器(Bi-T-LSTM autoencoder, Bi-T-LSTM AE), 将前向T-LSTM和后向T-LSTM组合, 更好捕捉双向的记录信息, 从两个方向学习患者时序记录的单一表示, 然后使用K-means算法进行聚类, 输出针对当前疾病的患者亚型分类. 患者疾病分型研究涉及的时间序列数据, 包含患者的病历记录、治疗过程、病情变化等信息, 而BT-DST模型能够有效地捕捉时间间隔不规律的时序特征, 在学习表示时考虑时间因素, 在处理时序数据上具有较好的泛化性能. 基于BT-DST模型在处理时序数据上的优势和适应性将其用于患者疾病分型研究, 该模型结合了时间感知性和双向性, 能够有效地学习时间序列数据的复杂结构和时序依赖关系, 这种高性能的表示学习能力使得在患者疾病分型研究中能够更好地发现数据中的隐藏模式和关联信息, 提高对患者疾病亚型分类的准确性.
1 方法 1.1 Bi-T-LSTM 1.1.1 循环神经网络RNN是一种对序列数据进行建模的神经网络, 其结构包含输入层、隐含层和输出层. 以输入层的输入和上一时刻隐含层的输出作为当前隐含层的输入, 即RNN会将之前输出的信息进行记忆并应用于当前输出中. RNN能处理任意长度的序列数据, 但是若对于长时间序列数据的输入, 后一时间节点对之前时间节点的感知能力将逐渐下降, 若当前输出与非常久远的时间序列有关, 则RNN难以学习, 造成梯度消失或梯度爆炸问题.
1.1.2 长短期神经网络LSTM将RNN隐藏层的简单节点改进成记忆单元, 利用记忆单元记住长期的历史信息, 利用门机制进行管理. LSTM单元由遗忘门、输入门和输出门控制单元状态, 门结构不提供信息, 仅用于限制信息. 输入门控制信息输入, 遗忘门控制单元历史状态信息保留, 输出门控制信息输出. 但是LSTM结构隐含序列元素之间时间均匀分布的假设, 对于数据中可能出现的不规律时间间隔却并未考虑.
1.1.3 时间感知长短期神经网络序列数据中时间间隔并不总是均匀分布, 不同的时间间隔可被视为是序列数据中所包含信息的一部分, 因此应在处理数据时加入此部分信息, 挖掘序列中连续元素之间不规律时间间隔的隐含信息. 两连续记录间的时间间隔可能是几天, 也可能是几年, 若时间间隔相隔过大, 则当前输出对于前一记录的依赖程度极低, 则前一单元的记忆对于当前单元的贡献应该被忽略. T-LSTM不将长期记忆完全丢弃, 而是对于前一时间步的记忆进行子空间分解, 将短期记忆按照时间步
首先, 式(1)通过网络获取前一单元的长期记忆
| CSt−1=tanh(WdCt−1+bd) | (1) |
式(2)使用非递增函数
| ˆCSt−1=CSt−1×g(Δt) | (2) |
式(3)为长期记忆
| CTt−1=Ct−1−CSt−1 | (3) |
| C∗t−1=CTt−1+ˆCSt−1 | (4) |
后接LSTM标准门控体系结构, 具体公式如下.
| ft=σ(Wfxt+Ufht−1+bf) | (5) |
| it=σ(Wixt+Uiht−1+bi) | (6) |
| ˜C=tanh(Wcxt+Ucht−1+bc) | (7) |
| Ct=ft×C∗t−1+itטC | (8) |
| ot=σ(Woxt+Uoht−1+bo) | (9) |
| ht=ot×tanh(Ct) | (10) |
其中,
| g(Δt)=1Δt | (11) |
| g(Δt)=1log(e+Δt) | (12) |

|
图 1 T-LSTM单元内部结构 |
1.1.4 双向时间感知长短期神经网络
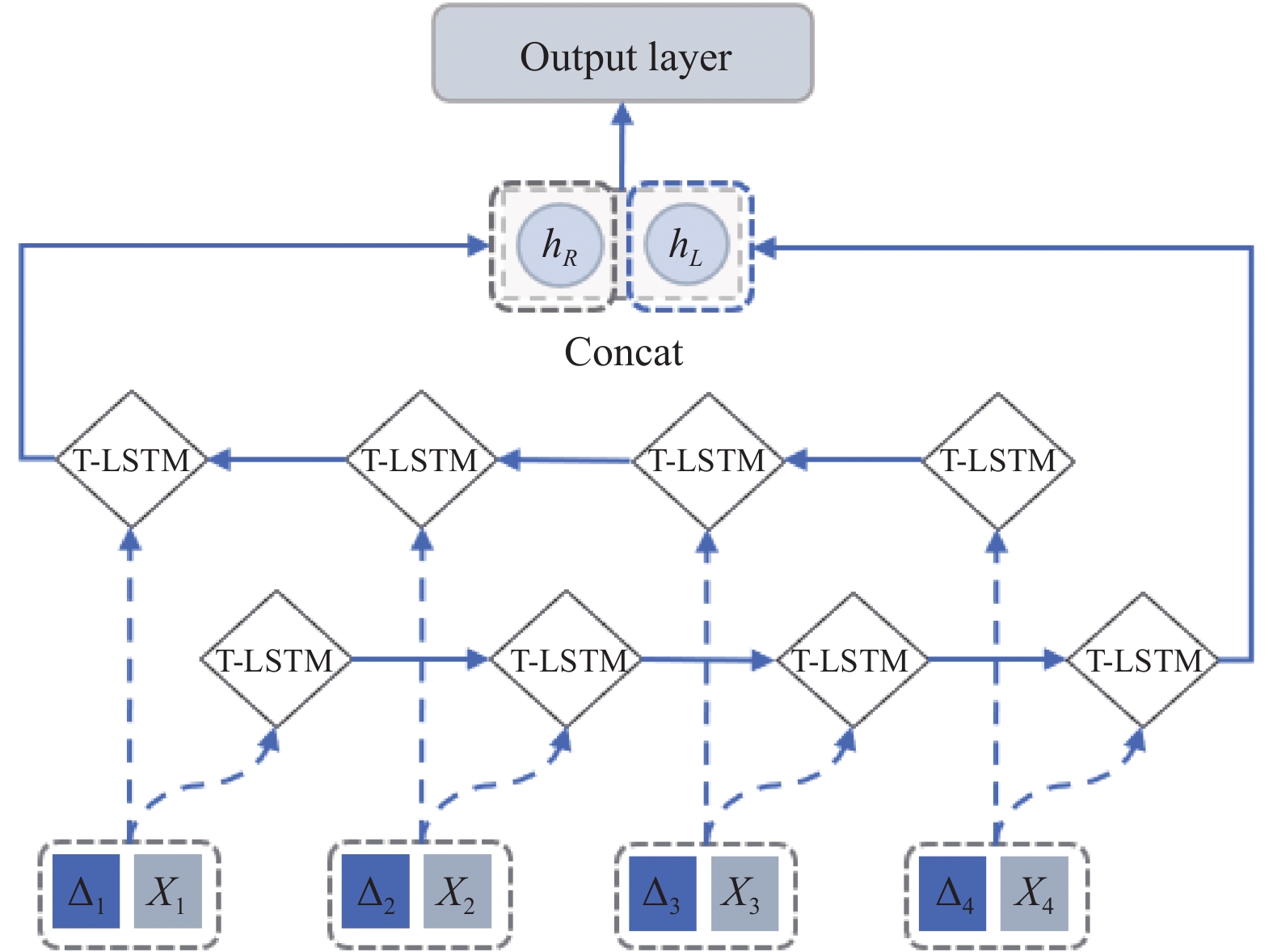
传统的LSTM仅能从前向后的传递消息, 对于当前记录之后的记录信息则无法利用, 通常对于一组时间序列数据进行处理时, 因其不依赖因果关系, 仅利用当前记录前向信息无法做出完善详尽的决策, 这在很多任务中都有局限性, 充分考虑时序数据的正反向信息规律, 进一步挖掘当前记录同过去及未来时刻记录的内在联系, 加深对于原序列特征提取的层次可以提高模型精度. 如图2所示, Bi-T-LSTM能够将前向和后向T-LSTM的输出拼接, 相对于单向T-LSTM来说, 能够同时获取从前向后和从后向前两个方向的信息.
| H=[hL⊕hR] | (13) |
|
图 2 Bi-T-LSTM模型结构 |
其中,
在医学上存在共同医学特征的集合称为一个亚型, 本研究通过处理患者电子病历数据, 在某种疾病范畴内, 根据其特征、病理机制、临床表现等方面的差异, 将患者进行聚类, 区分不同的亚型. 对于真实世界的疾病数据集没有关于患者队列内的任何先验信息, 疾病本身的复杂性和异质性使得某些疾病可能存在多个隐含的亚型, 而且这些亚型在现有的临床分类系统中尚未被充分认识或明确定义, 因此将患者亚型划分为无监督聚类问题. 从大规模患者就诊记录的电子病历中提取疾病的亚型可以指导下一代个性化医疗. 然而患者电子病历数据集的一个重要性质就是其序列中连续记录的时间间隔是不规律的, 其中患者记录的频率和数量是非结构化的, 其纵向数据缺少信息, 无法得知患者未就诊的时间间隔其疾病的发展情况, 且仅利用当前就诊记录前向信息无法做出完善详尽的决策, 需同时考虑双向记录获取过去及未来对于疾病当前发展状况的内在联系, 使用现有的临床数据进行聚类分析可能无法完全捕捉到其存在的亚型, 因此, 使用一种能够克服这种时间间隔不规律, 并充分考虑就诊记录时序信息的模型结构学习患者疾病特征, 将由此学习到的患者表示用于聚类能够更为精确地得出疾病的亚型分类.
本文提出了一种基于Bi-T-LSTM构造的双向自动编码器(Bi-T-LSTM autoencoder, Bi-T-LSTM AE)和K-means算法组成的疾病分型模型, 简称BT-DST. 使用此无监督框架处理时序电子病历数据, 通过自动编码器模型重构患者数据, 在就诊记录存在时间不规则的情况下, 双向考虑就诊时序, 训练自动编码器重构数据, 直至重构误差最小. 然后将患者序列重新输入至已保存训练权重的编码器部分, 由编码器学习到的输出即为患者记录的有效表示, 此表示将患者信息转换为低维潜在向量, 保持了原始序列随时间变化的时间动态, 能够从双向抓住患者因就诊间隔不同而暗含的疾病状况, 然后使用K-means算法对于上述编码器部分的输出所得患者数据的判别表示进行聚类, 将拥有相似特征的患者分为一组, 被聚类为一组的患者为一个亚型, 这样的特征提取能力可以帮助模型发现患者疾病数据中的关键模式和特征, 从而提高分型任务的性能. 当为一位新患者判断亚型分类时, 将新患者列数据输入至已保存训练权重的编码器结构中, 输出患者的单一表示, 将新加入患者与各个聚类子类型的质心距离进行比较, 将其加入距离最近的质心所在组即为该患者的亚型分类.
图3为BT-DST模型整体框架, 其由Bi-T-LSTM AE部分以及聚类部分构成, Bi-T-LSTM AE使用两个T-LSTM编码器和两个T-LSTM解码器组成, 其中编码器分别为前向编码器和后向编码器, 前向编码器和后向编码器均由多个T-LSTM单元组成, T-LSTM单元的个数由当前输入的单个患者的序列记录数决定, 解码器与编码器结构相同, 通过联合学习使双向重构误差最小化, 以T-LSTM单元构建自动编码器能够将不规律的时间间隔信息加入数据依赖学习中, 捕获长期和短期记忆间的关系, 更好的学习到输入序列的单一表现形式, 用此单一表现形式进行无监督聚类得到更准确的分类效果.

|
图 3 BT-DST模型架构 |
前向T-LSTM以
| Er=∑Li=1‖Xi−ˆXi‖22+∑1j=L‖Xj−ˆXj‖22 | (14) |
其中,
图3中将n位患者的单一表示
本实验由于无法得知真实世界的疾病数据集聚类情况的基本事实, 因此分别对已知数据分布及分类情况的人工生成的电子病历数据集和真实世界的电子病历数据集进行实验. 首先在人工生成数据集使用所提出Bi-T-LSTM模型在有监督情况下进行数据分类, 检验其对于挖掘序列电子病历数据蕴含信息的能力. 在无监督情况下, 使用BT-DST对人工生成的患者数据进行学习获得的单一表示, 检验其是否能更精准的进行聚类得到不同的亚型分型. 其次在真实世界的数据集SEER上进行无监督聚类, 验证本文提出的患者疾病分型模型是否能够对于胃癌患者的亚型分类产生积极影响. 本实验中所有权重的学习均以数据驱动方式同时进行, 将患者时序数据按照相同序列长度分批, 使用固定迭代次数, 以小批量Adam optimizer优化器进行优化.
2.1 评价指标针对有监督分类实验采用3种在医疗领域研究中被广泛使用的分类性能评估指标对于不同模型的性能进行评估, 分别为准确率(Accuracy)、F1-score以及AUC (area under curve), 而对于无监督聚类实验则使用聚类算法的常用评价指标Rand指数(Rand index, RI). 令 TP 表示预测为正类的正样本; TN 为预测为负类的负样本; FP 为预测为正类的负样本; FN 为预测为负类的正样本.
各个评估指标的定义以及计算公式如下.
Accuracy (准确率)是分类任务中最常用的指标, 它表示的是分类正确的预测数在总预测数中占据的比例, 其计算公式为:
| Accuracy=TP+TNTP+TN+FP+FN | (15) |
然而当数据不平衡时, Accuracy评估方法的缺陷尤为显著. 因此, 我们需要引入Precision (精确率), Recall (召回率)和F1-score评估指标, Precision是所有被预测为正的样本中实际为正的样本的比例, 计算公式如下:
| Precision=TPTP+FP | (16) |
Recall是实际为正的样本中被预测为正样本的比例, 计算公式如下:
| Recall=TPTP+FN | (17) |
而F1-score则是Precision和Recall二者的调和平均, 其计算公式为:
| F1-score=2×Precision×RecallPrecision+Recall | (18) |
受试者工作特征曲线(receiver operating characteristic, ROC)是通过不断移动分类器的“截断点”来生成曲线上一系列关键点的. 在此引入两个公式TPR (真阳性率)和FPR (假阳性率).
| TPR=TP(TP+FN) | (19) |
| FPR=FP(FP+TN) | (20) |
ROC 曲线是以假阳率为横轴, 以真阳率为纵轴的二维平面曲线, 其能在数据集出现类不平衡现象时不受干扰, ROC曲线越接近左上角, 该分类器性能越好. 由于ROC很难反映出模型之间的差异, 因此使用ROC曲线下面积进行对比, 即AUC.
Rand指数用于衡量聚类结果与数据的外部标准类之间的一致程度, 评估聚类方法所学习表示的辨别能力.
| RI=TP+TNTP+FP+FN+TN | (21) |
RI 取值范围为[0, 1], 值越大意味着聚类结果与真实情况越吻合, 划分的一致程度越高, 当Rand指数值取1时, 划分完全一致.
2.2 人工生成数据集 2.2.1 数据处理此人工生成的电子病历数据集中包含10万多名患者病历数据, 其中包括患者入院时间, 实验室测量结果, 用药日志, 病情诊断等多种特征, 虽然其数据为人工生成, 但参考其生成方法[14]可知, 此数据集中数据分布具有与真实世界电子病历数据相似的特征, 故可用其进行研究.
有监督分类实验选取其中与糖尿病相关的患者767例, 将数据中缺失值大于90%的特征列删除, 剩余存在缺失值的特征列, 连续变量使用均值填充, 分类变量使用“Not recorded”填充进行独热编码后得到数据维度为529. 将数据集按照7:3的比例划分为训练集和验证集, 其中训练集536例, 验证集231例.
无监督聚类实验中使用具有与患者电子病历数据相同结构的4种不同均值和相同协方差的正态分布聚类数据进行实验, 患者序列数据被随机丢弃, 以形成存在不规律时间间隔的数据分布, 其分批序列长度为4, 6, 18, 22, 30, 输入数据维度为5.
2.2.2 有监督实验结果实验比较Bi-T-LSTM与仅考虑正向序列信息的单层T-LSTM模型, 常用于处理电子病历等时序数据的未加入不规律时间间隔信息的传统单层LSTM模型, 以及不考虑时序信息的传统logistic回归分类器的性能差别, 验证Bi-T-LSTM在同时考虑当前时间步之前和之后的上下文信息以及序列记录随时间变化的时间动态后, 对于有监督分类实验性能是否得到明显提升, 能否捕捉更丰富上下文信息, 从而更全面地理解序列数据. 本实验分别使用Bi-T-LSTM、T-LSTM、LSTM、logistic回归模型对于数据进行分类, 其中神经网络模型的参数设置如表1. 由于逻辑回归模型为线性分类模型, 不考虑时间信息, 因此将每个患者的就诊记录进行汇总, 作为逻辑回归模型的输入, 其学习率为1E–3, 迭代次数为100. 实验结果如表2所示.
| 表 1 神经网络模型参数设置 |
表2展示了4种不同模型对于此数据集进行分类的准确率和AUC值. 从表2结果, 逻辑回归分类器的Accuracy, AUC, F1-score均为最低可知, 另外3种神经网络模型能够有效地建模序列信息, 捕捉到序列数据中的时间依赖关系, 且电子病历数据中的每个观测结果都可能与前面的观测结果相关联, 具有上下文信息, 传统的逻辑回归分类器只能考虑当前观测结果的特征, 无法有效地利用上下文信息, 因此在对数据进行分类时, 神经网络模型相较于不考虑时间信息的传统逻辑回归分类器的分类效果有明显优越性; T-LSTM比之LSTM在准确率上提升了7.25%, AUC提升了6.6%, F1-score提升了8.93%, T-LSTM通过引入时间注意力机制, 在输入中加入了不规律时间间隔成分后能够比LSTM获取更多与时间间隔相关信息, 使得模型能够自适应地学习不同时间间隔的重要性权重, 从而更好地学习患者病历数据中的动态变化, 提高模型精度; 由Bi-T-LSTM比T-LSTM的准确率提升了0.95%, AUC提升了1.15%, F1-score提升了0.84%, 可知Bi-T-LSTM模型从双向分析数据能够更好地抓住记录的双向信息, 捕捉时序数据中的上下文和依赖性, 该模型在进行双向特征拼接时充分利用了电子病历数据的前向和后向信息, 从而更全面地学习患者病情的变化, 这种双向建模的能力使得模型更为适应序列数据的建模任务, 更好地理解电子病历数据中的复杂关系, 使之具有更优秀的分类效果.
| 表 2 人工数据集下不同模型分类结果 |
2.2.3 无监督实验结果
为验证BT-DST在患者疾病分型任务中学习表示的表达能力. 为此, 使用一个已知基本事实的合成数据集, 对其进行亚型聚类, 并对聚类结果进行评估. 与有监督实验相同, 同样分别使用单层单向T-LSTM、LSTM构造模型中自动编码器部分, 后加K-means算法, 分别组成T-LSTM AE-KM模型和 LSTM AE-KM模型, 以及使用在医疗领域常用于表示学习的Deep Patient神经网络模型[15]进行数据表征学习能力的对比.
本实验中T-LSTM AE-KM模型和 LSTM AE-KM模型输入均为正向数据序列及时间间隔, 编码器末端单元输出的隐藏状态和单元记忆分别作为解码器首个单元的初始隐藏状态和上一单元记忆. 解码器的首个单元数据输入和间隔时间输入均被设置为零. 各模型参数设置如表3所示, 训练自动编码器使得重构误差最小化, 然后仅保留编码器部分的输出用于输入至K-means算法中进行亚型聚类. BT-DST模型中Bi-T-LSTM AE隐藏层维度均设为2, 双向向量拼接得到4维表示向量, T-LSTM AE和LSTM AE隐藏层维度均设置为4, 同样得到4维表示向量用于聚类. Deep Patient采用3层自动编码器, 每层神经元数均为50, 将每个患者的就诊记录进行汇总作为输入, 输出层维度为4. 各模型训练迭代次数均为2 000次.
由于我们知道合成数据的基本事实, 因此计算聚类的Rand指数(RI), 以观察所学习到表征的判别能力. Rand指数的较大值表明学习到的表征聚类接近基本事实. 启发式衰减函数选用式(12), K-means聚类的K值设为4, 每组均进行10次重复实验, 使用平均Rand指数评估各个模型对于数据的聚类效果. 各模型对比RI值如表4所示, 将4种模型所得二维表示进行聚类可视化如图4所示.
| 表 3 模型参数设置 |
| 表 4 人工数据集下不同模型聚类结果 |
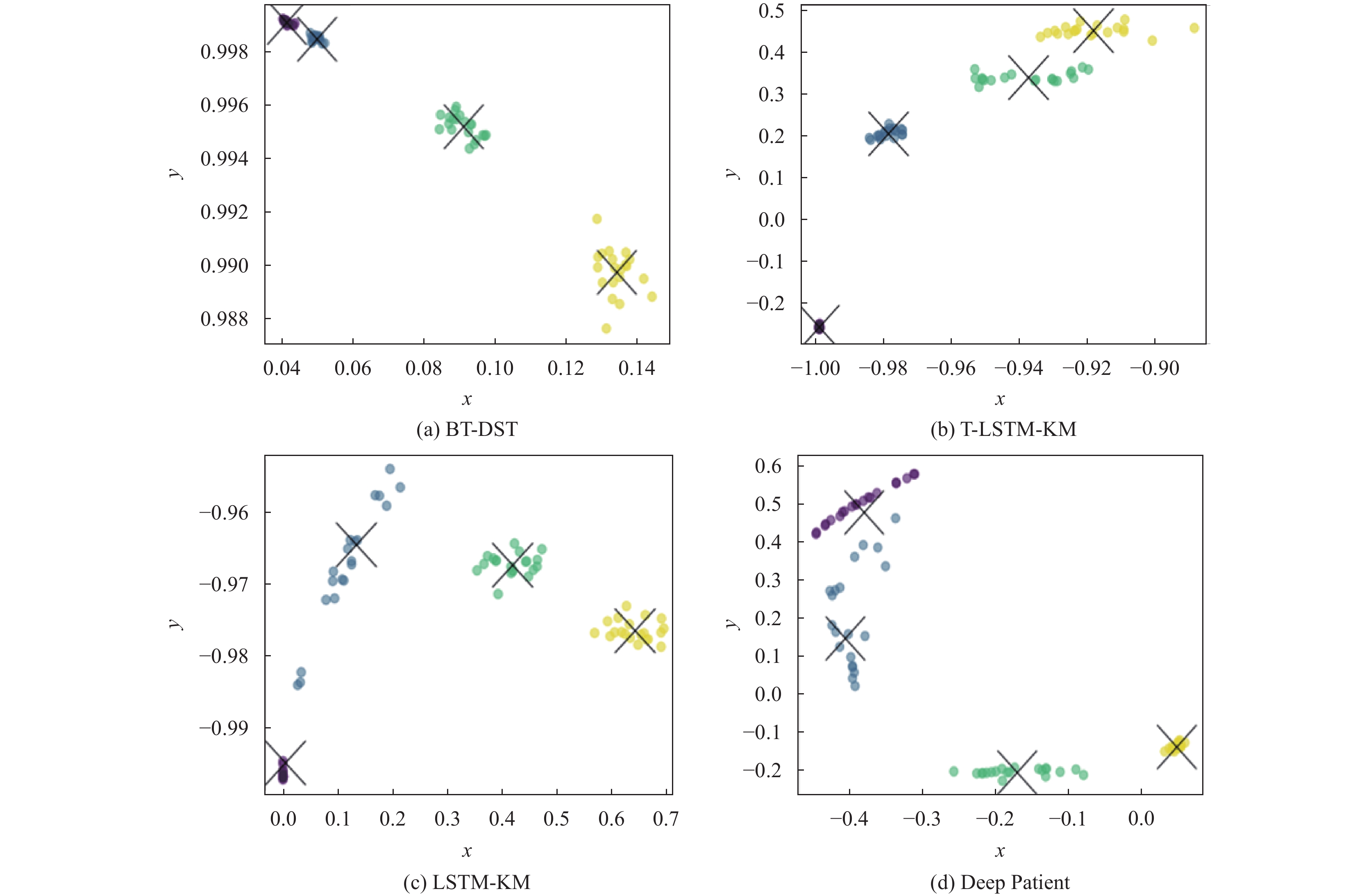
|
图 4 不同模型聚类可视化对比 |
由表4结果可知, 不考虑序列时间特征的Deep Patient的平均Rand指数相较于其他3种用于处理时间序列数据的模型表现最差, 由于电子病历数据中的时序特征反映了患者在不同时间点的医疗情况和变化趋势, 因此通过建模这种时间依赖性, 可以更好地捕捉到疾病的演变过程、治疗效果以及患者的健康状态变化等. 表4中BT-DST的平均Rand指数比标准LSTM AE-KM高0.0783, 比T-LSTM AE-KM高0.0081, 可知BT-DST聚类性能优于其他两种表示学习方法, 其聚类结果与真实标签的一致性最高, 能够有效地将相似的样本归为一类, 此类拥有不规律时间间隔的序列数据对于此不规律性较为敏感, BT-DST具有良好的时间建模能力, 在学习数据的同时, 捕捉了数据内在的结构和模式, 有助于更好地理解时间序列数据的动态演变过程, 从而提高了患者疾病分型的准确性, 不但能够更为全面的学习到在数据中存在非结构化时间间隔时数据的隐含信息, 同时能够捕捉输入序列所携带的前后信息, 能够利用时间上的依赖关系, 提取时间上的特征, 更全面地理解患者疾病数据. 观察图4可知, BT-DST对于输入序列的学习表示能够在二维空间中产生更为紧凑的组, 更能够捕捉到数据中的内在结构和类别之间的差异, 反映了数据的更好可分性, 能够很好地捕捉到数据的内在异质性, 并将其反映在聚类结果中, 使其更为准确, 这对于理解数据的结构和属性分布、揭示数据之间的关联关系具有重要意义.
2.3 真实数据集 2.3.1 数据处理本研究将利用美国国家癌症研究所(National Cancer Institute, NCI)建立的权威监测、流行病学及肿瘤患者预后随访数据库(surveillance, epidemiology and end results, SEER), SEER公开数据集的数据存储格式与上述人工生成电子病历相同, 均为非结构化纵向时序数据. 选取序列长度不小于3的胃癌患者记录为研究队列, 统计特征缺失值百分比, 删除缺失率大于90%的特征列, 共保留226维特征. 对于数值型特征使用平均值进行插值, 对于分类特征使用“Not recorded”填充, 然后进行独热编码, 最终得到809例患者时序数据, 共12833条数据, 数据维度为953, 其中655例胃癌临床N1期患者和154例胃癌临床N0期患者对照[16].
2.3.2 实验结果在本实验中, 由于学习到的表示与原始输入相比维数较低, 由高维学习到低维空间的映射需要更多的复杂性, 以便捕获高维输入序列的更多细节, 单层自编码器需要更多的迭代次数来最小化重构误差. 在本实验中, 由于上述原因, 均使用两层T-LSTM和两层LSTM构造模型中自动编码器部分, 其中第1层的输出是第2层的输入, 后加K-means算法, 分别组成T-LSTM AE-KM模型和 LSTM AE-KM模型与BT-DST进行对比实验. 本实验中各模型参数设置如表5所示, BT-DST的隐藏层维度设为2, 经双向训练后拼接的表示向量维度为4, T-LSTM AE-KM和 LSTM AE-KM的两层隐藏层维度分别为32和4, 各模型训练迭代次数均为3 000次. 然后将编码器部分学习到的单一表示用于K-means聚类.
| 表 5 模型参数设置 |
分别对数据分析患者聚类的子类型, 由于无法得知聚类的基本事实, 因此通过统计分析了解聚类结果间接知悉聚类性能.
尝试使用多个K值进行K-means聚类, 观察结果发现常见两种主要聚类, 因此设置K-means聚类参数为K=2. 为研究聚类产生的子类型分类性能, 对于聚类结果进行解释, 将分类特征使用卡方检验, 连续特征使用f检验进行聚类比较, 评估特征与聚类结果之间的相关性, 当聚类结果的特征具有小于0.05的显著性水平(即p-value小于0.05)时, 意味着特征与聚类结果之间的相关性是显著的, 这表明特征在区分不同的聚类簇时起到了重要的作用, 将对应的p-value小于0.05的特征认定为存在显著群体效应的相关特征, 表6列出了使用3种模型分别进行聚类所得结果, 表中仅列出p-value小于0.05的显著特征及其分别在各自类群中的均值. 由表6可知, LSTM AE-KM聚类结果中无显著相关性特征, 大多数患者被归为一个类群(null表示空值); BT-DST较之T-LSTM AE-KM多识别出positive lymph node以及lymph nodes removed两种特征. p-value小于0.05的特征越多, 表示这些特征在不同聚类簇之间具有较大的差别, 能够更好地区分不同簇之间的差异和相似性, 这些特征在聚类分析中对区分不同聚类簇的能力更强, 聚类结果更可靠. 比较BT-DST所得两聚类均值, 子类型1的positive lymph node 值显著高于子类型2的患者, N0期和N1期的胃癌患者在淋巴结转移的程度上有明显区别, N0期患者没有淋巴结转移, 癌细胞局限在原发肿瘤部位, 而N1期患者存在淋巴结转移, 癌细胞已经扩散到胃周围的淋巴结, 胃癌转移至淋巴结可能会影响癌症的治疗策略和预后, 这种区别对于指导治疗方案的选择和预后评估非常重要. 因此可知处于N1期的患者主要集中于子类型1, 处于N0期的患者主要集中于子类型2中. BT-DST产生了更多具有小p-value的显著特征, 意味着该方法在真实世界患者数据集上, 对于存在未知基本事实的数据进行患者亚型聚类分析, 能够通过加入非结构化时间间隔和通过双向获取电子病历信息提供更为精确的患分型结果.
| 表 6 真实数据集下不同模型聚类结果分析 |
3 总结
疾病的发展和演变通常受到时间的影响, 不同时间点上的观测值可能对患者疾病分型有不同的重要性. 本文使用一种能够将时间序列数据中存在的不规律时间间隔信息嵌入处理的T-LSTM单元构建Bi-T-LSTM模型, 使用Bi-T-LSTM结构分别在人工生成的数据以及真实世界的数据上分别进行有监督分类和无监督聚类实验, 捕捉时间上的依赖关系, 从而更好地表达患者疾病的动态变化. 实验结果表明使用Bi-T-LSTM结构构建的BT-DST模型通过学习患者电子病历数据的压缩表示来捕捉数据的重要特征进行聚类时, 不但能够通过加入到输入中的时间间隔信息来挖掘数据中因两连续记录间隔时间长短产生的影响, 同时利用过去和未来的观测值, 联合进行更为全面的数据学习, 增强对患者疾病发展的建模能力. BT-DST所输出的患者单一表示提取出对疾病分型具有较高区分能力的特征, 更深刻的抓住了患者信息, 为研究疾病子类型的聚类提供了极大的积极影响, 从而产生更为精确的分型结果. 本文所提出的BT-DST模型被设计用于处理患者电子病历数据, 模型所使用的T-LSTM单元决定其在处理具有不规律时间间隔特征的时序数据时具有突出表现, 擅长于捕捉时间依赖性和动态变化的建模任务. 因BT-DST模型具有的强大的特征提取能力, 将BT-DST模型应用于如病程预测, 病因分析等其他业务或领域时, 具有一定的适应性和泛化性, 需确保该领域数据与本论文所用数据具有相似时间特性, 领域数据的时间序列信息和连续记录之间的间隔对模型的性能产生重大影响, 针对模型进行特定的调整后, 在其应用场景中进行研究, 以确保其在不同领域的有效性.
| [1] |
Che ZP, Kale D, Li WZ, et al. Deep computational phenotyping. Proceedings of the 21th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. Sydney: ACM, 2015. 507–516.
|
| [2] |
Jensen PB, Jensen LJ, Brunak S. Mining electronic health records: Towards better research applications and clinical care. Nature Reviews Genetics, 2012, 13(6): 395-405. DOI:10.1038/nrg3208 |
| [3] |
Lopez-Martinez-Carrasco A, Juarez JM, Campos M, et al. A methodology based on trace-based clustering for patient phenotyping. Knowledge-based Systems, 2021, 232: 107469. DOI:10.1016/j.knosys.2021.107469 |
| [4] |
Landi I, Glicksberg BS, Lee HC, et al. Deep representation learning of electronic health records to unlock patient stratification at scale. npj Digital Medicine, 2020, 3: 96. DOI:10.1038/s41746-020-0301-z |
| [5] |
Chaudhary K, Poirion OB, Lu LQ, et al. Deep learning-based multi-omics integration robustly predicts survival in liver cancer. Clinical Cancer Research, 2018, 24(6): 1248-1259. DOI:10.1158/1078-0432.CCR-17-0853 |
| [6] |
Lu WJ, Ma L, Chen H, et al. A clinical prediction model in health time series data based on long short-term memory network optimized by fruit fly optimization algorithm. IEEE Access, 2020, 8: 136014–136023.
|
| [7] |
赵奎, 闫玉芳, 曹吉龙, 等. 融合规范化判断的双向循环神经网络诊疗预测模型. 小型微型计算机系统, 2022, 43(6): 1278-1284. |
| [8] |
Bai T, Zhang SS, Egleston BL, et al. Interpretable representation learning for healthcare via capturing disease progression through time. Proceedings of the 24th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining. London: ACM, 2018. 43–51.
|
| [9] |
Zhang Y, Yang X, Ivy J, et al. ATTAIN: Attention-based time-aware LSTM networks for disease progression modeling. Proceedings of the 28th International Joint Conference on Artificial Intelligence. Macao: AAAI Press, 2019. 4369–4375.
|
| [10] |
Hochreiter S, Schmidhuber J. Long short-term memory. Neural Computation, 1997, 9(8): 1735-1780. DOI:10.1162/neco.1997.9.8.1735 |
| [11] |
Baytas IM, Xiao C, Zhang X, et al. Patient subtyping via time-aware LSTM networks. Proceedings of the 23rd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. Halifax: ACM, 2017. 65–74.
|
| [12] |
Bengio Y, Courville A, Vincent P. Representation learning: A review and new perspectives. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2013, 35(8): 1798-1828. DOI:10.1109/TPAMI.2013.50 |
| [13] |
Cho K, van Merriënboer B, Gulcehre C, et al. Learning phrase representations using RNN encoder-decoder for statistical machine translation. Proceedings of the 2014 Conference on Empirical Methods in Natural Language Processing (EMNLP). Doha: Association for Computational Linguistics, 2014. 1724–1734.
|
| [14] |
Kartoun U. A methodology to generate virtual patient repositories. arXiv:1608.00570, 2016.
|
| [15] |
Miotto R, Li L, Kidd BA, et al. Deep patient: An unsupervised representation to predict the future of patients from the electronic health records. Scientific Reports, 2016, 6: 26094. DOI:10.1038/srep26094 |
| [16] |
国家卫生健康委员会. 胃癌诊疗规范(2018年版). 中华消化病与影像杂志(电子版), 2019, 9(3): 118-144. |
 2024, Vol. 33
2024, Vol. 33



