2. 中山火炬开发区人民医院 放射科, 中山 528400
2. Radiology Department, Zhongshan Torch Development Zone People’s Hospital, Zhongshan 528400, China
目前, 测量脂肪分数需要人工选择肝实质中的感兴趣区域(region of interest, ROI). 这一策略非常耗时, 因为需要多个ROI才能获得可重复性的结果. 人工全肝分割的诊断性能与不同的人工ROI采样方法结果显示出质子密度脂肪分数有很强的相关性[1,2]. 在可重复性条件下, 人工ROI和人工全肝分割获得了相似的观察者间一致性, 但是这两种方法都导致了影像组学测量的差异. 目前Martí-Aguado等人[3]提出的基于卷积神经网络(convolutional neural network, CNN)的全肝自动分割方案, 获得与手动分割相似的诊断准确性, 且自动分割具有稳定结果. 文献[3]前瞻性研究证实了基于深度学习的全肝脏分割算法, 在测量MRI质子密度脂肪分数时, 诊断准确性与手工采样ROI进行评估得到的结果相似. 深度学习方法与手工勾勒的ROI估计值相比, 变异性误差和不确定性程度更低, 性能更优越. 准确的MRI自动全肝分割方法可以被扩展并无缝整合到临床实践工作流程中, 能简化肝脏脂肪定量分析流程.
1.2 相关工作肝脏MRI影像的脂肪定量标准化过程中, 传统方法常需要对肝脏感兴趣区域进行手工采样, 通过对多个手工勾勒的感兴趣区域(通常3−4个)进行脂肪定量分析求取均值. 但手工采样感兴趣区域时需要对肝脏中血管部分的进行排除, 防止血管部分对结果计算产生影响, 这种策略耗时且因采样的区域不同而导致结果多变. 目前大部分的肝脏分割相关任务主要集中在肝脏及其病变分割上, 陆雪松等人[4]针对腹部 MR中呈现出浸润现象、与相邻器官灰度值相似、边界较为模糊的问题, 提出了一种基于深度卷积编解码的迭代网络结构. 它将前次分割输出的概率图引入到网络浅层, 与来自编码器的低水平特征图融合, 弥补下采样时丢失的细节信息, 最终得到93%的Dice系数. Amin等人[5]提出了一种由两个主要步骤组成的方法: 首先, 采用局部拉普拉斯滤波提高图像质量; 其次, 提出语义分割模型, 将特征提取到预训练好的VGG16模型中, 传递到U型网络中, 所提出的分割框架能精确地分割受感染的肝脏区域. 该方法在3D-IRCADB和LiTS17两个数据集上进行了性能测试, 达到98%的Dice系数. 对于肝脏中剔除血管的分割研究较少, Martí-Aguado等人[3]利用CNN进行全肝自动分割方案, 该方案验证了基于深度学习方法的全肝分割与手工勾勒的感兴趣区域在进行脂肪定量分析时, 获得与手动分割相似的诊断准确性, 且自动分割具有稳定结果. 王志明[6]构建了剔除血管的肝脏私有数据集, 并设计并实现一种基于U-Net+网络[7], 用于肝脏MR图像的分割, 该方法Dice系数还到93.8%.
上述分割任务通常采用与U-Net类似的编码器-解码器架构, 其中编码器学习图像的分层低维表示, 解码器将学习到的表示映射到体素进行分割. 基于 CNN的方法在编码器和解码器中分别使用卷积和反卷积, 由于局限的局部感受野而难以获得更为准确的分割结果. 随着Transformer结构[8]被用到计算机视觉领域, 这种本质上是全局操作的网络, 展现了具有竞争力的性能.
本文主要探索CNN及Transformer混合架构, 以结合卷积和自注意力各自的优点来实现更好的分割.
1.3 本文贡献本文对基于Transformer为主的UNETR++网络进行改进, 用于肝脏分割, 后续用来替换肝脏脂肪定量分析中手工选取ROI步骤. 创新点如下: (1) 本文模型是一种CNN及Transformer相混合的编码器、解码器架构, 在UNETR++基出上引入CNN结构分支用于捕获更多的局部特征, 用于提升模型的分割准确性. (2) 引入改进的注意力门控模块, 用于融合CNN结构及Transformer结构提取的特征, 使模型能隐式学习, 抑制输入特征中的无关区域的同时, 使分割能按注意力权重聚焦到目标区域.
2 方法CNN结构的成功依赖于其两个固有的归纳偏置, 即平移不变性和局部相关性, 而视觉Transformer结构通常缺少这种特性, 导致通常需要大量数据才能超越CNN的表现, CNN在小数据集上的表现通常比纯Transformer结构要好. 但CNN感受野有限导致很难捕获全局信息, 而Transformer可以捕获长距离依赖关系, 本文尝试将CNN和Transformer结合, 使得网络结构能够继承CNN和Transformer各自的优点, 并且最大程度保留全局和局部特征.
2.1 网络整体结构本文提出的改进后UNETR++[9]分割网络结构如图1所示, 模型采用分层设计, 是一种CNN-Transformer相混合的编码器、解码器架构. 编码器有两条分支, 一条基于Transformer结构, 另一条基于CNN结构. 编码器Transformer结构分支有4个阶段, 其第1阶段包括patch嵌入(patch embedding), 其将体积块输入划分为3D patches, 然后是新颖的高效配对注意力 (efficient paired-attention, EPA) 块. 在patch嵌入中, 将每个3D输入
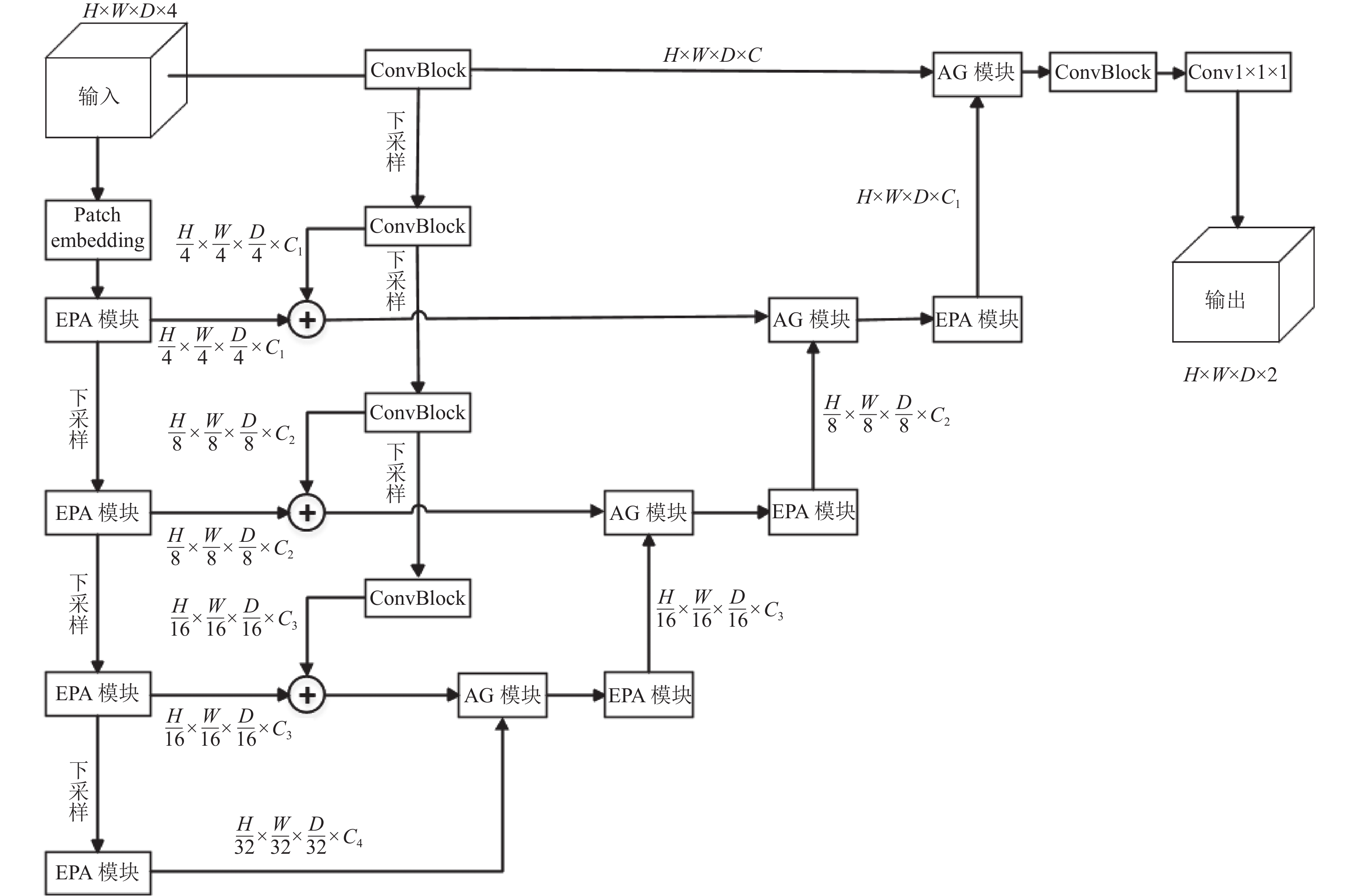
|
图 1 改进的UNETR++架构 |
与编码器类似, 解码器也包括4个阶段, 其中每个解码器阶段都包含注意力门控(attention gate, AG)模块[10], 在AG模块中集成了上采样操作, 通过使用反卷积将解码器部分的EPA块生成的特征图的分辨率提高两倍. 编码器层级通过跳跃连接与CNN分支结构生成的特征图进行相加操作, 得到融合后的特征. 再经过跳跃连接输入至同解码器层级的EPA模块捕获长距离依赖关系. 对输出的特征输入至AG模块, 通过注意力门控机制使图像分割时能按注意力权重聚焦到目标区域. 每两个解码器之间的通道数量减少两倍, 因此, 最后一个解码器的输出与卷积特征图融合以恢复空间信息并增强特征表示. 最后将生成的输出, 经3×3×3 和 1×1×1 卷积块以生成体素方面的最终分割掩码预测.
接下来对EPA模块和AG模块进行详细介绍.
2.2 EPA模块EPA模块是UNETR++基于最近的UNETR框架[11]进行的改进, 通过在两个分支中应用空间注意力和通道注意力, 有效地捕捉了丰富的相互依存的空间和通道特征. 如图2所示, 给出EPA模块的结构图, 每个EPA块包括两个注意模块, 通过使用共享keys-queries方案, 对空间和通道维度的信息进行编码来有效地学习空间通道特征表示. patch嵌入生成的特征图被直接送到EPA块中, 随后是3个编码器层. Q和K线性层的权重在两个注意力模块之间共享, 每个注意力模块使用不同的V层. 两个注意力模块计算如下:
| $ \hat {{X_{{s}}}} = {\textit{SA}}({Q_{{\mathrm{shared}}}}, {K_{{\mathrm{shared}}}}, {V_{{\mathrm{spatial}}}}) $ | (1) |
| $ \hat {{X_c}} = CA({Q_{{\mathrm{shared}}}}, {K_{{\mathrm{shared}}}}, {V_{{\mathrm{channel}}}}) $ | (2) |
其中,

|
图 2 EPA模块 |
空间注意力: 该部分通过将复杂度从 O(n2) 降低到 O(np)来有效地学习空间信息, 其中n是token的数量, p是投影向量的维度, p<<n. 给定HWD×C形状的归一化张量X, 使用3个线性层计算Qshared、Kshared和Vspatial投影, 得出Qshared=WQV, Kshared=WKV, Vspatial=WVX, 维度为HWD×C, 其中WQ、WK 和 WV 分别是Qshared、Kshared和Vspatial的投影权重. 然后, 执行3个步骤: 首先, 将Kshared和Vspatial层从HWD×C投影到形状为p×C的低维矩阵中. 其次, 通过将Qshared层乘以投影Kshared的转置来计算空间注意力图, 然后使用Softmax来计算每个特征与其余空间特征之间的相似度. 第三, 将这些相似性乘以投影的Vspatial层, 以生成HWD×C形状的最终空间注意力图. 空间注意力的定义如下:
| ${ {\hat {{X}}} _p} = {\textit{Softmax}} \left( {\frac{{{Q_{{\mathrm{shared}}}}K_{{\mathrm{shared}}}^{\rm T}}}{{\sqrt d }}} \right) \cdot {{\mathop V\limits^ \sim} _{{\mathrm{spatial}}}} $ | (3) |
通道注意力: 该部分通过在通道value层和通道注意力图之间的通道维度中应用点积运算来捕获特征通道之间的相互依赖关系. 使用与空间注意力模块相同的Qshared和Kshared, 计算通道的value层, 并使用线性层学习互补特征, 得到Vchannel=WVX, 维度为HWD×C, 其中WV是Vchannel的投影权重. 通道注意力定义如下:
| ${ \hat X }_p= {\textit{Softmax}} \left( {\frac{{{Q_{{\mathrm{shared}}}}K_{{\mathrm{shared}}}^{\rm T}}}{{\sqrt d }}} \right) \cdot {{\mathop V\limits^ \sim} _{{\mathrm{spatial}}}}$ | (4) |
其中, Vchannel、Qshared、Kshared分别表示通道value层、共享query和共享key, d是每个向量的大小. 最后, 执行融合, 并通过卷积块变换两个注意力模块的输出, 以获得丰富的特征表示. EPA模块的最终输出如下所示:
| $ \hat X = Con{v_1}(Con{v_3}(\hat {{X_s}} + \hat {{X_c}} )) $ | (5) |
Attention gate (AG)是一种注意力门控模块, 最早应用于医学图像分割任务之中, 该模块能够自动学习聚焦不同形状和大小的目标结构. 使用AG训练的模型能隐式学习, 抑制输入图像中的无关区域, 同时突出对特定任务有用的显著特征.
本文改进的AG模块结构如图3所示, 其中

|
图 3 改进的AG模块 |
2.4 损失函数
损失函数基于常用soft Dice损失[13]和交叉熵损失的总和, 以同时利用两个互补损失函数的优势. 其定义为:
| $ {{\mathcal{L}}}(Y, P) = 1 - \displaystyle\sum\limits_{i = 1}^I {\left( \begin{gathered} \frac{{2 \times \displaystyle\sum\nolimits_{v = 1}^V {{Y_{v, i}} \cdot {P_{v, i}}} }}{{\displaystyle\sum\nolimits_{v = 1}^V {Y_{v, i}^2 + \displaystyle\sum\nolimits_{v = 1}^V {P_{v, i}^2} } }} + \displaystyle\sum\limits_{v = 1}^V {{Y_{v, i}}\log {P_{v, i}}} \\ \end{gathered} \right)} $ | (6) |
其中,
本研究通过广东中山火炬开发区医院伦理委员会批准. 从病历中收集患者的临床、影像学和组织病理学资料, 共筛选出95例MRI数据进行研究. 每个患者采用 1.5T MR 扫描仪进行上腹部 MR 平扫, 使用 T1 VIBE Dixon 序列, 取仰卧位进行检查. 扫描参数: 回波时间(TE)=2.39 ms, 重复时间(TR)=7.03 ms, 反转角度为 10, 带宽=±490 kHz, 层厚 3.0 mm, 切片数: 72. 于检查前对受试者进行呼吸训练, 以确保受试者每次吸气末屏气幅度尽可能一致. 一次屏气扫描完成后即可同时获得4幅图像, 同相位, 反向位, 脂肪相及水相. 将数据随机分为训练集(70例)、验证集(15例)和测试集(10例).
标注的制定: 目前公开的相关腹部器官分割数据集中, 肝脏标签图像并未对其中的血管组织进行剔除, 本研究将构建分割数据集. 数据集由3名放射科医师共同制定, 其中由一名有10年腹部影像经验的放射科医师应用ITK-SNAP软件在T1横断面勾画肝脏区域并剔除其中较大的血管组织; 另一名放射科医师(在腹部影像方面有10年经验)检查和调整标签, 如果对某些区域的标注没有一致意见, 则由第3名医师(在肝脏成像方面有15年的经验)进行检查和调整, 并做出最终决定, 横断面切片上的肝脏分割(剔除血管)标签如图4所示.

|
图 4 肝脏分割(剔除血管)标签示例 |
3.2 实验配置
实验环境配置如表1所示. 训练过程中使用了Adam作为优化器, 初始学习率为0.001, 每50个epoch衰减到原始值的一半. 将总训练时间点设置为500个, 批次大小为4个, 并保留训练期间在验证集中表现最好的模型用于预测.
| 表 1 实验环境 |
3.3 评估指标
在医学图像分割的评估指标中, Dice相似性系数(Dice similarity coefficient, DSC)常用来评估分割性能, 其又称为重叠指数, 表示两个物体相交的面积点总面积的比值, DSC评价指标公式如下:
| $ {\textit{DSC}} = \frac{{2\left| {X \cap Y} \right|}}{{\left| X \right| + \left| Y \right|}} $ | (7) |
其中,
95%豪斯多夫距离(95% Hausdorff distance, HD95)表示预测分割区域边界与真实区域边界之间的距离, 其值越小代表预测边界分割误差越小. 定义如下:
| $ H{D_{95}}(X, Y) = \max \{ {d_{XY}}, {d_{YX}}\} $ | (8) |
其中,
本文将提出的算法与3D U-Net、3D V-Net[14]、UNETR、UNETR++模型进行了对比实验, 结果如表2所示.
| 表 2 模型比较 |
所有对比的实验方法均为3D分割网络. 在训练轮次达到 500 个 epoch 后, 3D U-Net、3D V-Net、UNETR、UNETR++的平均 DSC系数分别为93.52%、93.89%、94.25%、95.17%, HD95分别为6.34、6.01、5.47. 改进的模型DSC系数及HD95指标, 均比其他模型更优. 对AG模块的验证, 从表2中可得到, 加入AG模块后, 改进的模型DSC系数提高了0.14%, HD95减少0.17. 根据实验结果, 本文提出的方法与其他方法相比在全局信息的提取和局部细节保留上更具有优势, 在目前最先进的 UNETR ++的基础上其分割效果DSC提升了0.56%, HD95减小0.68, 证实了本文提出模型在肝脏分割任务上的有效性.
不同模型预测结果如图5所示, 相比本文改进模型, 3D U-Net、3D V-Net、UNETR及UNETR++等模型肝脏中血管部分有的未分割出, 且分割细节不精确, 本文模型能最大程度地排除掉血管组织.

|
图 5 不同模型的分割结果对比 |
4 结论与展望
在本文中, 我们基于UNETR++模型提出了一种改进的结构, 用于自动分割肝脏, 该模型的性能优于目前最先进的UNETR++网络, 证明了我们的方法在肝脏分割任务上的有效性. 模型不足之处主要集中在提高了分割精度的同时, 增加了模型的参数和计算量. 同时, 由于构建数据集时对肝脏中的血管组织的剔除标注较为困难, 实验构建出的数据集较小, 后续研究需要进一步扩充数据集大小. 肝脏的准确分割是后续定量分析的重要前提, 后续可以利用深度学习方法进行自动全肝区域提取来替换手工ROI选取肝脏区域, 极大地帮助简化肝脏脂肪定量分析操作流程. 在未来的研究中, 我们计划通过在更大的数据样本中使用本文的解决方案, 并将自动分割算法扩展到其他器官, 如胰腺等其他腹腔组织.
| [1] |
Procter AJ, Sun JY, Malcolm PN, et al. Measuring liver fat fraction with complex-based chemical shift MRI: The effect of simplified sampling protocols on accuracy. BMC Medical Imaging, 2019, 19(1): 14. DOI:10.1186/s12880-019-0311-y |
| [2] |
Song JJ, Yu XL, Song WL, et al. MRI-based radiomics models developed with features of the whole liver and right liver lobe: Assessment of hepatic inflammatory activity in chronic hepatic disease. Journal of Magnetic Resonance Imaging, 2020, 52(6): 1668-1678. DOI:10.1002/jmri.27197 |
| [3] |
Martí-Aguado D, Jiménez-Pastor A, Alberich-Bayarri Á, et al. Automated whole-liver MRI segmentation to assess steatosis and iron quantification in chronic liver disease. Radiology, 2022, 302(2): 345-354. DOI:10.1148/radiol.2021211027 |
| [4] |
陆雪松, 闫书豪. 基于迭代卷积神经网络的肝脏MRI图像分割. 中南民族大学学报(自然科学版), 2022, 41(3): 319-325. |
| [5] |
Amin J, Anjum MA, Sharif M, et al. Visual geometry group based on U-shaped model for liver/liver tumor segmentation. IEEE Latin America Transactions, 2023, 21(4): 557-564. DOI:10.1109/TLA.2023.10128927 |
| [6] |
王志明. 基于U-Net+的全身脂肪及肝脏MR图像分割算法研究[硕士学位论文]. 武汉: 华中师范大学, 2021.
|
| [7] |
Çiçek Ö, Abdulkadir A, Lienkamp SS, et al. 3D U-Net: Learning dense volumetric segmentation from sparse annotation. Proceedings of the 19th International Conference on Medical Image Computing and Computer-assisted Intervention. Athens: Springer, 2016. 424–432.
|
| [8] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 6000–6010.
|
| [9] |
Shaker A, Maaz M, Rasheed H, et al. UNETR++: Delving into efficient and accurate 3D medical image segmentation. arXiv:2212.04497, 2022.
|
| [10] |
Deng WJ, Shi Q, Li J. Attention-gate-based encoder–decoder network for automatical building extraction. IEEE Journal of Selected Topics in Applied Earth Observations and Remote Sensing, 2021, 14: 2611-2620. DOI:10.1109/JSTARS.2021.3058097 |
| [11] |
Hatamizadeh A, Tang YC, Nath V, et al. UNETR: Transformers for 3D medical image segmentation. Proceedings of the 2022 IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2022. 1748–1758.
|
| [12] |
Chen YP, Dai XY, Liu MC, et al. Dynamic ReLU. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 351–367.
|
| [13] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [14] |
Lei Y, Wang TH, Wang B, et al. Ultrasound prostate segmentation based on 3D V-Net with deep supervision. Proceedings of the 2019 Medical Imaging Conference on Ultrasonic Imaging and Tomography. San Diego: SPIE, 2019. 198–204.
|
 2024, Vol. 33
2024, Vol. 33


