胶质瘤是脑肿瘤中最常见类型, 按肿瘤恶性程度可划分为高级别胶质瘤(high-grade gliomas, HGG)和低级别胶质瘤(low-grade gliomas, LGG)[1]. 其中, 高级胶质瘤生长速度迅速、治愈率极低、危害性更大, 是一种恶性肿瘤. 核磁共振成像(magnetic resonance imaging, MRI)因其成像清晰且对人体无伤害的特点, 已被广泛应用于临床医学的诊断和治疗[2]. 由于脑肿瘤为渗透性肿瘤, 其外观高度异质、位置随机并且各子区域体素数量相差悬殊[3], 现有工作仍无法在临床MRI脑影像分割上取得满意效果[4]. 因此, 获得高精度的MRI脑肿瘤图像自动化分割仍具有很大挑战.
自2014年来, MRI脑肿瘤的分割工作随着卷积神经网络(convolutional neural network, CNN)的火热逐渐形成以深度学习为主流架构的研究趋势. 早期, Zikic等人[5]探索了将CNN应用于脑肿瘤分割, 其没有对输出图像进行后处理操作便取得了较好的分割效果. 考虑到CNN在医学图像分割上的局限性, 受全卷积网络(fully convolutional network, FCN)方法[6]的启发, Ronneberger等人[7]于2015年提出了U-Net模型, 很好地适应了医学图像分割任务, 也成为脑肿瘤分割的主流算法之一. 其中, Isensee等人[8]提出了一种用于脑肿瘤分割的改进的U-Net, 通过修改Dice损失函数和进行数据增强来防止过拟合以提高分割精度. 研究者也将Dense块[9], 空洞卷积[10]引入来进一步扩展U-Net并获得了更精确的脑肿瘤分割结果. 考虑到基于二维U-Net架构的分割方法在进行输入图像切片操作时不可避免地会丢失脑肿瘤的空间信息, 为了更好地从原始三维数据中提取丰富的特征信息, Milletari等人[11]提出了U-Net的变体三维版本, 并构建新的Dice coefficient损失函数来更好应对类别不平衡问题. 基于三维U-Net模型, Chen等人[12]在上采样部分通过聚合多尺度的预测结果来对网络添加约束, 以此增强模型性能; Liu等人[13]在网络中添加了一个额外的精细类预测, 以防止网络进行过度分割; Tang等人[14]将三维U-Net与变分自动编码器进行结合, 以此来提高网络的分割性能; Zhang等人[15]则通过使用多个编码器来降低提取特征的难度, 进一步提高模型性能; 在此基础上, Huang等人[16]增加了一个距离变换的解码器, 可使解码器生成分割轮廓更加准确的分割图; Zhang等人[17]通过引入不对称的期望最大化注意力来增加分割的准确程度. Guo等人[18]和Cheng等人[19]在此基础上把重心放在对损失函数的修改, Guo等人[18]通过加权处理来更好的约束网络, Cheng等人[19]则提出采用混合损失来监督网络. 此外, Li等人[20]将级联思想用到脑肿瘤分割中, 将前一阶段的粗分割结果作为下一阶段网络先验信息来获得更精细的分割; Cheng等人[21]在级联网络的基础上通过减少通道数并使用多尺度特征模块来增强特征的表现力使网络在使用更少计算资源的情况下还可以保持不错的精度. 近年, 随着Transformer在自然语言处理任务中的成功应用, Chen等人[22]提出的TransUNet将Transformer引入医学图像分割领域中, 来弥补CNN在显式建模长程依赖关系方面的局限性; 从三维角度出发, Wang等人[23]将Transformer引入到脑肿瘤分割任务中, 结合Transformer和三维U-Net网络来提升分割任务精度.
借鉴三维U-Net在脑肿瘤分割任务中的优异表现, 考虑到网络感受野被卷积核大小限制和易忽略全局特征问题, 着重于来提升网络的全局特征提取能力和表达能力. 首先, 从加强全局其他信息着手, 将非局部自注意力(non-local)模块[24]引入网络中; 此外, 为加强卷积的特征提取能力, 将下采样部分的普通卷积替换为金字塔卷积[25], 可在不增加参数的情况下进一步提升网络的分割性能. 综上, 本文构建了融合非局部自注意力和金字塔卷积的多尺度非局部自注意力脑肿瘤分割网络(PyCSAU-Net). 本文主要贡献如下: 1) 以经典脑肿瘤分割网络架构U-Net为基础, 从改善网络因卷积核大小受限导致的长距离建模能力不足问题角度着手, 提出了一种基于非局部自注意力机制的新脑肿瘤分割网络PyCSAU-Net. 2) PyCSAU-Net在网络第4层横向连接中引入扩展的三维非局部注意力模块以提高网络捕获长距离依赖特征的能力, 同时将网络中下采样阶段将普通卷积替换为具有多尺度特点的三维金字塔卷积来提取更具判别性的脑肿瘤深度特征, 实现在整体上提升网络的脑肿瘤分割性能. 3) 在公开的BraTS 2019和BraTS 2020数据集上对所提出方法进行了充分验证, 消融实验和对比实验结果证明了所提出方法的有效性和竞争力.
1 网络架构及算法原理 1.1 网络架构在U-Net网络的下采样模块中, 由于输入图像的尺寸不断减小, 不可避免地会丢失一些空间信息和细节信息. 鉴于脑肿瘤本身存在形状复杂、大小不一和边界模糊等问题, 导致U-Net在小尺寸肿瘤的分割任务中分割效果上还有所不足. 对于所构建的多尺度非局部自注意力脑肿瘤分割网络PyCSAU-Net, 是以三维U-Net网络作为主体框架, 在网络第4层横向连接中引入扩展的三维非局部注意力模块, 同时在下采样阶段采用具有多尺度特点的三维金字塔卷积操作, 来形成可有效获取全局的特征信息和多尺度局部精细特征信息的脑肿瘤分割网络, 网络整体架构如图1所示.
具体来说, PyCSAU-Net沿用传统U-Net网络架构, 由编码器部分和解码器部分组成, 两者中间通过横向连接操作进行连接. 网络输入图像大小为4×128×128×128像素, 其中4为通道数, 128×128×128为输入脑肿瘤图像的大小. 在编码部分, 使用金字塔卷积模块替换U-Net中的标准卷积模块. 此外, 将步长为2的三维最大池化操作单元置于下采样层, 经过一次下采样操作后, 脑肿瘤图像大小减半, 通道数增加一倍.

|
图 1 PyCSAU-Net的网络框架图 |
网络解码部分由4个上采样模块组成, 使用4个标准卷积模块进行特征图的恢复. 在上采样的过程中, 通过反卷积使特征图大小增加一倍, 通道数量减半, 以恢复特征图的空间信息和输入的原始大小. 将非局部自注意力机制模块嵌入到第4层横向连接, 计算单个样本元素之间的相互作用, 可以增强细小特征提取和表达能力, 促进模型更加关注小尺寸脑肿瘤分割. 在解码器的最后部分, 通过使用1×1×1卷积和上采样操作将3个解码器块的输出进行融合, 作为最终的脑肿瘤分割结果.
1.1.1 非局部自注意力模块自注意力机制是一种具有较强全局关注能力的方法, 相较于传统的卷积操作, 其通过简单查询与赋值可获取特征图全局空间信息. 其中, 非局部自注意力机制是自注意力的变体, 可以有效捕捉长远依赖关系来提高模型的性能和表达能力, 更好表征脑肿瘤图像中其他像素对当前肿瘤像素的重要关系. 因此, 借助非局部自注意力模块去有效进行脑肿瘤分割任务. 同时, 为适应三维U-Net架构, 将非局部注意力模块从二维扩展至三维, 扩展后模块具体结构如图2所示.

|
图 2 非局部自注意力结构图 |
在PyCSAU-Net中, 三维非局部自注意力模块的计算可分为3步. 首先, 将在U-Net下采样提取的紧凑特征图
| Q=Unfold(XWQ),K=Unfold(XWK),V=Unfold(XWV) | (1) |
其中,
然后, 经过矩阵
| A=Softmax(QTK√C),O=AV | (2) |
其中, 输出矩阵
然后, 使用残差连接将输入特征图
| Y=Conv1(fold(O))+X | (3) |
其中,
非局部自注意力模块的主要作用是实现脑肿瘤图像像素之间的长距离依赖性, 克服传统卷积操作的限制, 更直接地捕获像素全局信息, 有利于在模型得到更加精确的脑肿瘤分割结果.
1.1.2 金字塔卷积模块金字塔卷积是包含多个级别, 每个级别包含不同类型的核的金字塔结构卷积, 主要致力于扩大图像的感受野大小, 通过并行增加内核大小来处理图像. 它在脑肿瘤分割任务中可以捕获不同级别、多个尺度上的局部精细特征与小尺寸肿瘤. 在不增加计算成本或模型复杂性的情况下, 通过在不同核尺度上处理输入, 提高模型性能. 金字塔卷积模块结构如图3所示.
为能够在金字塔卷积模块的每个级别上使用不同深度的内核, 将输入的特征图分为不同组, 并对每个输入特征图独立应用不同大小的内核. 这被称为分组卷积, 图4展示了其中两个例子. 在这些例子中, 有8个输入和输出特性映射. 图4(a)显示了一组输入特征图组成的情况. 其中内核的深度等于输入特征图的数量, 每个输出特征图都被连接到所有的输入特征图. 在图4(b)中, 可观察到输入特征图被分成两组, 并且将内核独立地应用于每个组, 这种技术被称为分组卷积, 它使得内核深度减少了2倍. 当组数增加时, 内核的连通性会随之减小.

|
图 3 金字塔卷积结构图 |

|
图 4 分组卷积示例图[25] |
在三维U-Net架构中嵌入金字塔卷积模块, 可以在多个尺度上解析输入, 并捕获更详细的特征信息. 同时, 结合非局部自注意力模块计算全局特征的相似性, 获得全局的特征信息. 将局部精细特征与全局的特征信息相结合之后, 生成更多有利于分割的特征表示, 有助于脑肿瘤分割性能的进一步提升.
1.2 损失函数在损失函数上, 采用Dice损失函数
| LDC=1−2∑Ni=0yiˆyi∑Ni=0(yi+ˆyi) | (4) |
| LCE=−∑i∈N∑l∈Lyilogˆyi | (5) |
组合函数
| LDCE=αLDC+(1−α)LCE | (6) |
类别不平衡问题是脑肿瘤分割任务中长期存在的挑战, 采用Dice损失和Cross-Entropy损失的组合损失函数
所提出的脑肿瘤分割模型采用PyTorch深度学习框架实现, 并在搭载有双卡24 GB内存的GTX 3090 GPU的硬件环境下进行训练和测试. 实验中采用Adam优化器, 初始学习率为0.001, 动量为0.95, 权重衰减为1E–5, 批量大小为4, 训练轮数为500轮.
2.2 数据集与预处理为评估PyCSAU-Net在脑肿瘤分割任务中的性能, 采用BraTS 2019和BraTS 2020两个公开数据集进行评估. BraTS 2019数据集包含训练数据集和验证数据集, 其中训练数据集包括335例胶质瘤患者, 其中259例为高级别胶质瘤(HGG)病例, 76例为低级别胶质瘤(LGG)病例; 验证数据集则包含125例未知级别患者病例. 在BraTS 2020数据集中, 训练数据集扩展了到369例但不再区分HGG和LGG, 而验证数据集保留了125例未知等级病例.
在两个数据集中, 每个患者的MRI扫描包括原生T1加权(T1)、对比后T1加权(T1CE/T1Gd)、T2加权(T2)和液体减毒反转恢复(FLAIR) 4种模态. 图5给出了数据库中典型脑肿瘤的样本示例情况. 图5中, 从左至右依次是FLAIR、T1、T1CE/T1Gd、T2这4种模态和真实标签(ground truth). 其中, 每个图像的真实标签由多年相关经验的专业医师手动分割标记, 每一种颜色分别代表着一种标签, 红色(标签1)代表着坏死和非增强区域, 绿色(标签2)代表着水肿区域, 黄色(标签4)代表着增强肿瘤. 分割评估指标考虑的是有各子区域组成的3个肿瘤区域, 即整个肿瘤(WT, 标签1、2和4的组合区域)、肿瘤核心(TC, 标签1、4的组合区域)和增强肿瘤(ET, 标签4). 训练数据集的真实标签由BraTS的组织者提供, 由于验证数据集的标签不对公众开放, 需通过提交官方网站对模型的预测结果进行评估以确保结果的权威性和公正性. 在脑肿瘤图像处理上, 为解决类别不平衡问题, 将原始的数据大小裁剪为128×128×128, 这样可以消除大量无关背景和无用的像素的影响. 并且对每张图像进行Z值归一化处理、将图像在[−10°, +10°]范围内进行随机旋转并且进行[−0.1, 0.1]之间的随机强度偏移.
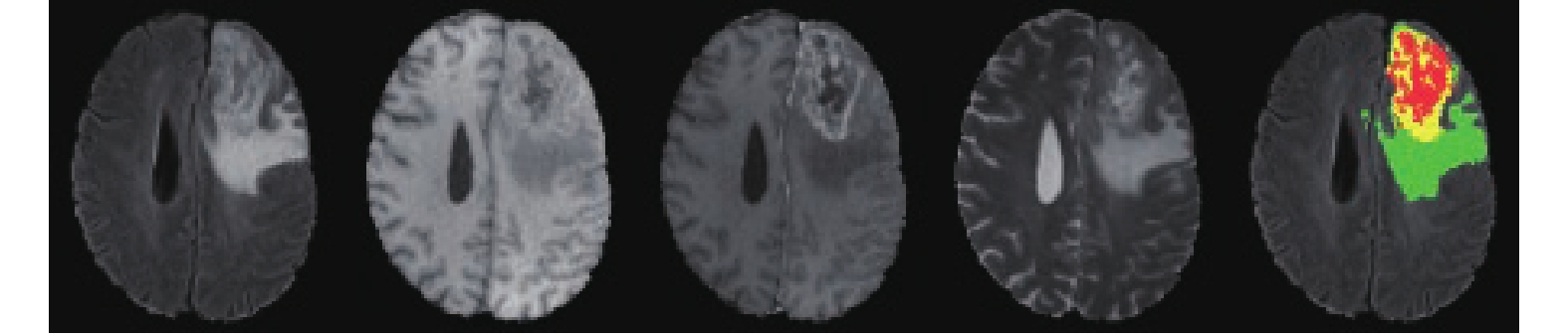
|
图 5 MRI脑肿瘤多模态图像及标签示例 |
2.3 评价标准
评估网络模型的分割精度主要通过Dice相似度分数(DSC)和95% Hausdorff距离(HD95)这两个指标来衡量, 它们将用于3个肿瘤子区域: 完全肿瘤(WT)、增强肿瘤(ET)和肿瘤核心(TC)的分割评估. DSC是测量网络模型的体素分割预测和真实标签之间的重合度(相似性), 其定义如式(7)所示:
| DSC(Y,P)=2×|Y∩P||Y|∪|P|=2×Y⋅PY2+P2 | (7) |
其中,
HD95通常用作基于边界的度量来测量体素分割预测的边界与真实值之间距离, 其定义如式(8)所示:
| HD95(Y,P)=max | (8) |
其中,
为验证PyCSAU-Net的有效性, 采用BraTS 2019和BraTS 2020数据集进行模型评估. 首先, 在BraTS 2020训练和验证数据集上进行金字塔卷积(PyConv)和非局部自注意力(SA)模块的消融实验, 以评估对脑肿瘤分割的有效性, 进而在BraTS 2019和BraTS 2020验证数据集上完成对比实验.
2.4.1 消融实验结果在BraTS 2020训练数据集上, 利用五折交叉验证来测试金字塔卷积和非局部自注意力模块及其组合在脑肿瘤分割任务上的效果, 即使用三维U-Net作为基线模型, 将金字塔卷积模块单独嵌入到U-Net下采样过程中形成PyConvU-Net, 将非局部自注意力模块单独加入到第4层的横向连接中形成SAU-Net, 将两个模块组合来形成PyCSAU-Net进行测试比较, 具体消融实验结果如表1所示.
| 表 1 BraTS 2020训练集DSC指标的消融实验结果 |
表1可见, PyCSAU-Net在表中列出的4个模型中DSC得分最高, 在完全肿瘤、增强肿瘤和肿瘤核心分别实现了0.910、0.792、0.846的DSC分割精度, 分别超过其基线方法0.9%、0.9% 和2.3%. 此外, 对于单独引入金字塔卷积模块和非局部自注意力模块形成的SAU-Net和PyConvU-Net模型, 其中SAU-Net 在完全肿瘤、增强肿瘤和肿瘤核心分别实现了0.906、0.790、0.836的DSC分割精度, 与基线方法相比分别增加0.5%, 0.7% 和 1.4%, 特别是在肿瘤核心上的提升结果, 可有效证明非局部自注意力模块可以更加关注小尺寸肿瘤分割. 同样, PyConvU-Net在完全肿瘤、增强肿瘤和肿瘤核心分别实现了0.909、0.788、0.834的DSC分割精度, 与基线方法分别增加0.8%, 0.5%和 1.1%, 也说明金字塔卷积模块在脑肿瘤分割的有效性. 综上, 实验结果可说明金字塔卷积模块和非局部自注意力模块对于脑肿瘤分割任务的有效性.
为进一步验证PyCSAU-Net各个模块的有效性, 继续在BraTS 2020验证数据集上进行消融实验, 利用369张MRI图像在BraTS 2020训练数据集上训练模型, 然后将模型预测的125个验证数据集结果提交到官方BraTS网站进行统一评估. 实验结果如表2所示.
| 表 2 BraTS 2020验证集DSC指标的消融实验结果 |
从表2中可以看出, PyCSAU-Net在BraTS 2020验证数据集上也取得了最好的结果, 这与在训练集上的结果保持了一致. 实验结果表明, PyCSAU-Net在完全肿瘤、增强肿瘤和肿瘤核心分别实现了0.902、0.774、0.824的DSC分割精度, 与基线方法相比分别增加0.8%, 1.2% 和 2.5%, 再一次证明了PyCSAU-Net在脑肿瘤分割上的有效性. 在BraTS 2020验证数据集上, 增强肿瘤和肿瘤核心的分割性能也得到了改进, 这进一步表明, 金字塔卷积模块和非局部自注意力模块的组合可以使整个网络模型更加关注到细节特征信息, 以准确分割肿瘤区域.
2.4.2 对比实验结果此后, 在BraTS 2019和BraTS 2020验证数据集上与先进方法进行比较, 来进一步验证所提出模型的泛化性和有效性. 表3和表4分别显示所提出网络模型在这两个数据集上的比较结果.
| 表 3 BraTS 2019验证集DSC和HD95指标的比较结果 |
| 表 4 BraTS 2020验证集DSC和HD95指标的比较结果 |
从表3中可看出, PyCSAU-Net在完全肿瘤、增强肿瘤和肿瘤核心的DSC值分别为90.4%、78.1%和82.5%. 很明显, PyCSAU-Net在完全肿瘤和增强肿瘤的DSC值中优于其他模型得分, 进一步说明所提出模型更加关注脑肿瘤图像细节特征, 有助于增强肿瘤的分割. 在HD95指标上, PyCSAU-Net在完全肿瘤、增强肿瘤和肿瘤核心分别得到了4.54, 2.71和6.01的结果. 同样, PyCSAU-Net在增强肿瘤分割上取得了最好的结果. 因此, 上述实验结果再次证实了PyCSAU-Net模型在脑肿瘤分割任务上的有效性.
同时, 也在BraTS 2020验证数据集进行了与领域代表性方法的比较实验, 实验结果如表4所示. 从表4中可以看出, PyCSAU-Net在完全肿瘤、增强肿瘤和肿瘤核心的DSC值分别为90.1%、77.4%和82.4%, 在完全肿瘤和肿瘤核心上的DSC值中优于其他模型. 在6个所比较模型中, Huang等人[16]、Wang等人[23]以及Jiang等人[26]引入由CBAM模块和Transformer架构的自注意机制等组成的注意模块来构建分割网络, 其余3个模型属于非注意方法. 从实验结果来看, 引入注意力机制的模型平均DSC值要高于无注意机制的模型结果, 这也说明了注意机制对脑肿瘤分割任务有效性. 在HD95指标上, PyCSAU-Net在肿瘤核心分割上获得了最优结果, 在另外两个任务上也表现出较好竞争性. Wang等人将三维Transformer引入到U-Net结构中, 利用Transformer来捕获肿瘤全局信息, 获得了更高的增强肿瘤分割结果, 但在3个任务总体上与所提出模型结果仍然相当. 因此, 比较结果显示了PyCSAU-Net的良好竞争性能.
2.4.3 结果可视化为更加直观说明所提出模型的有效性, 还进行了脑肿瘤分割结果的可视化实验, 如图6所示. 在图6中, 从左到右分别为ground truth (真实标签), 基线方法和PyCSAU-Net分割结果, 此外将基线方法和PyCSAU-Net分割结果放大. 在脑肿瘤分割图像中, 用不同颜色表示不同肿瘤类别, 黄色区域代表增强肿瘤, 绿色区域表示水肿, 红色区域为坏死和非增强.

|
图 6 网络模型分割结果对比图 |
通过观察分割结果示例图可知, 在与真值图像进行对比后, 可以发现PyCSAU-Net和三维U-Net (baseline)在这几个肿瘤图像上均能够获得较好的分割结果, 表明这两个方法在脑肿瘤分割任务中均是有效的. 通过观察放大区域可以发现, PyCSAU-Net模型获得了优于基线的脑瘤分割结果, 尤其是在小尺寸脑肿瘤的分割结果上, 这也在一定程度说明了注意力机制在该医学图像任务上的效果.
3 结论本文将非局部自注意力模块和金字塔卷积模块嵌入到三维U-Net中构建出了PyCSAU-Net脑肿瘤分割模型, 该模型在三维U-Net横向连接位置引入扩展的三维非局部注意力模块来提升U-Net的长距离特征建模能力, 并在网络下采样阶段以金字塔卷积模块替代传统卷积来更好地捕获脑肿瘤图像多个尺度上的局部精细特征, 从而在整体上提升脑肿瘤分割模型的性能. 广泛的消融实验和对比实验结果也证明了所提出模型在脑肿瘤分割任务上的良好竞争力. 在未来工作中将考虑在包括器官分割、脊柱分割等其他典型医学分割任务上来评测所提出PyCSAU-Net模型的泛化能力; 另一方面, 也将探索引入更先进的Transformer自注意力模块来进一步提升脑肿瘤分割网络的性能.
| [1] |
Bakas S, Akbari H, Sotiras A, et al. Advancing the cancer genome atlas glioma MRI collections with expert segmentation labels and radiomic features. Scientific Data, 2017, 4: 170117. DOI:10.1038/sdata.2017.117 |
| [2] |
张腾达, 吕晓琪, 任晓颖, 等. 基于空间模糊核聚类的脑肿瘤图像分割方法. 控制工程, 2017, 24(10): 2107–2111. |
| [3] |
赵奕名, 李锵, 关欣. 组卷积轻量级脑肿瘤分割网络. 中国图象图形学报, 2020, 25(10): 2159–2170. |
| [4] |
江宗康, 吕晓钢, 张建新, 等. MRI脑肿瘤图像分割的深度学习方法综述. 中国图象图形学报, 2020, 25(2): 215–228. |
| [5] |
Zikic D, Ioannou Y, Brown M, et al. Segmentation of brain tumor tissues with convolutional neural networks. Proceedings of the 2014 MICCAI workshop on Multimodal Brain Tumor Segmentation Challenge. Boston, 2014. 36–39.
|
| [6] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015. 3431–3440.
|
| [7] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [8] |
Isensee F, Kickingereder P, Wick W, et al. Brain tumor segmentation and radiomics survival prediction: Contribution to the brats 2017 challenge. Proceedings of the 3rd International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Quebec City: Springer, 2018. 287–297.
|
| [9] |
Shaikh M, Anand G, Acharya G, et al. Brain tumor segmentation using dense fully convolutional neural network. Proceedings of the 3rd International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Quebec City: Springer, 2018. 309–319.
|
| [10] |
Liu D, Zhang H, Zhao MM, et al. Brain tumor segmention based on dilated convolution refine networks. Proceedings of the 16th IEEE International Conference on Software Engineering Research, Management and Applications. Kunming: IEEE, 2018. 113–120.
|
| [11] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision. Stanford: IEEE, 2016. 565–571.
|
| [12] |
Chen ML, Wu YZ, Wu JH. Aggregating multi-scale prediction based on 3D U-Net in brain tumor segmentation. Proceedings of the 5th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Shenzhen: Springer, 2020. 142–152.
|
| [13] |
Liu SA, Guo XN. Improving brain tumor segmentation with multi-direction fusion and fine class prediction. Proceedings of the 5th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Shenzhen: Springer, 2020. 349–358.
|
| [14] |
Tang JR, Li TF, Shu H, et al. Variational-autoencoder regularized 3D MultiResUNet for the BraTS 2020 brain tumor segmentation. Proceedings of the 6th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Lima: Springer, 2021. 431–440.
|
| [15] |
Zhang WB, Yang G, Huang H, et al. ME-Net: Multi-encoder net framework for brain tumor segmentation. International Journal of Imaging Systems and Technology, 2021, 31(4): 1834-1848. DOI:10.1002/ima.22571 |
| [16] |
Huang H, Yang G, Zhang WB, et al. A deep multi-task learning framework for brain tumor segmentation. Frontiers in Oncology, 2021, 11: 690244. DOI:10.3389/fonc.2021.690244 |
| [17] |
Zhang JX, Jiang ZK, Liu DW, et al. 3D asymmetric expectation-maximization attention network for brain tumor segmentation. NMR in Biomedicine, 2022, 35(5): e4657. DOI:10.1002/nbm.4657 |
| [18] |
Guo D, Wang L, Song T, et al. Cascaded global context convolutional neural network for brain tumor segmentation. Proceedings of the 5th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Shenzhen: Springer, 2020. 315–326.
|
| [19] |
Cheng K, Hu CH, Yin PY, et al. Glioma sub-region segmentation on Multi-parameter MRI with label dropout. Proceedings of the 6th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Lima: Springer, 2021. 420–430.
|
| [20] |
Li XY, Luo GN, Wang KQ. Multi-step cascaded networks for brain tumor segmentation. Proceedings of the 5th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Shenzhen: Springer, 2020. 163–173.
|
| [21] |
Cheng XC, Jiang ZK, Sun QL, et al. Memory-efficient cascade 3D U-Net for brain tumor segmentation. Proceedings of the 5th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Shenzhen: Springer, 2020. 242–253.
|
| [22] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [23] |
Wang WX, Chen C, Ding M, et al. TransBTS: Multimodal brain tumor segmentation using Transformer. Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention. Strasbourg: Springer, 2021. 109–119.
|
| [24] |
Wang XL, Girshick R, Gupta A, et al. Non-local neural networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7794–7803.
|
| [25] |
Duta IC, Liu L, Zhu F, et al. Pyramidal convolution: Rethinking convolutional neural networks for visual recognition. arXiv:2006.11538, 2020.
|
| [26] |
Jiang Y, Zhang Y, Lin X, et al. SwinBTS: A method for 3D multimodal brain tumor segmentation using Swin Transformer. Brain Sciences, 2022, 12(6): 797. DOI:10.3390/brainsci12060797 |
 2024, Vol. 33
2024, Vol. 33



