2. 福建省大数据挖掘与应用技术重点实验室, 福州 350118;
3. 闽江学院 计算机与大数据学院, 福州350121;
4. 福建省信息处理与智能控制重点实验室, 福州 350121
2. Fujian Provincial Key Laboratory of Big Data Mining and Applications, Fuzhou 350118, China;
3. School of Computer and Big Data, Minjiang University, Fuzhou 350121, China;
4. Fujian Provincial Key Laboratory of Information Processing and Intelligent Control, Fuzhou 350121, China
肝脏是人体的主要器官, 对新陈代谢、消化吸收和免疫功能有着重要作用. 目前, 肝癌是6大常见癌症, 也是3大死因之一[1]. 临床上, 计算机断层扫描(computed tomography, CT)是对肝脏的形状、纹理和局部病变评估的重要工具[2]. 肝脏的形状和纹理异常以及CT中可见的病变是原发性和继发性肝肿瘤疾病进展的重要生物标志物[3]. 此外, 来自其他腹部器官(如结肠、直肠和胰腺)以及远处器官(如乳腺和肺部)的癌症在疾病期间往往转移至肝脏. 因此, 常规分析肝脏及其病变对于全面的肿瘤分期非常重要. 然而, 在临床医学中需要医生手动标注肝脏的病变和形状, 这个过程非常费力且耗时. 因此, 借助计算机辅助诊断系统, 进行CT图像的自动肝脏病变检测有助于减少医生的工作量以提高诊断效率.
最近, 基于有监督的深度学习方法在肝部病灶分割中取得不错的进展[4–6], 这些方法需要利用大量标记的腹部CT数据集进行训练. 例如, 马力等人[7]在UNETR++基础上引入门控单元相关技术进行肝脏的分割. Yan等人[5]在Mask R-CNN框架上探索多模态的联合检测系统. Araújo等人[6]则通过U-Net为主导的技术探索肝脏病变分割任务. 然而, 腹部CT图像所呈现的器官不止肝部, 在进行病灶检测时需要在公开腹部CT图像上把肝部单独分割出来再进行检测. 因此, 这些方法的模型复杂程度通常较高. 并且, 大量标记的、可用的数据难以获取, 这促使了无监督异常检测方法的研究.
最近, 无监督异常检测方法通常仅使用正常数据进行训练, 力图让模型充分拟合正常数据. 其可分为两种范式[8]: 基于特征嵌入的方法和数据重建的方法. 前者通过分析特征嵌入空间中的抽象表示来推断异常. 而后者则通过比较数据空间中重构数据与原始数据之间的差异来识别潜在的异常. 然而, 基于特征嵌入的方法[9–11]往往需要强大的预训练网络, 其带来的冗余特征信息导致此类方法并不适用于语义单一的医学影像[12]. 此外, 以VAE和GAN为基础的数据重建方法会面临将正常和异常图像都较好重建的情况, 这导致了较大的误报率. 由此, 许多方法被提出以解决上述问题[13–16]. 这些方法在工业领域已经取得一些突破, 尤其是在MVTec数据集[17]上, 但在医学领域中少有对肝部异常检测的探索[5,6,18–20]. 最近, 随着基于合成异常的自监督方法逐渐流行, 许多方法被提出并在医学领域获得不错的效果[20–23], 这类方法将预测合成异常作为代理任务, 首先通过解决预测任务来学习自监督表示, 然后在学习到的表示上构建一个异常检测器, 以区分异常样本和正常样本. 但这些方法仍面临着模型过拟合合成异常数据的问题. 例如, 对于肝部CT数据集而言, 异常信息通常集中在肝部区域, 现有方法将面临在背景区域创建异常, 从而导致生成异常的数据分布和真实异常的数据分布差距过大. 此外, 利用U-Net结构进行肝部肿瘤病灶定位时, 可能由于病灶过于细微而导致训练前期的性能挣扎, 影响模型对肝部异常检测的性能[24].
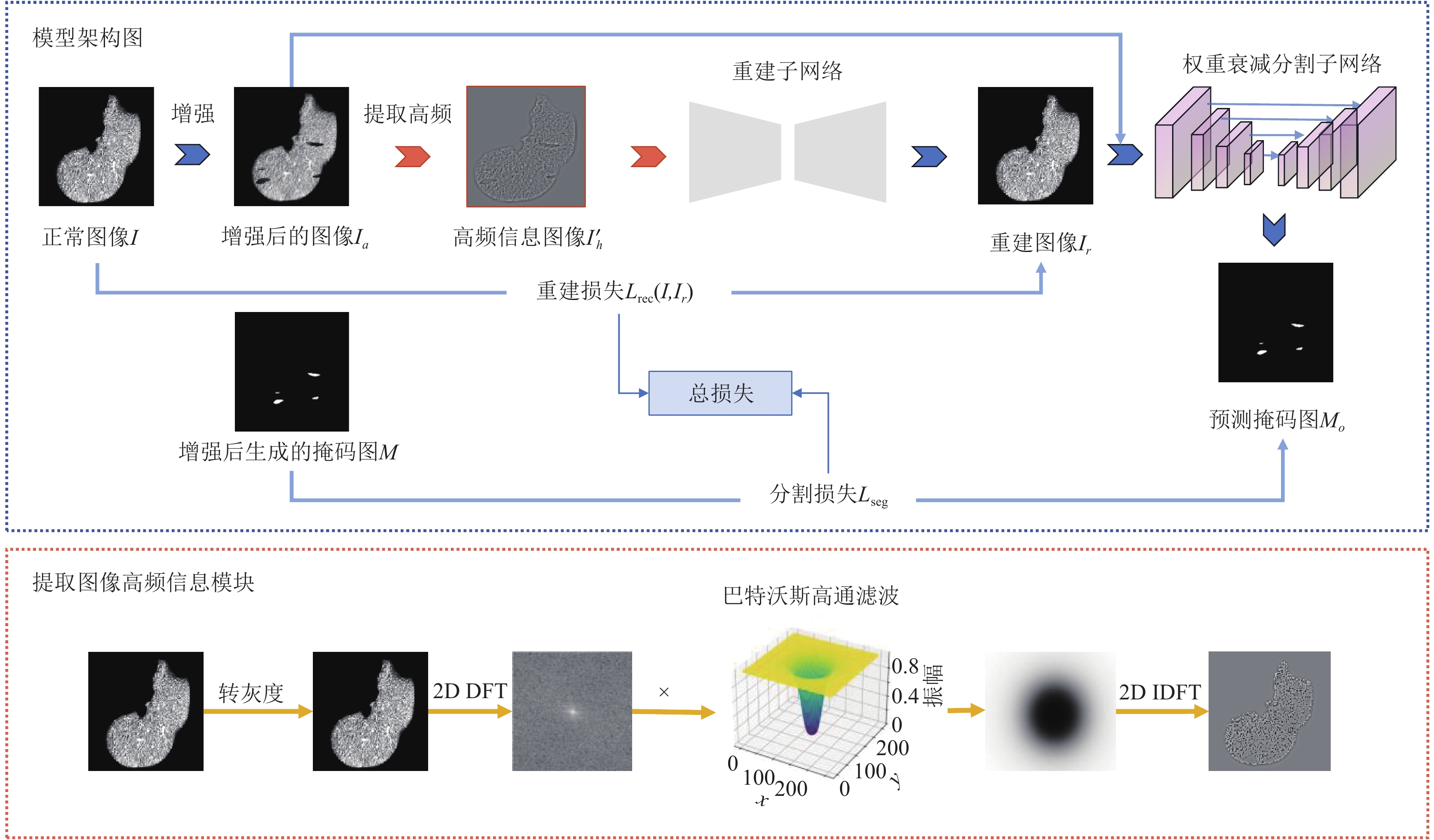
因此, 为了解决上述问题, 本文提出了一个基于频率感知图像恢复的自监督算法以实现肝部病灶的精确检测和定位. 该方法由3部分构成: 第1部分包括合成异常和提取图像高频信息; 第2部分为重建网络对提取高频图像的重建; 第3部分则是分割网络对异常图像的识别与定位. 具体而言, 方法首先设置更加广泛且适宜的合成异常策略, 以创建局部不规则模式作为真实异常图像的粗略近似, 该策略保留的丰富局部特征, 生成类似密度阴影的异常, 增加了模型寻找局部不规则性的难度, 激励了模型学习正常样本的聚类边界, 以达到抑制模型对合成异常拟合的问题. 其次, 针对重建网络对合成肝部异常图像进行隐式检测和消除时仍会将异常信息进行重建的问题, 在合成图像传入重建网络之前引入提取图像高频信息模块, 对合成异常图像信息进行局部破坏, 以抑制重建网络对异常的敏感性. 此外, 为了增加模型的训练难度, 方法采取重建子网络和分割子网络共同训练的范式, 通过在重建和原始外观以及重建子空间上训练判别模型, 以避免模型学习到单一的异常信息. 最后, 在分割网络中采用权重衰减的训练策略, 以减少U-Net架构范式网络中的跳跃链接在训练前期学习到恒等映射函数的风险. 本文通过大量实验证明了所提出方法的有效性和先进性. 综上所述, 方法的贡献可总结如下.
(1) 设计了一个自监督框架以用于肝部病灶检测, 并提出更加广泛适宜的合成异常策略, 作为真实异常图像的粗略近似, 通过生成覆盖广泛范围的异常, 以激励模型学习正常样本的聚类边界, 从而减少训练中对合成肝部异常图像的过拟合问题.
(2) 引入一个提取图像高频信息模块, 以解决重建网络训练时期对合成肝部异常图像的异常信息敏感问题.
(3) 采用权重衰减训练策略以训练分割子网络, 从而减少模型训练前期因跳跃链接所导致的琐碎解问题.
(4) 在真实数据集所开展的广泛实验证明, 本文所提出的方法相较于其他异常检测方法拥有更好的结果, 能够提升肝肿瘤异常检测的准确率.
1 相关模型和技术现有的医学异常检测领域中可把检测方法大致分为3类: 基于有监督学习的方法, 基于图像重建的方法和基于自监督学习的方法.
1.1 基于有监督学习的方法有监督的方法能够在有标签数据的指导下, 从全局到局部学习异常图像的特征分布. 目前, 该方法依赖于足够多的有标签数据, 在不同的计算机视觉领域取得成功. 最近, 已经提出了多种用于CT图像中肝脏病变检测的有监督方法. 例如, Ben-Cohen等人[18]使用全连接网络将轴向切片输入得到一个病变的概率图, 然后利用超像素稀疏分类的局部补丁级别分析, 判断每个局部超像素分类为病变或非病变. Yan等人[5]提出了一种多任务通用病变分析网络(MULAN) 用于联合检测、标注和分割各种身体部位的病变. MULAN是基于改进的Mask R-CNN框架, 具有3个头分支和三维特征融合策略. Araújo等人[6]提出了一种自动分割CT图像中肝脏病变的方法, 所提出的方法采用了两个深度卷积神经网络(CNN)模型, 包括5个主要步骤: 图像获取、图像预处理、使用RetinaNet进行初始分割、使用U-Net进行病变分割、分割的精化. 然而, 基于有监督学习的方法仅从标注的异常图像中学习特征, 面临着有限训练数据的问题. 并且, 在已标注的大部分异常图像中往往是一些常见的例子(如常见疾病), 有监督的方法可能无法检测其分布外的异常.
1.2 基于图像重建的方法基于图像重建方法的常用基准模型包括自编码器, 变分自编码器和生成对抗网络等. 通常这类方法以重建模型能很好地重建正常图像为前提, 并用重建误差来判别图像是否异常. 例如, Schlegl等人[25]提出AnoGAN, 模型中判别器被过度拟合到正常图像分布. 其主要思想是通过迭代的过程找到一个潜在的特征, 生成与查询图像最类似的图像, 如果两者有很大差异则判定为异常. 然而, 该方法存在测试耗时的问题. 因此, Schlegl等人[19]进一步利用编码器学习从视网膜OCT图像到潜在空间的映射, 并提出了AnoGAN的快速版本, 称为f-AnoGAN. 此外, Li等人[26]提出一种基于生成对抗网络的无监督肝脏病变检测框架, 仅使用正常和伪病变数据进行训练, 其中伪病变数据由生成对抗网络生成. Frid-Adar等人[27]利用生成对抗网络合成医学影像并用于数据增强以提升CNN网络对肝脏病变的分割能力. 然而, 这些基于GAN的方法可能会面临有时也将异常图像很好地重建的问题, 这导致了模型的误判. 为了避免异常图像也被较好重建, 一些方法[13,16]利用存储训练正常样本的潜在表示内存库来规范化测试样本的表示. 尽管这些方法有效, 但内存库同样引入了额外的计算开销.
此外, 许多工作还采用合成的伪异常样本来训练去噪模型[21,28]或共同训练额外的分割模型[21], 从而抑制只用正常数据集训练的缺陷: 由于模型并没有学习到异常图像特征分布, 导致在检测分布外的异常时性能低下. 然而, 这些合成的纹理异常可能无法处理接近真实分布的异常[29]. 因此, 文献[29]提出了一种利用量化特征生成伪异常的方法. 值得注意的是, 上述重建模型在测试时采用原始图像, 尽管去噪模型在训练时会破坏图像, 但测试时仍使用原始图像. 这种设计保留了测试图像的所有信息. 然而, 同样保留的异常信息可能会在最终输出中经历恒等映射, 从而导致性能欠佳[30]. 为此, 一些方法[31,32]将传统的图像重建任务转化为图像恢复任务, 本文提出的方法也受此启发.
1.3 基于自监督学习的方法自监督学习(self-supervised learning, SSL)是一种通过预训练任务以学习任务相关的语义特征, 而无需手动标注的方法. 对于医学影像领域, 基于自监督学习的异常检测任务, 其主要挑战在于如何设计有效的预训练代理任务. 目前, 许多学者已经提出了各种SSL方法用于自然图像, 例如对比学习[33]、旋转预测[34]、几何变换[35]. 此外, 自监督学习方法的一个独特子类依赖于合成异常样本[36,37], CutPaste[36]通过随机裁剪和粘贴进行数据增强, 而AugPaste[37]使用MixUp从病变库中混合和粘贴补丁来合成异常. 尽管这类方法已经应用于医学影像, 但仅进行了未经修改的初步应用, 这类增强预测方法会生成明显与其他部分不同的补丁, 特别是在腹部CT扫描图像上, 因为形状对齐不良和亮度变化, 这种差异更为明显. 由于这类自监督任务过于简单, 容易过拟合明显的差异, 降低了学习到的特征的有用性. 因此, 为了合成更有助于医学图像任务的异常, Meissen等人提出AutoSeg[38]用于生成类似于真实世界病理特征的多样化人工异常. PII[22]和FPI[23]则设计了关注局部病灶的自监督任务: FPI在图像的随机补丁中合成细微的缺陷; PII则在FPI的基础上, 引入泊松图像编辑使合成的异常更加自然. 相应的像素级标签可帮助网络在给定上下文区域对异常进行定位, 这些方法在空间对齐的脑部MRI和腹部CT数据上表现出良好性能. 此外, Schlüter等人提出的NSA[20]将Poisson图像编辑与缩放、平移和基于Gamma分布的补丁形状采样策略相结合, 以合成自然且多样化的异常. Li等人[39]提出了一个自监督的增强任务CropMixPaste用于生成类似于密度阴影的异常, 以促使模型检测肺部CT扫描图像的局部不规则性. 然而, 许多医学图像具有复杂的结构, 并且需要专业的医学知识, 这使得确定异常变得具有挑战性, 需要针对性设计具体的自监督任务. 因此, 基于这项挑战 本文在DRAEM[21]提出的合成异常基础上加以改进 并探索合成异常方法在肝部图像上的应用.
2 频率感知图像恢复模型 2.1 概述与定义异常检测是指在有限甚至没有异常先验知识的情况下识别和定位异常[40]. 对于肿瘤检测任务, 本文仅使用正常数据集
| ϕi={1,ifScore(Mo)⩾t0,otherwise | (1) |
其中,
本文提出的方法如图1所示. 模型由合成异常与提取图像高频模块, 重建子网络和权重衰减分割子网络3部分组成. 首先, 将正常图像进行增强得到伪异常肝部图像, 以解决肝部病灶数据难以获取的问题. 具体而言, 合成方法分为两个步骤: 定位肝部区域和异常合成. 接着, 为了抑制重建网络对异常信息的敏感性, 在将合成图像传入重建网络之前, 引入提取图像高频信息模块, 即通过高通滤波器破坏图像局部信息, 从图像高频角度重建(恢复)图像. 其中, 重建网络被训练用于将破坏后的图像恢复为原始正常图像. 最后, 将重建图像和原始图像合并输入到权重衰减的分割网络中进行异常的识别与定位, 以增强模型的训练难度从而缓解过拟合现象.
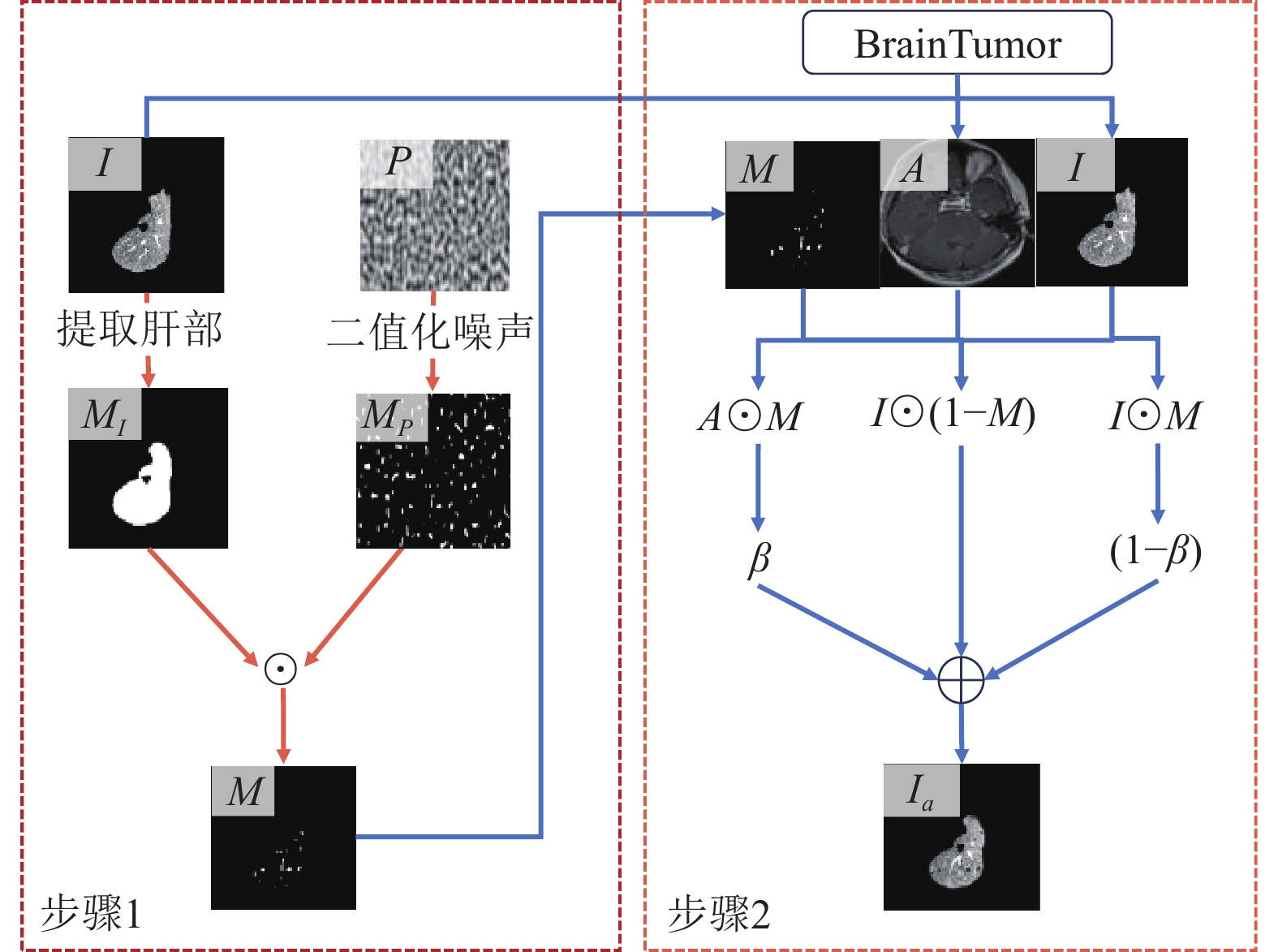
2.2 异常数据合成模块最近, 基于自监督学习的方法在异常检测领域展示了优异的性能. 这些方法通过设置代理任务让模型学习有利于区分异常和正常的特征, 再对异常和正常进行分类和定位. 由于不同的自监督学习方法对不同领域的泛化性能不佳, 且模型可能过拟合基于数据增强代理任务合成的外观, 在缺少先验异常知识背景下设置适宜且广泛的数据增强代码任务是一大挑战. 因此, 受到DRAEM[21]启发, 本文设计了一种有效的数据增强代理任务来模拟肝部肿瘤异常. 值得注意的是, 尽管合成的异常病变可能与某些真实病变相似, 但本文提出的方法仍然不是对真实异常病变的完美模拟. 在设计增强任务时, 尽力保留了原图像的丰富性, 并模拟了不同程度的密度变化. 然而, 考虑到CT扫描特征的多样性和异常病变的未知性, 合成的目标仅是生成与真实异常病变相符的局部细微不规则模式(即局部类别变化). 这激发了模型关注局部的不规则性, 并将检测能力推广到未见过的真实异常病变. 具体而言, 通过合成异常的代理任务, 使得重建子网络学习到有利于区分异常和正常特征信息. 并结合分割子网络对异常进行识别和定位以实现自监督学习的训练. 如图2所示, 所提出的异常合成方法主要分为两个步骤, 具体如下.
噪声掩码图生成: 首先, 通过二维Perlin噪声生成器[41]随机生成一个噪声图像
|
图 1 方法模型架构图 |
|
图 2 异常合成方法图 |
伪异常图像合成: 与DRAEM[21]不同的是, 该阶段不再使用DTD[42]数据集作为合成异常的外分布数据集. 由于肝部肿瘤区域像素通常偏暗(如图3所示) 直接采用DTD数据集生成的异常图像会严重增加与真实异常样本分布的差异. 因此, 本阶段选择使用外源数据集BrainTumor[43]. 首先, 将得到的掩码图
| Ia=(1−M)⊙I+(1−β)(M⊙I)+β(M⊙A) | (2) |
其中,
抑制重建网络对异常信息的敏感性是一大难题. 基于这一挑战, 受到文献[30]启发, 引入提取图像高频信息模块, 以在保留图像信息的同时减少图像异常区域对重建网络的影响. 首先, 方法将合成的异常图像
| F(μ,ν)=m−1∑x=0n−1∑y=0f(x,y)e−i2π(μxm+νyn) | (3) |
其中,
| HB(μ,ν)=11+[D0D(μ,ν)]2 | (4) |
其中,
| I′h=f(x,y)h=1mnm−1∑μ=0n−1∑ν=0F(μ,ν)hej2π(μxm+νyn) | (5) |
其中,
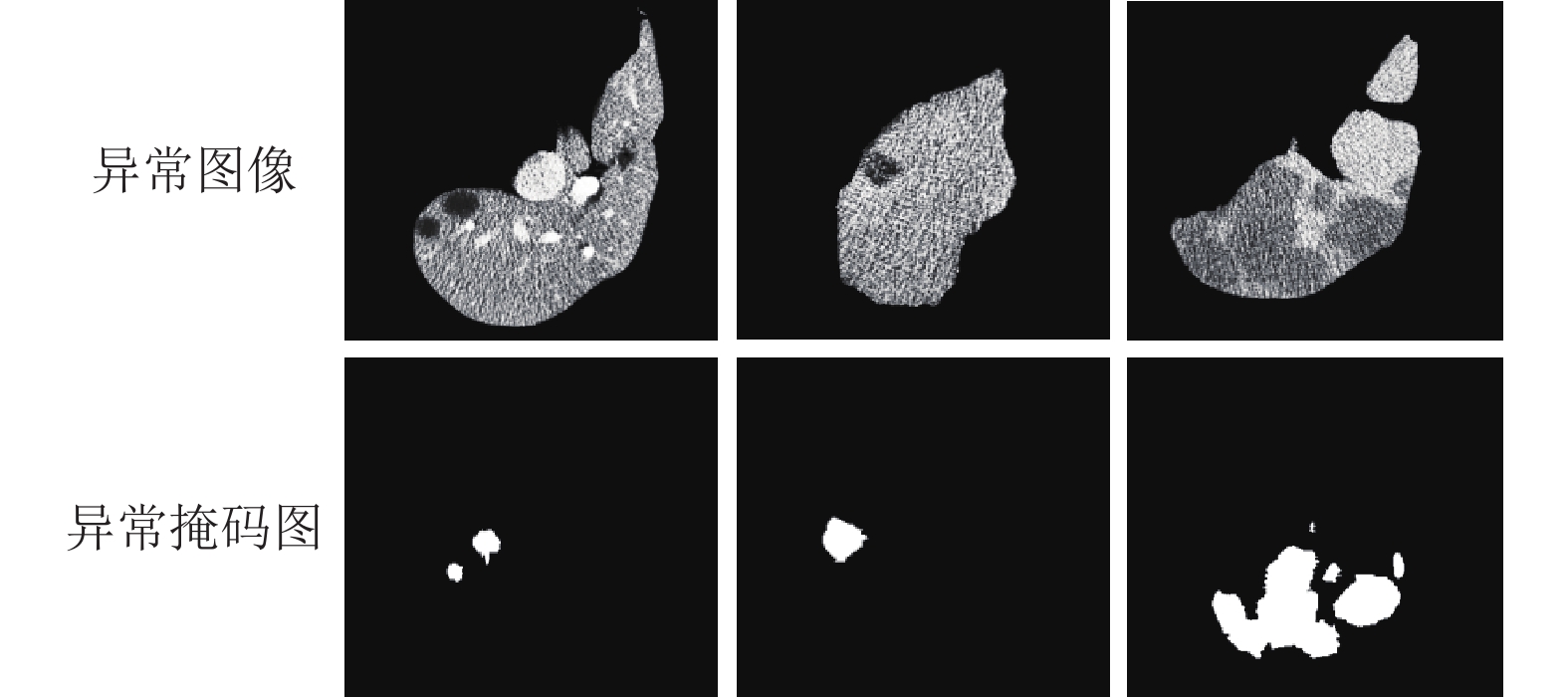
|
图 3 真实异常示例图 |
2.4 重建子网络
通过异常数据合成模块及提取图像高频信息模块所生成的高频图像
| Lrec(I,Ir)=λLSSIM(I,Ir)+L2(I,Ir) | (6) |
其中,
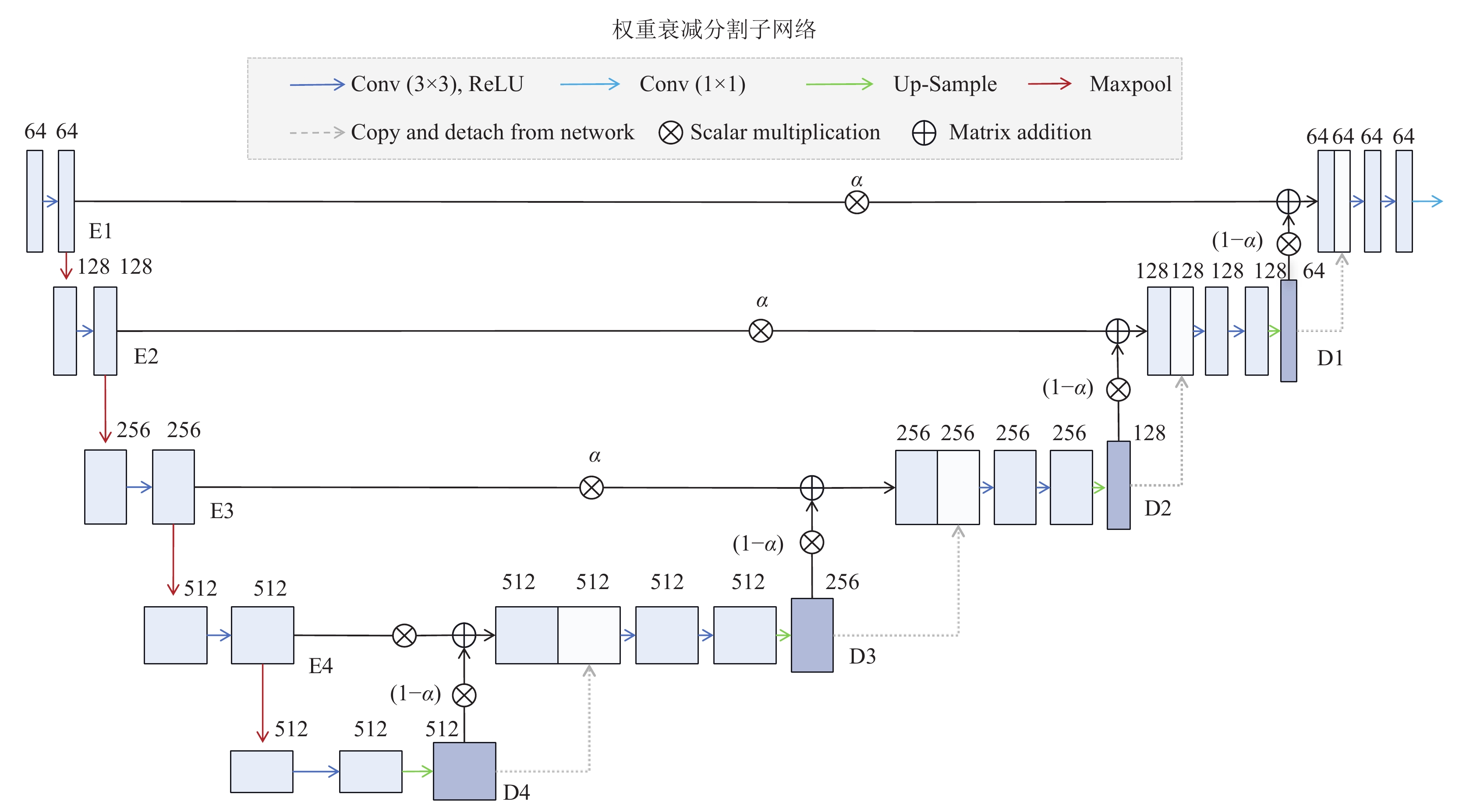
最近, U-Net架构通过在编码器和解码器之间引入跳跃链接来弥补两者的语义差距, 在恢复目标主体的细粒度细节方面非常有效, 尤其是分割任务中[45]. 然而, 跳跃链接可能会让模型通过“捷径”绕开低层次的特征而学习到恒等映射, 这将影响模型对肝部肿瘤检测的性能. 基于这种困境, 受到文献[45]启发, 本文在重建子网络后引入了一个基于权重衰减的分割子网络, 在网络训练过程中逐步减弱U-Net的跳跃链接从而减少前期跳跃链接带来的影响, 以对异常图像的识别与定位. 具体结构如图4所示.
首先, 将重建子网络所输出的重建图像
| Mi=(α⊗Ei)⊕((1−α)⊗Di) | (7) |
其中,
其次, 对
| D′i=Concat(Mi,ˉDi) | (8) |
其中,
在该模型训练阶段, 式(6)中的权重因子
最后, 在该任务中使用
| L1=‖M−M0‖1 | (9) |
| Lf=−αt(1−ρt)γlog(ρt) | (10) |
其中,
| Lseg=L1+Lf | (11) |
结合式(5)和式(10), 整个网络模型训练目标如下:
| Lall=Lseg+Lrec(I,Ir) | (12) |
|
图 4 权重衰减分割子网络架构图 |
2.6 测试阶段
如图5所示, 与训练阶段不同, 测试时不再采用数据增强方法生成异常图像, 而是直接将测试图像作为模型的输入图像. 具体来说, 将测试图像提取高频成分信息后输入到重建网络以得到重建正常图像, 最后由权重衰减分割子网络预测掩码以定位异常区域. 在异常分数计算中, 输出的预测掩码图像
| Score(M0)=max | (13) |
其中,
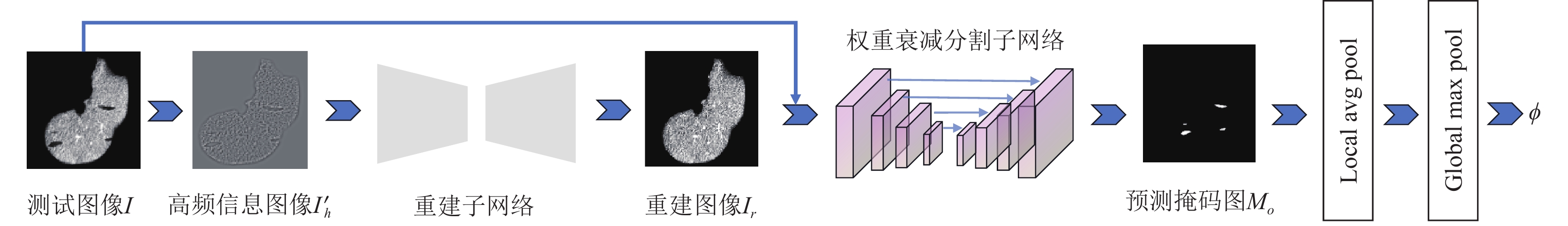
|
图 5 测试阶段图 |
3 实验分析
实验将从使用数据集、实施方案、对比方法的介绍、性能对比、消融实验5部分展开.
3.1 数据集本文使用由文献[12]提供的数据集测试所提出方法, 该数据由肝肿瘤的异常分割数据集LiTS[47]和无异常的腹部CT多器官分割数据集BTCV[48]所组成. 其中, LiTS来自MICCAI 2017挑战赛, 包含131个患者的腹部CT扫描. BTCV则来自MICCAI 2015挑战赛, 是腹部CT多器官分割的基准数据集. 具体而言, 对于这两个数据集, 3D扫描的Hounsfield单位(HU)被转换为以腹部窗口为基础的灰度图像. 然后, 将扫描切割为2D轴向切片, 并根据提供的器官注释提取出肝脏的感兴趣区域. 如表1所示,
| 表 1 数据统计表 |
3.2 实验实施方案
在实验中, 方法使用Python 3.8和PyTorch 1.9.0进行实现, 所有实验都在带有NVIDIA Tesla V100 GPU的Ubuntu 18.04 LTS操作系统上进行训练. 除了极个别网络在前期就呈现收敛因此单独设置训练批次外, 其余对比方法均设置训练批次为230个epoch. 所提出方法的学习率设置为
本文将通过与多个先进的无监督学习算法和自监督学习算法的比较以证明所提出方法的有效性. 其中, RD++[10], RD[9], SQUID[49], DRAEM[21], WDMT[45]为无监督学习方法, CutPaste[36], FPI[23], PII[22], NSA[20], FARI[30]为自监督学习方法. 对比方法的详细介绍如下.
RD: 该方法是一个新的T-S模型, 由一个教师编码器和一个学生解码器组成, 并相应地引入了一个简单而有效的“反向蒸馏”范式. 其中, 学生网络不直接接收原始图像, 而是将教师模型的单类嵌入作为输入和目标并恢复教师的多尺度表示.
RD++: 该方法在RD的基础加以改进, 通过加入伪异常机制和多尺度投影层以联合多个损失共同训练, 提升了模型在基准数据上的性能.
SQUID: 该方法是一种用于放射成像图像修复和异常检测的空间感知内存队列方法. 该方法将根深蒂固的解剖结构分类为重复模式, 并在推理中识别图像中的异常(未见过/修改的模式).
DRAEM: 该方法通过重建网络和分割网络的联合训练, 使得模型学习异常图像及其无异常重建的联合表示, 同时学习正常和异常样本之间的决策边界.
WDMT: 该方法是一种权重衰减训练策略, 通过逐步禁用U-Net的跳跃链接, 从而有效地将U形网络运用于异常检测任务.
CutPaste: 该方法是一种专门针对工业场景的异常检测方法. 它通过从正常图像中切割一部分并将其粘贴到同一图像的另一个位置来生成异常图像.
FPI/PII: 这两种方法通过在图像随机补丁中合成细微的缺陷, 从而利用足够的伪异常图像训练模型区分合成伪异常和正常图像. PII则在FPI的基础上, 引入泊松图像编辑使合成的异常更加自然真实, 从而提升模型的检测精度.
NSA: 该方法将Poisson图像编辑与缩放、平移和基于Gamma分布的补丁形状采样策略相结合, 以合成自然且多样化的异常.
FAIR: 该方法通过利用正常和异常重建误差之间的不同频率偏差, 以更好地缓解重建保真度和异常重建可区分性的权衡问题.
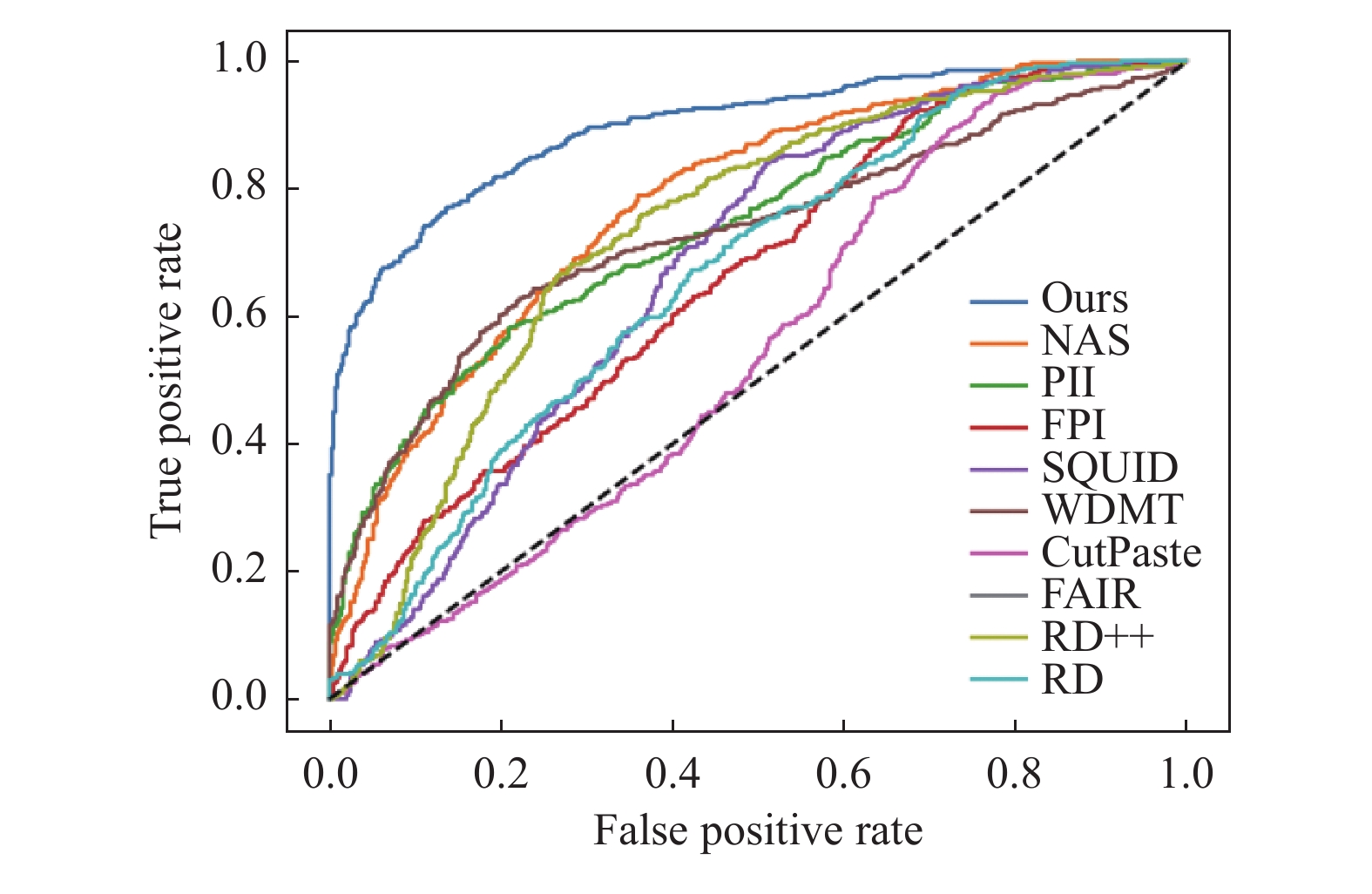
3.4 与先进方法的性能对比肿瘤检测的性能对比: 首先, 将所提出的方法与基于无监督学习的异常检测方法进行对比. 表2定量地展示了方法在检测肿瘤的优越性, 其中最佳结果用粗体表示, 并且在图6中绘制了该曲线图, 较好的结果倾向于向左上角弯曲. 结果显示, 所提出方法的Image AUROC为89.6%, 分别超过了当前最先进的方法WDMT和DRAEM 16%和12%的性能. 其次, 所提出方法相比于RD++, RD, SQUID的Image AUROC均有较大提升. 这是由于肝部异常图像和正常图像在视觉上相差无几, 未针对性设计的无监督方法可能陷入过拟合的困境而无法有效区分正常和异常的边界.
其次, 与自监督学习方法对比中, 所提出的方法分别比最先进的方法NSA, FAIR高了11%和15% Image AUROC, 比FPI, PII分别高了22%和14%. 其中, 导致FPI检测性能低下的主要原因是, 该方法过拟合了所合成的异常, 而所提出方法通过精心设计可以避免此类情况. 此外, CutPaste通过简单的粘贴复制以合成异常的方法, 可能产生与真实异常样本分布差异较大的合成异常, 这同样导致方法无法很好拟合真实图像分布.
| 表 2 与先进算法比较结果 (%) |
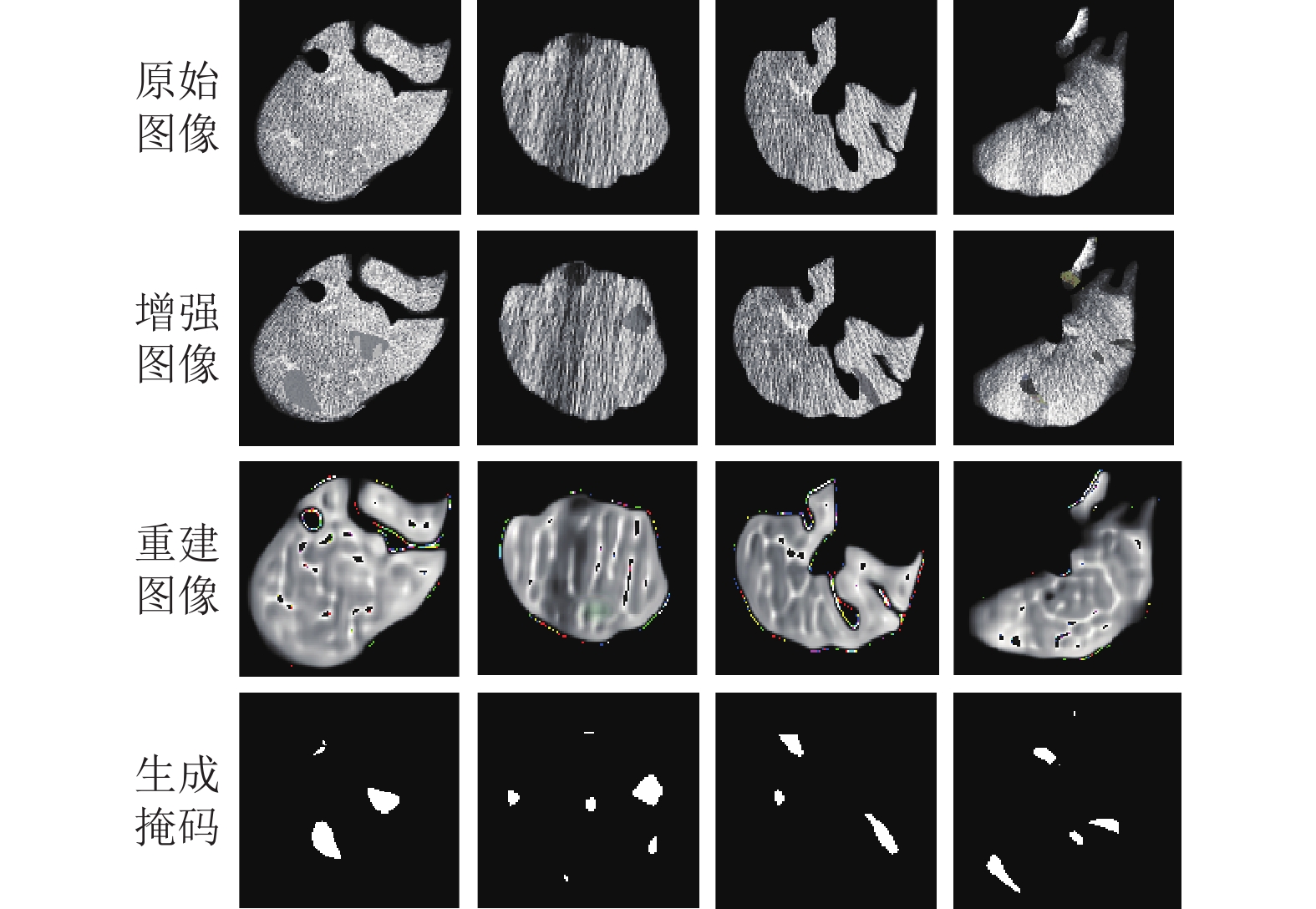
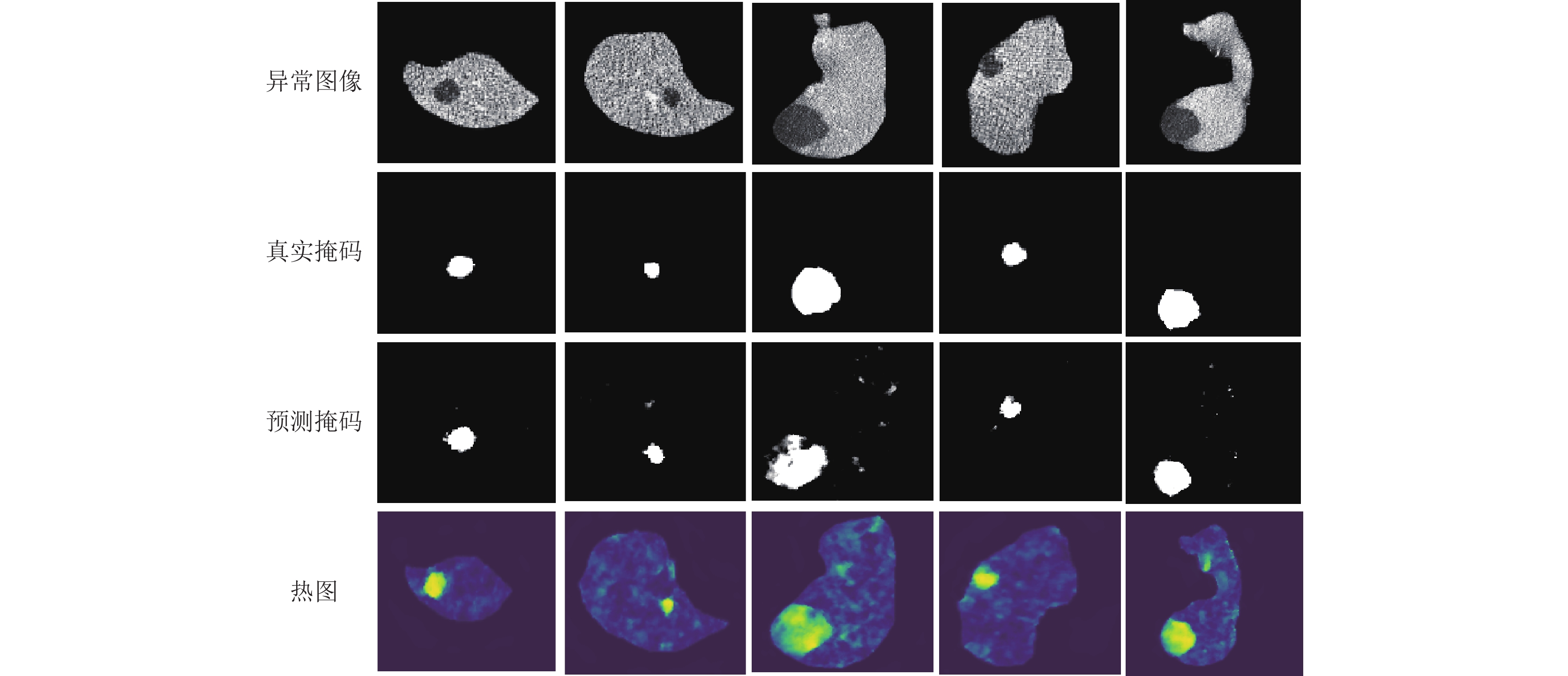
肿瘤定位的性能对比: 进一步地, 在表2中使用Pixel AUROC评估所提出方法的肿瘤定位性能. 所提出方法能够获得99.1%的Pixel AUROC, 这一结果定量地展示了所提出方法在肿瘤定位性能上的优越性. 具体来说, 所提出的方法的定位性能超过了当前最优RD方法1.3%, 并与最优的自监督学习方法FAIR性能相当. 另外, 本文还通过图7数据增强效果图和图8推理实例图分别展示了一些本文生成异常图像的肿瘤图和推理结果.
|
图 6 方法在LiTS与BTCV数据集的ROC曲线下面积结果图 |
|
图 7 数据增强效果图 |
|
图 8 推理实例图 |
值得注意的是, Image AUROC和Pixel AUROC在所有方法上都存在较大的数值差异. 这是因为两个指在肝部CT测试基准当中, 背景的大部分像素由黑色组成, 加之正常和异常样本分布不均衡, 因此数值存在偏差. 这同样也是各类方法在像素级定位上指标相差不多, 而图像级差距大的原因之一. 尽管如此, 本文方法都取得了先进的结果.
3.5 消融实验为了进一步验证方法的有效性, 本文开展了广泛的消融实验. 实验分别从模型架构的评估、合成异常模式选择两部分展开. 模型架构评估在于验证模型框架是否合理, 合成异常模式的选择在于认证异常合成方法是否有效.
模型架构的评估: 为了评估整体模型的有效性, 将单独训练重建子网络(Re.SNet)和分割子网络(WD.SSNet.), 并与完整模型架构(Ours)做对比.
首先对于重建子网络的单独训练, 将重建子网络按照基于自动编码器的异常检测范式进行训练. 在测试时, 将重建子网络的输出与输入图像使用最近提出的MS-GMS函数[32]进行比较, 生成异常图. 最后结果显示在表3中的实验RE-MS (仅单独训练重建子网络)中. 与完整方法相比, 其异常检测性能大幅下降, 定位性能也有所降低. 从结果定量的分析, 单独训练重建子网络时异常的检测能低下的原因在于网络无法很好地将图像进行重建, 重建图像在图7中显示. 由图7可知重建子网络输出图像尽管能丢失异常信息, 但是图像许多正常信息也一并丢失, 从而输出的图像缺少许多细节. 然而, 采取将重建图像和合成异常图像进行合并输入到分割子网络中, 这些丢失的信息将被弥补, 这也是完整架构有效的原因之一.
| 表 3 模型评估消融实验结果 (%) |
其次, 单独训练分割子网络的结果在表3实验WD(仅单独训练分割子网络)中, 与完整架构相比, 检测和定位性能都有所下降, 其原因可能由于权重衰减分割子网络对生成的异常图像过拟合, 而生成的异常并不能完全代表真实异常.
合成异常模式选择: 在确定方法完整架构有效性之后, 为了进一步探究合成异常方法对最后性能的影响, 将合成异常方法分为两部分进行消融, 其一为肝部的定位(Local), 其二为是否提取图像高频成分以破坏图像(Break). 在完整模型架构(REWD)下进行实验. 并于完整方法做对比(Ours), 其结果如表4所示.
| 表 4 合成异常模型的消融实验结果 (%) |
首先在合成异常中, 当仅采取肝部定位时, 其实验结果如表4 REWD-L (完整模型架构下仅对肝部进行定位)所示, 结果表明与完整的方法相比性能有所下降. 这印证了完整方法的有效性.下降的原因在于, 重建网络有时将异常信息进行重建导致模型在测试时的误判.
此外, 在仅采取提取图像高频成分时, 其实验结果在表4 REWD-B (完整模型架构下不采用定位方法仅对图像提取高频)中. 不难发现, 在和完整方法相比时检测性能和定位性能有了大幅度的下降, 造成其结果的主要原因在于如果直接将Perlin噪声生成的掩码作为最终合成异常掩码图时, 难免会在背景区域创建异常, 增加了合成异常数据分布和真实异常数据分布上的差异.
为了进一步验证本文合成异常方法的有效性, 将合成异常方法修改为和DRAEM一样的形式(不采用肝部定位和提取高频模块), 其实验结果在表4 REWD中, 与完整方法做对比, 检测性能和定位性能都有所下降. 定量验证了本文合成异常方法的有效性. 此外, 对于不采取合成异常方法训练模型, 实验结果如表5 REWD-NOs (剔除了本文合成异常方法)所示, 通过实验结果可以观察到, 模型已经退化到做出随机预测, 定量验证了合成异常方法对本文方法的重要性.
| 表 5 合成异常方法删除的实验结果 (%) |
4 结论与展望
在本文中, 为增强算法细微病灶检测能力, 首先, 设计了适合肝部肿瘤检测的数据增强自监督策略以激励模型学习正常样本的聚类边界, 解决了异常数据难以收集地挑战, 模型对合成异常拟合的问题. 此外, 引入了提取图像高频信息模块以抑制重建网络中对异常的敏感性. 最后采用权重衰减分割策略, 训练结果能比较准确的分割出病灶区域. 与其他方法相比, 本文方法取得了领先的性能, 但是在训练阶段相对于其他方法所花时间成本要多, 模型参数量比较大. 未来, 将探索更多方法以更一致的方式优化训练.
| [1] |
Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 |
| [2] |
Hann LE, Winston CB, Brown KT, et al. Diagnostic imaging approaches and relationship to hepatobiliary cancer staging and therapy. Seminars in Surgical Oncology, 2000, 19(2): 94-115. DOI:10.1002/1098-2388(200009)19:2<94::AID-SSU3>3.0.CO;2-X |
| [3] |
Heimann T, Van Ginneken B, Styner MA, et al. Comparison and evaluation of methods for liver segmentation from CT datasets. IEEE Transactions on Medical Imaging, 2009, 28(8): 1251-1265. DOI:10.1109/TMI.2009.2013851 |
| [4] |
黄泳嘉, 史再峰, 王仲琦, 等. 基于混合损失函数的改进型U-Net肝部医学影像分割方法. 激光与光电子学进展, 2020, 57(22): 221003. |
| [5] |
Yan K, Tang YB, Peng YF, et al. MULAN: Multitask universal lesion analysis network for joint lesion detection, tagging, and segmentation. Proceedings of the 22nd International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI). Shenzhen: Springer, 2019. 194–202. [doi: 10.1007/978-3-030-32226-7_22]
|
| [6] |
Araújo JDL, Da Cruz LB, Ferreira JL, et al. An automatic method for segmentation of liver lesions in computed tomography images using deep neural networks. Expert Systems with Applications, 2021, 180: 115064. DOI:10.1016/j.eswa.2021.115064 |
| [7] |
马力, 王骏, 梁羡和, 等. 基于改进UNETR++的肝脏分割. 计算机系统应用, 2024, 33(2): 246-252. DOI:10.15888/j.cnki.csa.009414 |
| [8] |
张颖, 仇大伟, 刘静. 生成对抗网络在肝脏肿瘤图像分割中的应用综述. 计算机工程与应用, 2022, 58(16): 18-30. DOI:10.3778/j.issn.1002-8331.2203-0229 |
| [9] |
Deng HQ, Li XY. Anomaly detection via reverse distillation from one-class embedding. Proceedings of the 2022 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). New Orleans: IEEE, 2022. 9727–9736. [doi: 10.1109/CVPR52688.2022.00951]
|
| [10] |
Tien TD, Nguyen AT, Tran NH, et al. Revisiting reverse distillation for anomaly detection. Proceedings of the 2023 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Vancouver: IEEE, 2023. 24511–24520. [doi: 10.1109/CVPR52729.2023.02348]
|
| [11] |
Roth K, Pemula L, Zepeda J, et al. Towards total recall in industrial anomaly detection. Proceedings of the 2022 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). New Orleans: IEEE, 2022. 14298–14308. [doi: 10.1109/CVPR52688.2022.01392]
|
| [12] |
Bao JA, Sun HS, Deng HQ, et al. BMAD: Benchmarks for medical anomaly detection. arXiv:2306.11876, 2024.
|
| [13] |
Gong D, Liu LQ, Le V, et al. Memorizing normality to detect anomaly: Memory-augmented deep autoencoder for unsupervised anomaly detection. Proceedings of the 2019 IEEE/CVF International Conference on Computer Vision. Seoul: IEEE, 2019. 1705–1714. [doi: 10.1109/ICCV.2019.00179]
|
| [14] |
Hou JL, Zhang YY, Zhong QY, et al. Divide-and-assemble: Learning block-wise memory for unsupervised anomaly detection. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 8771–8780. [doi: 10.1109/ICCV48922.2021.00867]
|
| [15] |
Wu K, Zhu L, Shi WH, et al. Self-attention memory-augmented wavelet-CNN for anomaly detection. IEEE Transactions on Circuits and Systems for Video Technology, 2023, 33(3): 1374-1385. DOI:10.1109/TCSVT.2022.3211839 |
| [16] |
Bae J, Lee JH, Kim S. PNI: Industrial anomaly detection using position and neighborhood information. Proceedings of the 2023 IEEE/CVF International Conference on Computer Vision. Paris: IEEE, 2023. 6350–6360. [doi: 10.1109/ICCV51070.2023.00586]
|
| [17] |
Bergmann P, Fauser M, Sattlegger D, et al. MVTec AD—A comprehensive real-world dataset for unsupervised anomaly detection. Proceedings of the 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Long Beach: IEEE, 2019. 9584–9592. [doi: 10.1109/CVPR.2019.00982]
|
| [18] |
Ben-Cohen A, Klang E, Kerpel A, et al. Fully convolutional network and sparsity-based dictionary learning for liver lesion detection in CT examinations. Neurocomputing, 2018, 275: 1585-1594. DOI:10.1016/j.neucom.2017.10.001 |
| [19] |
Schlegl T, Seeböck P, Waldstein SM, et al. f-AnoGAN: Fast unsupervised anomaly detection with generative adversarial networks. Medical Image Analysis, 2019, 54: 30-44. DOI:10.1016/j.media.2019.01.010 |
| [20] |
Schlüter HM, Tan J, Hou B, et al. Natural synthetic anomalies for self-supervised anomaly detection and localization. Proceedings of the 17th European Conference on Computer Vision. Tel Aviv: Springer, 2022. 474–489.
|
| [21] |
Zavrtanik V, Kristan M, Skočaj D. DRAEM—A discriminatively trained reconstruction embedding for surface anomaly detection. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision (ICCV). Montreal: IEEE, 2021. 8310–8319.
|
| [22] |
Tan J, Hou B, Day T, et al. Detecting outliers with Poisson image interpolation. Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI). Strasbourg: Springer, 2021. 581–591.
|
| [23] |
Tan J, Hou B, Batten J, et al. Detecting outliers with foreign patch interpolation. arXiv:2011.04197, 2022.
|
| [24] |
Ji W, Yu S, Wu JD, et al. Learning calibrated medical image segmentation via multi-rater agreement modeling. Proceedings of the 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Nashville: IEEE, 2021. 12336–12346. [doi: 10.1109/CVPR46437.2021.01216]
|
| [25] |
Schlegl T, Seeböck P, Waldstein SM, et al. Unsupervised anomaly detection with generative adversarial networks to guide marker discovery. Proceedings of the 25th International Conference on Information Processing in Medical Imaging. Boone: Springer, 2017. 146–157.
|
| [26] |
Li H, Iwamoto Y, Han XH, et al. An accurate unsupervised liver lesion detection method using pseudo-lesions. Proceedings of the 25th International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI). Singapore: Springer, 2022. 214–223.
|
| [27] |
Frid-Adar M, Diamant I, Klang E, et al. GAN-based synthetic medical image augmentation for increased CNN performance in liver lesion classification. Neurocomputing, 2018, 321: 321-331. DOI:10.1016/j.neucom.2018.09.013 |
| [28] |
Collin AS, De Vleeschouwer C. Improved anomaly detection by training an autoencoder with skip connections on images corrupted with stain-shaped noise. Proceedings of the 2021 International Conference on Pattern Recognition (ICPR). Milan: IEEE, 2021. 7915–7922.
|
| [29] |
Zavrtanik V, Kristan M, Skočaj D. DSR—A dual subspace re-projection network for surface anomaly detection. Proceedings of the 17th European Conference on Computer Vision. Tel Aviv: Springer, 2022. 539–554. [doi: 10.1007/978-3-031-19821-2_31]
|
| [30] |
Liu TK, Li B, Du X, et al. FAIR: Frequency-aware image restoration for industrial visual anomaly detection. arXiv:2309.07068, 2023.
|
| [31] |
Pirnay J, Chai K. Inpainting transformer for anomaly detection. Proceedings of the 21st International Conference on Image Analysis and Processing. Lecce: Springer, 2022. 394–406.
|
| [32] |
Zavrtanik V, Kristan M, Skočaj D. Reconstruction by inpainting for visual anomaly detection. Pattern Recognition, 2021, 112: 107706. DOI:10.1016/j.patcog.2020.107706 |
| [33] |
Chen T, Kornblith S, Norouzi M, et al. A simple framework for contrastive learning of visual representations. Proceedings of the 37th International Conference on Machine Learning. ICML, 2020. 1597–1607.
|
| [34] |
Dosovitskiy A, Springenberg JT, Riedmiller M, et al. Discriminative unsupervised feature learning with convolutional neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. Montreal: ACM, 2014. 766–774.
|
| [35] |
Golan I, El-Yaniv R. Deep anomaly detection using geometric transformations. Proceedings of the 32nd International Conference on Neural Information Processing Systems. Montréal: ACM, 2018. 9781–9791.
|
| [36] |
Li CL, Sohn K, Yoon J, et al. CutPaste: Self-supervised learning for anomaly detection and localization. Proceedings of the 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Nashville: IEEE, 2021. 9659–9669. [doi: 10.1109/CVPR46437.2021.00954]
|
| [37] |
Huang WK, Huang YJ, Tang XY. AugPaste: One-shot anomaly detection for medical images. Proceedings of the 9th International Workshop on Ophthalmic Medical Image Analysis. Singapore: Springer, 2022. 1–11.
|
| [38] |
Meissen F, Kaissis G, Rueckert D. AutoSeg—Steering the inductive biases for automatic pathology segmentation. Biomedical Image Registration, Domain Generalisation and Out-of-distribution Analysis. Strasbourg: Springer, 2021. 127–135. [doi: 10.1007/978-3-030-97281-3_19]
|
| [39] |
Li W, Liu GH, Fan HY, et al. Self-supervised multi-scale cropping and simple masked attentive predicting for lung CT-scan anomaly detection. IEEE Transactions on Medical Imaging, 2024, 43(1): 594-607. DOI:10.1109/TMI.2023.3313778 |
| [40] |
Tao X, Gong XY, Zhang X, et al. Deep learning for unsupervised anomaly localization in industrial images: A survey. IEEE Transactions on Instrumentation and Measurement, 2022, 71: 5018021. |
| [41] |
Perlin K. An image synthesizer. ACM SIGGRAPH Computer Graphics, 1985, 19(3): 287-296. DOI:10.1145/325165.325247 |
| [42] |
Cimpoi M, Maji S, Kokkinos I, et al. Describing textures in the wild. Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Columbus: IEEE, 2014. 3606–3613. [doi: 10.1109/CVPR.2014.461]
|
| [43] |
Cai Y, Chen H, Yang X, et al. Dual-distribution discrepancy with self-supervised refinement for anomaly detection in medical images. Medical Image Analysis, 2023, 86: 102794. DOI:10.1016/j.media.2023.102794 |
| [44] |
Bergmann P, Löwe S, Fauser M, et al. Improving unsupervised defect segmentation by applying structural similarity to autoencoders. Proceedings of the 14th International Joint Conference on Computer Vision, Imaging and Computer Graphics Theory and Applications. Prague: VISAPP, 2019. 372–380.
|
| [45] |
Zhang WT, Sun X, Li YX, et al. A multi-task network with weight decay skip connection training for anomaly detection in retinal fundus images. Proceedings of the 25th International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI). Singapore: Springer, 2022. 656–666.
|
| [46] |
Lin T Y, Goyal P, Girshick R, et al. Focal loss for dense object detection. Proceedings of the 2017 International Conference on Computer Vision. Venice: IEEE, 2017. 2999–3007.
|
| [47] |
Bilic P, Christ P, Li HB, et al. The liver tumor segmentation benchmark (LiTS). Medical Image Analysis, 2023, 84: 102680. DOI:10.1016/j.media.2022.102680 |
| [48] |
Cao H, Wang YY, Chen J, et al. Swin-UNet: UNet-like pure Transformer for medical image segmentation. Proceedings of the 2022 European Conference on Computer Vision. Tel Aviv: Springer, 2022. 205–218. [doi: 10.1007/978-3-031-25066-8_9]
|
| [49] |
Xiang TG, Zhang YX, Lu YY, et al. SQUID: Deep feature in-painting for unsupervised anomaly detection. Proceedings of the 2023 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Vancouver: IEEE, 2023. 23890–23901.
|
 2024, Vol. 33
2024, Vol. 33



