2. 山东浪潮优派科技教育有限公司, 济南 250101
2. Shandong Inspur-UPTEC Education Co. Ltd., Jinan 250101, China
皮肤癌是由异常细胞形成引起的, 这些细胞根据其性质和强度的不同, 可能会渗透或扩散到身体的不同部位[1]. 皮肤癌病变的诊断因良性和恶性黑素瘤的多种类型而变得复杂. 鳞状细胞癌、基底细胞癌和黑色素瘤是临床实践中常见的不规则皮肤细胞的主要形式[2]. 此外, 皮肤癌基金会[3]区分出3种较不常见的异常细胞类型, 即默克尔细胞癌、日光性角化病和非典型痣.
在所有类型的皮肤癌中, 黑色素瘤的发病率最高, 但如果及时诊断, 通常是可以治愈的[4]. 根据一项研究, 及时发现早期皮肤癌可以将死亡率降低90%[5]. 然而, 正确皮肤病诊断是具有挑战性的, 需要利用各种视觉线索, 如个体病变形态, 身体部位分布, 颜色, 病变的缩放和排列, 以促进诊断. 即使有丰富经验的医生在诊断皮肤癌过程中也会出现误诊和漏诊[6]. 医生诊断的局限性是它严重依赖主观判断, 并且诊断结果在不同医生之间差别很大. 相比之下, 计算机辅助诊断(CAD)系统更为客观. 现代以CAD为基础的技术已经广泛应用了机器学习. 而深度学习是机器学习的一个广泛而迅速发展的分支, 使计算机能够自动学习和发展, 而无需明确编程[7]. 该技术源于模式识别和计算学习理论的研究, 并在近几十年的大量应用中找到了一席之地.
深度学习作为目前应用最广泛的学习算法, 在图像分类、图像分割和目标检测等任务中取得了显著成就. 其中, 卷积神经网络(CNN)更是成为图像处理领域最为成功的应用之一. 近年来, 深度学习算法在癫痫、糖尿病视网膜病变、皮肤癌等疾病领域[8–10]发挥着重要作用, 通过捕捉大规模数据中的局部特征并将其放入堆叠的网络结构中进行推理, 从而实现对复杂图像的分类和识别. 在皮肤癌领域, 深度学习不仅提高了诊断的准确性和效率, 还减少了对人工标注的依赖. 通过利用大量的皮肤镜图像, 深度学习算法可以自动提取有意义的特征, 进行病变检测和分类, 大大提升了早期诊断的可能性. 此外, 深度学习模型的可解释性和自适应学习能力使其在处理皮肤癌复杂的异质性和多样性方面具有独特优势, 为临床诊断提供了重要的参考价值.
对于皮肤癌分类诊断, 国内外文献[11–14]是近期发表的几篇优秀综述, 如张杰等人[11]在他们的研究中, 从病灶分割和病灶分类两个方面对卷积神经网络在黑色素瘤检测中的应用进行了综述. 而Bhatt等人[14]总结了最先进机器学习技术在黑色素瘤皮肤癌中的研究进展. 但以上文献都未在深度学习及传统机器学习领域对全部种类的皮肤癌分类进行系统概述. 为了给皮肤癌辅助诊断相关研究领域的未来工作提供参考, 本文整理了皮肤癌分类诊断常用的数据集及评价指标, 从机器学习和深度学习的角度阐述不同的分类模型, 对比分析不同模型的特点及优劣, 最后在此基础上对本文进行总结, 针对现有技术分析在皮肤病分类中存在的问题, 并对未来的研究方向进行展望.
1 数据集及评价指标皮肤癌分类领域的数据集对于算法的训练和评估至关重要, 提供丰富的皮肤镜图像和相关临床注释, 其中包括日光性角化病、非典型痣、基底细胞癌、黑色素瘤、默克尔细胞癌和鳞状细胞癌, 如图1所示. 这些类别涵盖了从癌前病变到各种常见和罕见的皮肤癌类型, 支持皮肤癌诊断算法的研究和开发.
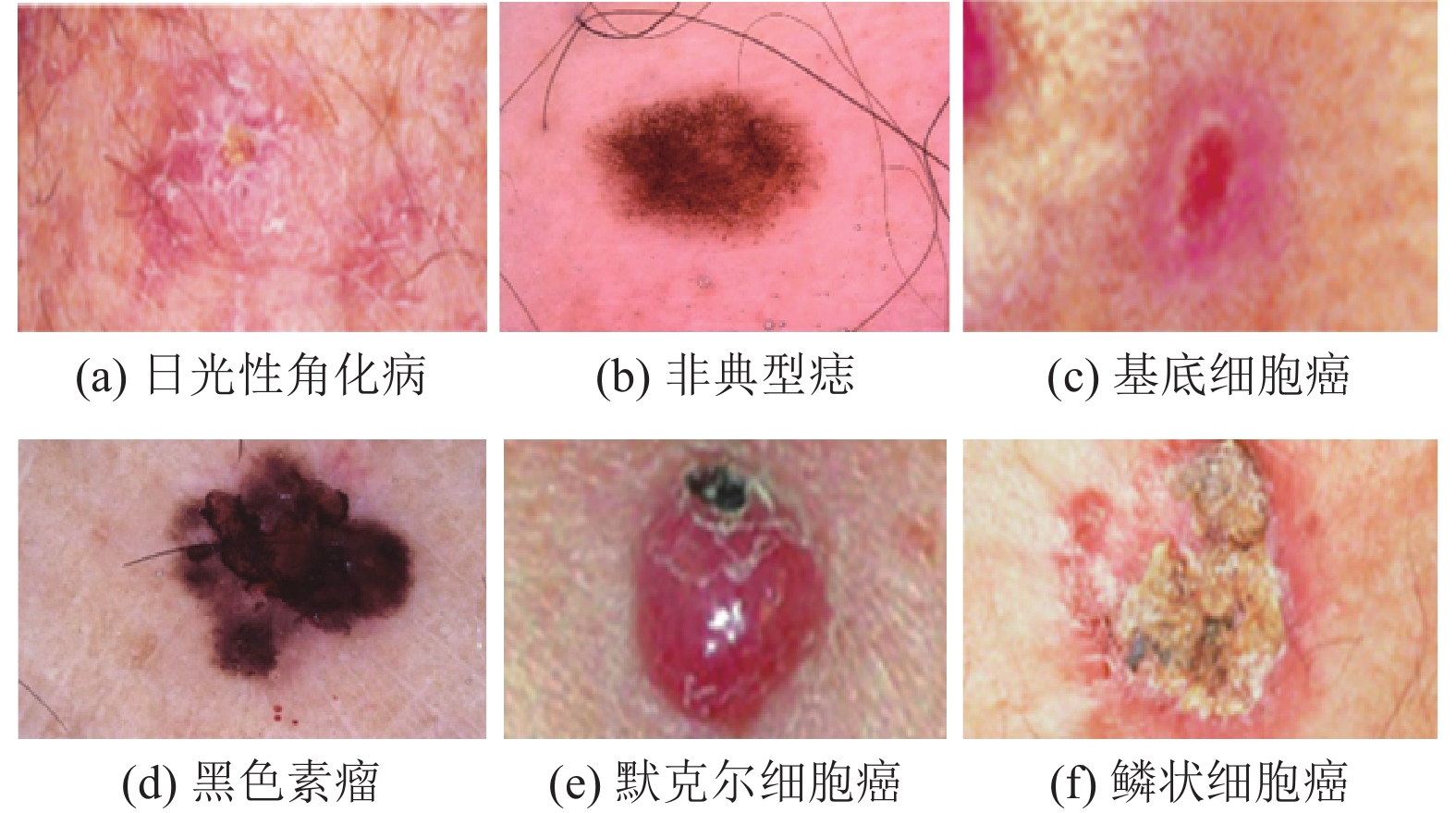
|
图 1 不同种类皮肤病 |
1.1 PH2数据集
Mendonca等人在2003年建立了公开可用的皮肤镜图像数据集PH2 [15]. 该数据集包含80个普通痣、80个非典型痣和40个黑色素瘤样本. 这些皮肤镜图像是在葡萄牙马托辛霍斯的医院皮肤科服务下以相同条件下通过Tuebinger痣分析系统获取的, 使用了20倍的放大倍数. 这些图像是8位RGB彩色图像, 分辨率为768×560像素. 数据集包括所有图像的医学注释, 包括病变的医学分割、临床和组织学诊断, 以及对几个皮肤镜标准的评估(例如颜色、色素网格、点、条纹、退行区域、蓝白色薄纱). 由于该数据集包含了全面的元数据, 因此经常被用作评估黑色素瘤诊断算法的基准数据集.
1.2 ISIC数据集国际皮肤成像协作组织聚合了一个大规模的公开可用的皮肤镜图像数据集[16]. 该数据集包含来自国际领先的临床中心的超过20 000张图像, 这些图像是通过各个中心使用的不同设备获取的. ISIC数据集首次在2016年被公开用于皮肤镜图像分析的公共基准挑战[17,18]. 挑战的目标是提供一个数据集, 促进黑色素瘤自动诊断算法在分割、皮肤镜特征检测和分类方面的发展. 2017年, ISIC举办了挑战的第2期, 同时扩展了数据集. 扩展数据集提供了2 000张用于训练的图像, 其中包括分割的蒙版、用于皮肤镜特征提取的超像素蒙版和用于分类的注释[19]. 这些图像被分为3类, 即黑色素瘤、脂溢性角化病和痣. 黑色素瘤是一种恶性皮肤肿瘤, 而其他两种是来源于不同细胞的良性皮肤肿瘤. 此外, ISIC提供了一个额外的验证集, 包含额外的150张图像用于评估.
1.3 HAM10000数据集由Tschandl等人发布的HAM10000数据集[20]包括不同人群的皮肤镜图像, 这些图像通过不同的模式获取和存储. 该数据集通过ISIC存档公开提供, 包含10 015张皮肤镜图像. 病例包括在色素病变领域中所有重要诊断类别的代表性集合. 所有黑色素瘤的诊断均通过组织学检查的活检进行验证, 而痣的诊断则通过组织学检查(24%)、专家共识(54%)或其他诊断方法(例如一系列未显示时间变化的图像, 占22%)进行.
1.4 Dermofit数据集Dermofit数据集[21]是一个包含
MoleMap[22]是一个包含102 451张图像的数据集, 涵盖了25种皮肤病症状. 具体来说, 癌性病变包括黑色素瘤、基底细胞癌和鳞状细胞癌. 每个病变都有两张图像: 一张是距离病变10 cm远拍摄的特写图像, 另一张是病变的皮肤镜图像. 图像的选择基于以下4个标准: 1)每张图像都有特定的疾病诊断(例如, 蓝色痣); 2)至少有100张具有相同诊断的图像; 3)图像质量可接受(例如, 对比度良好); 4)病变占据了图像大部分区域, 周围组织较少.
1.6 Dermnet数据集Dermnet[23]是最大的独立皮肤科照片资源, 致力于通过文章、照片和视频进行在线医学教育. Dermnet通过新媒体提供各种皮肤病状的信息. 它包含超过23 000张皮肤病图像. 用户可以通过点击放大图像, 并通过浏览图像类别或使用搜索引擎来定位图像. 这些图像和视频可以免费获取, 用户还可以购买高分辨率图像的副本用于出版目的. 表1总结了常用的皮肤癌数据集.
| 表 1 皮肤病分类常用数据集 |
1.7 评价指标
皮肤癌分类任务的评价指标包括灵敏度、特异度、准确率、F1分数、召回率以及ROC曲线下的AUC值. 表2是关于皮肤癌分类的评价指标. 灵敏度和特异度分别衡量了分类器在检测阳性和阴性样本方面的能力, 准确率反映了分类器正确分类的整体比例. F1分数综合了灵敏度和精确度, 对分类器综合性能进行评估. 召回率衡量了分类器对阳性样本的检测能力. ROC曲线和AUC值提供了分类器在不同阈值下的性能评估, 并可比较不同分类器的性能. 这些评价指标有助于确定皮肤癌分类模型的准确性、可靠性和适用性, 为临床诊断提供重要的参考依据.
| 表 2 分类模型的评价指标 |
2 机器学习算法在皮肤癌分类中的应用
在皮肤癌分类领域, 机器学习算法依然具有一定的参考使用价值. K最近邻算法(K-nearest neighbor, KNN)和支持向量机(support vector machine, SVM)是两种备受关注的机器学习方法. KNN以其简单的原理和对数据分布假设的免疫性而闻名, 适用于分类和回归问题, 并且在处理异常值时表现出较强的稳健性. 相比之下, SVM通过寻找最优超平面来分隔不同类别的皮肤病, 尤其在处理多种特征类型和小数据集时表现突出. 接下来, 我们将详细介绍它们在皮肤癌分类中的应用及其优劣势. 表3是对本节方法的总结.
| 表 3 基于机器学习的皮肤病分类诊断算法比较 |
2.1 KNN
KNN作为一种惰性学习算法, 它的基本原理简单, 能够处理分类和回归问题, 并且不需要对数据进行假设, 因而对异常值不敏感. 这些特性使得KNN在皮肤癌分类领域具有广泛的应用潜力. 然而, KNN也存在一些挑战, 如目标区域的模糊边界和特征提取的困难.
Hatem[24]通过实施KNN方法来区分正常皮肤和恶性皮肤病变, 取得了良好的分类准确率. 随后, Pal[25]将KNN与随机森林(random forest, RF)算法进行比较, 发现KNN在准确率和F1分数方面均优于RF. 这表明KNN在分类任务中具有显著的优势. 在处理模糊边界的目标区域时, Abbes等人[26]引入了模糊决策本体论, 以提高KNN模型的性能. 通过生成推理过程的痕迹, 他们最大程度地还原边界框架, 改进了诊断过程的模拟效果. 这一方法增强了KNN在处理模糊边界数据时的能力. 由于KNN的非参数特性, 难以将其与特征提取器有效结合. 为此, Zhuang等人[27]在特征提取器的学习过程中, 强制每个训练样本与其K个近邻属于同一类. 在处理多类和类不平衡医学图像数据集的实验中, 深度KNN显示出优于传统KNN的性能. 这种改进提升了KNN在复杂数据集上的分类效果.
综上所述, KNN系统存在精度不高和无法与特征提取器学习相结合的问题. 一些研究通过引入本体论和端到端学习策略来提高KNN模型的性能, 但KNN的非参数特性仍然限制了其在特征提取器学习中的有效应用. 虽然KNN能够有效区分皮肤癌病变, 但在处理大型和复杂数据集时可能存在计算成本高和模型泛化能力不足的挑战.
2.2 SVMSVM通过寻找最优的超平面来有效分离不同类别的皮肤病[34]. SVM不仅适用于线性可分的数据, 还可以通过使用径向基函数(RBF)等非线性核函数将数据映射到更高维空间, 从而实现更好的分类效果[35].
Raju等人[28]通过模糊集分割算法对皮肤损伤区域进行分割, 并提取颜色、灰度共现矩阵(GLCM)[36]纹理和形状特征, 再使用SVM-BWO算法进行分类. 尽管取得了较高的分类准确率, 但该方法存在过拟合和鲁棒性不足的问题. 为解决过拟合问题, Balamurugan等人[29]利用GLCM提取图像纹理和颜色特征, 并将这些特征输入SVM分类器, 他们显著提高了系统的鲁棒性, 减少了过拟合的风险. 在这个基础上, Hasanah等人[30]将GLCM方法和多类SVM算法结合, 提取纹理特征中的对比度、相关性、能量和同质性值, 再通过多类SVM进行分类, 这种改进不仅提高了分类准确率, 还进一步降低了模型的过拟合程度. 这些研究展示了多特征提取方法在增强SVM分类性能方面的潜力. 提高了分类准确率并降低了过拟合.
在降低诊断的计算成本方面, Nawar等人[31]提出了一种基于SVM的皮肤病紧急识别系统. 他们在系统中结合了两个特征集, 应用GLCM算法和SVM分类器, 成功识别了8种皮肤病, 准确率达到94.79%. 这种方法有效减少了计算成本, 提高了识别效率, 展示了SVM在实际应用中的可行性.
此外, Arora等人[32]将特征袋法与加速鲁棒特征(SURF)结合, 通过二次SVM进行分类, 将病变精确分类为癌症或非癌症. 与其他机器学习方法相比, 这一方法将性能精度提高了3%. 这表明, 精确的特征提取技术对于提高分类效果至关重要. 随着技术的发展和数据集的不断扩大, 传统SVM局限性逐渐显现. Lingaraj等人[33]提出了一种新的分类器真实支持向量机(VSVM). VSVM通过在监督学习下训练, 利用高阶特征向量对医学图像的原始像素进行分类, 基于医学图像历史信息提取典型特征, 构建能够组合不同特征组的有效模型. VSVM不仅利用医学图像的原始像素构造最佳分类器, 还进一步提升了分类效果, 展示了其在处理复杂数据集时的优势.
综上所述, SVM在皮肤癌分类中的优势在于能够处理多种特征类型, 并且在小数据集上表现良好, 具有较高的准确率. 然而, SVM在处理大规模数据时的计算成本较高, 且对参数的敏感性较强, 需要仔细调整以避免过拟合.
3 深度学习算法在皮肤癌分类中的应用近年来, 深度学习在皮肤癌分类任务中的应用取得了显著进展. 针对这一领域的挑战, 研究者们开发了多种先进的神经网络架构, 包括卷积神经网络(convolutional neural network, CNN)、循环神经网络(recurrent neural network, RNN)、生成对抗网络(generative adversarial network, GAN)和Transformer. 这些方法各自发挥着独特的优势, 通过优化网络结构和引入创新算法, 有效提升了皮肤癌分类的准确性和泛化能力. 本文将深入探讨这些深度学习方法在皮肤癌分类中的具体应用及其关键技术创新点. 表4总结了本节的技术与方法.
| 表 4 基于深度学习的皮肤病分类诊断算法比较 |
3.1 CNN
CNN是一种深度学习技术和一类人工神经网络, 被广泛应用于皮肤癌分类任务中. 与传统的机器学习算法相比, CNN在皮肤癌分类任务中表现出卓越的性能, 能够有效地学习训练集中的特征, 并用于区分测试集中的不同皮肤癌类别[75]. CNN通常由卷积层、池化层和全连接层等组成, 这些层依次出现, 构成了网络的基本结构[76].
减少参数和降低计算成本是CNN在实际应用中的关键挑战之一. Allugunti[37]结合数学专业知识, 开发了一个基于密集卷积网络的模型, 显著减少了参数计算量的同时, 准确率达到了86.6%. 同样的, 张荣梅等人[38]通过将ResNet50中残差网络结构的卷积层、池化层用Inception结构代替, 在减少模型的训练参数数量, 降低时间复杂度的同时, 也提升了识别准确率. 这两种方法都通过优化网络结构, 实现了参数的高效利用.
与此同时, Maduranga等人[39]开发了一个实现CNN模型的Android移动应用程序, 使用MobileNet[77]预训练网络进行微调. 虽然该模型在检测受影响区域方面表现出色, 但仍然存在数据集局限的问题, 只能识别7种类型的皮肤病. 为解决这一问题, Srinivasu等人[40]设计了一种结合MobileNet V2和LSTM的方法, 使得模型能够识别更多种类的皮肤癌, 并取得较高的分类准确率.
在提高模型准确性方面, 引入注意力机制和优化超参数是有效的途径. Karthik等人[41]在CNN检测皮肤疾病的模型中引入高效通道注意模块, 构建Eff2Net模型, 通过ECA块替代标准挤压和激励块. 模型流程图如图2所示. 王师玮等人[42]在ResNet50的基础上对模型进行改进, 并引入轻量型注意力模块SimAM, 该模块使模型更好的聚焦病变区域提炼关键特征, 因此可以将准确率提高到90.95%. 然而, 单纯引入注意力机制尚未表现出对模型准确性的显著增益.
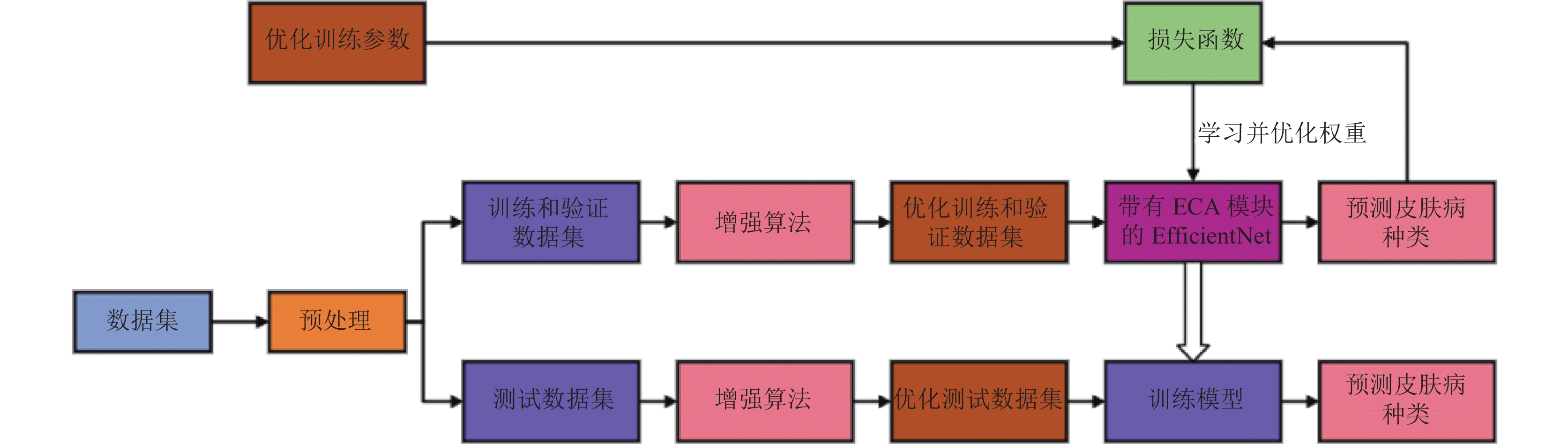
|
图 2 EfficientNetv2-ECA模型总体流程 |
相比之下, 通过调整超参数来优化模型效果的策略显得更为有效. Junayed等人[43]提出一个深度CNN模型, 通过超参数优化, 取得了比预训练模型GoogLeNet[78]和MobileNet更高的准确率. 为了验证不同优化器对分类的影响, Fu’adah等人[44]通过比较不同优化器(SGD、RMSprop、Adam和Nadam)的性能, 发现Adam优化器在皮肤癌和良性肿瘤病变分类中表现最好, 分类准确度高达99%.
迁移学习技术在CNN模型的应用中发挥了重要作用. 陈明海等人[79]采用部分迁移学习技术降低了负迁移现象, 同时他们将ResNet作为特征提取模型, 丰富提取的特征, 又用Focal Loss损失函数解决数据集存在的图像类别之间数量的严重失衡的问题. 这种设计的实验结果比传统迁移学习方法有了显著提高. 此外Anand等人[45]在迁移学习的基础上改进Xception模型, 显著提升了分类效果. 迁移学习不仅提高了模型的准确性, 还减少了训练时间和计算成本.
为了增加模型的可解释性, 展现CNN与传统机器学习算法结合的明显优势. Gupta等人[46]先使用VGG16、VGG19和Inception V3[80]模型提取特征, 然后将图像传递给多个机器学习分类器, 结果显示Inception V3与NN分类器的组合效果最佳. Hameed等人[47]使用AlexNET提取特征, 并将结合线性纠错输出码技术的SVM分类器应用于特征分类. 但由于AlexNET[81]模型结构简单, 无法准确捕捉到更复杂的特征, 所以Janoria等人[48]用VGG16模型提取特征, 结合KNN分类器进行分类, 进一步提升了分类精度, 达到了97.63%的准确度.
综上所述, CNN在皮肤癌分类中起到了关键作用, 通过结合迁移学习、注意力机制等方法, 提高了模型的准确性和性能. 同时, 将CNN与机器学习算法结合, 如使用VGG、Inception等模型提取特征, 并通过SVM、KNN等分类器进行分类, 进一步提升了分类精度. 它是皮肤癌分类领域中应用最为广泛的网络模型, 其优点如下: (1) CNN的模型结构可以根据任务的需要进行灵活设计和调整. 可以通过增加卷积层、池化层或调整全连接层的数量和结构来改变模型的复杂度和性能, 从而更好地适应不同的皮肤癌分类任务. (2)由于CNN能够学习到抽象的特征表示, 因此在训练集之外的数据上通常具有较好的泛化能力. 这对于皮肤癌分类任务来说至关重要. (3) CNN能够自动从原始图像数据中学习特征, 而不需要手动设计特征提取器. 这使得CNN在处理皮肤镜图像等复杂数据时尤其有优势. (4) CNN可以进行端到端的学习, 即直接从原始数据到最终的输出结果, 无需人工提取特征或设计复杂的流程. (5) CNN在设计上具有平移不变性, 即无论目标在图像中的位置如何变化, CNN都能够识别出相同的特征. 这使得CNN在处理皮肤癌图像时能够应对不同的图像变换和干扰. 但也存在以下问题: (1) CNN通常需要大量的标记数据进行训练, 特别是在处理复杂的皮肤病分类任务时. (2) CNN作为深度学习模型, 通常被认为是黑盒模型, 即难以解释其决策过程, 这可能会限制其在临床实践中的应用. (3)在训练数据不足或者标记有噪声的情况下, CNN可能会倾向于过拟合, 即在训练集上表现良好但在测试集上性能下降. (4) CNN主要关注提取和学习局部特征, 而皮肤癌分类可能需要考虑全局性质, 这导致CNN在捕获全局信息方面的不足. 因此, 继续改进CNN模型的结构和算法, 以及拓展多样化的数据集, 将有助于进一步提高皮肤癌分类的准确性和泛化能力.
3.2 RNNRNN是一种深度学习技术, 特别适用于处理序列数据或具有时间关联的数据, 因此被广泛应用于皮肤癌图像分类任务. 其循环结构能够捕捉序列中的上下文信息, 但传统RNN在处理长时间依赖问题时可能存在挑战[82]. 为此, 长短时记忆网络(LSTM)和门控循环单元(GRU)等改进结构被提出.
Senthilkumaran等人[49]提出了一种增强自适应混合RNN, 尽管其在皮肤癌分类中表现出较高的准确率, 但仍存在优化算法不足的问题. 为了提升模型性能, Dabbu等人[50]引入了深度递归神经网络, 并结合水波优化算法和原子搜索优化, 设计了一种新型水原子搜索优化算法. 这种方法有效地对皮肤癌数据集进行了降维特征分类, 显著提升了分类效果.
此外, Divya等人[51]借鉴了人类狩猎行为, 通过一系列方程式描述位置的更新和搜索行为, 迭代优化模型参数, 从而减少了实际结果和测量结果之间的误差. Shukla等人[52]则利用猫群智能生成循环神经网络来优化参数设置, 其中关键的是用维纳滤波器消除采集到的原始图像和用布尔滤波器组增强提取到的图像特征, 这样设计可以确保模型更全面的理解序列数据中的上下文信息. 然而, 使用这种技术可能需要更多的处理时间, 在处理高维特征和增强算法的收敛速度方面仍存在挑战.
为解决训练时间长和收敛速度慢的问题, Gomathi 等人[53]使用 RNN 混合束状虫群算法来优化模型的权重. 研究结果显示, 该模型的特异性分别比SVM和传统 RNN 方法提高了 22.67% 和 26.67%. 这一发现表明, 优化算法的选择对于提升模型性能具有重要作用. 在此背景下, Vinoth 等人[54] 提出了另一种基于rider chicken优化算法的 RNN 大数据分类方法. 该方法在 Spark 架构下有效选择特征, 并在主节点上实现皮肤癌分类, 成功优化了模型参数并提升了效率. 然而, 值得注意的是, 该方法在处理不同数据集和应用场景时, 容易出现梯度消失和梯度爆炸的问题, 这限制了其在大规模数据集上的应用效果.
传统RNN在处理输入数据时常面临梯度消失或爆炸的问题. Yunandar等人[55]利用进化的RNN递归神经网络LSTM, 提高了皮肤癌检测的准确性. 基于此, Ahmad等人[56]提出深度卷积神经网络和堆叠BLSTM网络的混合分类方法. 其中最关键的是BLSTM网络通过两个平行层相反方向传递输入信息, 在一定程度上缓解了梯度消失问题, 显著提高了皮肤癌分类的平均准确率.
综上所述, RNN在皮肤癌分类中发挥着重要作用. 其记忆能力和对长期依赖关系的捕捉, 显著提高了模型的准确性. 通过结合启发式优化算法和调整权重, 可以加速模型的收敛速度并提高分类性能. 然而, RNN的训练难度较大, 且容易出现梯度消失或爆炸的问题. 因此, 需要进一步优化网络结构和参数设置, 以克服这些挑战并提升皮肤癌分类的准确性和泛化能力.
3.3 GANGAN是一种深度学习模型, 常用于生成与训练数据相似的新皮肤癌图像样本. GAN由生成器和判别器组成, 二者相互竞争, 使生成器逐渐提升生成样本的逼真程度, 同时判别器不断努力区分真实和生成的皮肤病图像样本, 从而促使生成的图像样本更加贴近真实数据分布的特征[83].
为了解决过拟合和泛化不良的问题, Medi等人[57]提出了通过训练带有梯度惩罚的GAN生成合成数据的方法. 这种方法的特点是通过使用梯度惩罚, 紧密地模拟了Lipschitz函数, 确保了距离的准确计算, 从而优化了模型的泛化能力. 在此基础上, Wang等人[58]利用CycleGAN进行数据增强, 并提出了一种新的图像配对策略. 即通过将多数类图像和少数类图像配对进行训练, 生成少数类图像来平衡数据集. 然而, 由于无法将预训练权值加载到ImageNet上, 这种方法可能导致梯度下降的初始方向产生偏差, 影响精度.
进一步地, 为了改善所存在的问题, Teodoro等人[59]将深度卷积GAN与EfficientNet结合, 采用固定缩放系数代替随机缩放, 从而在相同层中根据需求对模型进行缩放, 提高了模型的性能和效率. 与此同时, Zhao等人[60]设计了一种皮肤病变增强样式GAN的皮肤病变图像分类框架, 其特点是通过重新设计生成器的样式控制和噪声输入结构, 有效合成高质量的皮肤病变图像. 然而, 该方法在处理不同病变重叠和多阶段皮肤癌时增加了模型复杂度, 可能导致误分类.
为应对上述问题, Ahmad等人[61]引入了带有辅助分类器的GAN, 通过在重尾学生t分布中采样噪声向量, 增加了生成图像的多样性, 提高了皮肤病变分类任务的性能, 大大减少误分类的可能性. 相似地, Abdelhalim等人[62]使用自关注渐进生长GAN (SPGGANs)生成细粒度的256×256皮肤病变图像, 并将双时间尺度更新规则(TTUR)应用于SPGGAN, 以提高模型的稳定性和生成图像的质量.
此外, 赵宸等人[63]在StyleGAN结构的基础上引入了自注意力机制, 重新设计生成器的控制样式和噪声输入结构, 从而有效的生成高质量的皮肤癌病变图像. 而Qin等人[64]在该结构的基础上, 用风格控制方法对该结构进行修改, 调整合成网络结构, 并重新设计生成器和判别器的架构, 进一步优化了生成图像的质量. 为了降低计算成本并提高分类效率, Behara等人[65]提出了一种改进的深度卷积生成对抗网络(DCGAN)的皮肤病变综合分类模型, 该模型的特点是利用深度卷积神经网络作为生成器和判别器模型, 并采用能够稳定训练生成器的模型配置和训练方法, 大大地降低了计算成本. 而Alsaidi等人[66]提出了带有训练辅助分类器的GAN架构(AC-GAN), 这种架构是DCGAN的扩展, 其独特之处在于可以生成特定类的合成数据, 以平衡次要类数据. 尽管这种方法在生成特定类数据时表现出色, 但仍面临生成图像可能不属于特定病变类别的挑战.
综上所述, GAN在皮肤癌分类中有以下优点: (1) GAN可以生成逼真的合成皮肤癌图像, 从而扩展训练数据集, 帮助模型更好地学习皮肤癌特征, 提高分类性能. (2) GAN生成的图像具有多样性, 可以模拟不同类型的皮肤病变, 有助于训练模型更全面地识别和分类各种皮肤癌类型. (3) GAN的生成器和判别器之间的对抗性学习机制可以推动模型学习更丰富和更具判别性的特征, 有助于提高皮肤癌分类模型的鲁棒性和性能. 但也有以下缺点: (1) GAN可能会出现模式崩溃的问题, 即生成器倾向于生成类似的图像, 导致合成图像缺乏多样性, 从而影响模型的泛化能力和分类效果. (2) GAN的训练过程通常比较复杂, 容易出现训练不稳定的情况, 如模式震荡和模型崩溃等问题, 需要花费大量时间和计算资源来优化. (3) GAN的潜在空间往往较为复杂, 难以理解和控制, 这使得生成的图像可能不易满足特定的分类需求. 因此, 需要进一步研究和改进GAN的模型结构和训练方法, 以提高其在皮肤病分类中的性能和可靠性.
3.4 TransformerTransformer是一种基于自注意力机制的神经网络架构, 近年来, 在皮肤癌分类领域取得了显著的成功. Transformer能够同时考虑序列数据中的所有位置, 使其在处理皮肤癌图像序列时更为高效. 其主要特点包括引入了自注意力机制, 有助于捕捉皮肤癌图像中的长距离依赖关系; 无需循环的处理方式使得模型的训练过程高度并行化, 提高了训练效率; 多头注意力机制允许模型学习不同层次的特征表示; 位置编码则为模型提供了处理序列数据的位置信息[84].
传统的卷积神经网络(CNN)由于卷积算子的固有局部性, 缺乏捕捉上下文信息和远程依赖的能力. 为了提高模型性能, Xin等人[67]改进了Transformer网络, 他们使用多尺度和重叠滑动窗口对图像进行序列化, 再将多尺度块嵌入, 从而提高对多尺度特征的关注度. 通过对比学习, 使得皮肤癌相似数据的编码相似, 不同数据的编码结果尽可能地不同, 从而取得了较好的实验效果. 然而, 该模型在处理高分辨率图像时速度较慢. Lungu-Stan等人[68]通过知识蒸馏技术, 在Transformer的每个级别添加分类头, 这样就可以增强不同空间位置特征图之间的相关性, 在提升处理速度的同时也提升了基础模型的平衡多类准确性.
近年来, CNN和Transformer分别被用来学习病灶图像的局部特征和全局特征. 然而, 单一网络的特征提取能力有限. 为解决这一问题, Rezaee等人[69]提出了交叉融合全局和局部特征的方法, 通过特征融合架构合并分支之间的并行系统, 生成细粒度特征, 从而提高分类性能. 同样的, Hao等人[70]结合ConvNeXt和Swin Transformer的优势, 设计了一种融合模型ConvNeXt-ST-AFF, 通过注意力特征融合子模块进行特征融合, 并采用去噪模块提高图像对比度, 从而提供了更全面和有效的皮肤病变分析解决方案.
在面对较低的检测精度和模型泛化能力差的问题时, Aladhadh等人[71]开发了一个双阶段的框架. 他们在第1阶段采用数据增强方法增加图像样本数量, 第2阶段使用医学Vision Transformer进行皮肤癌分类. 该方法优点是将输入图像分割为图像块, 再以序列结构将这些块馈送到Transformer中, 从而提高了模型的泛化能力. 然而, 其检验精度仍有待提高. Yang等人[72]通过对图像进行类别重新平衡和分块预处理, 采用Transformer编码器处理展平的图像块, 显著提高了分类准确度.
为了减少模型处理时间和提升分类效率, He等人[73]提出了一种用于皮肤癌病变分析的全Transformer网络, 采用SPT计算特征, 显著减少了计算和内存使用. 实验结果表明, 这种改进网络在计算效率和可调参数数量方面优于CNN, 并突出了重要特征的价值, 抑制了噪声特征. 类似的, Abbas等人[74]设计了一种结合Squeeze-Net和深度可分离CNN的新型可分离视觉转换器架构, 减少了参数量的同时缩短了处理时间.
综上所述, Transformer在皮肤癌分类中的作用是通过提取远程上下文信息和全局特征来改善模型的性能. 相对于传统的CNN模型, Transformer在皮肤癌图像分类中具有更强的长距离依赖关系捕获能力, 同时还能够有效地捕获全局特征, 从而提高了分类准确度和泛化能力. 与RNN和GAN算法相比, Transformer的优势在于高效的并行化计算、更好的参数效率和模型解释性. 然而, Transformer也存在一些挑战, 如计算复杂度较高、模型参数较多以及对大量标记数据的依赖性. 因此, 需要进一步优化Transformer的结构和训练方法, 以提高其在皮肤病分类中的效率和可靠性.
4 算法总结及展望通过对传统机器学习算法和深度学习算法在皮肤癌病分类诊断中的回顾, 两种算法在该领域也是备受关注, 为了让读者更直观地理解两种算法在分类中的优势和不足, 本文对两种算法进行归纳总结, 如表5所示.
| 表 5 皮肤病分类方法总结 |
首先是机器学习, 传统机器学习算法如KNN和SVM在皮肤癌分类中具有简单易用、处理小样本数据能力强、对异常值不敏感、可解释性强等优势, 但随着深度学习技术的发展, 该算法在面对复杂的非线性关系和大规模数据集时表现不佳, 传统机器学习算法在皮肤癌分类中的地位逐渐被深度学习所取代.
而深度学习在皮肤癌分类中具有显著优势, CNN通过结合迁移学习、注意力机制等方法, 提高了模型的准确性和性能. 尽管传统机器学习算法在某些应用中已逐渐被深度学习所取代, 但结合深度学习方法进行研究仍是当前的趋势. 如使用VGG、Inception等模型提取特征, 并通过SVM、KNN等分类器进行分类, 进一步提升了分类精度. RNN的记忆特性和对长期依赖关系的利用有助于提高模型准确性, 并且结合启发式优化算法和权重调整可以加速模型收敛, 提高分类性能. 与CNN相比, RNN在序列数据建模和长期依赖性捕捉方面具有更强的能力, 这归功于其递归结构和内部记忆单元, 使其能够有效地处理时间序列数据并捕获序列中的长距离相关性. 相较于CNN和RNN算法, GAN在皮肤病图像分类中的优势在于生成合成数据以提高模型泛化能力, 平衡数据集中的类别分布. 但GAN存在模式崩溃、训练不稳定、合成图像质量不高以及潜在空间理解困难等问题.
近年来, Transformer在计算机视觉领域的广泛应用展现出了其在皮肤癌分类方面的巨大潜力. 其独特之处在于不依赖固定大小图像的输入, 能够轻松处理不同尺度的皮肤图像. 其自注意力机制能够提供对分类决策的解释, 使得模型的预测结果更具可解释性. 此外, Transformer还具有并行化处理图像像素的能力, 加速了模型训练和推断的过程. 未来, Transformer可能会与其他深度学习技术结合, 如半监督学习、迁移学习或元学习, 进一步提高分类性能和适用性, 为皮肤癌分类提供更可靠的解决方案.
尽管两种学习方法在皮肤病辅助诊断中有出色的表现, 但仍面临一些挑战, 包括以下7个方面.
(1) 现有皮肤癌数据集中病例的多样性不足, 大多数病例来自白皮肤个体, 而深皮肤个体的病例较少.
(2) 皮肤癌分类需要大量高质量的标记数据来训练模型, 而且这些数据通常难以获取和标记, 导致数据质量和数量不足的问题.
(3) 很少有研究明确如何为特定的皮肤癌诊断任务选择合适类型的深度神经网络.
(4) 皮肤癌分类中各种类型的病变样本数量可能存在严重的不平衡, 这会导致模型对于少数类别的分类性能下降.
(5) 皮肤癌病变具有多样性和复杂性, 不同类型的病变在形状、颜色、纹理等方面差异巨大, 这增加了分类任务的难度.
(6) 对于医疗领域而言, 模型的解释性和可解释性至关重要, 但深度学习模型通常被认为是“黑盒子”, 难以解释其预测过程, 这限制了其在临床实践中的应用.
(7) 皮肤癌分类模型需要具有良好的泛化能力和鲁棒性, 即使在面对未见过的数据或者噪声干扰时也能够做出准确的预测, 但两种学习模型在泛化能力和鲁棒性方面仍然存在挑战.
为了解决皮肤癌辅助诊断面临的挑战, 未来的发展方向可能包括如下几个方面.
(1) 开发高度多样性的皮肤病数据集至关重要, 需包含不同人种、年龄、性别和病变类型的图像, 以确保模型准确分类和诊断.
(2) 利用迁移学习和数据增强技术, 通过在其他领域预训练的模型来初始化皮肤病分类模型, 并结合合成数据和自动生成数据的方法来扩充数据集.
(3) 有必要研究皮肤病特征及相应数据, 为特定任务设计具备领域知识的深度网络. 这样可望取得更佳性能.
(4) 采用类别平衡的损失函数或采样技术来平衡数据集中的各类别样本数量, 或者引入类别加权的损失函数来关注少数类别的训练效果.
(5) 结合多模态信息或多尺度特征, 利用注意力机制或多任务学习来捕获不同类型病变的多样性和复杂性, 从而提高分类性能.
(6) 采用解释性模型或增强模型解释性的方法, 如可视化注意力机制、解释性对抗训练等, 以提高深度学习模型的可解释性.
(7) 引入正则化技术、集成学习方法或对抗性训练来提高模型的泛化能力和鲁棒性, 同时加强数据清洗和预处理以降低噪声对模型的影响.
未来在皮肤病分类领域的发展趋势将是深度学习和传统机器学习的融合, 利用深度学习模型如CNN、RNN、GAN、Transformer等的强大特征提取能力和传统机器学习算法的高效性能, 以解决皮肤病分类中的挑战. 同时, 随着医学影像技术的不断进步, 可能会出现更高分辨率、更丰富的皮肤病图像数据, 为模型训练和分类提供更丰富的信息, 从而实现更准确、高效的皮肤病分类和诊断.
5 结束语深度学习技术展现出了巨大的潜力和应用前景. 传统机器学习算法在特征工程和模型解释性方面具有优势, 但在复杂特征提取和大规模数据处理方面存在局限性. 而深度学习方法则能够自动学习特征表示, 并在大规模数据上展现出强大的分类性能, 但其模型的可解释性和泛化能力仍然是需要持续改进的方面. 未来的发展方向将聚焦于提高模型的泛化能力、解释性和个性化医疗水平, 以更好地服务于临床实践, 为皮肤癌患者提供更准确、更可靠的诊断和治疗方案.
| [1] |
Kumari S, Choudhary PK, Shukla R, et al. Recent advances in nanotechnology based combination drug therapy for skin cancer. Journal of Biomaterials Science, Polymer Edition, 2022, 33(11): 1435-1468. DOI:10.1080/09205063.2022.2054399 |
| [2] |
Anand V, Gupta S, Nayak SR, et al. An automated deep learning models for classification of skin disease using dermoscopy images: A comprehensive study. Multimedia Tools and Applications, 2022, 81(26): 37379-37401. DOI:10.1007/s11042-021-11628-y |
| [3] |
Skin Cancer Foundation. Skin cancer information. http://www.skincancer.org/skin-cancer-information. (2016-08-20).
|
| [4] |
CSCO黑色素瘤专家委员会. 中国黑色素瘤诊治指南. 北京: 人民卫生出版社, 2012.
|
| [5] |
Cazzato G, Mangialardi K, Falcicchio G, et al. Preferentially expressed antigen in melanoma (PRAME) and human malignant melanoma: A retrospective study. Genes, 2022, 13(3): 545. DOI:10.3390/genes13030545 |
| [6] |
Mohakud R, Dash R. Skin cancer image segmentation utilizing a novel EN-GWO based hyper-parameter optimized FCEDN. Journal of King Saud University—Computer and Information Sciences, 2022, 34(10): 9889-9904. DOI:10.1016/j.jksuci.2021.12.018 |
| [7] |
Habif TP, Dinulos JG, Chapman MS, et al. Skin Disease: Diagnosis and Treatment. 4th ed., Amsterdam: Elsevier, 2017.
|
| [8] |
张汉明, 马金刚, 张宁宁, 等. 深度学习在癫痫检测中的应用进展. 计算机工程与应用, 2023, 59(10): 35-47. DOI:10.3778/j.issn.1002-8331.2207-0062 |
| [9] |
孙石磊, 李明, 刘静, 等. 深度学习在糖尿病视网膜病变分类领域的研究进展. 计算机工程与应用, 2024, 60(8): 16-30. DOI:10.3778/j.issn.1002-8331.2307-0330 |
| [10] |
殷文君. 基于深度学习的医学影像分类与分割算法研究[硕士学位论文]. 桂林: 桂林电子科技大学, 2023. [doi: 10.27049/d.cnki.ggldc.2023.000366]
|
| [11] |
张杰, 赵惠军, 李贤威, 等. 基于深度学习方法检测皮肤镜图像中黑色素瘤的研究进展与展望. 医疗卫生装备, 2018, 39(11): 90-95. |
| [12] |
杨聚加, 段然, 吴亚光, 等. 数据分类算法智慧诊疗模型在皮肤痤疮诊断中的研究与应用. 重庆医学, 2022, 51(3): 507-511. DOI:10.3969/j.issn.1671-8348.2022.03.032 |
| [13] |
Li HF, Pan YN, Zhao J, et al. Skin disease diagnosis with deep learning: A review. Neurocomputing, 2021, 464: 364-393. DOI:10.1016/j.neucom.2021.08.096 |
| [14] |
Bhatt H, Shah V, Shah K, et al. State-of-the-art machine learning techniques for melanoma skin cancer detection and classification: A comprehensive review. Intelligent Medicine, 2023, 3(3): 180-190. DOI:10.1016/j.imed.2022.08.004 |
| [15] |
Mendonça T, Ferreira PM, Marques JS, et al. PH2—A dermoscopic image database for research and benchmarking. Proceedings of the 35th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC). Osaka: IEEE, 2013. 5437–5440.
|
| [16] |
Codella NCF, Gutman D, Celebi ME, et al. Skin lesion analysis toward melanoma detection: A challenge at the 2017 international symposium on biomedical imaging (ISBI), hosted by the international skin imaging collaboration (ISIC). Proceedings of the 15th IEEE International Symposium on Biomedical Imaging (ISBI 2018). Washington: IEEE, 2018. 168–172.
|
| [17] |
Gutman D, Codella NC, Celebi E, et al. Skin lesion analysis toward melanoma detection: A challenge at the international symposium on biomedical imaging (ISBI) 2016, hosted by the international skin imaging collaboration (ISIC). arXiv:1605.01397, 2016.
|
| [18] |
Marchetti MA, Codella NCF, Dusza SW, et al. Results of the 2016 international skin imaging collaboration international symposium on biomedical imaging challenge: Comparison of the accuracy of computer algorithms to dermatologists for the diagnosis of melanoma from dermoscopic images. Journal of the American Academy of Dermatology, 2018, 78(2): 270–277.
|
| [19] |
Li YX, Shen LL. Skin lesion analysis towards melanoma detection using deep learning network. Sensors, 2018, 18(2): 556. DOI:10.3390/s18020556 |
| [20] |
Tschandl P, Rosendahl C, Kittler H. The HAM10000 dataset, a large collection of multi-source dermatoscopic images of common pigmented skin lesions. Scientific Data, 2018, 5(1): 180161. DOI:10.1038/sdata.2018.161 |
| [21] |
Dermofit image library. https://licensing.edinburgh-innovations.ed.ac.uk/i/software/dermofit-image-library.html. (2019-09-11).
|
| [22] |
Giotis I, Molders N, Land S, et al. MED-NODE: A computer-assisted melanoma diagnosis system using non-dermoscopic images. Expert Systems with Applications, 2015, 42(19): 6578-6585. DOI:10.1016/j.eswa.2015.04.034 |
| [23] |
Dermnet. http://www.dermnet.com/. (2019-09-11).
|
| [24] |
Hatem MQ. Skin lesion classification system using a K-nearest neighbor algorithm. Visual Computing for Industry, Biomedicine, and Art, 2022, 5(1): 7. DOI:10.1186/s42492-022-00103-6 |
| [25] |
Pal OK. Skin disease classification: A comparative analysis of K-nearest neighbors (KNN) and random forest algorithm. Proceedings of the 2021 International Conference on Electronics, Communications and Information Technology (ICECIT). Khulna: IEEE, 2021. 1–5.
|
| [26] |
Abbes W, Sellami D, Marc-Zwecker S, et al. Fuzzy decision ontology for melanoma diagnosis using KNN classifier. Multimedia Tools and Applications, 2021, 80(17): 25517-25538. DOI:10.1007/s11042-021-10858-4 |
| [27] |
Zhuang JX, Cai JB, Wang RX, et al. Deep KNN for medical image classification. Proceedings of the 23rd International Conference on Medical Image Computing and Computer Assisted Intervention. Lima: Springer, 2020. 127–136.
|
| [28] |
Raju DN, Shanmugasundaram H, Sasikumar R. Fuzzy segmentation and black widow-based optimal SVM for skin disease classification. Medical & Biological Engineering & Computing, 2021, 59(10): 2019-2035. |
| [29] |
Balamurugan M, Periasamy JK, Akash M, et al. An efficient mechanism to detect skin disease using SVM. Journal of Green Engineering, 2020, 10(10): 9506-9516. |
| [30] |
Hasanah RL, Riana D. Classification of dermoscopic image of skin cancer using the GLCM method and multi-SVM algorithm. Rekayasa, 2021, 14(3): 407-415. DOI:10.21107/rekayasa.v14i3.12213 |
| [31] |
Nawar A, Sabuz NK, Siddiquee SMT, et al. Skin disease recognition: A machine vision based approach. Proceedings of the 7th International Conference on Advanced Computing and Communication Systems (ICACCS). Coimbatore: IEEE, 2021. 1029–1034.
|
| [32] |
Arora G, Dubey AK, Jaffery ZA, et al. Bag of feature and support vector machine based early diagnosis of skin cancer. Neural Computing and Applications, 2022, 34(11): 8385-8392. DOI:10.1007/s00521-020-05212-y |
| [33] |
Lingaraj M, Senthilkumar A, Ramkumar J. Prediction of melanoma skin cancer using veritable support vector machine. Annals of the Romanian Society for Cell Biology, 2021, 25(4): 2623-2636. |
| [34] |
Battineni G, Sagaro GG, Chinatalapudi N, et al. Applications of machine learning predictive models in the chronic disease diagnosis. Journal of Personalized Medicine, 2020, 10(2): 21. DOI:10.3390/jpm10020021 |
| [35] |
Cervantes J, Garcia-Lamont F, Rodríguez-Mazahua L, et al. A comprehensive survey on support vector machine classification: Applications, challenges and trends. Neurocomputing, 2020, 408: 189-215. DOI:10.1016/j.neucom.2019.10.118 |
| [36] |
Mohanaiah P, Sathyanarayana P, GuruKumar L. Image texture feature extraction using GLCM approach. International Journal of Scientific and Research Publications, 2013, 3(5): 1-5. |
| [37] |
Allugunti VR. A machine learning model for skin disease classification using convolution neural network. International Journal of Computing, Programming and Database Management, 2022, 3(1): 141-147. DOI:10.33545/27076636.2022.v3.i1b.53 |
| [38] |
张荣梅, 张琦, 刘院英. 基于Inception深度残差网络的皮肤黑色素癌图像分类算法. 计算机系统应用, 2021, 30(7): 142-149. DOI:10.15888/j.cnki.csa.007962 |
| [39] |
Maduranga MWP, Nandasena D. Mobile-based skin disease diagnosis system using convolutional neural networks (CNN). International Journal of Image, Graphics Signal Process, 2022, 14(3): 47-57. DOI:10.5815/ijigsp.2022.03.05 |
| [40] |
Srinivasu PN, SivaSai JG, Ijaz MF, et al. Classification of skin disease using deep learning neural networks with MobileNet V2 and LSTM. Sensors, 2021, 21(8): 2852. DOI:10.3390/s21082852 |
| [41] |
Karthik R, Vaichole TS, Kulkarni SK, et al. Eff2Net: An efficient channel attention-based convolutional neural network for skin disease classification. Biomedical Signal Processing and Control, 2022, 73: 103406. DOI:10.1016/j.bspc.2021.103406 |
| [42] |
王师玮, 陈俊, 易才键. 基于改进ResNet50的皮肤病变图像分类. 软件工程, 2023, 26(6): 50-54. DOI:10.19644/j.cnki.issn2096-1472.2023.006.011 |
| [43] |
Junayed MS, Anjum N, Noman A, et al. A deep CNN model for skin cancer detection and classification. Proceedings of the 2021 International Conference in Central Europe on Computer Graphics, Visualization and Computer Vision. 2021. 71–80.
|
| [44] |
Fu’adah YN, Pratiwi NKC, Pramudito MA, et al. Convolutional neural network (CNN) for automatic skin cancer classification system. IOP Conference Series: Materials Science and Engineering, 2020, 982: 012005. DOI:10.1088/1757-899X/982/1/012005 |
| [45] |
Anand V, Gupta S, Koundal D, et al. Multi-class skin disease classification using transfer learning model. International Journal on Artificial Intelligence Tools, 2022, 31(2): 2250029. DOI:10.1142/S0218213022500294 |
| [46] |
Gupta S, Panwar A, Mishra K. Skin disease classification using dermoscopy images through deep feature learning models and machine learning classifiers. Proceedings of the 19th International Conference on Smart Technologies. Lviv: IEEE, 2021. 170–174.
|
| [47] |
Hameed N, Shabut AM, Hossain MA. Multi-class skin diseases classification using deep convolutional neural network and support vector machine. Proceedings of the 12th International Conference on Software, Knowledge, Information Management & Applications (SKIMA). Phnom Penh: IEEE, 2018. 1–7.
|
| [48] |
Janoria H, Minj J, Patre P. Classification of skin disease from skin images using transfer learning technique. Proceedings of the 4th International Conference on Electronics, Communication and Aerospace Technology (ICECA). Coimbatore: IEEE, 2020. 888–895.
|
| [49] |
Senthilkumaran N. Efficient skin cancer detection using enhanced adaptive hybrid RNN classification for bio medical application. Journal of Pharmaceutical Negative Results, 2022, 13(10): 2804–2814.
|
| [50] |
Dabbu M, Karuppusamy L, Pulugu D, et al. Water atom search algorithm-based deep recurrent neural network for the big data classification based on spark architecture. International Journal of Machine Learning and Cybernetics, 2022, 13(8): 2297-2312. DOI:10.1007/s13042-022-01524-8 |
| [51] |
Divya D, Ganeshbabu TR. Fitness adaptive deer hunting-based region growing and recurrent neural network for melanoma skin cancer detection. International Journal of Imaging Systems and Technology, 2020, 30(3): 731-752. DOI:10.1002/ima.22414 |
| [52] |
Shukla A, Shyam GK, Shree R, et al. Skin cancer identification using cat swarm-intelligent generative RNN algorithm. International Journal of Intelligent Systems and Applications in Engineering, 2023, 11(8s): 447-454. |
| [53] |
Gomathi P. Skin cancer prediction with optimized RNN via tunicate swarm algorithm. International Journal of Advanced Research in Engineering and Technology (IJARET), 2020, 11(7): 929–940.
|
| [54] |
Vinoth R, Ananth JP. Rider chicken optimization algorithm-based recurrent neural network for big data classification in spark architecture. The Computer Journal, 2022, 65(8): 2183–2196.
|
| [55] |
Yunandar R, Irianto SY. Skin cancer clasification using region growing & recurrent neural network. Proceedings of the 2022 International Conference on Information Technology and Business. Lampung: Institut Informatika dan Bisnis Darmajaya, 2022. 58–63.
|
| [56] |
Ahmad B, Usama M, Ahmad T, et al. An ensemble model of convolution and recurrent neural network for skin disease classification. International Journal of Imaging Systems and Technology, 2022, 32(1): 218-229. DOI:10.1002/ima.22661 |
| [57] |
Medi PR, Nemani P, Pitta VR, et al. SkinAid: A GAN-based automatic skin lesion monitoring method for iomt frameworks. Proceedings of the 19th OITS International Conference on Information Technology (OCIT). Bhubaneswar: IEEE, 2021. 200–205.
|
| [58] |
Wang H, Qi QQ, Sun WJ, et al. Classification of skin lesions with generative adversarial networks and improved MobileNetV2. International Journal of Imaging Systems and Technology, 2023, 33(5): 1561-1576. DOI:10.1002/ima.22880 |
| [59] |
Teodoro AAM, Silva DH, Rosa RL, et al. A skin cancer classification approach using GAN and RoI-based attention mechanism. Journal of Signal Processing Systems, 2023, 95(2–3): 211-224. DOI:10.1007/s11265-022-01757-4 |
| [60] |
Zhao C, Shuai RJ, Ma L, et al. Dermoscopy image classification based on StyleGAN and DenseNet201. IEEE Access, 2021, 9: 8659-8679. DOI:10.1109/ACCESS.2021.3049600 |
| [61] |
Ahmad B, Jun S, Palade V, et al. Improving skin cancer classification using heavy-tailed student t-distribution in generative adversarial networks (TED-GAN). Diagnostics, 2021, 11(11): 2147. DOI:10.3390/diagnostics11112147 |
| [62] |
Abdelhalim ISA, Mohamed MF, Mahdy YB. Data augmentation for skin lesion using self-attention based progressive generative adversarial network. Expert Systems with Applications, 2021, 165: 113922. DOI:10.1016/j.eswa.2020.113922 |
| [63] |
赵宸, 帅仁俊, 马力, 等. 基于Self-Attention StyleGAN的皮肤癌图像生成与分类. 计算机工程与应用, 2022, 58(18): 111-121. DOI:10.3778/j.issn.1002-8331.2102-0092 |
| [64] |
Qin ZW, Liu Z, Zhu P, et al. A GAN-based image synthesis method for skin lesion classification. Computer Methods and Programs in Biomedicine, 2020, 195: 105568. DOI:10.1016/j.cmpb.2020.105568 |
| [65] |
Behara K, Bhero E, Agee JT. Skin lesion synthesis and classification using an improved DCGAN classifier. Diagnostics, 2023, 13(16): 2635. DOI:10.3390/diagnostics13162635 |
| [66] |
Alsaidi M, Jan MT, Altaher A, et al. Tackling the class imbalanced dermoscopic image classification using data augmentation and GAN. Multimedia Tools and Applications, 2023, 83(16): 49121-49147. DOI:10.1007/s11042-023-17067-1 |
| [67] |
Xin C, Liu ZF, Zhao KY, et al. An improved Transformer network for skin cancer classification. Computers in Biology and Medicine, 2022, 149: 105939. DOI:10.1016/j.compbiomed.2022.105939 |
| [68] |
Lungu-Stan VC, Cercel DC, Pop F. SkinDistilViT: Lightweight vision Transformer for skin lesion classification. Proceedings of the 32nd International Conference on Artificial Neural Networks. Heraklion: Springer, 2023. 268–280.
|
| [69] |
Rezaee K, Zadeh HG. Self-attention Transformer unit-based deep learning framework for skin lesions classification in smart healthcare. Discover Applied Sciences, 2024, 6(1): 3. DOI:10.1007/s42452-024-05655-1 |
| [70] |
Hao SN, Zhang LG, Jiang YY, et al. ConvNeXt-ST-AFF: A novel skin disease classification model based on fusion of ConvNeXt and swin Transformer. IEEE Access, 2023, 11: 117460-117473. DOI:10.1109/ACCESS.2023.3324042 |
| [71] |
Aladhadh S, Alsanea M, Aloraini M, et al. An effective skin cancer classification mechanism via medical vision Transformer. Sensors, 2022, 22(11): 4008. DOI:10.3390/s22114008 |
| [72] |
Yang G, Luo SH, Greer P. A novel vision Transformer model for skin cancer classification. Neural Processing Letters, 2023, 55(7): 9335-9351. DOI:10.1007/s11063-023-11204-5 |
| [73] |
He XZ, Tan EL, Bi HW, et al. Fully Transformer network for skin lesion analysis. Medical Image Analysis, 2022, 77: 102357. DOI:10.1016/j.media.2022.102357 |
| [74] |
Abbas Q, Daadaa Y, Rashid U, et al. Assist-Dermo: A lightweight separable vision Transformer model for multiclass skin lesion classification. Diagnostics, 2023, 13(15): 2531. DOI:10.3390/diagnostics13152531 |
| [75] |
Kassem MA, Hosny KM, Damaševičius R, et al. Machine learning and deep learning methods for skin lesion classification and diagnosis: A systematic review. Diagnostics, 2021, 11(8): 1390. DOI:10.3390/diagnostics11081390 |
| [76] |
文成林, 吕菲亚. 基于深度学习的故障诊断方法综述. 电子与信息学报, 2020, 42(1): 234-248. DOI:10.11999/JEIT190715 |
| [77] |
Sinha D, El-Sharkawy M. Thin MobileNet: An enhanced mobilenet architecture. Proceedings of the 10th IEEE Annual Ubiquitous Computing, Electronics & Mobile Communication Conference (UEMCON). New York: IEEE, 2019. 280–285.
|
| [78] |
Zhong ZY, Jin LW, Xie ZC. High performance offline handwritten chinese character recognition using GoogLeNet and directional feature maps. Proceedings of the 13th International Conference on Document Analysis and Recognition (ICDAR). Tunis: IEEE, 2015. 846–850.
|
| [79] |
陈明海, 贺建峰. 基于部分迁移学习的皮肤病图像分类. 生物医学工程与临床, 2022, 26(3): 272-277. DOI:10.13339/j.cnki.sglc.20220421.002 |
| [80] |
Xia XL, Xu C, Nan B. Inception-v3 for flower classification. Proceedings of the 2nd International Conference on Image, Vision and Computing (ICIVC). Chengdu: IEEE, 2017. 783–787.
|
| [81] |
Iandola FN, Han S, Moskewicz MW, et al. SqueezeNet: AlexNet-level accuracy with 50× fewer parameters and <0.5 MB model size. arXiv:1602.07360, 2016.
|
| [82] |
吴甜, 刘海华, 童顺延. 基于深度反馈的卷积神经网络的图像分类. 计算机与现代化, 2023(9): 82-86. DOI:10.3969/j.issn.1006-2475.2023.09.013 |
| [83] |
曾懋胜. 基于GAN的图像生成研究 [硕士学位论文]. 杭州: 杭州电子科技大学, 2023. [doi: 10.27075/d.cnki.ghzdc.2023.000696]
|
| [84] |
黄倩, 崔静雯, 李畅. 基于骨骼的人体行为识别方法研究综述. 计算机辅助设计与图形学学报, 2024, 36(2): 173-194. |
 2024, Vol. 33
2024, Vol. 33



