抑郁症, 这一日益严重的精神障碍, 正逐渐成为全球关注的焦点. 它无情地侵蚀着患者的心理健康, 对其生理机能造成深远影响, 导致记忆力减退、睡眠质量下滑等症状频发, 严重降低了患者的生活质量和社会功能[1]. 这些复杂多变的临床表现使得抑郁症的诊断成为医学界亟待攻克的难题. 准确诊断抑郁症不仅关乎患者的及时治疗和康复, 更是提升精神健康领域研究水平的关键.
然而, 当前抑郁症的诊断方法主要依赖医生的经验和患者的自我描述, 存在主观性和不确定性. 为了克服这一局限性, 我们亟需探索一种客观、有效的诊断方法. 近年来, 神经科学和人工智能技术的迅猛发展为我们提供了新的思路. 特别是基于生理信号的抑郁症诊断方法, 其中睡眠脑电信号[2]因其非侵入性、易获取和高时间分辨率等优点而备受关注. 通过对睡眠脑电信号进行深入分析, 我们可以提取出与抑郁症相关的特征信息[3], 从而实现对其的准确识别.
尽管已有研究表明, 睡眠脑电信号中包含了与抑郁症相关的特征信息, 但目前对于如何有效地利用这些信息进行抑郁症诊断的研究仍然不足. 而准确的睡眠分期能够明确反应抑郁症的睡眠特征, 进而精确利用睡眠脑电信号中与抑郁症相关的特征信息. 因此, 为了更准确地利用睡眠信号的特征信息, 从而进一步提高诊断的准确性和客观性, 本文结合了睡眠分期技术, 采用CNN-BiLSTM混合架构算法对睡眠脑电数据进行处理与分析. 该算法融合了CNN和BiLSTM的优势, 能够实现对睡眠阶段的高精度划分. 我们重点关注快速眼动(REM)睡眠期的脑电信号, 该阶段信号与抑郁症的病理生理机制密切相关, 具有独特的频谱特征. 为此, 我们引入了DepNet2D模型, 通过构建针对频谱信息的二维网络, 深入剖析不同频段间的关联与影响, 从而精准提取出抑郁症患者的特征性脑电频谱信息. 这种基于睡眠分期的抑郁症诊断方法不仅提高了诊断准确率, 还增强了其客观性和说服力, 为抑郁症的精准治疗提供了有力支持.
1.2 研究现状近年来, 随着深度学习技术的快速发展, 其在医疗领域的应用也逐渐广泛. 尤其在基于脑电信号的睡眠分期和抑郁症诊断方面, 深度学习技术展现出了巨大的潜力. 在基于脑电信号的抑郁症分类的研究现状方面, 研究者们通过精心设计深度学习模型, 在抑郁症识别领域取得了显著的进展. Acharya等人[4]采用CNN模型对抑郁症患者与正常人的脑电数据进行了分类研究, 成功验证了深度学习在抑郁症诊断中的有效性, 然而, 该研究受限于较小的样本量(仅30名受试者), 可能导致模型泛化能力不足, 难以在新样本中准确区分抑郁症患者与正常人的脑电信号差异. Li等人[5]则在此基础上更进一步, 他们结合了CNN与迁移学习技术, 深入探索了脑电信号中不同层面信息在抑郁症识别中的重要作用, 为脑电信号识别抑郁症提供了更坚实的理论基础, 然而, 他们在特征选择和提取上具有一定的局限性, 这在一定程度上影响了模型的分类性能. Kwon等人[6]提出了一种创新的深度学习方法, 该模型基于前额叶脑电的不对称图像来实现对抑郁症患者的初步筛选, 但可能由于对模型的选取考虑不周, 导致模型不能很好地拟合, 出现测试集预测精度波动较大的情况. 卢官明等人[7]开发的具有脑电信号情感识别功能的组合模型, 在特征提取和处理动态时序信息方面展现出优势, 准确率达到86.08%, 但其研究指出需要较大的样本量来支持模型训练. 李梅等人[8]通过将基于算法的卷积神经网络(CNN)与无线传感器结合的方法, 实现了98.00%的高准确率, 但缺点是其成本过于昂贵. Qayyum等人[9]展示了卷积神经网络与GRU结合的方法, 在短序列学习方面表现出优于LSTM的特性, 准确率为90.45%, 但这种方法仍在时间窗口分段长度处理方面有所不足. Spencer等人[10]的研究采用了基于22层ResNet的卷积神经网络模型, 这一选择确实为开发分类器和应用算法权重分配提供了便利. 然而, 在测试数据集上的分类准确率仅为78.94%, 这一结果相较于当前领域内其他先进模型而言相对较低. 综上, 尽管上述研究在抑郁症诊断的深度学习应用方面取得了进展, 但普遍面临样本量不足、成本高昂、模型准确性和泛化能力有待提高等问题.
为进一步精确地诊断抑郁症, 许多学者分析了众多脑电信号与抑郁症的关联, 其中, 睡眠信号作为一种稳定的脑电信号, 备受关注. 研究者们通过深入探究睡眠脑电信号与抑郁症之间的关系, 发现其存在紧密关联. Steiger等人[11]的研究表明, 长期服用抗抑郁药物会对抑郁症患者的睡眠脑电信号特征产生显著影响, 这一发现为基于睡眠脑电信号的抑郁症诊断方法提供了坚实的理论基础. Tao 等人[12]则使用网络分析方法构建了抑郁、焦虑与睡眠障碍之间的相互作用关系, 证明三者具有一定的相关性. Yasugaki等人[13]的研究则指出抑郁症患者进入快速眼动(REM)睡眠的潜伏期缩短和非快速眼动睡眠期(NREM)间脑电图δ功率降低. Mirchandaney 等人[14]也指出失眠和夜间昼夜节律偏好是抑郁症的独特危险因素, 治疗睡眠障碍可以改善整体结果.
综上所述, 基于脑电信号的抑郁症诊断方法已经取得了一定的研究进展. 然而, 现有研究的准确率尚未达到临床应用的理想水平, 其次, 深度学习模型通常被视为“黑箱”操作, 其决策过程缺乏足够的透明度, 导致对诊断结果的解释性不足. 这种缺乏可解释性的特性使得医生和其他医疗专业人士难以完全信任模型输出的结果, 也阻碍了模型在临床实践中的广泛应用, 而睡眠分期则可以在一定程度上反映出抑郁症状, 有利于医疗人士分析其中原因. 因此, 我们受研究者们关于睡眠脑电和抑郁症关系的研究的启发, 进一步探索和完善了其中的方法, 提出了基于睡眠分期的抑郁症诊断方法, 为抑郁症的诊断和治疗提供更为准确、可靠的技术支持.
2 基于睡眠生理信号的抑郁症筛查算法本文基于睡眠脑电信号实现对抑郁症患者的筛查. 睡眠障碍是大多数抑郁症患者的典型症状, 其中, REM 潜伏期的缩短、REM 睡眠持续时间的增加和 REM 密度的增加被认为是抑郁症的生物学标志物[15]. 而 REM 期睡眠改变可能先于抑郁症的临床表现[16], 有助于抑郁症患者的识别与筛查. 因此, 本文使用REM期的睡眠脑电数据进行抑郁症的筛查. 为了提取REM期的睡眠脑电数据, 本文先使用CNN-BiLSTM网络模型对睡眠脑电数据进行睡眠分期. CNN-BiLSTM网络模型具有权值共享网络结构和对时间序列数据信息提取的优势, 能够构建准确的自动睡眠分期网络模型, 可以获得更为精确的REM期的睡眠脑电数据.
伪迹的存在可能影响基于睡眠脑电数据的抑郁症筛查的准确性, 且对于抑郁症患者的脑电信号数据, 可能存在多种不同的信号源, 因此, 本文使用基于独立成分分析的伪迹减少方法将多个信号源混合在一起的数据分离成原始的独立成分, 把分离后的独立成分用作特征提取.
睡眠脑电数据信号脑电图的每个频带都与大脑中的一些机制有关, 相关研究表明[17]抑郁症患者的相关脑电频段反映了其某些脑电区活动与正常人有所不同. 对此, 本文提出DepNet2D模型, 能够对REM期的睡眠脑电数据的频谱特征进行充分的特征提取, 挖掘其中隐藏的信息, 构建基于睡眠脑电频谱信息的二维网络模型, 以实现抑郁症的筛查.
2.1 基于CNN-BiLSTM 的睡眠分期 2.1.1 睡眠脑电数据的预处理本文使用的睡眠脑电数据来源于Sleep-EDF公开数据集, 该数据集在睡眠分期领域被广泛使用. 对睡眠脑电信号数据的预处理如下.
(1) 划分数据样本: 以帧(epoch)为判读的基本单位, 把连续30 s的睡眠数据划分为一个睡眠数据样本.
(2) 设置睡眠分期标签: 根据美国睡眠医学协会(American Academy of Sleep Medicine, AASM)提出的AASM睡眠分期标准[18], 把睡眠周期分为: 清醒期、N1期、N2期、N3期和REM期, 设置对应睡眠分期标签为0、1、1、1、2, 对应清醒期、深睡期和REM期3个阶段.
(3) 对睡眠脑电数据滤波处理: 为降低伪影的影响, 使用截止频率是0.1 Hz和50 Hz的巴特沃斯带通滤波器对2个通道的脑电信号滤波. 并对每5段滤波后的数据样本和判别标签构成一个组别, 构造成特征矩阵.
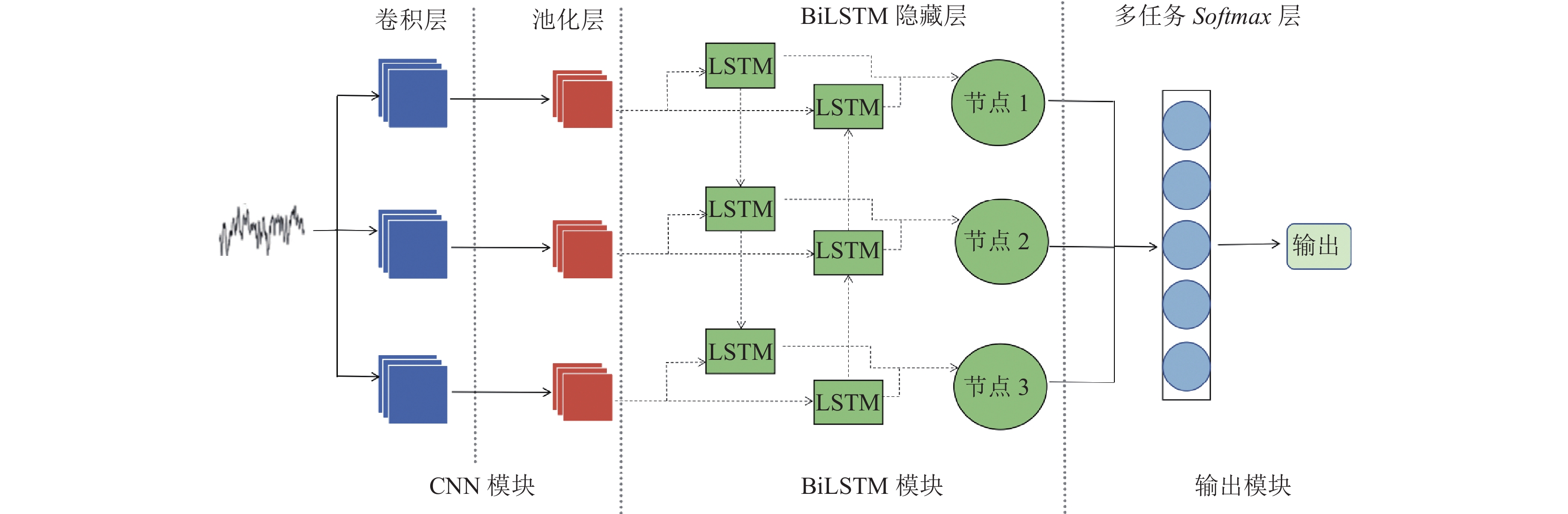
2.1.2 基于CNN-BiLSTM网络模型的睡眠分期由于睡眠脑电数据的非平稳性和非线性性, 对于睡眠脑电数据的频域特征以及从非线性过程派生的特征, 我们可以使用卷积神经网络(CNN)框架来处理这种联合分类问题[19], 提高模型的泛化性, 以实现睡眠脑电数据的特征提取. 而由于睡眠脑电数据在是时域上包含着大量的时间序列数据信息, 为了提取其中隐含的时域特征信息, 本文使用双向长短时记忆网络(BiLSTM), 能够更好捕捉双向时序数据信息的关联和依赖性. 对此, 本文使用CNN-BiLSTM 网络模型结合CNN特征提取和BiLSTM学习时序信息的优势. 这种结合能够非常有效地进行睡眠脑电数据的特征提取与睡眠分期. 本文使用的CNN-BiLSTM网络模型的结构图如图1所示.
|
图 1 CNN网络结构 |
本文使用的CNN-BiLSTM网络结构由4层组成: 卷积层、池化层、基于LSTM的BiLSTM隐藏层和多任务Softmax层, 这种体系结构有4个主要特征: 首先, 它的卷积层同时容纳3个不同大小的卷积核, 因此能够学习睡眠脑电数据不同分辨率上的特征, 并减少数据特征提取过程重新构建数据的繁杂, 有利于构建自动睡眠分期模型; 其次, 与常见的子采样池化相比, 池化层的
| $ {\textit{Softmax}}({\textit{z}}) = \frac{{\mathop {\mathrm{e}}\nolimits^{\mathop {\textit{z}}\nolimits_j } }}{{\displaystyle\sum\limits_{j = 1}^k {\mathop {\mathrm{e}}\nolimits^{\mathop {\textit{z}}\nolimits_j } } }} $ | (1) |
如式(1)所示, 对于输入向量
同时, 为解决睡眠数据类别不均衡问题, 本模型使用各个子任务的交叉熵误差之和作为训练的分类和预测误差, 如式(2)所示, 其中,
| $ \mathop E\nolimits^{(i)} (\theta ) = \sum\limits_{n = {n_i} - \mu }^{{n_i} + \mu } {y_n^{(i)}\log (\hat y_n^{(i)}(\theta ))} $ | (2) |
在临床上, 脑电频谱分析可用于诊断和监测各种疾病, 有利于客观的分析各种疾病, 提高可解释性与准确率. 抑郁症患者在脑电信号中可能表现出特定的频谱特征, 在特定脑区或特定频段出现异常的脑电活动模式[20]. 对此, 本文提出了DepNet2D模型来实现以抑郁症筛查的功能. DepNet2D模型充分利用REM期睡眠脑电数据的频谱信息, 构建频谱信息的二维网络模型, 能够从高维数据中提取脑电信号的隐藏信息, 有助于提取REM期睡眠脑电数据中的空间特征, 捕捉抑郁症患者大脑活动相关的特定频率范围或空间的特征模式. DepNet2D模型是一种广泛的数据学习方法, 能够将抑郁脑电图信号与健康对照的脑电图信号进行分类, 并它们返回最先进的精度的信号以实现提取特征. 同时, DepNet2D可以分析不同频段之间的关联性和互相影响, 对比正常人的脑电频谱信息, 可以有效地提取抑郁症患者的脑电频谱特征, 并根据识别的特征对抑郁症患者实现高精度筛查分类.
2.2.1 睡眠脑电数据的预处理本文使用分别记录了正常人的睡眠EEG和抑郁症患者的EEG两种不同的睡眠脑电信号. 图2是本文对睡眠脑电数据的预处理流程.

|
图 2 睡眠脑电数据预处理流程 |
首先取出REM期的原始睡眠脑电数据进行数据解析与数据转换, 使用零相位滤波器(FIR)把频率限制在0.5–45 Hz, 可以帮助减少不必要的噪音, 避免引起相位失真. 而伪迹的存在可能导致误解、不准确和扭曲的定量结果. 因此本文使用基于独立成分分析(ICA)的伪迹减少方法减少混合在一起的信号分离成独立的成分, 有效地提取出潜在的脑电活动源, 并可以减少伪迹的影响, 提高信号的质量和准确性, 使得睡眠时不同脑区神经活动模式表现更为显著. 最后以30 s的时间窗口对过滤后的REM-SLEEP-EEG进行裁剪, 再将脑电信号的频谱信息进一步提取, 最终提取得到REM期睡眠脑电数据的频谱信息.
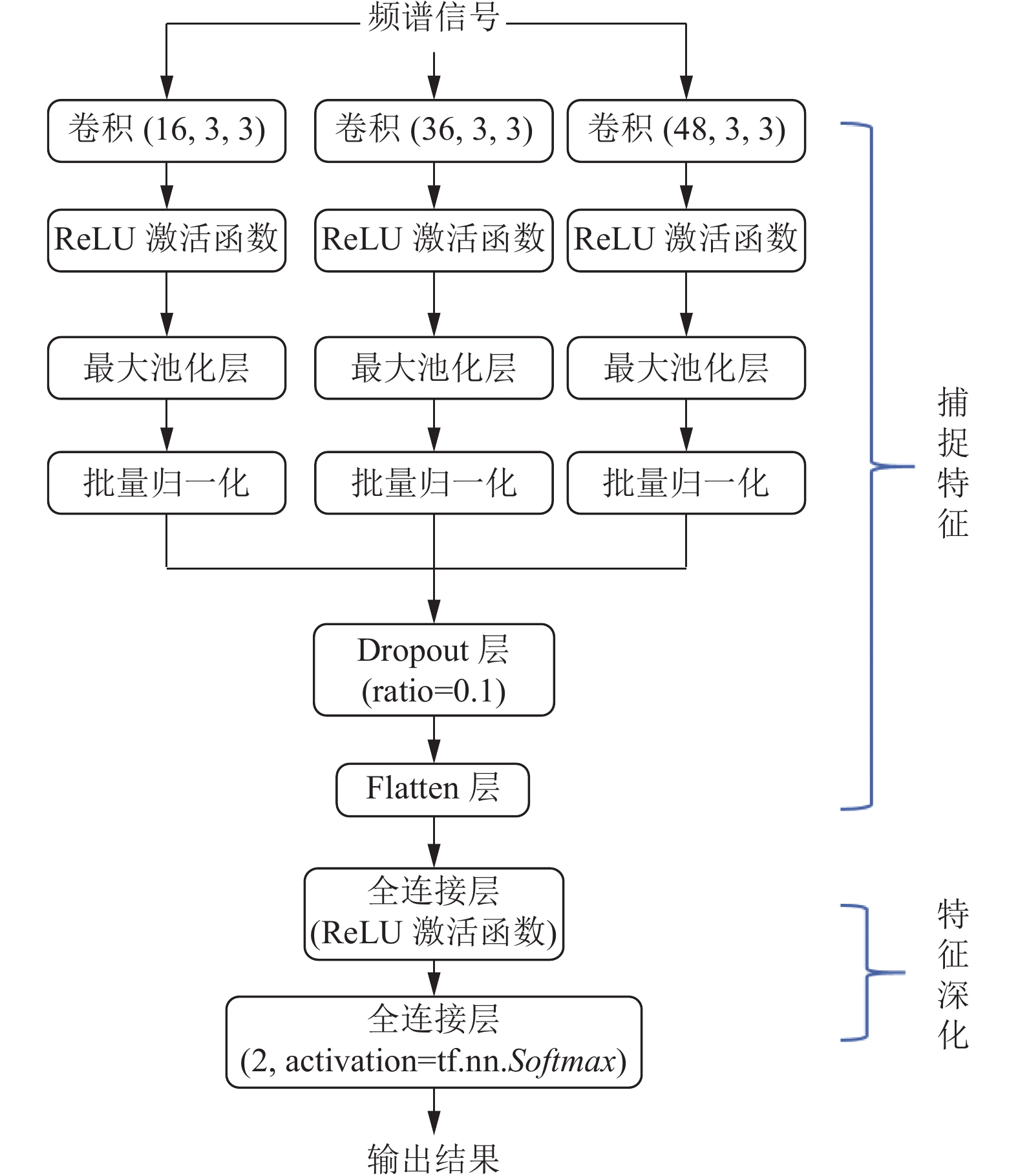
2.2.2 构建DepNet2D模型睡眠脑电频谱信息反映了大脑睡眠时不同脑区神经的活动模式, 涵盖了很多频率范围. 而抑郁症患者的睡眠脑电频谱信息中隐藏了大量与抑郁症相关联的信息. 在大量睡眠脑电信号数据面前, 对此, 本文提出的DepNet2D能基于二维空间有效地学习睡眠脑电频谱信息的空间特征, 更为方便地捕获局部空间上的相关性, 并能泛化到新的睡眠脑电信号样本. 相比于传统的卷积神经网络, 本文提出的DepNet2D模型能够充分利用频谱信息中包含的抑郁症患者在不同脑区的活动模式下的特征与联系, 对睡眠频谱信息进行高效地进行特征提取, 提高抑郁症筛查的准确率. DepNet2D模型构建如图3所示.
首先将提取出来的REM期的原始睡眠脑电的频谱信息, 取其中两个通道(EEG Fpz-Cz和EEG Pz-Oz), 将频谱信息输入到模型中, 输入层的大小为(150, 150, 3), 使用堆叠多个卷积层进行特征提取, 以构建出具有更强大表征能力的模型, 本文使用3个二维卷积(Conv2D), 第1个卷积层的卷积核个数为16, 第2个卷积层的卷积核个数为36, 第3个卷积层的卷积核个数为48, 3个卷积层的卷积核大小都为(3, 3). 在经过卷积层后, 分别通过ReLU激活函数和大小为(2, 2)的最大池化层(MaxPooling2D), 再进行批量归一化、降采样操作以减小特征图的大小, 并保留最显著的特征; 为了减少神经网络过拟合并提高模型的性能, 再把Dropout层(ratio=0.1)连接到每一个池化层之后. 连接Flatten层, 将卷积层和池化层提取的特征展平成一维向量. 最后经过两次全连接层(Dense), 通过这种多次全连接层的组合, 模型可以学习更高级别的特征表示, 并且将这些特征表示映射到输出类别的概率分布. 其中, 第1个全连接层具有64个神经元, 使用ReLU激活函数 学习输入数据的非线性特征表示, 在学习特征表示时更具表达能力. 第2个全连接层(Dense(2, activation=tf.nn.Softmax))具有 2 个神经元, 使用Softmax 激活函数, 将神经网络的输出转换为表示每个类别概率的分布.
|
图 3 模型构建图 |
DepNet2D模型的主要特征是通过3个卷积层和2个全连接层. 使用3个卷积层增加了模型的表征能力和特征提取的深度. 每个卷积层都可以学习不同级别和抽象程度的特征, 从而使模型能够更好地捕捉输入数据的非线性特征的复杂性和变化. 经过3个卷积层, 本模型可以逐渐学习到更高级别的特征表示. 而最后通过两个全连接层, 本模型能够对前面卷积和池化层提取的特征进行更深入的组合和抽象, 从而更好地区分不同类别, 从而提高模型对抑郁症筛查结果的准确性, 并把最后一层卷积层提取的特征表示映射到输出类别的概率分布上, 使得模型输出的结果更加直观和可解释.
本文使用二元交叉熵函数(binary cross-entropy loss)如式(3)所示, 对于二元分类, 该损失函数能够衡量模型输出的概率分布与真实标签之间的差异, 从而指导模型参数的优化过程. 通过使用Adam优化器, 能够根据每个参数的梯度的一阶矩估计和二阶矩估计动态调整学习率. 相比于传统的梯度下降算法, Adam具有更快的收敛速度和更好的性能表现. 并使用了准确率(accuracy)作为评估指标, 能够直观地反映模型的分类性能.
| $ L = - \frac{1}{N}\left\{ {\sum\limits_{i = 1}^N {\left[ {{t_j}\log ({p_i}) + (1 - {t_j})\log (1 - {p_j})} \right]} } \right\} $ | (3) |
对于
本文采用了Sleep-EDF数据库作为数据源, 该数据库收录了197个全夜的多参数睡眠记录(PSG), 覆盖了脑电图(EEG)、眼动图(EOG)、颏下肌电图及相关事件的标注. 其中部分资料还包含了呼吸和体温的记录. 所有睡眠阶段图表, 即催眠图, 都是根据Rechtschaffen与Kales的指南, 由接受过专业培训的技术人员手动进行评分的. 在1987–1991年间, 有一项研究收集了包含153个来自年龄介于25–101岁的健康白人的睡眠数据(被标记为SC, 即Sleep Cassette) 与22位健康的白人男性和女性的睡眠数据.
本实验设计并实施了一种结合了CNN和LSTM的混合架构算法, 专门针对两个EEG通道(EEG Fpz-Cz和EEG Pz-Oz)数据进行睡眠阶段分类. 通过对数据进行分段、生成频谱图、模型训练和性能评估等一系列步骤, 对模型的性能进行了全面测试. 选择了数据库中70%的数据作为训练集来训练神经网络模型; 15%的数据作为验证集, 用于模型性能的评估和迭代训练; 剩余的15%数据被用作测试集, 以评价模型的泛化能力.
3.1.2 评价方法为了验证本文方法的有效性, 采用了测试集上的分类结果准确率、召回率和精确率等作为性能评估指标. 旨在验证模型的准确性和泛化性能.该模型旨在基于两通道EEG数据对睡眠阶段进行分类, 分为清醒、NREM (非快眼动睡眠)和REM (快眼动睡眠). 具体的性能指标计算公式如下:
| $ ACC = \frac{{{N_{{\mathrm{true}}}}}}{{{N_{{\mathrm{predict}}}}}} $ | (4) |
其中,
| $ Recall = \frac{{TP}}{{TP + FN}} $ | (5) |
其中,
| $ Precision = \frac{{TP}}{{TP + FP}} $ | (6) |
其中,
通过这些指标, 可以全面评估模型在不同方面的性能, 包括其正确识别睡眠阶段的能力(准确率), 在识别特定睡眠阶段(如REM阶段)时的敏感性(召回率), 以及其预测的可靠性(精确率).
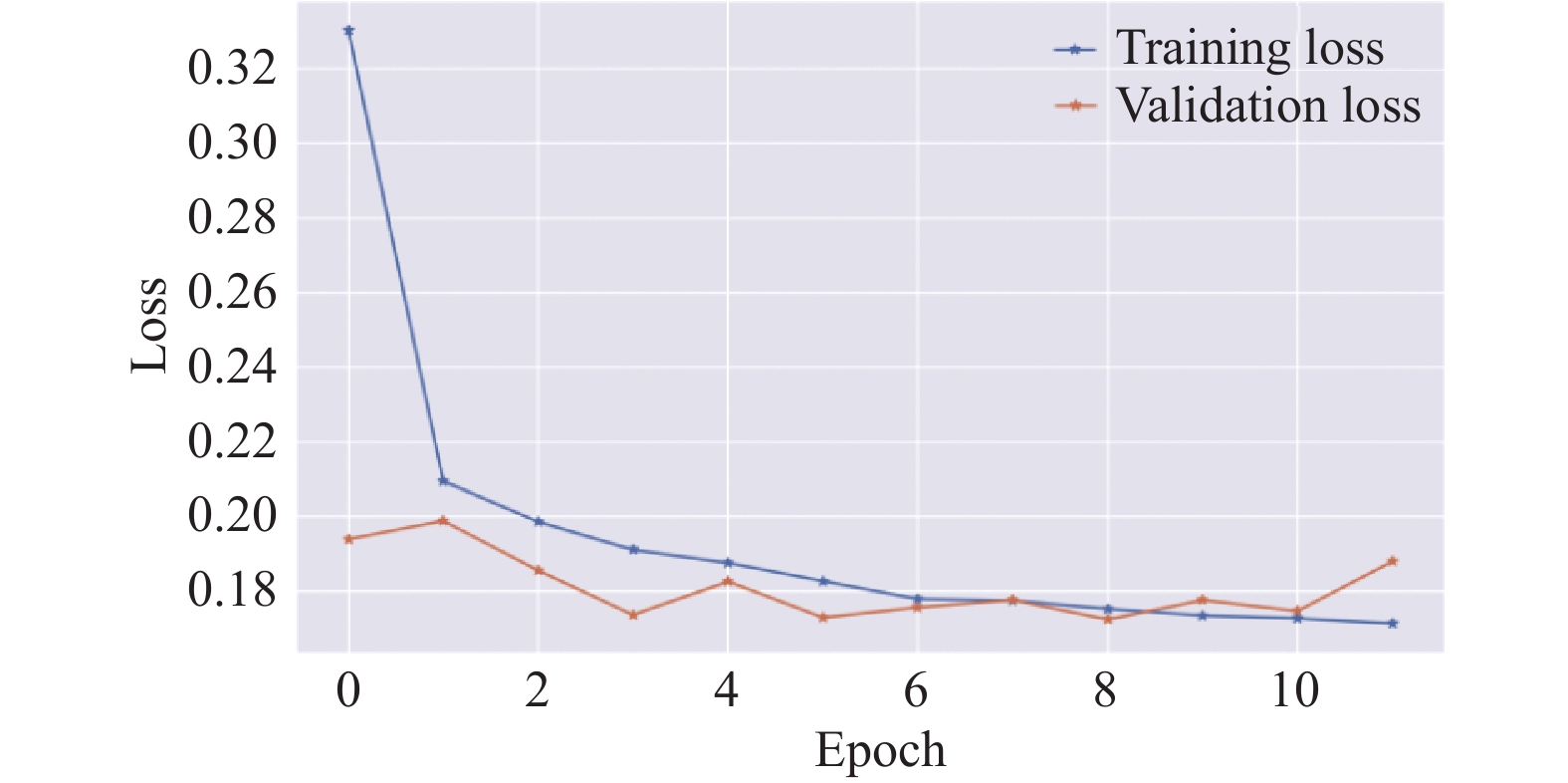
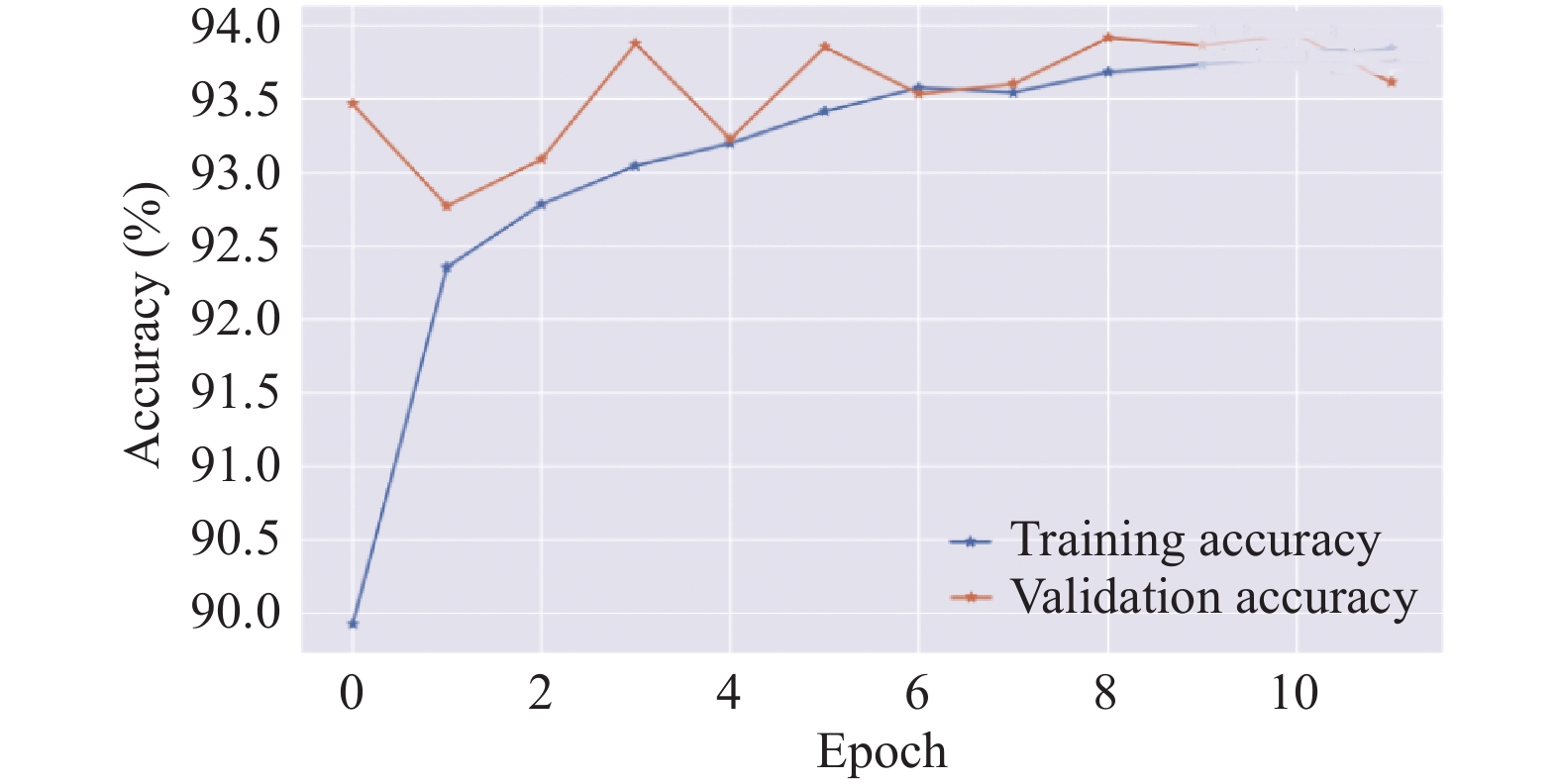
3.1.3 实验结果与分析在模型训练过程中, 15%的数据被用作验证集, 并引入了基于验证损失的早停机制以避免过拟合. 训练和验证过程中的损失值及准确率变化如图4和图5所示, 可以看出验证损失在第8轮后不再下降, 因此最终的模型基于第8轮的结果.
训练过程中的损失折线图(图4)展示了模型在不同训练周期(epochs)的训练损失和验证损失的变化情况. 这张折线图显示了一个模型在训练过程中的损失值变化, 包括训练损失(蓝色线)和验证损失(橙色线). 由图4可知, 在最初的几个训练周期(epochs)中, 训练损失迅速下降, 这表明模型在这个阶段快速学习. 随着训练的进行, 训练损失和验证损失都趋向于稳定. 两者的差距在第2–10个周期里相对较小, 这表明模型的泛化能力相对稳定, 没有显著的过拟合迹象. 在后期周期, 特别是从第8个周期开始, 两条线都显示出损失的稳定性, 训练损失和验证损失的变化不大.
|
图 4 训练过程中的损失折线图 |
而训练过程中的分类准确率折线图则显示了模型在训练和验证数据上的准确率百分比. 如图5所示, 这张折线图展示了模型在训练过程中的分类准确率, 包括训练准确率(蓝色线)和验证准确率(橙色线). 从图5中可以观察到, 在最初几个训练周期内, 训练准确率迅速提升, 这通常意味着模型在快速学习和适应训练数据. 随着更多训练周期的进行, 训练准确率和验证准确率都趋于稳定. 从第6个周期开始, 两者似乎收敛在93.5%准确率值附近. 在最后几个周期内, 两条线看起来非常接近, 均保持在93.75%上下, 这说明模型在训练和验证数据上具有一致的性能, 并且没有出现显著的过拟合现象.
|
图 5 训练过程中的分类准确率折线图 |
通过图4和图5, 我们可以直观地观察到模型在训练过程中损失的降低和准确率的提高, 这些都是模型学习的积极迹象. 同时模型也没有出现过拟合的情况, 即训练损失持续下降而验证损失不降反升的情况. 而对比训练和验证曲线, 选择最佳的模型参数和停止训练的时机, 模型可以在未见数据上有着良好的泛化能力.
如图6所示, 混淆矩阵可以帮助理解模型在每个类别上的表现, 包括正确和错误预测的次数. 在此混淆矩阵中有3个类别: 清醒(Wake)、非快速眼动睡眠(NREM)、和快速眼动睡眠(REM). 模型正确预测了
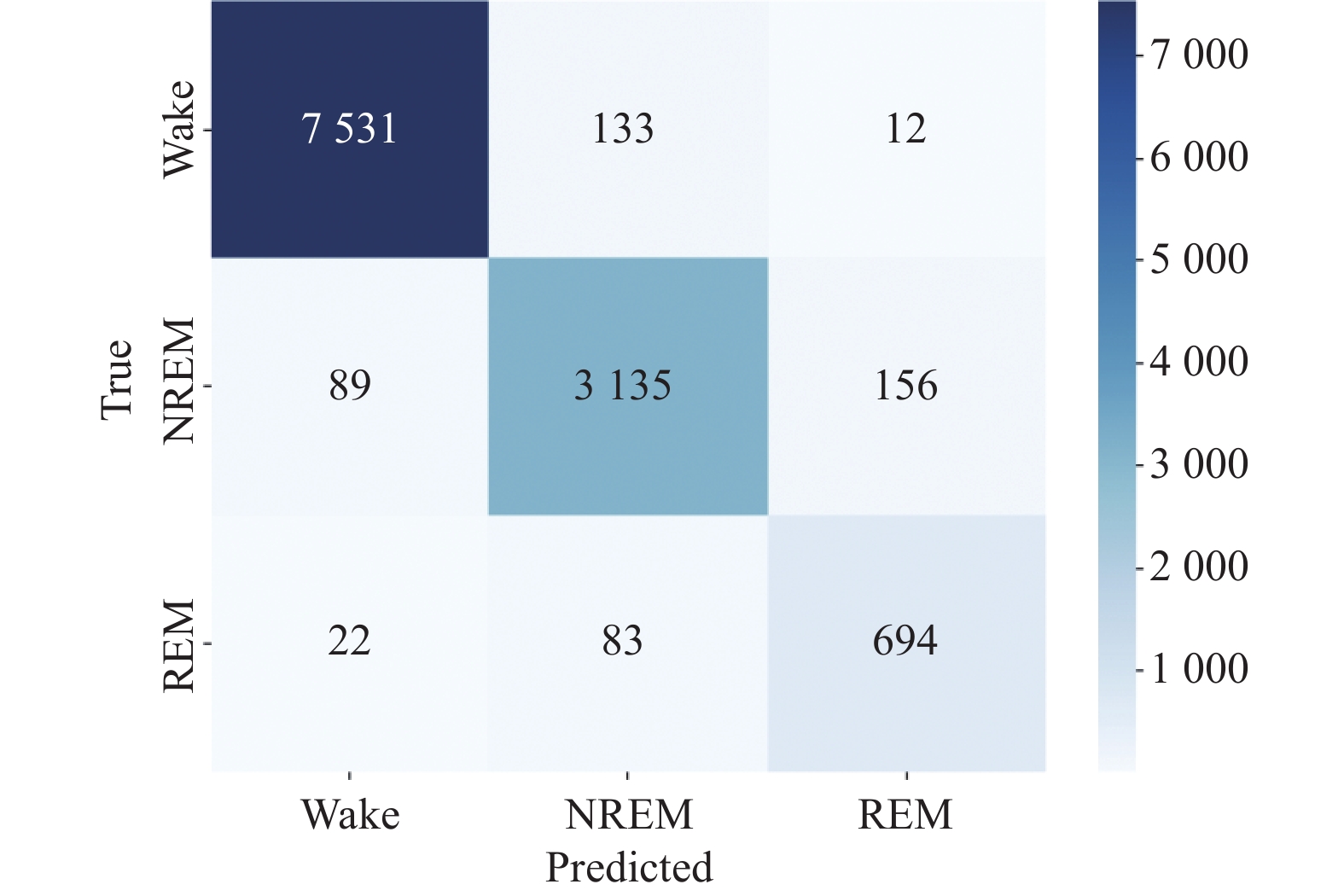
|
图 6 睡眠分期任务混淆矩阵 |
| 表 1 模型性能表 (%) |
这些结果表明, 该模型在Wake和NREM阶段的精确率和召回率都相当高. 综上, 模型在训练过程中表现出了良好的学习能力, 并且泛化到验证集上也表现出了一定的稳健性. 为了全面评价本文提出的睡眠分期方法的先进性. 本文还比较了近年来不同学者在同一数据集上的表现. 这一比较涉及多个不同的模型, 包括基于不同特征提取技术和分类器的方法, 以及它们在分类睡眠阶段任务上的准确率指标, 以此与本文模型性能进行对比. 相关的结果如表2所示.
从表2中可知, 本文开发的睡眠分期模型展示了卓越的性能, 在测试数据集上达到了95.82%的准确率, 本研究的测试准确率已在表2中加粗. 这一结果不仅显著优于当前大多数先进方法, 而且还进一步证实了本文模型在处理睡眠数据上的有效性. 以基于时间注意力机制结合Bi-GRU的自动睡眠分期算法为例, 该模型在Sleep-EDF数据集上实现了92.18%的准确率, 而本文研究的模型相比之下提高了3.64%. 此外, XGBoost算法在睡眠分期任务中的表现也非常出色, 准确率达到93.1%. 尽管这一结果与本文模型的性能相当接近, 但本文模型在准确率上仍有近2.72%优势. 这些比较结果说明我们的CNN-BiLSTM架构能够更有效地从EEG信号中提取和学习睡眠阶段的区分特征. 与近年提出的方法相比, 本文提出的深度学习方法提供了更深层次的特征表示和更复杂的决策边界, 从而提高了分类的准确性.
| 表 2 2021年以后常见分类算法睡眠分期效率 (%) |
3.2 抑郁症分类实验结果 3.2.1 实验数据及说明
这项研究中, 使用来自 Mumtaz等人[29]收集的实验数据, 包括34名MDD患者(其中女性18人, 平均年龄40.33岁, 标准偏差±12.861)和30名健康受试者(其中女性9人, 平均年龄38.227岁, 标准偏差±15.64)的脑电数据. 根据诊断和统计手册-IV (DSM-IV), 符合MDD诊断标准的MDD患者是从马来西亚理科大学医院(HUSM)的门诊招募的. MDD患者已通过单相抑郁症的诊断标准, 没有任何精神病症状. MDD患者和健康参与者都是志愿者, 并签署了参与同意书. 医院伦理委员会已经批准了向参与者简要介绍的实验设计. 对健康参与者的临床症状进行了检查, 以排除任何身体和精神残疾的可能性, 并确认正常.
在这项实验中, 我们利用睡眠分期中判别为REM期的脑电信号来对模型进行测试. 从数据集中选择了70%的数据作为训练集, 用于训练神经网络模型; 15%的数据作为验证集, 用于评估模型的表现并进行迭代训练; 最后, 剩下的15%的数据被用作测试集, 用于评估模型的泛化能力.
3.2.2 实验结果与分析本文采用CNN-BiLSTM对数据集进行睡眠分期, 并筛选REM期的脑电信号片段, 将其输入DepNet2D网络结构实现分类, 并利用测试集作对训练完成的模型进行精度监测, 最终预测结果的混淆矩阵如图7所示.
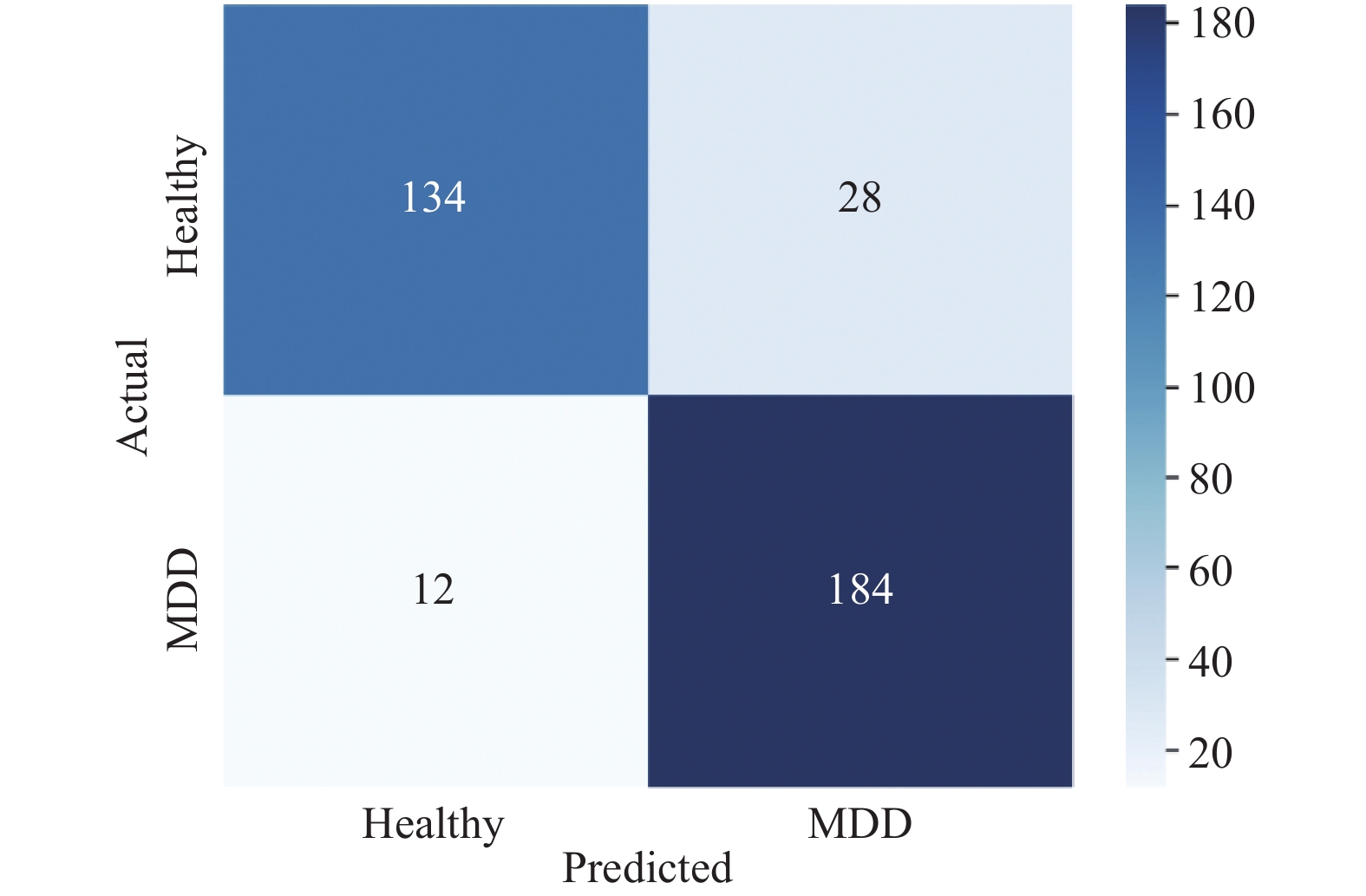
|
图 7 抑郁症分类任务混淆矩阵 |
混淆矩阵在分类任务中扮演着重要角色, 它能够直观地展示模型的分类准确性. 对于二分类任务而言, 混淆矩阵是一个2×2的矩阵, 其中对角线上的元素表示正确分类的样本数, 非对角线上的元素表示错误分类的样本数. 从图7可得测试集中实际为健康状态的数据共有162个, 模型将其中28个数据分类为抑郁症状态; 测试集中实际为抑郁症状态的数据共有196个, 模型将其中12个数据分类为健康状态. 从图中可以看出, 模型对于大部分数据都能够准确的预测. 从混淆矩阵中计算得出, 模型在该分类任务中, 准确率能达到88.82%, 除此之外, 还得到了模型的错误率、召回率、精确率, 通过这些指标, 能更直观地展示模型的有效性. 其结果如表3所示.
| 表 3 模型性能表 (%) |
实验结果表明在本文提出的抑郁症分类模型中, 准确率达到88.82%, 错误率为11.17%, 召回率为94.90%, 精确率达86.11%, 该结果说明本文提出的抑郁症检测方法拥有较高的分类性能, 同时也表明模型的泛化能力较强. 目前通过EEG信号进行抑郁症分类的研究已经受到广泛关注, 为了能够更加全面的分析本文的抑郁症分类方法是否具有先进性, 本文还对比了其他学者提出的算法以及一些常见深度学习分类模型在本数据集上的表现, 并列出其准确率指标, 以便与本文的模型进行了对比分析, 如表4所示.
| 表 4 常见分类模型在抑郁症领域分类效率 (%) |
从表4的实验结果可以看到, 与另外9种算法相比, 本文方法获得了最高的平均准确率88.82%, 与其他在Mumtaz数据集的实验结果相比, 本文方法取得了更高的平均准确率, 说明随着数据集的增大, 模型能够学习到更多的特征. 但值得注意的是, DenseNet169、ResNet152以及Xception在Mumtaz数据集取得了更低的平均识别率, 这可能是因为所选模型没有能力学习脑电信号的复杂特征, 或者发生过拟合现象而导致的. 从上述分析可以得出, 我们的研究方法具有较高的准确性, 所提出的模型在抑郁症分类领域有一定的参考价值.
4 结语本文基于目前EEG信号在抑郁症分类上的应用以及关于睡眠与抑郁症的研究, 提出基于睡眠分期的抑郁症诊断方法, 为今后抑郁症的客观诊断提供了一种新的解决方案. 使用在Sleep-EDF数据集得到验证的CNN-BiLSTM网络实现睡眠分期, 并筛选出REM期的脑电数据输入本文搭建的DepNet2D网络中进行抑郁症分类, 在公共数据集中进行实验并进行详细的可视化分析, 最终结果表明, 本文方法对比目前许多现有方法具有更高的分类准确率.
在未来的工作中, 我们计划引入更多有关睡眠脑电信号的抑郁症数据进行研究, 进一步优化算法模型, 并提升算法的泛化性能.
| [1] |
陈贵伟, 白银霞. 儿童青少年抑郁症现况调查. 临床医学进展, 2024, 14(2): 3611-3615. |
| [2] |
熊馨, 杨鑫亮, 罗剑花, 等. 睡眠呼吸暂停综合症患者脑电微状态发生改变. 中国生物医学工程学报, 2023, 42(5): 563-571. DOI:10.3969/j.issn.0258-8021.2023.05.006 |
| [3] |
王域枫, 冯伟, 杭文龙. 基于自适应多视角深度神经网络的脑电识别. 计算机应用与软件, 2024, 41(1): 97-104. DOI:10.3969/j.issn.1000-386x.2024.01.015 |
| [4] |
Acharya UR, Oh SL, Hagiwara Y, et al. Automated EEG-based screening of depression using deep convolutional neural network. Computer Methods and Programs in Biomedicine, 2018, 161: 103-113. DOI:10.1016/j.cmpb.2018.04.012 |
| [5] |
Li X, La R, Wang Y, et al. EEG-based mild depression recognition using convolutional neural network. Medical & Biological Engineering & Computing, 2019, 57(6): 1341–1352.
|
| [6] |
Kwon H, Kang S, Park W, et al. Deep learning based pre-screening method for depression with imagery frontal EEG channels. Proceedings of the 2019 International Conference on Information and Communication Technology Convergence (ICTC). Jeju: IEEE, 2019. 378–380.
|
| [7] |
卢官明, 丛文康, 魏金生, 等. 基于CNN和LSTM的脑电信号情感识别. 南京邮电大学学报(自然科学版), 2021, 41(1): 58-64. |
| [8] |
李梅, 宁德军, 郭佳程. 基于注意力机制的CNN-LSTM模型及其应用. 计算机工程与应用, 2019, 55(13): 20-27. DOI:10.3778/j.issn.1002-8331.1901-0246 |
| [9] |
Qayyum A, Razzak I, Mumtaz W. Hybrid deep shallow network for assessment of depression using electroencephalogram signals. Proceedings of the 27th International Conference on Neural Information Processing. Bangkok: Springer, 2020. 245–257.
|
| [10] |
Spencer BF, Hoskere V, Narazaki Y. Advances in computer vision-based civil infrastructure inspection and monitoring. Engineering, 2019, 5(2): 199-222. DOI:10.1016/j.eng.2018.11.030 |
| [11] |
Steiger A, Kimura M. Wake and sleep EEG provide biomarkers in depression. Journal of Psychiatric Research, 2010, 44(4), 242–252.
|
| [12] |
Tao YQ, Hou WX, Niu HQ, et al. Centrality and bridge symptoms of anxiety, depression, and sleep disturbance among college students during the COVID-19 pandemic—A network analysis. Current Psychology, 2024, 43(15): 13897-13908. DOI:10.1007/s12144-022-03443-x |
| [13] |
Yasugaki S, Okamura H, Kaneko A, et al. Bidirectional relationship between sleep and depression. Neuroscience Research. https://www.sciencedirect.com/science/article/pii/S0168010223000871. (published online). [doi: 10.1016/j.neures.2023.04.006]
|
| [14] |
Mirchandaney R, Asarnow LD, Kaplan KA. Recent advances in sleep and depression. Current Opinion in Psychiatry, 2023, 36(1): 34-40. DOI:10.1097/YCO.0000000000000837 |
| [15] |
Pandi-Perumal SR, Monti JM, Burman D, et al. Clarifying the role of sleep in depression: A narrative review. Psychiatry Research, 2020, 291: 113239. DOI:10.1016/j.psychres.2020.113239 |
| [16] |
Arıkan MK, Uysal Ö, Gıca Ş, et al. REM parameters in drug-free major depressive disorder: A systematic review and meta-analysis. Sleep Medicine Reviews, 2024, 73: 101876. DOI:10.1016/j.smrv.2023.101876 |
| [17] |
de Aguiar Neto FS, Rosa JLG. Depression biomarkers using non-invasive EEG: A review. Neuroscience & Biobehavioral Reviews, 2019, 105: 83-93. |
| [18] |
Berry RB, Brooks R, Gamaldo C, et al. AASM scoring manual updates for 2017 (version 2.4). Journal of Clinical Sleep Medicine, 2017, 13(5): 665-666. DOI:10.5664/jcsm.6576 |
| [19] |
Mahmud T, Khan IA, Mahmud TI, et al. Sleep apnea detection from variational mode decomposed EEG signal using a hybrid CNN-BiLSTM. IEEE Access, 2021, 9: 102355-102367. DOI:10.1109/ACCESS.2021.3097090 |
| [20] |
Keren H, O’Callaghan G, Vidal-Ribas P, et al. Reward processing in depression: A conceptual and meta-analytic review across fMRI and EEG studies. American Journal of Psychiatry, 2018, 175(11): 1111-1120. DOI:10.1176/appi.ajp.2018.17101124 |
| [21] |
Feng LX, Li X, Wang HY, et al. Automatic sleep staging algorithm based on time attention mechanism. Frontiers in Human Neuroscience, 2021, 15: 692054. DOI:10.3389/fnhum.2021.692054 |
| [22] |
Liu C, Tan B, Fu MY, et al. Automatic sleep staging with a single-channel EEG based on ensemble empirical mode decomposition. Physica A: Statistical Mechanics and its Applications, 2021, 567: 125685. DOI:10.1016/j.physa.2020.125685 |
| [23] |
Zhao RQ, Xia Y, Zhang YL. Unsupervised sleep staging system based on domain adaptation. Biomedical Signal Processing and Control, 2021, 69: 102937. DOI:10.1016/j.bspc.2021.102937 |
| [24] |
Wang H, Guo HB, Zhang K, et al. Automatic sleep staging method of EEG signal based on transfer learning and fusion network. Neurocomputing, 2022, 488: 183-193. DOI:10.1016/j.neucom.2022.02.049 |
| [25] |
He ZL, Du LD, Wang P, et al. Single-channel EEG sleep staging based on data augmentation and cross-subject discrepancy alleviation. Computers in Biology and Medicine, 2022, 149: 106044. DOI:10.1016/j.compbiomed.2022.106044 |
| [26] |
Fu MY, Wang YT, Chen ZX, et al. Deep learning in automatic sleep staging with a single channel electroencephalography. Frontiers in Physiology, 2021, 12: 628502. DOI:10.3389/fphys.2021.628502 |
| [27] |
Fiorillo L, Favaro P, Faraci FD. DeepSleepNet-Lite: A simplified automatic sleep stage scoring model with uncertainty estimates. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2021, 29: 2076-2085. DOI:10.1109/TNSRE.2021.3117970 |
| [28] |
Jiang X, Zhao JH, Du B, et al. Self-supervised contrastive learning for EEG-based sleep staging. Proceedings of the 2021 International Joint Conference on Neural Networks (IJCNN). Shenzhen: IEEE, 2021. 1–8.
|
| [29] |
Mumtaz W, Xia LK, Ali SSA, et al. Electroencephalogram (EEG)-based computer-aided technique to diagnose major depressive disorder (MDD). Biomedical Signal Processing and Control, 2017, 31: 108-115. DOI:10.1016/j.bspc.2016.07.006 |
| [30] |
Mahato S, Paul S. Detection of major depressive disorder using linear and non-linear features from EEG signals. Microsystem Technologies, 2019, 25(3): 1065-1076. DOI:10.1007/s00542-018-4075-z |
| [31] |
Mumtaz W, Xia LK, Mohd Yasin MA, et al. A wavelet-based technique to predict treatment outcome for major depressive disorder. PLoS One, 2017, 12(2): e0171409. DOI:10.1371/journal.pone.0171409 |
| [32] |
Li XW, La R, Wang Y, et al. EEG-based mild depression recognition using convolutional neural network. Medical & Biological Engineering & Computing, 2019, 57(6): 1341-1352. |
 2024, Vol. 33
2024, Vol. 33


