生物医学命名实体识别(biomedical named entity recognition, BioNER)[1]和命名实体规范化(biomedical named entity normalization, BioNEN)[2]是生物医学文本挖掘的关键任务, 分别用于从文献中识别实体和将实体映射到标准化概念. 这些基础任务对医学研究和临床决策至关重要. 由于传统的流水线模型存在错误传播和灵活性不足的问题, 因此联合模型越来越受到重视, 它通过一个统一框架同时处理两个任务, 能减少错误传播并提升处理效率. 然而, 实际应用中联合模型仍然面临任务关联性和组件独立性等挑战.
生物医学命名实体识别和规范化是两个互补的过程, 它们共同构成了从生物医学文本中提取和利用关键信息的基础. 为此, 本文设计了一个联合标注方案, 可以同时编码实体信息和相应的概念信息, 将两种信息结合起来共同标注生物医学文本中的实体. 在此基础上, 本文提出了一种面向生物医学命名实体识别和规范化地多粒度融合模型(multi-granularity feature fusion approach for biomedical named entity recognition and normalization, MGFFA). MGFFA模型通过整合字符级、词级、概念级的文本信息, 显著增强了模型的学习能力. 模型包含一个动态更新的记忆库, 用于存储和综合不同层次信息, 以实现对实体及其规范化标签间复杂关系的深入理解. 通过预训练模型BioBERT和CharBERT的配合使用, MGFFA不仅捕捉了文本的粗粒度语义表示, 还细致分析了构词层面的特征, 并融合了概念级信息, 从而全面提升了对长跨度实体的识别准确率.
1 相关工作 1.1 生物医学命名实体识别近年来, 生物医学命名实体识别任务主要围绕循环神经网络[3]、长短期记忆网络[4]、条件随机场[5]等模型结构展开. Guan等人[6]提出了一个名为BioByGANS的生物医学命名实体识别模型, 该模型结合了前缀特征和注意力图判别融合技术, 旨在提高命名实体识别任务的性能. 通过实验验证, 该方法在多个数据集上均表现出了优越的性能, 为生物医学领域的命名实体识别任务提供了新的解决方案. Malmasi等人[7]构建了一个MultiCoNER的大规模多语言数据集, 用于命名实体识别. MultiCoNER数据集的构建旨在提供一个广泛覆盖多语言的资源, 以促进复杂命名实体识别模型的发展和评估. 该数据集为研究人员提供了丰富的语言材料, 以支持多语言命名实体识别任务的进展. Li等人[8]针对NER任务提出了一种新的方法W2NER, 将其建模为词与词之间的关系分类任务. 实验证明, 该方法在多个基准数据集上均表现出优于基线模型的性能, 为命名实体识别任务的研究提供了新的思路和解决方案.
1.2 生物医学命名实体规范化生物医学命名实体规范化是自然语言处理领域的另一个关键任务. 传统方法依赖于领域特定的词典[9]、手工制定的规则以及特征工程[10,11], 而最近深度学习方法因其强大的特征自动提取能力被广泛应用. Mehmood等人[12]提出了一种多任务模型MTM-CNN, 用于提高生物医学实体规范化任务的性能, 强调了知识转移技术在生物医学NER中的重要性, 以及利用外部资源和领域知识的关键性. 此外, 该模型还针对每个特定数据集进行了微调, 以将其从通用特征表示定制为专用特征表示. Sung等人[13]提出了一种用于实体规范化的模型BioSyn, 通过纳入同义词信息来增强生物医学实体的表示. 通过边缘化同义词的影响, 模型可以生成更健壮和准确的生物医学实体表示. Peng等人[14]也提出了一种用于实体规范化的模型IA-BIOSYN. 该模型基于交互式同义词边缘化的方法, 将多个语义特征整合在一起, 以增强模型捕获实体同义性的能力. 该模型在NCBI-Disease、BC5CDR-Disease和BC5CDR-Chemical数据集上验证了其优秀的性能表现, 并优于其他基线模型.
1.3 联合生物医学命名实体识别和规范化随着技术的发展, 越来越多的研究开始使用联合模型来克服流水线模型的弊端. Zhou等人[15]研究提出了一种名为MTAAL的模型, 结合了多任务学习和对抗性主动学习. MTAAL模型通过对抗性主动学习, 模型能够有效地利用标签有限的数据来训练, 并且在命名实体识别和规范化任务上取得了显著的性能提升. 在Zhou等人[16] 的进一步研究中, 提出了一个名为E2EMERN的端到端渐进式多任务学习模型. 该模型采用了渐进式的多任务学习方法, 通过逐步学习不同任务的相关知识, 逐步提升模型的性能. 实验结果表明, 该模型在医学命名实体识别和规范化任务上取得了显著的性能提升. Ji等人[17] 提出的NeuJoRN模型采用了神经过渡式联合框架, 将生物医学命名实体识别和规范化任务转换为一系列动作预测任务, 并通过注意力机制充分利用词汇表中每个候选概念的详细信息, 有效地支持了命名实体是被任务. 另一方面, Zhao等人[18]提出了一种神经多任务学习模型MTL-MEN&MER_feedback, 用于医学命名实体识别和规范化的联合建模. 该方法采用循环神经网络(RNN)和门控循环单元(GRU), 并利用多任务学习策略来整合实体识别和规范化任务, 同时引入了共享的表示层和任务特定的输出层. 这种方法在多个医学领域的基准数据集上取得了显著的性能提升.
2 方法 2.1 联合标注方案为了从文本中提取精确且一致的生物医学信息, 生物医学命名实体识别和规范化任务的融合显得尤为重要. 本文提出了一种独特的标注方案, 旨在同时编码实体提及及其对应的规范概念, 将BioNER和BioNEN的联合任务转化为一个序列标注问题[19]. 联合标注方案不仅能够消除级联错误, 还加强了BioNER和BioNEN两个任务的紧密结合, 使得模型能够更好地识别出实体, 并将其准确地映射到概念标签上.
图1 给出了所提出的联合标注方案的示例. 这一方案中, B-Disease、E-Disease和S-Disease等实体标签用于指示命名实体提及的位置和类型; 而D013921和D06493等概念标签则作为来自参考词典的概念标识符. 通过为实体的每个Token分配一个概念标签, 并将其与相应的实体标签进行连接, 这种方法成功地集成了实体位置信息和概念标识符于单一的联合标签中. 以图1中词元HIT为例, 联合标签B-D013921表示该词元是在提及概念D013921的开头. 同时, 为了使联合标签更加的简洁, 本节省去了类型Disease, 因为其已经包含在了概念标识符中了.
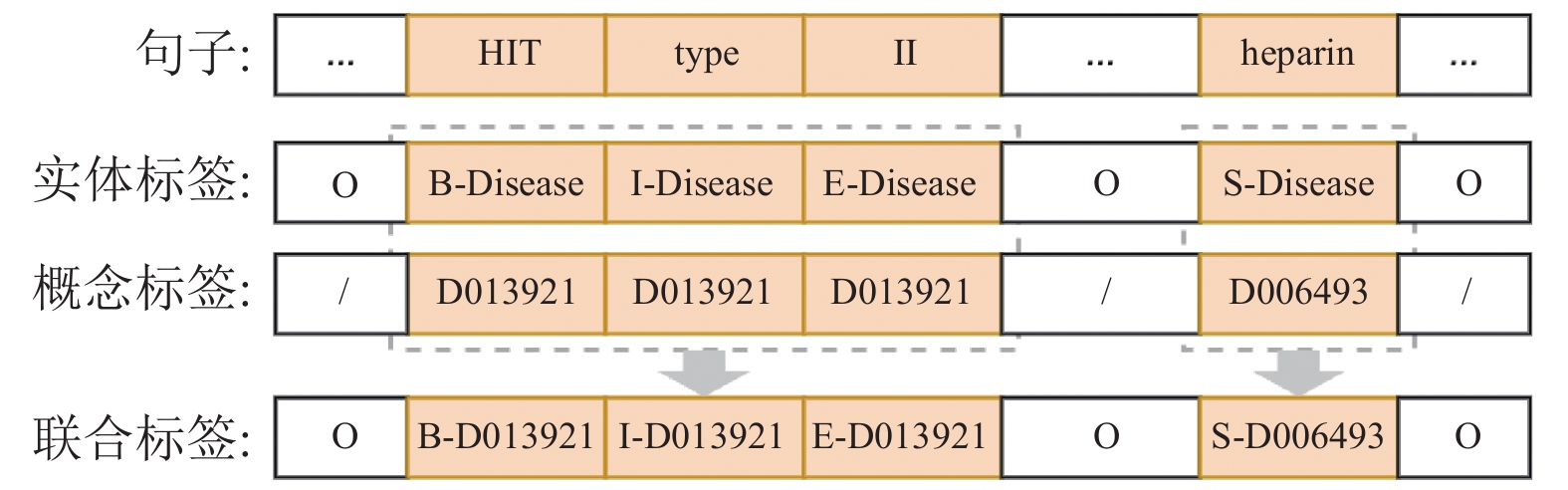
|
图 1 联合标注方案示例 |
2.2 多粒度特征融合方法(MGFFA)
本文提出的MGFFA模型用于生物医学命名实体识别和规范化的联合抽取任务. 如图2所示, MGFFA模型主要由以下几个模块组成: 单词表示模块、字符表示模块、多粒度融合模块、记忆模块和标签解码模块, 本节将对这几个模块进行详细介绍.
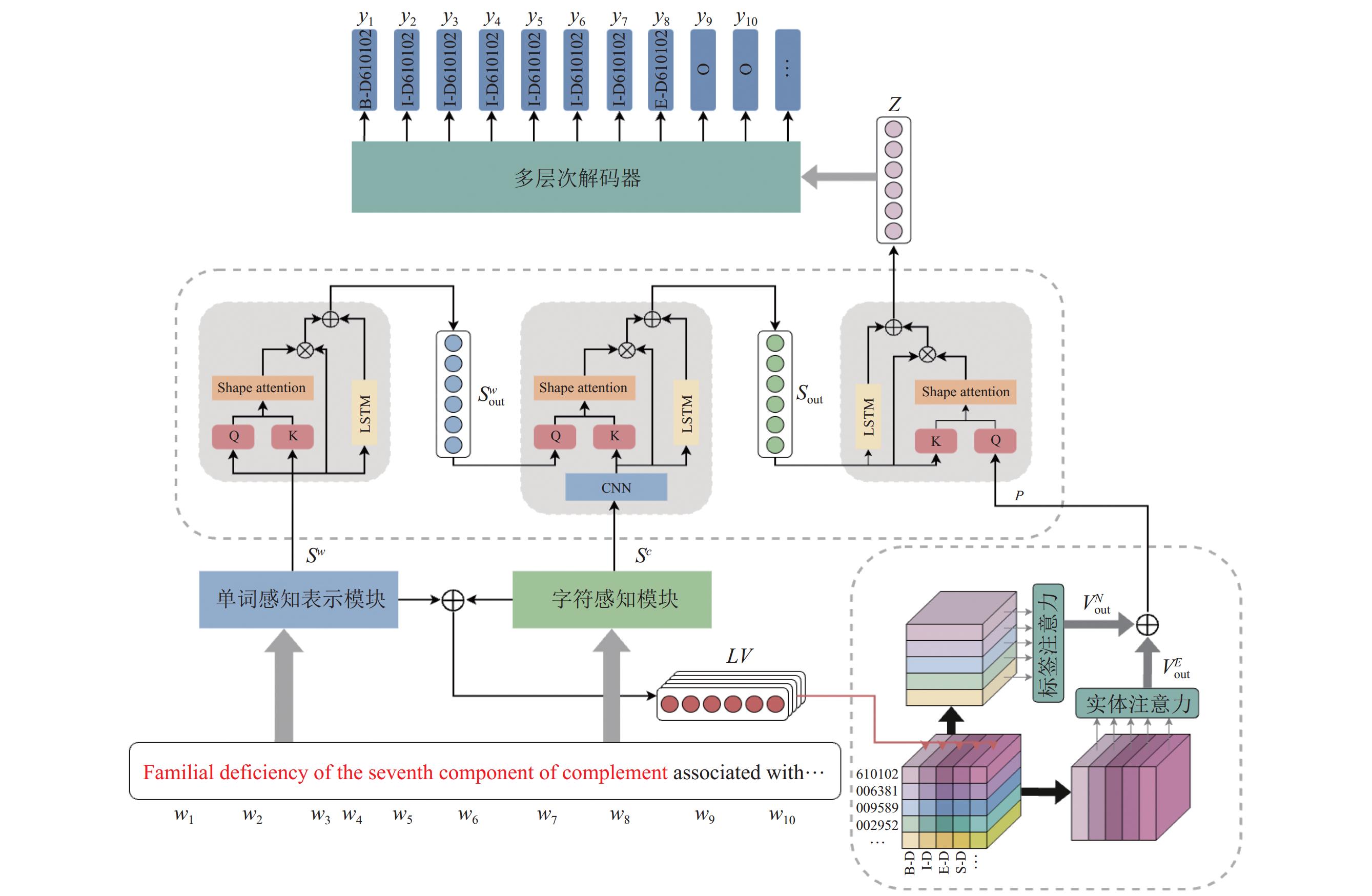
|
图 2 MGFFA整体模型架构 |
2.2.1 单词和字符表示模块
成功的神经序列标注模型很大程度上依赖于输入序列的综合表示. 考虑到基于 BERT[20] 的预训练语言模型在多种任务上所展现的卓越性能[21,22], 本文选择了BioBERT[23]作为输入句子的基础编码器. BioBERT是一种专门在生物医学文本上预训练的BERT模型, 能够通过捕获文本中单词间的上下文依赖关系, 构建出丰富的单词表示. 然而, 生物医学文本中的单词构成通常遵循特定的规则, 如前缀、后缀和词根对于理解生物医学术语的语义至关重要. 传统的BERT系统, 包括BioBERT在内, 往往难以充分捕获这种构词学特征. 鉴于此, 本文进一步引入了一种字符感知的预训练语言模型CharBERT[24], 它通过融合字符级信息, 产生更为细粒度的单词表示.
在MGFFA模型中, 每个句子被赋予两种类型的表示: 单词感知表示和字符感知表示. 具体来说, 对于一个包含
记忆模块是一种外部的记忆存储器, 可以被显式地读写[25,26], 使得模型能够灵活地存储和检索信息. 记忆模块使用更加轻量化的设计, 在联合标签矩阵生成以后, 将其在纵向和横向方面融合, 生成实体记忆部分和标签记忆部分.
(1) 联合标签矩阵. 联合标签是由实体标签和规范化标签组成, 如图3所示. 例如, 联合标签B-D610102表示编号610102的疾病实体开头, 可以将其拆分为实体表示B-D和规范化表示610102, 并嵌入到坐标为
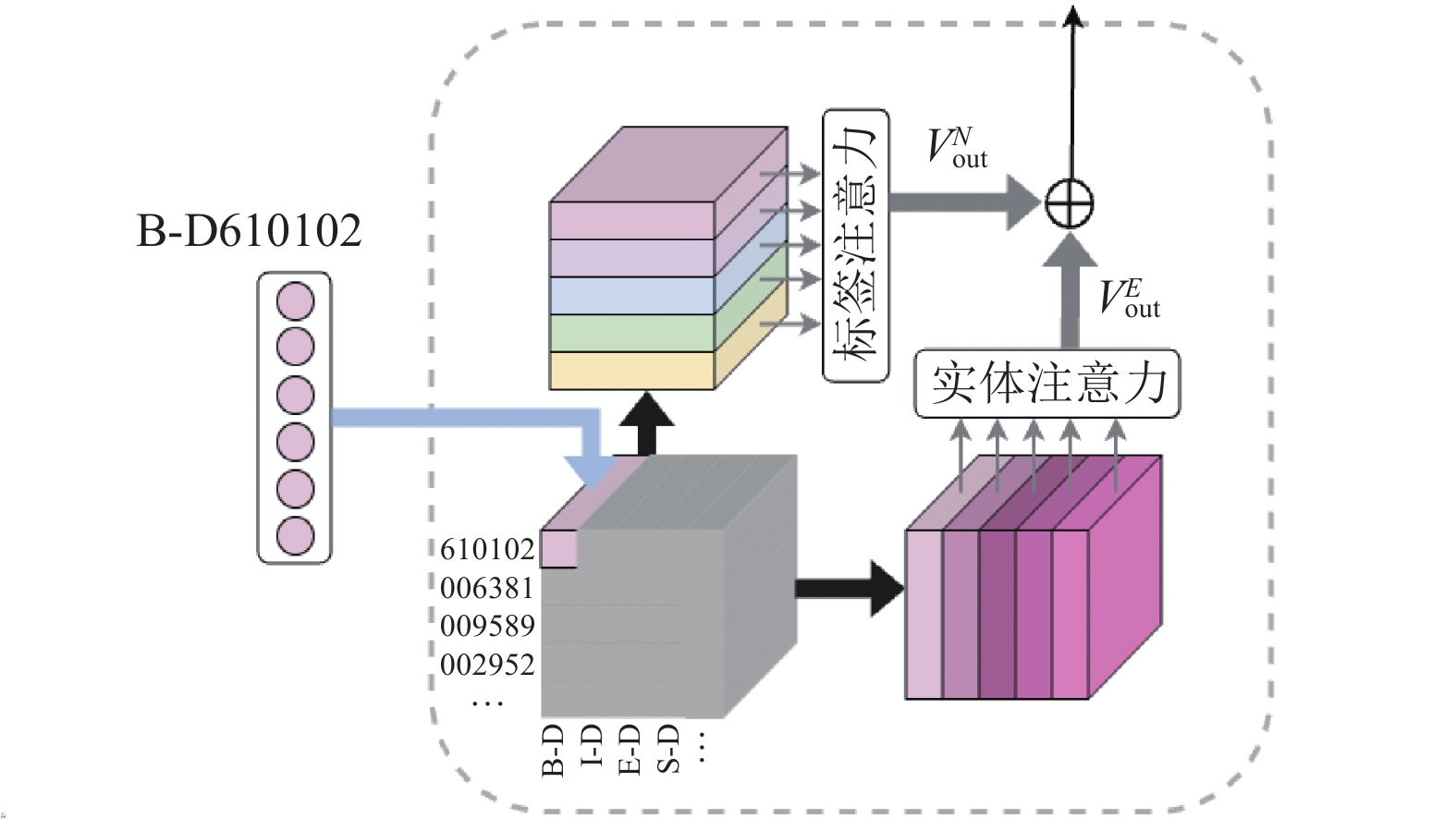
|
图 3 记忆标签 |
本质上联合标签矩阵
| $ LV = {S^w} \oplus {S^c} $ | (1) |
其中,
(2) 实体记忆部分. 联合标签矩阵在纵向上形成
| $ \left\{\begin{gathered} T_i^E = {\mathit{Softmax}}\left( {\frac{1}{{\sqrt {{d_k}} }}V_i^E{W_i^Q}({{W_i^K})^{{\mathrm{T}}}}(V{{_i^E})^{{\mathrm{T}}}}} \right) \\ {{{V_{{\rm out}i}^E}}} = \left( {{\alpha _i}{I_i} + {\beta _i}T_i^E - {\gamma _i}C} \right)V_i^E \\ \end{gathered} \right.$ | (2) |
其中,
(3) 标签记忆部分. 联合标签矩阵在横向上形成
| $\left\{ \begin{gathered} T_j^N = {\mathit{Softmax}}\left( {\frac{1}{{\sqrt {{d_k}} }}{V_j^N}{W_j^Q}({{W_j^K})^{\rm T}}({{V_j^N})^{\rm T}}} \right) \\ {{V^N_{{\rm out}j}}} = \left( {{\alpha _j}{I_j} + {\beta _j}T_j^N - {\gamma _j}C} \right)V_j^N \\ \end{gathered} \right.$ | (3) |
其中,
再对
| $ {T^E} = {\mathit{Softmax}}\left( {\frac{1}{{\sqrt {{d_k}} }}{V_{\rm out}^E}{W_1^Q}{({W_1^K})^{\rm T}}{({V_{\rm out}^E}})^{\rm T}} \right) $ | (4) |
| $ V_{\rm out}^E = \left( {{\alpha _1}{I_1} + {\beta _1}{T^E} - {\gamma _i}C} \right){V_{\rm out}^E} $ | (5) |
| $ {T^N} = {\mathit{Softmax}}\left( {\frac{1}{{\sqrt {{d_k}} }}{V_{\rm out}^N}{W_2^Q}{({W_2^K})^{\rm T}}{(V_{\rm out}^N)}^{\rm T}} \right) $ | (6) |
| $ V_{\rm out}^N = \left( {{\alpha _2}{I_2} + {\beta _2}{T^N} - {\gamma _j}C} \right){V_{\rm out}^N} $ | (7) |
| $ P = V_{\rm out}^E \oplus V_{\rm out}^N $ | (8) |
标签概念向量、词向量和字符向量分别代表生物医学文本中的标签、单词和字符, 属于3种不同粒度的信息. 这些向量能捕捉数据的不同尺度和层次信息, 对生物医学命名实体识别和规范化任务具有不同的重要性. 本文借鉴简化版Transformer[27]的概念, 提出了一种并行Transformer模块, 优化利用多粒度信息. 此并行块通过Transformer优化和参数调整, 有效提升模型运行效率, 同时保持高性能, 证明了在增加效率的同时, 模型性能未受影响.
(1) 对于并行Transformer模块, 假定输入序列
| $ {A_h}\left( X \right) = {\mathit{Softmax}}\left(\frac{1}{{\sqrt {{d_k}} }}X{W_h^Q}{(W_h^K)^{\rm T}}{X^{\mathrm{T}}}\right) $ | (9) |
其中,
构建一个新的Shaped Attention矩阵, 使之等价于单位矩阵. 这样带来的效果就是注意力权重分配机制中, Token更多的是关乎自身, 非常类似于残差网络的机制, 因此信号可以在很深的网络中良好传播, 更容易训练. 可以表示为:
| $ {\widetilde {Attn}_h}\left( X \right) = \left( {{\alpha _h}{I_T} + {\beta _h}{A_h}\left( X \right) - {\gamma _h}C} \right){X_h} $ | (10) |
其中,
对于
| $ \widetilde {MHA}\left( X \right) = concat\left( {{{\widehat {Attn}}_1}\left( X \right), \cdots , {{\widehat {Attn}}_H}\left( X \right)} \right) $ | (11) |
其中,
在生物医学文本中常使用大量缩写词, 很多时候缩写词的完整含义只能通过上下文来确定. 为了更好地捕捉生物医学文本序列中的长时记忆和上下文信息, 本节使用LSTM来处理序列数据. 在注意力机制中使用LSTM使模型能够更有效地融合不同部分的信息, 从而提高对复杂序列数据的建模能力. 在引入LSTM后, 使之与
| $ \begin{gathered} {\mathit{STB}}\left( X \right) = {\beta _{FF}}{\mathit{LSTM}}\left( {Norm\left( X \right)} \right) + {\beta _{SA}}\widetilde {MHA}\left( {Norm\left( X \right)} \right) \end{gathered} $ | (12) |
(2) 使用
首先使用
| $ out_1^w = \mathit{STB}_1^w\left( {{S^w}} \right) $ | (13) |
接下对于第
| $ ou{t_m} = \mathit{STB}_m^w\left( {out_{m - 1}^w} \right) $ | (14) |
最终单词加权和向量表示为:
| $ S_{\rm out}^w = \mathit{STB}_{Lw}^w\left( {out_{Lw - 1}^w} \right) $ | (15) |
其中,
(3) 使用CNN来捕捉字符表示
| $ S_L^c = {\mathrm{CNN}}\left( {{S^c}} \right) $ | (16) |
接下来使用
| $ {A_h}\left( X \right) = \mathit{Softmax}\left(\frac{1}{{\sqrt {{d_k}} }}S_{\rm out}^w{W_h^Q}{(W_h^K){^{\mathrm{T}}}}{X^{\mathrm{T}}}\right) $ | (17) |
对于第1层
| $ out_1^c = \mathit{STB}_1^c\left( {{S^w}} \right) $ | (18) |
接下对于第
| $ out_m^c = \mathit{STB}_m^c\left( {out_{m - 1}^w} \right) $ | (19) |
最后单词和字符这两种不同粒度的加权和向量表示为:
| $ {S_{\rm out}} = \mathit{STB}_{Lc}^c\left( {out_{Lc - 1}^c} \right) $ | (20) |
其中,
(4) 再次使用
| $ {A_h}\left( X \right) = \mathit{Softmax}\left(\frac{1}{{\sqrt {{d_k}} }}P{W_h^Q}{(W_h^K){^{\mathrm{T}}}}{X^{\mathrm{T}}}\right) $ | (21) |
最终标签概念、单词和字符这3种不同粒度的加权和向量表示为:
| $ Z = \mathit{STB}\left( {{S_{\rm out}}} \right) $ | (22) |
为了解决联合标签集数量庞大的问题, 本文设计了一个新的多层次标签解码器, 如图4所示. 联合标签解码器每一层簇的数量都不相同, 并且下一层簇的数量是上一层的整数倍. 为了使得不同层之间的向量可以进行运算, 本文定义了一种向量扩展方式, 即向量

|
图 4 联合标签解码器 |
(1) 首先第1层, 使用两个多层感知机(MLP)来处理纬度高且复杂的非线性关系数据
| $\left\{ \begin{gathered} F{O_1} = {\mathrm{MLP}}\left( Z \right) \\ F{O_2} = {\mathrm{MLP}}\left( {F{O_1}} \right) \\ \end{gathered} \right.$ | (23) |
其中,
得到第1层所有聚类簇的得分
| $ {\mathit{Score}_1} = expand\left( {F{O_2}, {K_1}} \right) $ | (24) |
(2) 第2层, 使用MLP处理记忆模块输出
| $ {{\mathit{SO}}_1} = {\mathrm{MLP}}\left( Z \right) $ | (25) |
然后与第1层的
| $ {{\mathit{SO}}_2} = {{\mathit{SO}}_1} \oplus F{O_1} $ | (26) |
其中,
再经过一个MLP层:
| $\left\{ \begin{gathered} {{\mathit{SO}}_3} = {\mathrm{MLP}}\left( {{{\mathit{SO}}_2}} \right) \\ {{\mathit{SO}}_4} = {{\mathit{SO}}_3} \odot {{\mathit{Score}}_1} \\ \end{gathered} \right.$ | (27) |
其中,
得到第2层所有聚类簇的得分, 然后将这些簇的得分扩展到原有的
| $ {{\mathit{Score}}_2} = expand\left( {{{\mathit{SO}}_4}, {K_2}} \right) $ | (28) |
(3) 第3层表示为:
| $\left\{ \begin{gathered} T{O_1} = {\mathrm{MLP}}\left( Z \right) \\ T{O_2} = {\mathit{SO}_2} \oplus T{O_1} \\ T{O_3} = {\mathrm{MLP}}\left( {T{O_2}} \right) \\ T{O_4} = T{O_3} \odot {\mathit{Score}_2} \\ \end{gathered} \right.$ | (29) |
最后经过一个Softmax后得到最终的联合标签预测结果
| $ Y = {\mathit{Softmax}}\left( {T{O_4}} \right) $ | (30) |
本节模型MGFFA使用BCEWithLogitsLoss损失函数. 假设有
| $ \begin{split} & \mathrm{BCEWithLogitsLoss} = \\ & \frac{1}{N}\sum\limits_{i = 1}^N {({y_i} \cdot \log (\sigma ({p_i})) + (1 - {y_i}) \cdot \log (1 - \sigma ({p_i})))} \end{split} $ | (31) |
其中,
本文选取了两个广泛应用于生物医学实体识别和关系提取任务的公开生物医学文献数据集: NCBI疾病语料库和BC5CDR数据集.
表1提供了两个关键生物医学文献数据集的详细信息概览: NCBI疾病语料库由美国国家生物技术信息中心(NCBI)提供, 包含来自PubMed摘要的793篇文章, 共标注了
| 表 1 BC5CDR和NCBI数据集的相关统计信息 |
3.2 实验设置
本文使用MGFFA模型处理BioNER和BioNEN, 输入句长为50. 记忆矩阵采用12头8层的注意力机制, 多粒度融合模块结合8头5层的注意力机制和2层LSTM. 联合标签解码器包含64、512和
如果标签为非实体标签的话, 则不进行统计. 在此基础上使用F1分数最为评估指标, 表示为:
| $ F1 = 2 \cdot \frac{{precision \cdot recall}}{{precision + recall}} $ | (32) |
(1) TaggerOne[28]是一个在训练和预测期间联合BioNER和BioNEN的机器学习模型, 由半马尔可夫结构化线性分类器组成.
(2) Transiton-based model[29]是一种用于医学领域的疾病命名实体识别和规范化的联合模型, 该模型通过在处理文本时进行状态转换, 动态的识别和标准化疾病名称, 并提高了两个任务的性能.
(3) MTL-MEN&MER_feedback[18]是一个多任务神经网络学习模型, 能够同时处理BioNER和BioNEN两个任务, 并且通过反馈策略来提升两项任务的性能.
(4) NeuJoRN[17]是一个联合模型, 将BioNER和BioNEN任务转变为一种动作序列预测任务.
(5) MTAAL[15]是一个用于BioNER和BioNEN的多任务对抗主动学习模型, 可以有效地选择有助于模型改进的数据样本, 以提高训练效率和模型性能.
(6) E2EMERN[16]是一种端到端的模型, 通过增量任务设置来减少错误传播. 该模型还利用上下文特征来丰富实体提及的语义信息, 以增强BioNEN的性能.
3.5 对比实验及其结果分析MGFFA模型在NCBI和BC5CDR数据集上与基线模型对比的实验结果如表2所示.
| 表 2 各模型在NCBI 和 BC5CDR 数据集上的F1分数 |
表2显示了TaggerOne和Transition-based两种机器学习方法在生物医学命名实体识别和规范化任务的性能, 其中Transition-based由于使用了非局部特征, 总体表现更佳. 表2中还比较了4种深度学习方法, 联合模型在两个数据集上普遍优于流水线模型. 其中, MTL-MEN&MER_feedback在NCBI和BC5CDR数据集上的F1分数分别为0.874/0.882和0.876/0.891, 显示其在BioNEN任务上的显著优势. NeuJoRN在BioNER和BioNEN任务上的表现至少优于MTL-MEN&MER_feedback 0.57%和0.59%. MTAAL通过确保任务特定特征的独立性, 并在查询样本多样性方面优化, 提升了性能和主动学习效率. MTAAL在BC5CDR的BioNER任务中表现最佳, 并在两数据集的BioNEN任务上至少优于NeuJoRN 3.9%和1.1%, 在NCBI的BioNEN任务中达到最高92.7%. E2EMERN在BC5CDR的BioNER任务中也表现突出, 整体优于其他基线模型.
这些基线模型都是通过参数共享将BioNER和BioNEN任务相关联, 两个任务是松散相关的, 有可能会出现级联错误. 本文提出的MGFFA模型使用联合标记方案, 可以避免这种错误的发生, 实现真正的联合输出. MGFFA模型在NCBI数据集和BC5CDR数据集的生物医学命名实体识别任务上分别取得了93%和92.1%的最高F1得分.
3.6 消融实验本文通过消融实验来进一步评估 MGFFA 模型中各个组成部分的贡献, 如表3所示.
| 表 3 消融实验的结果 |
–Memory: 去除整个记忆模块, 使用
–CF: 去除多粒度融合模块的概念融合部分, 使用
–Fusion: 去除整个多粒度融合模块, 将预训练词感知表示
–JTD: 去除联合标签解码器, 使用Softmax代替.
首先, 去除记忆模块后的结果显示, 模型在NCBI数据集上两个任务的F1得分分别下降了3.4%和2.8%, 而在BC5CDR数据集上下降了3.3%和4.1%, 这表明了记忆模块对于模型的正面影响, 在提升了模型的泛化能力而不会引入额外的复杂度.
其次, 去除多粒度融合模块的概念融合部分的结果显示, NCBI数据集上生物医学命名实体识别和规范化的F1得分分别降低了7.6%和6.7%, BC5CDR数据集上降低了5.4%和5.9%. 不难看出去除多粒度融合模块的概念融合部分的影响要比去除记忆模块的影响要大. 这是因为概念融合部分在被去除掉后, 记忆模块产生的概念无法被充分利用, 还会对模型产生干扰.
然后尝试移除整个多粒度融合模块. 结果显示, 模型在NCBI数据集上的生物医学命名实体识别和规范化任务中的F1得分分别下降了9.7%和11.7%, 在BC5CDR数据集上下降了9.8%和12.3%. 在未引入多粒度融合信息的情况下, 模型主要依赖于单一粒度的信息处理, 限制了其对于实体边界和内在属性的识别.
最后, 去除联合标签解码器的结果显示, 模型在NCBI数据集上的两个任务中的F1得分分别下降了12.9%和12%, 在BC5CDR数据集上下降了9.1%和10.4%. 联合标签解码器的移除导致模型分别处理每个任务, 从而增加了信息隔阂并降低了处理效率.
综合消融实验的结果得出: MGFFA模型中的每一个组件都对模型的最终性能有着显著的影响, 这些组件的协同工作使得MGFFA模型在生物医学文本的命名实体识别和规范化任务中达到了优异的效果.
4 结论与展望在本文中, 我们深入探讨了 BioNER 和BioNEN 任务之间的内在联系, 并提出了一种新颖的多粒度特征融合方法MGFFA. MGFFA模型的设计允许我们同时提取BioNER 和BioNEN 两个任务的标签, 并通过引入记忆模块形成一种对文本标签的概念级理解, 而多粒度融合模块能够充分利用不同粒度的文本信息来增强模型学习能力. 在 BC5CDR 和NCBI 数据集上的实验结果验证了 MGFFA的有效性. 未来, 我们考虑融合文本以外的其他模态数据, 如图像和声音, 来进一步丰富实体识别和规范化的上下文信息.
| [1] |
Luo L, Wei CH, Lai PT, et al. AIONER: All-in-one scheme-based biomedical named entity recognition using deep learning. Bioinformatics, 2023, 39(5): 310–319. DOI:10.1093/bioinformatics/btad310 |
| [2] |
Jeon SH, Cho S. Edge weight updating neural network for named entity normalization. Neural Processing Letters, 2023, 55(5): 5597-5618. DOI:10.1007/s11063-022-11102-2 |
| [3] |
Dash A, Darshana S, Yadav DK, et al. A clinical named entity recognition model using pretrained word embedding and deep neural networks. Decision Analytics Journal, 2024, 10: 100426. DOI:10.1016/j.dajour.2024.100426 |
| [4] |
Rani S, Jain A, Kumar A, et al. CCheXR-Attention: Clinical concept extraction and chest X-ray reports classification using modified Mogrifier and bidirectional LSTM with multihead attention. International Journal of Imaging Systems and Technology, 2024, 34(1): e23025. DOI:10.1002/ima.23025 |
| [5] |
Dai CQ, Zhuang XB, Cai JX. Chinese electronic medical record named entity recognition based on Bi-RNN-LSTM-RNN-CRF. Proceedings of the 11th International Conference on Computing and Pattern Recognition. Beijing: ACM, 2022. 577–583.
|
| [6] |
Guan ZY, Zhou XB. A prefix and attention map discrimination fusion guided attention for biomedical named entity recognition. BMC Bioinformatics, 2023, 24(1): 42. DOI:10.1186/s12859-023-05172-9 |
| [7] |
Malmasi S, Fang AJ, Fetahu B, et al. MultiCoNER: A large-scale multilingual dataset for complex named entity recognition. Proceedings of the 29th International Conference on Computational Linguistics. Gyeongju: International Committee on Computational Linguistics, 2022. 3798–3809.
|
| [8] |
Li JY, Fei H, Liu J, et al. Unified named entity recognition as word-word relation classification. Proceedings of the 36th AAAI Conference on Artificial Intelligence. AAAI, 2022. 10965–10973.
|
| [9] |
Toral A, Muñoz R. A proposal to automatically build and maintain gazetteers for named entity recognition by using Wikipedia. Proceedings of the 2006 Workshop on NEW TEXT Wikis and Blogs and Other Dynamic Text Sources. 2006. 56–61.
|
| [10] |
Lample G, Ballesteros M, Subramanian S, et al. Neural architectures for named entity recognition. Proceedings of the 2016 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. San Diego: Association for Computational Linguistics, 2016. 260–270.
|
| [11] |
Ganea OE, Hofmann T. Deep joint entity disambiguation with local neural attention. Proceedings of the 2017 Conference on Empirical Methods in Natural Language Processing. Copenhagen: Association for Computational Linguistics, 2017. 2619–2629.
|
| [12] |
Mehmood T, Serina I, Lavelli A, et al. On the use of knowledge transfer techniques for biomedical named entity recognition. Future Internet, 2023, 15(2): 79–106. DOI:10.3390/fi15020079 |
| [13] |
Sung M, Jeon H, Lee J, et al. Biomedical entity representations with synonym marginalization. Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Association for Computational Linguistics, 2020. 3641–3650.
|
| [14] |
Peng H, Xiong Y, Xiang Y, et al. Biomedical named entity normalization via interaction-based synonym marginalization. Journal of Biomedical Informatics, 2022, 136: 104238. DOI:10.1016/j.jbi.2022.104238 |
| [15] |
Zhou BH, Cai XR, Zhang Y, et al. MTAAL: Multi-task adversarial active learning for medical named entity recognition and normalization. Proceedings of the 35th AAAI Conference on Artificial Intelligence. AAAI, 2021. 14586–14593.
|
| [16] |
Zhou BH, Cai XR, Zhang Y, et al. An end-to-end progressive multi-task learning framework for medical named entity recognition and normalization. Proceedings of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th International Joint Conference on Natural Language Processing. Association for Computational Linguistics, 2021. 6214–6224.
|
| [17] |
Ji ZC, Xia T, Han M, et al. A neural transition-based joint model for disease named entity recognition and normalization. Proceedings of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th International Joint Conference on Natural Language Processing. Association for Computational Linguistics, 2021. 2819–2827.
|
| [18] |
Zhao SD, Liu T, Zhao SC, et al. A neural multi-task learning framework to jointly model medical named entity recognition and normalization. Proceedings of the 33rd AAAI Conference on Artificial Intelligence. Honolulu: AAAI, 2019. 817–824.
|
| [19] |
Yang J, Liang SL, Zhang Y. Design challenges and misconceptions in neural sequence labeling. Proceedings of the 27th International Conference on Computational Linguistics. Santa Fe: AAAI, 2018. 3879–3889.
|
| [20] |
Li YM, Tao W, Li ZH, et al. Artificial intelligence-powered pharmacovigilance: A review of machine and deep learning in clinical text-based adverse drug event detection for benchmark datasets. Journal of Biomedical Informatics, 2024, 152: 104621. DOI:10.1016/j.jbi.2024.104621 |
| [21] |
Xu H, Liu B, Shu L, et al. BERT post-training for review reading comprehension and aspect-based sentiment analysis. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. Minneapolis: Association for Computational Linguistics, 2019. 2324–2335.
|
| [22] |
Mayhew S, Nitish G, Roth D. Robust named entity recognition with truecasing pretraining. Proceedings of the 34th AAAI Conference on Artificial Intelligence. New York: AAAI, 2020. 8480–8487.
|
| [23] |
Lee J, Yoon W, Kim S, et al. BioBERT: A pre-trained biomedical language representation model for biomedical text mining. Bioinformatics, 2020, 36(4): 1234-1240. DOI:10.1093/bioinformatics/btz682 |
| [24] |
Ma WT, Cui YM, Si CL, et al. CharBERT: Character-aware pre-trained language model. Proceedings of the 28th International Conference on Computational Linguistics. Barcelona: International Committee on Computational Linguistics, 2020. 39–50.
|
| [25] |
Sukhbaatar S, Szlam A, Weston J, et al. End-to-end memory networks. Proceedings of the 28th International Conference on Neural Information Processing Systems. Montreal: MIT Press, 2015. 2440–2448.
|
| [26] |
Graves A, Wayne G, Reynolds M, et al. Hybrid computing using a neural network with dynamic external memory. Nature, 2016, 538(7626): 471-476. DOI:10.1038/nature20101 |
| [27] |
He B, Hofmann T. Simplifying Transformer blocks. Proceedings of the 12th International Conference on Learning Representations. Vienna, 2024. 1–29. He B, Hofmann T. Simplifying Transformer blocks. Proceedings of the 12th International Conference on Learning Representations. Vienna, 2024. 1–29. |
| [28] |
Leaman R, Lu ZY. TaggerOne: Joint named entity recognition and normalization with semi-Markov Models. Bioinformatics, 2016, 32(18): 2839-2846. DOI:10.1093/bioinformatics/btw343 |
| [29] |
Lou YX, Zhang Y, Qian T, et al. A transition-based joint model for disease named entity recognition and normalization. Bioinformatics, 2017, 33(15): 2363-2371. DOI:10.1093/bioinformatics/btx172 |
 2024, Vol. 33
2024, Vol. 33


