当前, 癌症研究正迎来一个充满挑战与机遇的新时代[1]. 随着癌症遗传学和癌症分子机制研究的不断发展, 研究人员逐渐开始认识到癌症驱动基因在癌症的发生、发展和转移过程中扮演着重要角色[2]. 识别癌症驱动基因不仅有助于更深刻地理解癌症的病理生理过程, 还为个性化治疗和精准医学的实现提供了关键支持[3].
然而, 癌症驱动基因的识别并非易事, 不同类型的癌症在遗传和分子机制上存在巨大的异质性[4]. 并且, 癌症发展涉及多种细胞类型、信号通路和调控机制的复杂相互作用, 在这个复杂的网络中, 单一基因突变的作用通常只是一个更为广泛的网络中的一部分, 这使得驱动基因的准确识别变得十分困难[5].
为此, 本文提出并成功实现了一种基于图自编码器与LightGBM的癌症驱动基因识别系统ACGAI, 旨在克服癌症驱动基因识别领域面临的异质性和复杂性问题. 相较于传统方法, 该系统充分利用图自编码器 (graph autoencoder)[6] 和LightGBM (light gradient boosting machine)[7,8] 的技术优势构建了GAELGBM模型, 该模型将生物分子网络的拓扑结构信息嵌入到编码器和解码器中, 以无监督学习的方式提取异质、复杂的生物分子网络特征; 接着, 将编码器的输出与原始基因特征拼接并输入至LightGBM, 通过梯度提升的方法进行训练, 进而使得测试结果表现出优于其他方法的识别性能, 有效地克服了癌症驱动基因识别领域面临的异质性和复杂性问题, 为该领域的研究提供了新的视角和解决思路; 最后, 该系统利用Web技术创建了一套用户友好且交互性强的可视化界面, 实现了可生物学解释的癌症驱动基因识别, 为癌症研究提供了便利和支持.
1 相关研究机器学习在癌症驱动基因识别方面的应用可分为基于传统机器学习的方法和基于深度学习的方法两大类别. 对于传统机器学习方法, 例如Nulsen等[9]提出的sysSVM2通过有效地整合癌症遗传变异的信息, 并结合基因系统级的特性进行癌症驱动基因预测. 而Yang等[10]开发的InDEP采用基于决策树的级联森林以及支持细粒度事后解释的KernelSHAP模块, 通过综合多组学数据来识别癌症驱动基因. 尽管这些基于传统机器学习的方法在癌症驱动基因识别方面表现出良好的性能, 但大多数未考虑到生物分子网络的拓扑结构, 因此在性能上存在一定限制. 而基于图神经网络 (GNN) 的深度学习方法部分解决了前述挑战[11,12]. 例如, Schulte-Sasse等[13]开发的EMOGI方法通过整合基因组学、转录组学和蛋白质组学等多组学信息, 运用图卷积网络(GCN)[14] 成功实现了癌症驱动基因的识别. Peng等[15]提出了MTGCN, 采用基于切比雪夫GCN[16]的多任务学习框架来提升癌症驱动基因识别性能. Zhang等[17]提出了HGDC方法, 通过采用个性化PageRank [18,19]技术对GCN进行扩展, 增强了模型对生物分子网络异质性的学习能力. 然而, 尽管以上方法在一定程度上能够提升癌症驱动基因识别的准确率, 但却未能充分利用复杂生物分子网络的多层次结构, 导致模型性能下降. 此外, 它们也缺乏高效的手段来对识别结果进行生物学解释.
2 系统设计与实现 2.1 系统架构如图1所示, ACGAI系统由控制层、数据层和用户层组成. 其中, 控制层和用户层服务于系统管理员(以下简称管理员) 和癌症驱动基因研究人员 (以下简称用户) 两个不同的用户群体. 控制层主要技术栈为Python+Streamlit, 涵盖了数据处理模块、模型训练模块以及识别与分析模块. 数据层主要通过MongoDB技术栈来提供数据支持, 它负责存取缓存数据、应用数据和计算结果数据, 为控制层和用户层提供可靠的数据存储和检索功能. 用户层采用Python+FastAPI+Vue作为主要技术栈, 由数据接口、交互与可视化界面构成, 该层的设计旨在为用户提供操作界面与数据接口, 以便进行癌症驱动基因的识别和分析. ACGAI系统通过上述的层次化设计, 充分发挥了各个层次的特点, 使得系统在控制、数据存储和用户交互方面都能够实现高效而可靠的功能.
2.2 数据处理模块数据处理模块的主要任务是将管理员上传的生物分子网络数据处理成可供GAELGBM模型使用的pickle数据, 以供本研究进一步调用和分析. 具体而言, 该模块先将管理员上传的生物分子特征、相互作用边进行整理形成统一的Data对象, 之后将其序列化为pickle数据文件, 模型需要使用时, 只需将其反序列化为Data对象即可, 确保了系统数据处理流程的便捷性和整体一致性.
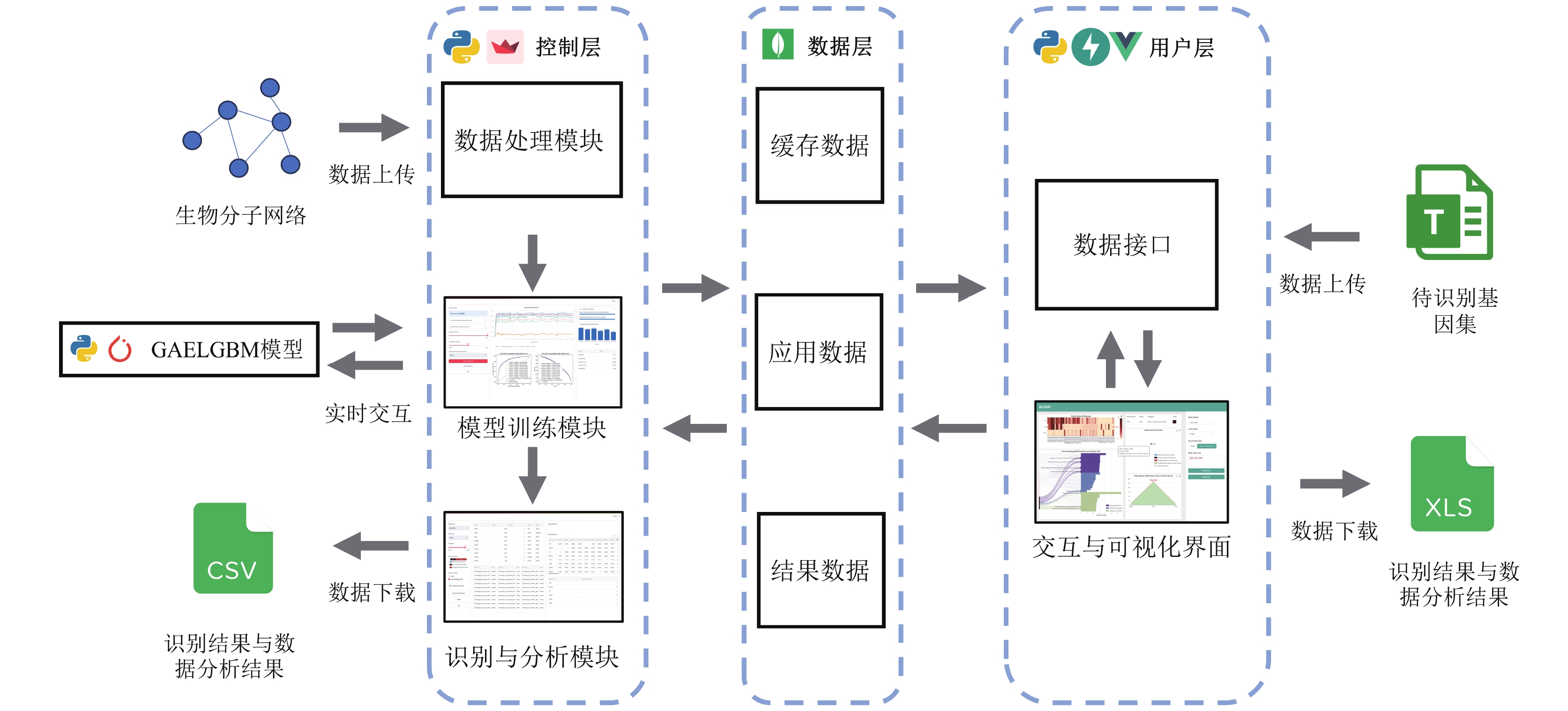
|
图 1 系统架构 |
2.3 模型训练模块
模型训练模块的主要功能是训练GAELGBM模型. 它利用Streamlit提供了一个实时可视界面, 允许管理员通过实时分析GAELGBM模型的训练效果, 调整模型的参数, 从而提升模型的识别性能. 该模块的运行流程为: 首先将生物分子网络数据输入GAELGBM模型进行训练, 随后在训练过程中利用回调函数将GAELGBM每一次迭代的训练与测试信息包括损失值、精度、召回率、准确率等实时传送给UI界面, 以进度条、折线图、受试者工作特性曲线图等方式进行实时的可视呈现, 最后待训练完成后将预测得分导出至数据层, 以供识别与分析模块进行下一步的工作.
2.3.1 GAELGBM模型如图2所示, 本研究提出的GAELGBM模型的运行流程如下.
(1) 首先, GAELGBM模型将生物分子网络输入图自编码器, 通过最小化编码器和解码器的重构误差来训练生成增强特征信息. 具体而言, 图自编码器由编码器、解码器、目标函数组成, 用于捕获生物分子网络中异质且复杂的网络结构并生成增强特征信息; 编码器和解码器结构相同, 均由两个图卷积层 (GCNConv) 叠加构成, 编码器的输出同时也是解码器的输入, 图卷积层的计算公式如下:
| H(l+1)=σ(ˆD−12ˆAˆD−12H(l)W(l)) | (1) |
其中,
| L1=1NN∑i=1‖xi−x′i‖2 | (2) |
其中,
(2) 接着, GAELGBM模型将增强特征信息与原始基因特征进行拼接, 其目的在于使LightGBM能够通过基因增强特征矩阵学习到网络结构信息, 该过程的具体计算公式为:
| Henh=[X,Z] | (3) |
其中,
(3) 最后, GAELGBM模型将基因增强特征矩阵输入至LightGBM, 利用梯度提升的方法得到每次迭代的预测得分向量
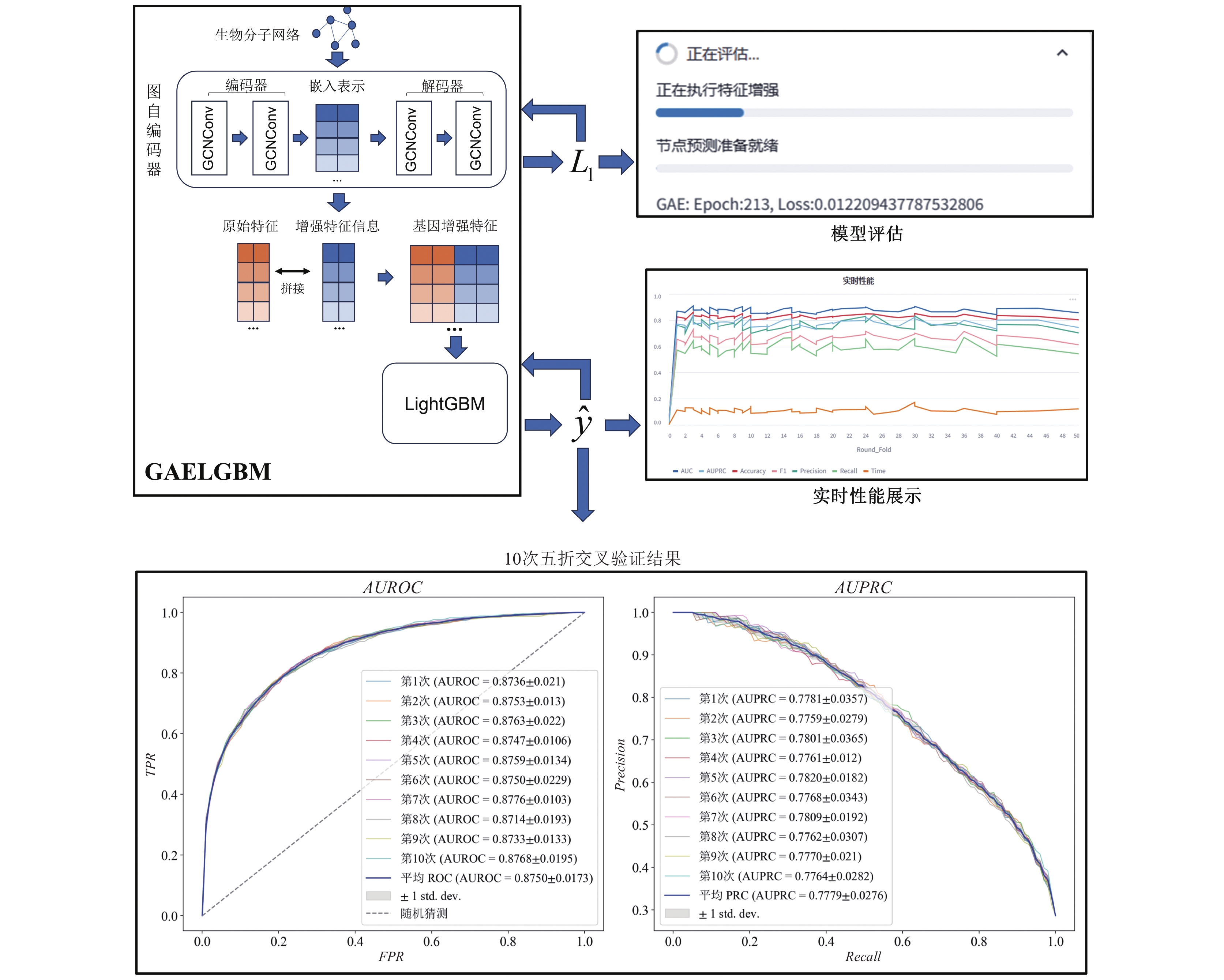
|
图 2 GAELGBM模型流程图 |
2.4 分析与识别模块
分析与识别模块的主要任务是进行癌症驱动基因的分析并为识别结果提供生物学解释. 具体而言, 该模块提供了相互作用网络分析、基因富集分析、基因特征分析3个数据分析功能. 同时, 为了增强使用体验, 该模块还提供了一个如图3所示的交互式可视界面, 使得管理员能够通过数据分析结果并结合模型训练模块的模型预测性能结果来优化分类阈值. 在ACGAI系统中, 通过设定适当的分类阈值, 分析与识别模块实现了基因的分类, 从而完成对癌症驱动基因的识别. 这一过程不仅允许管理员根据具体需求调整阈值, 而且有效地结合了模型预测性能的信息, 提高了系统对潜在癌症驱动基因识别的准确性和鲁棒性. 其计算过程可用公式表示为:
| c(t,yi)={0,yi⩽t1,yi>t | (4) |
其中,
管理员可以通过控制面板使用分析与识别模块中的各项功能, 这包括对数据的选择、阈值的调整、富集分析类型的选择以及可视模式的设定. 在数据选择方面, 管理员通过控制面板指定感兴趣的生物分子网络数据. 阈值调整功能允许管理员根据实际需求微调系统的敏感性和特异性, 以达到最佳的癌症驱动基因识别效果. 富集分析类型选择提供了多种选项, 使管理员能够在不同层次上深入分析基因的功能和关联. 可视化方式选择允许管理员按照个人偏好选择合适的可视模式, 当管理员关闭 [可视化方式] 开关, 系统将以表格的形式展示数据, 这个表格提供了数据下载、数据搜索和全屏显示等功能, 使得管理员能够更加灵活地利用数据. 当管理员点击 [导出数据] 按钮后, 系统将预测结果和特征数据存储到MongoDB数据库中, 以便用户层使用. 值得注意的是, 为了提供更丰富的数据分析体验, 系统生成的每个图表都是可交互且联动的; 管理员可以通过点击或将鼠标悬停在图表上, 选择特定基因或查看与之相关的信息. 这种交互性设计不仅提高了系统的易用性, 还使得管理员能够更深入地探索和理解数据, 从而更好地指导后续的决策和研究工作.

|
图 3 控制层分析与识别模块界面 |
2.4.1 相互作用网络分析
分析与识别模块通过计算生物分子网络中基因之间的相互作用关系和数量, 实现了相互作用网络分析. 其主要目标在于为管理员提供基于基因相互作用的判断依据, 以便更准确地选择适当的分类阈值. 具体而言, 该模块呈现了两个重要视图.
(1) 相互作用网络图
相互作用网络图是根据记录基因之间的相互作用构建的. 其中, 每一条边代表着基因之间有相互作用, 而每一个基因节点则根据其所属的基因类别进行划分. 如图 3 所示, 该图整体采用了力导向布局, 清晰地展示了得分超过特定阈值的基因节点之间的相互作用.
(2) 相互作用数量趋势图
相互作用数量趋势图以基因名称为横坐标, 每个基因与已知癌症驱动基因的相互作用数量为纵坐标. 通过折线面积图的方式, 直观描述了基因列表按预测得分从高到低排序的变化趋势, 从而呈现了预测得分与相互作用数量之间的关系.
2.4.2 基因富集分析分析与识别模块主要利用 KEGG (Kyoto encyclopedia of genes and genomes)[20] 和GO (gene ontology)[21] 两种富集分析方法进行基因富集分析. 其目标是为管理员提供对于预测得分超过选定阈值的基因在人类生物学通路、生物过程、细胞组分以及生物分子功能层面的解释. 通过KEGG富集分析, 模块能够揭示基因在生物通路上的聚集情况, 为管理员提供生物学通路洞察. 而通过GO富集分析, 模块将基因映射到生物学过程、细胞组分和分子功能的不同层面, 使得管理员能够全面理解基因在生物学上的多层面功能. 管理员可以通过点击 [富集分析类型] 单选按钮切换富集分析类型, 并通过点击 [启动富集分析] 按钮获取相应的富集分析图. 富集分析的具体过程如下:
(1) 使用GSEApy方法[22], 在设定cutoff值为0.05后向Enrichr[23,24]发送请求获取基因富集数据. 这一步骤的设计旨在通过GSEApy方法对基因富集进行有效处理, 并以设定的cutoff值过滤掉不显著的结果, 从而确保得到更为可信的生物学通路和功能注释信息.
(2) 基于富集数据中的
| P' = - {\log _{10}}(P) | (5) |
接着根据
(3) 使用桑基图与条形图组合生成富集分析图. 其中, 桑基图通过将富集分析的每个富集项均连接若干基因的方式, 呈现了基因在富集项上的富集关系. 而条形图通过富集项的
分析与识别模块通过基因特征热力图来实现基因特征分析. 在该图中, 基因的各项癌症生物特征, 包括突变频率 (MF)、甲基化水平(METH) 、基因表达 (GE) 以及拷贝数变异(CNA), 被设置为横坐标, 而基因名称则作为纵坐标, 呈现按预测得分从高到低排序的基因列表在不同癌症生物特征上的变化趋势. 这样的设计旨在描述预测得分与癌症生物特征之间的关系, 使管理员能够直观地了解不同生物特征对于基因预测得分的影响.
2.5 数据接口与交互可视界面用户层的数据接口和交互可视界面是前后端分离架构, 旨在为用户提供高效和界面友好的Web服务平台. 用户既可以选择使用系统提供的Web API, 也可以选择使用本系统提供的交互可视界面, 如图4所示.
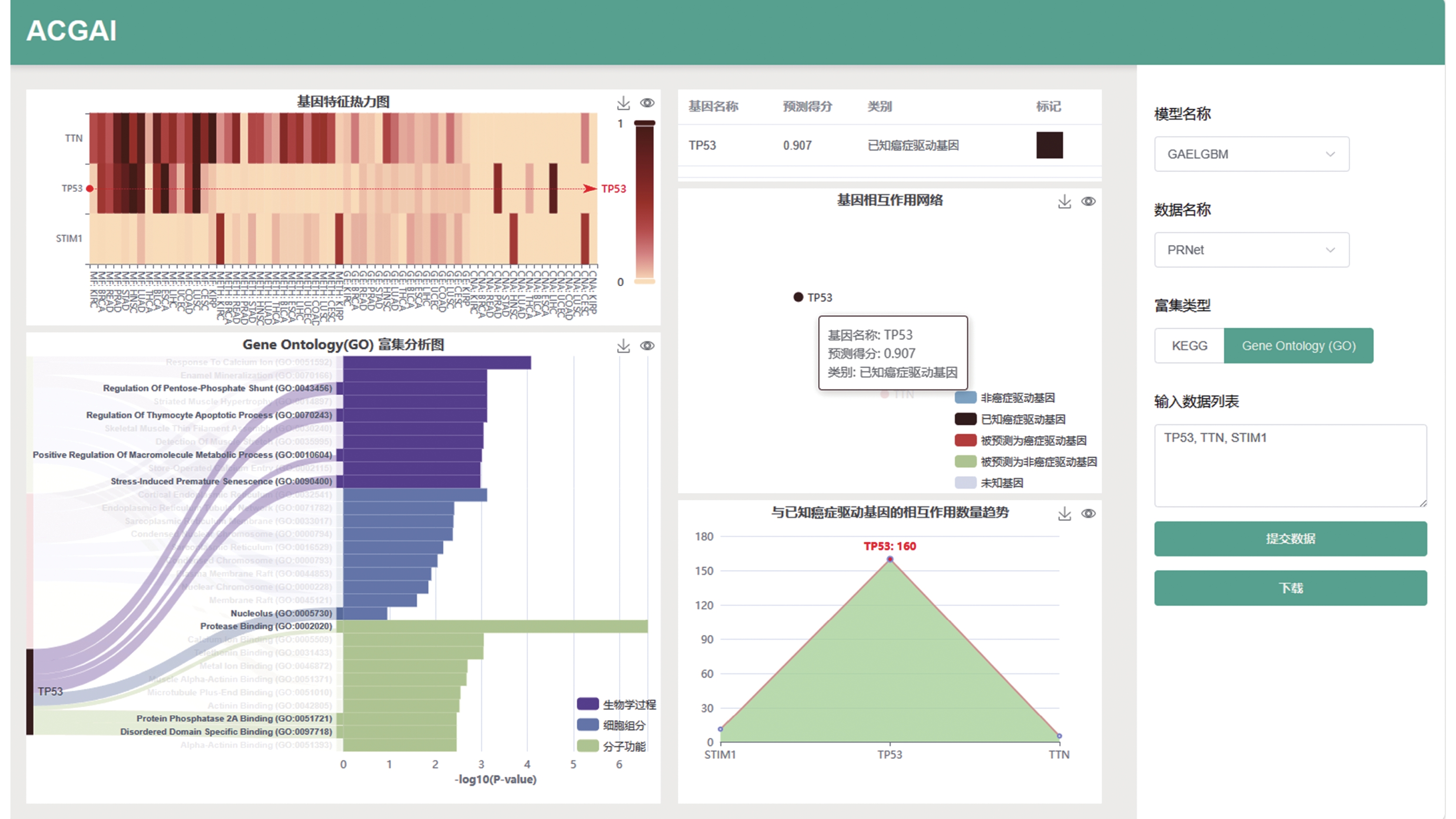
|
图 4 用户层交互可视界面 |
用户层交互可视界面继承了分析与识别模块的分析方法和图表设计, 但和后者不同的是, 前者主要分析的是用户提交的基因集, 而后者主要分析的是预测得分大于得分阈值的基因集. 该模块的运行流程为: 首先, 用户通过交互可视界面提交待识别的基因集, 数据接口接收到数据请求并通过格式检验后, 向数据缓存请求数据. 如果命中缓存, 数据接口就将数据直接返回, 否则使用和分析与识别模块相同的分析方式进行分析, 随后将分析结果进行缓存并返回给交互可视界面渲染可视图表和生成Excel表格供用户使用或下载. 这一流程的设计旨在确保数据的高效传输和处理, 使其在实现基因集中癌症驱动基因的识别和生物学解释的同时, 为用户提供更流畅的使用体验.
3 系统测试 3.1 功能测试为了验证ACGAI系统的功能是否符合用户需求, 本研究对其进行了功能测试, 测试流程如下.
(1) 依次启动控制层、数据层、用户层使用的软件服务.
(2) 在后台管理软件入口上传PRNet数据, 接着点击 [GAELGBM] 按钮, 进入模型训练模块界面, 设置好参数, 点击 [开始评估] 按钮启动实验, 实时观察模型的训练过程, 待训练完毕点击 [开始预测] 按钮实现癌症驱动基因预测, 待系统回应预测完毕后点击[退出]按钮退出该界面.
(3) 点击 [可视化] 按钮进入如图3所示的分析与识别模块界面, 设置好各项参数, 依次测试各个分析功能确认无误后, 点击 [导出数据] 按钮导出数据至MongoDB数据库, 待系统回应导出完毕后点击 [退出] 按钮退出该界面.
(4) 进入如图4所示的用户层交互可视界面, 设置好参数, 输入不同组待预测基因列表, 点击 [提交数据] 按钮. 待各个分析图表渲染完毕, 依次测试各个图表的功能. 确认无误后, 点击 [下载] 按钮, 下载含有识别结果和生物学解释结果的Excel文件, 并验证其内容是否符合要求.
(5) 根据用户层API文档, 使用Apifox逐一发送请求给接口地址, 检查响应结果中的数据是否满足期望.
经过以上测试流程, ACGAI系统功能能满足用户需求, 测试所使用的关键硬件设备如表1所示.
| 表 1 测试使用的关键硬件列表 |
3.2 GAELGBM模型性能测试 3.2.1 测试数据集
系统使用的原始数据(包括基因特征数据、已知的基因标签数据、生物分子网络数据)均收集自公开数据集. 具体而言, 基因特征数据与已知的基因标签数据来自EMOGI[13]. 其中, 基因特征数据包含16种癌症的突变频率特征、甲基化特征、基因表达特征和拷贝数变异特征, 已知的基因标签数据包括癌症驱动基因标签(正标签)和非癌症驱动基因标签(负标签). 生物分子网络数据来自STRING(v11.5)[25]的蛋白质相互作用网络和starBase(v2.0)的RNA-RNA交互网络[26]. 本研究基于这些数据进行处理并构建了PRNet数据集, 具体而言, 为了确保测试数据集具有高可信度和生物学意义, 本研究先从基因特征数据中提取基因列表, 然后根据基因列表在蛋白质相互作用网络中提取可信度得分大于800的边; 接着根据基因列表, 在RNA-RNA交互网络中提取类型是蛋白质编码的基因节点, 并删除匹配程度低于总RNA-RNA相互作用网络平均值的边; 最后, 整合以上过程提取的基因节点、边以及基因标签数据形成了PRNet数据集. 该数据集包含
由于测试数据属于类别不平衡数据, 所以本系统采用了对不平衡数据二分类任务更准确的评价指标, 包括受试者工作特性曲线下面积 (area under receiver operating characteristic, AUROC) 和Precision-Recall曲线下面积(area under the precision-recall curve, AUPRC), 两者的值越接近1代表性能越好, 越接近0.5代表性能越差. 具体而言, 为了减少因为数据分布不均衡而引起的评价结果的不稳定性, 本研究在测试时计算了模型在10次五折交叉验证下的平均AUROC和平均AUPRC. 该过程可用以下公式表示:
| {{Recall}} = {\mathit{TPR}} = \frac{{{{TP}}}}{{{{TP + FN}}}} | (6) |
| {\mathit{FPR}} = \frac{{{{FP}}}}{{{{FP + TN}}}} | (7) |
| {{Precision}} = \frac{{{{TP}}}}{{{{TP + FP}}}} | (8) |
| {\mathit{AUROC}} = \int_0^1 {\mathit{TPR(FPR)}} {\mathrm{d}}(\mathit{FPR}) | (9) |
| {\mathit{AUPRC}} = \int_0^1 {Precision(Recall)} {\mathrm{d}}(Recall) | (10) |
| {\mathit{AAUROC}} = \frac{1}{{10}}\sum\limits_{j = 1}^{10} {\left( {\frac{1}{5}\sum\limits_{i = 1}^5 {{\mathit{AUROC}_{i, j}}} } \right)} | (11) |
| {\mathit{AAUPRC}} = \frac{1}{{10}}\sum\limits_{j = 1}^{10} {\left( {\frac{1}{5}\sum\limits_{i = 1}^5 {{\mathit{AUPRC}_{i, j}}} } \right)} | (12) |
其中,
为了验证GAELGBM的优越性能, 本研究将其与目前本领域的SOTA模型和传统机器学习模型在PRNet数据集上进行了对比实验. 对比结果如表2所示, GAELGBM在平均AUROC上相比于排名第2的MTGCN提升了0.019 (约2.22%), 在平均AUPRC上相比于排名第2的HGDC提升了
为了验证GAELGBM每个模块均有助于提升模型性能, 本研究将GAELGBM进行了消融实验. 具体而言, 本研究在保持模型参数不变的情况下, 将GAELGBM的各个模块进行消融, 构造出以下变种模型, 并在PRNet数据集上进行了10次五折交叉验证.
GAESVM: 将模型中的LightGBM模块更换为SVM后获得的模型.
GAERF: 将模型中的LightGBM模块更换为RF后获得的模型.
LGBM: 移除图自编码器模块后所获得的模型.
| 表 2 模型性能对比表 |
如表3所示, 相对于其他变种模型, GAELGBM在性能方面表现最佳, 其平均AUROC比排名第2的LGBM高出
| 表 3 消融实验结果 |
4 结论与展望
针对当前癌症驱动基因识别领域所面临的异质性和复杂性问题, 本文设计并实现了一种基于图自编码器与LightGBM的癌症驱动基因识别系统ACGAI. 该系统提供的GAELGBM模型能够准确识别生物分子网络上的癌症驱动基因, 且该系统为系统管理员提供了端到端的UI界面, 涵盖了从数据上传、模型训练, 到癌症驱动基因识别, 再到生物学解释的全过程, 具有对用户友好的特点. 此外, ACGAI系统还开放了面向癌症驱动基因研究人员的交互式可视界面, 使得研究人员可通过Web服务获取目标基因集的识别结果, 并可通过系统提供的可视化图表深入了解识别结果的生物学解释. 最后, 经过系统功能测试和模型的性能测试, ACGAI系统能够满足癌症驱动基因识别需求且具有优于其他方法的识别性能, 但仍需注意的是, 正负样本不平衡会限制ACGAI系统的识别性能. 因此, 在未来的研究中, 我们将着力于提升模型的正负样本不平衡适应性, 以增强系统的癌症驱动基因识别能力.
| [1] |
Foulkes I, Sharpless NE. Cancer grand challenges: Embarking on a new era of discovery. Cancer Discovery, 2021, 11(1): 23-27. DOI:10.1158/2159-8290.Cd-20-1657 |
| [2] |
Martínez-Jiménez F, Muiños F, Sentís I, et al. A compendium of mutational cancer driver genes. Nature Reviews Cancer, 2020, 20(10): 555-572. DOI:10.1038/s41568-020-0290-x |
| [3] |
Vogelstein B, Papadopoulos N, Velculescu VE, et al. Cancer genome landscapes. Science, 2013, 339(6127): 1546-1558. DOI:10.1126/science.1235122 |
| [4] |
Ostroverkhova D, Przytycka TM, Panchenko AR. Cancer driver mutations: Predictions and reality. Trends in Molecular Medicine, 2023, 29(7): 554-566. DOI:10.1016/j.molmed.2023.03.007 |
| [5] |
Lawrence MS, Stojanov P, Polak P, et al. Mutational heterogeneity in cancer and the search for new cancer-associated genes. Nature, 2013, 499(7457): 214-218. DOI:10.1038/nature12213 |
| [6] |
Yi HC, You ZH, Huang DS, et al. Graph representation learning in bioinformatics: Trends, methods and applications. Briefings in Bioinformatics, 2022, 23(1): bbab340. DOI:10.1093/bib/bbab340 |
| [7] |
孟祥福, 田友发, 张霄雁. 基于LightGBM模型的肺腺癌免疫相关基因筛选与患者生存率预测. 生物医学工程学杂志, 2024, 41(1): 70-79. DOI:10.7507/1001-5515.202305038 |
| [8] |
Ke GL, Meng Q, Finley T, et al. LightGBM: A highly efficient gradient boosting decision tree. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 3149–3157.
|
| [9] |
Nulsen J, Misetic H, Yau C, et al. Pan-cancer detection of driver genes at the single-patient resolution. Genome Medicine, 2021, 13(1): 12. DOI:10.1186/s13073-021-00830-0 |
| [10] |
Yang H, Liu YW, Yang YJ, et al. InDEP: An interpretable machine learning approach to predict cancer driver genes from multi-omics data. Briefings in Bioinformatics, 2023, 24(5): bbad318. DOI:10.1093/bib/bbad318 |
| [11] |
Yue X, Wang Z, Huang JG, et al. Graph embedding on biomedical networks: Methods, applications and evaluations. Bioinformatics, 2020, 36(4): 1241-1251. DOI:10.1093/bioinformatics/btz718 |
| [12] |
Zhang ZW, Cui P, Zhu WW. Deep learning on graphs: A survey. IEEE Transactions on Knowledge and Data Engineering, 2022, 34(1): 249-270. DOI:10.1109/TKDE.2020.2981333 |
| [13] |
Schulte-Sasse R, Budach S, Hnisz D, et al. Integration of multiomics data with graph convolutional networks to identify new cancer genes and their associated molecular mechanisms. Nature Machine Intelligence, 2021, 3(6): 513-526. DOI:10.1038/s42256-021-00325-y |
| [14] |
吴博, 梁循, 张树森, 等. 图神经网络前沿进展与应用. 计算机学报, 2022, 45(1): 35-68. DOI:10.11897/SP.J.1016.2022.00035 |
| [15] |
Peng W, Tang Q, Dai W, et al. Improving cancer driver gene identification using multi-task learning on graph convolutional network. Briefings in Bioinformatics, 2022, 23(1): bbab432. DOI:10.1093/bib/bbab432 |
| [16] |
Defferrard M, Bresson X, Vandergheynst P. Convolutional neural networks on graphs with fast localized spectral filtering. Proceedings of the 30th Conference on Neural Information Processing Systems. Barcelona: NIPS, 2016.
|
| [17] |
Zhang T, Zhang SW, Xie MY, et al. A novel heterophilic graph diffusion convolutional network for identifying cancer driver genes. Briefings in Bioinformatics, 2023, 24(3): bbad137. DOI:10.1093/bib/bbad137 |
| [18] |
Iván G, Grolmusz V. When the Web meets the cell: Using personalized PageRank for analyzing protein interaction networks. Bioinformatics, 2011, 27(3): 405-407. DOI:10.1093/bioinformatics/btq680 |
| [19] |
Yang MJ, Wang HZ, Wei ZW, et al. Efficient algorithms for personalized pagerank computation: A survey. IEEE Transactions on Knowledge and Data Engineering. [doi: 10.1109/TKDE.2024.3376000]
|
| [20] |
Kanehisa M, Furumichi M, Sato Y, et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Research, 2023, 51(D1): D587-D592. DOI:10.1093/nar/gkac963 |
| [21] |
The Gene Ontology Consortium, Aleksander SA, Balhoff J, et al. The gene ontology knowledgebase in 2023. Genetics, 2023, 224(1): iyad031. DOI:10.1093/genetics/iyad031 |
| [22] |
Fang ZQ, Liu XY, Peltz G. GSEApy: A comprehensive package for performing gene set enrichment analysis in Python. Bioinformatics, 2023, 39(1): btac757. DOI:10.1093/bioinformatics/btac757 |
| [23] |
Kuleshov MV, Jones MR, Rouillard AD, et al. Enrichr: A comprehensive gene set enrichment analysis Web server 2016 update. Nucleic Acids Research, 2016, 44(W1): W90-W97. DOI:10.1093/nar/gkw377 |
| [24] |
Xie ZR, Bailey A, Kuleshov MV, et al. Gene set knowledge discovery with enrichr. Current Protocols, 2021, 1(3): e90. DOI:10.1002/cpz1.90 |
| [25] |
Szklarczyk D, Gable AL, Lyon D, et al. STRING v11: Protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 |
| [26] |
Li JH, Liu S, Zhou H, et al. starBase v2.0: Decoding miRNA-ceRNA, miRNA-ncRNA and protein—RNA interaction networks from large-scale CLIP-Seq data. Nucleic Acids Research, 2014, 42(D1): D92-D97. DOI:10.1093/nar/gkt1248 |
| [27] |
Breiman L. Random forests. Machine Learning, 2001, 45(1): 5-32. DOI:10.1023/A:1010933404324 |
| [28] |
Khalsan M, Machado LR, Al-Shamery ES, et al. A survey of machine learning approaches applied to gene expression analysis for cancer prediction. IEEE Access, 2022, 10: 27522-27534. DOI:10.1109/ACCESS.2022.3146312 |
 2024, Vol. 33
2024, Vol. 33



