2. 浙江大学医学院附属儿童医院 儿童保健科, 杭州 310003;
3. 国家儿童健康与疾病临床医学研究中心, 杭州 310003
2. Child Healthcare Department, Children’s Hospital of Zhejiang University School of Medicine, Hangzhou 310003, China;
3. National Clinical Research Center for Child Health, Hangzhou 310003, China
患病儿童接种疫苗后可能产生异常反应, 严重者甚至危及生命[1]. 2015年起我国范围内免疫接种后不良事件(adverse events following immunization, AEFI)报告呈上升趋势[2]. 受一些疫苗事件和报道的影响, 部分公众对疫苗产生怀疑态度, 甚至产生疫苗犹豫[3], 不利于疫苗接种工作的良性循环.
由于相关知识的不完全了解, 社区卫生服务工作者通常对于患病儿童的疫苗接种犹豫不决[1], 尤其对于经验较少的新手医生[4]而言. 目前, 专科医院开设了儿童保健科免疫接种咨询门诊, 专家医生凭借其临床经验为患病儿童诊断并给出疫苗接种处理建议. 通过学习高水平医院医生的经验来研发儿童疫苗接种预测模型, 从而帮助基层医疗机构医生快速筛查高风险患儿, 是一种可行的方法.
因此, 迫切需要一种智能化方法, 通过利用已有的医疗知识来辅助医生对疾病儿童健康状况进行评估, 从而给出合理的接种建议决策. 这将有助于提高医疗服务的质量和效率, 更好地满足病人的需求.
医学知识图谱是推进智慧医疗的关键技术, 也是医疗信息智能化管理的基础[5], 被广泛应用在医学辅助诊断、个性化推荐、药物间关系发现等方面. 医学知识图谱与人工智能技术相结合, 可以提高医生的诊断效率, 在智慧医疗建设中具有关键的作用. 从电子病历中抽取知识对构建知识图谱至关重要, 但是中文电子病历文本形式多样, 其中存在的实体信息具有语义模糊、上下文不确定等特点. 高质量的知识抽取工作一定程度上影响着知识图谱下游任务的开展.
在这一背景下, 本研究提出了一种基于预训练模型的儿童免疫接种咨询门诊电子病历的命名实体识别(named entity recognition, NER)方法ELECTRA-BiGRU-CRF. 在基于该方法构建出的中文疫苗接种知识图谱上, 本文提出一种基于预训练语言模型的显著性类别指导的疫苗接种推荐方法. 通过对比基线模型, 验证了本文所提出方法的有效性. 本文研究方法为免疫干预工作中的医生提供了宝贵的资源, 能够有效地辅助医生对儿童健康进行评估并给出合理的接种建议.
1 相关工作 1.1 疫苗接种风险分析研究随着免疫规划的不断发展, 对AEFI事件的关注逐渐增加, 尤其是在儿童预防接种工作中[6].
在预测儿童疫苗接种风险的做法上, 研究者们大多采用统计学方法. 例如, 胡丹丹等人[7]分析神经系统疾病儿童免疫接种现状、相关的神经系统反应以及禁忌证等, 提出了不同神经系统疾病儿童免疫接种策略. Wang等人[8]对疫苗接种与系统性红斑狼疮和风湿关节炎发病风险的关系进行了系统评价和分析.
统计学方法大多对接种疫苗后的儿童疫苗进行监测, 统计分析不良反应报告发生率, 以提出预测和规避疫苗接种不良反应的方法, 且取得了一定的进展. 但这种方法需要进行随访跟踪和系统分析, 耗费大量的人力物力, 同时也难以避免医疗资源紧张和浪费等问题.
1.2 知识图谱应用研究医学知识图谱的应用是目前垂直领域知识图谱的研究热点之一[9], 其旨在有组织地充分利用庞大的数据资源, 为医疗信息化提供支持.
越来越多的研究人员致力于医学领域知识图谱的开发与应用. 比如, Chandak等人[10]整合20个高质量的资源, 构建了疾病丰富和具有功能性的多模态知识图谱PrimeKG. Joshi等人[11]设计了一种深度神经网络, 用于预测药物在生产阶段或临床试验阶段引起的不良反应. Tao等人[12]提出了一种分类模型用于从个人健康知识库中进行知识挖掘来帮助发现潜在的疾病威胁.
医疗领域知识图谱的研究呈现出蓬勃发展的趋势, 而中文医疗知识图谱的发展面临着语言特殊、知识来源繁杂、开源程度不足等问题, 构建仍然存在着许多困难[13,14]. 构建医学知识图谱的关键在于图谱三元组的抽取, 而在三元组抽取工作中, 医学命名实体的抽取直接决定了构建的知识图谱质量.
1.3 预训练语言模型大规模预训练语言模型(pre-trained language model, PLM)[15]是近年来发展起来的重要技术, 在自然语言处理领域取得了显著进展. PLM通过大规模的无监督学习, 让模型在大量文本数据上进行预先训练, 从而学到丰富的语言表示. BERT[16]是PLM中的代表作之一, 通过预先训练双向Transformer编码器[17], 使得模型能够同时考虑上下文的信息, 从而更好地捕捉词汇之间的关系. BERT在问答、文本分类和命名实体识别等任务上取得了领先水平.
预训练语言模型的发展过程中涌现了一系列变体和改进方法, 通过引入不同的训练策略、更大的数据集或改进的架构, 进一步提升了在各种任务上的性能, 医学领域的预训练语言模型也应运而生. 目前PLM也面临着一些挑战, 模型巨大的参数规模和训练过程需要大量的计算资源, 在部分场景下限制了模型的应用范围. 其次医学文本术语的特殊性和敏感性也带来了应用的挑战.
1.4 实体识别方法研究NER任务旨在从医学文本中识别各种实体. 目前NER方法主要分为3种. 基于字典和规则的方法侧重通过匹配的方法去处理文本, 但对医学词典库的依赖性高且维护成本巨大. 随着监督学习的发展, 基于统计和机器学习的方法成为主流. 然而传统的机器学习算法需要复杂的特征工程, 且模型泛化能力受限. 相较之下, 深度学习方法的显著优势在于其端到端的训练过程, 无需手动设计特征.
近年来融合深度学习方法和机器学习模型的NER方法不断涌现, 用以提高效率和结果精度[18]. Wang等人[19]提出了两种扩展BiLSTM的不同架构和特征表示方案来处理中文电子病历实体识别任务. 自Transformer被提出以来, 各种预训练语言模型层出不穷. 有研究表明, 使用预训练语言模型作为词嵌入层可以提高实体识别模型的序列标注性能[20]. 将深度神经网络用于文本特征学习, 无需人工手动定义模板, 且具有较强的泛化能力, 已成为目前NER任务的主流方法.
2 关键技术研究 2.1 基于ELECTRA-BiGRU-CRF的实体识别方法在构建儿童疫苗接种知识图谱的过程中, 从电子病历中提取有价值的医疗信息至关重要. 本文提出了一种基于预训练语言模型ELECTRA[21]的医学信息实体识别模型ELECTRA-BiGRU-CRF. 该模型主要分为3层, 分别为ELECTRA层、BiGRU层以及CRF层. 模型结构图如图1所示. 模型实体识别的过程包括以下步骤.
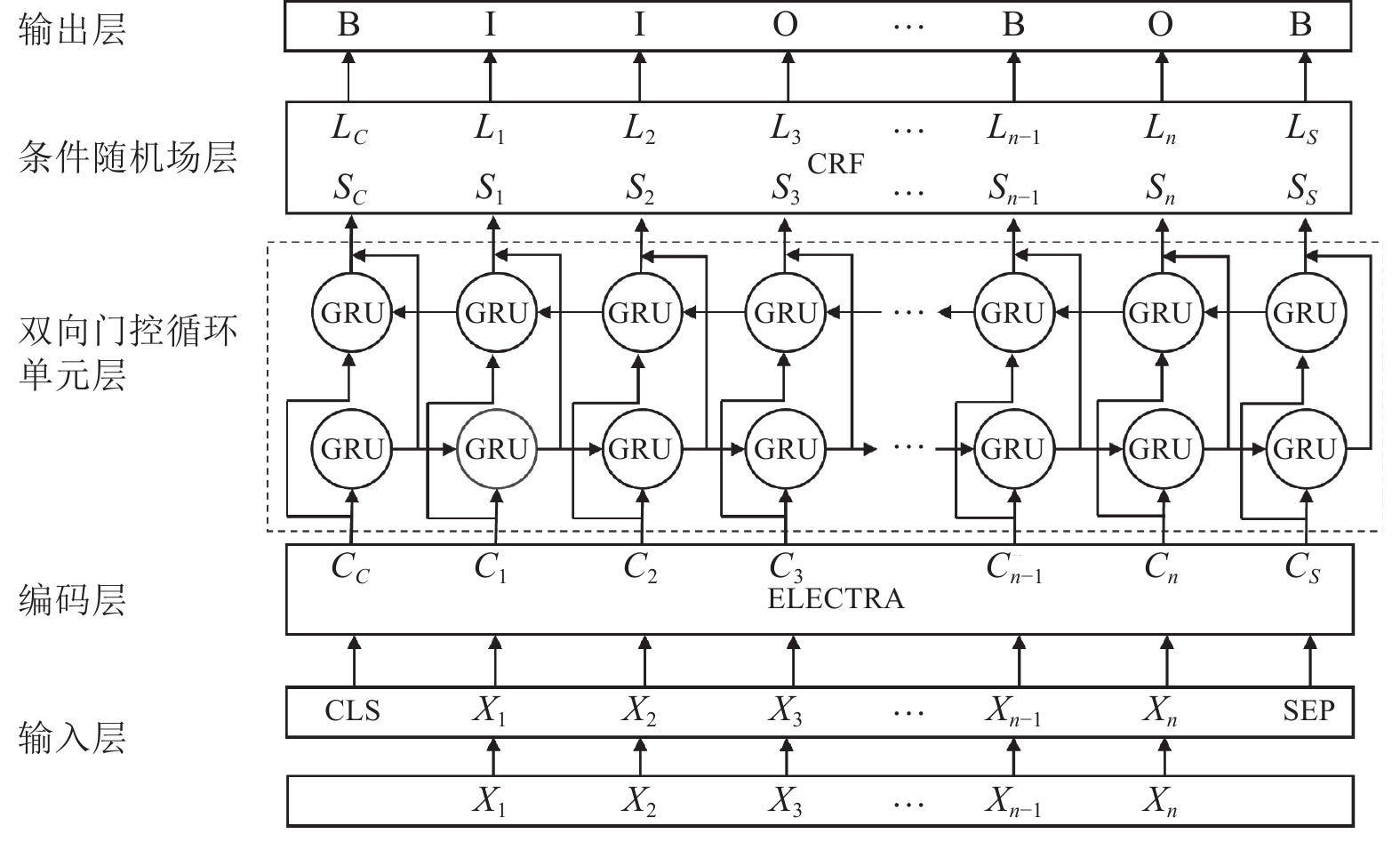
|
图 1 ELECTRA-BiGRU-CRF模型框架图 |
1) 对原始语料进行预处理, 将文本数据
2) 将序列
3) 将
4)最后利用条件随机场模型处理标签偏差, 学习标签之间的约束关系进而确定标签为某一实体类型的概率
在模型中所使用的预训练语言模型ELECTRA主要由生成器(Generator)和判别器(Discriminator)两部分组成. 其中, 生成器是一个与BERT相同的掩码语言模型. 在训练过程中, 生成器的训练方式是将输入序列
对于判别器来说, 其将经过生成器恢复后的输出序列当作输入序列, 对于每一个token, 判别器需要进行预测每个位置的token是否与原文一致. 具体来说, 对于恢复序列
ELECTRA的最终损失函数是由生成器和判别器的损失一同组成. 在预训练阶段, 生成器和判别器共享嵌入层, 并同时训练, 旨在让模型学会理解上下文和语义, 以及判断替换的token是否合理. 在微调阶段, 生成器和判别器二者继续合作. 通过这样的过程, 模型能够更好地理解和处理非结构化文本的NER任务.
2.2 基于Neo4j的疫苗接种知识图谱构建 2.2.1 本体设计本体设计是构建知识图谱的基础环节之一. 为获得良好得可视化效果, 本体设计需具有良好的可解释性. 本研究根据疫苗接种领域的需求, 参考相关标准和数据集, 确定了构建本体的目标和范围, 然后在Protégé上完成了对疫苗接种本体的构建工作.
目前疫苗接种领域尚未出现公开的特定本体模式, 但存在较多的涉及疫苗管理的数据集可供参考. 本文参照2017年实施的中华人民共和国卫生行业标准WS 375.19-2016《疾病控制基本数据集第19部分: 疫苗管理》对实体核心类、类之间的关系和属性进行定义. 对于实体类别的定义, 采取单一化的值类型, 例如诊断的内容属于疾病类型, 由医学专业名词术语构成, 命名具有统一性、规范性. 然而实体属性大多由各类型的实体及非专业名词术语构成, 表现为口语化描述性语句构成的长段文本, 难以进行枚举. 例如家族史的描述可能包括“奶奶白细胞偏低”或者“父亲有家族遗传哮喘”.
在本体构建时, 本文使用了当下流行的本体开发工具之一Protégé. 该工具提供了关于概念类、关系和属性等实例的构建方式, 便于用户构建和管理本体. 本研究关注的实体主要为7个核心大类: 患者、手术、药物、查体、诊断、暂缓疫苗、接种建议. 根据确定的实体, 定义了6种语义关系: 所做手术、所做诊断、所做查体、服用药物、接种建议、暂缓疫苗. 此外, 需要定义患者类的属性. 对于定义好的本体结构, 通过Ontograf进行可视化, 并在图2中进行展示.
2.2.2 Neo4j存储与图谱三元组表示Neo4j数据库是一种高性能的、可拓展图形数据库, 为知识图谱的存储、管理和可视化提供了技术基础[22]. Neo4j使用节点和关系的形式来组织和存储数据, 其中节点表示实体, 关系表示实体之间的连接和关联. 用户可以在节点和关系上定义属性和标签, 以更好地描述实体和关系的特征. Neo4j还提供了拥有图查询语言Cypher, 它是一种面向图分析的声明式查询语言, 专门用于在检索、操作和管理图数据库中的数据, 执行复杂的图查询和遍历操作.
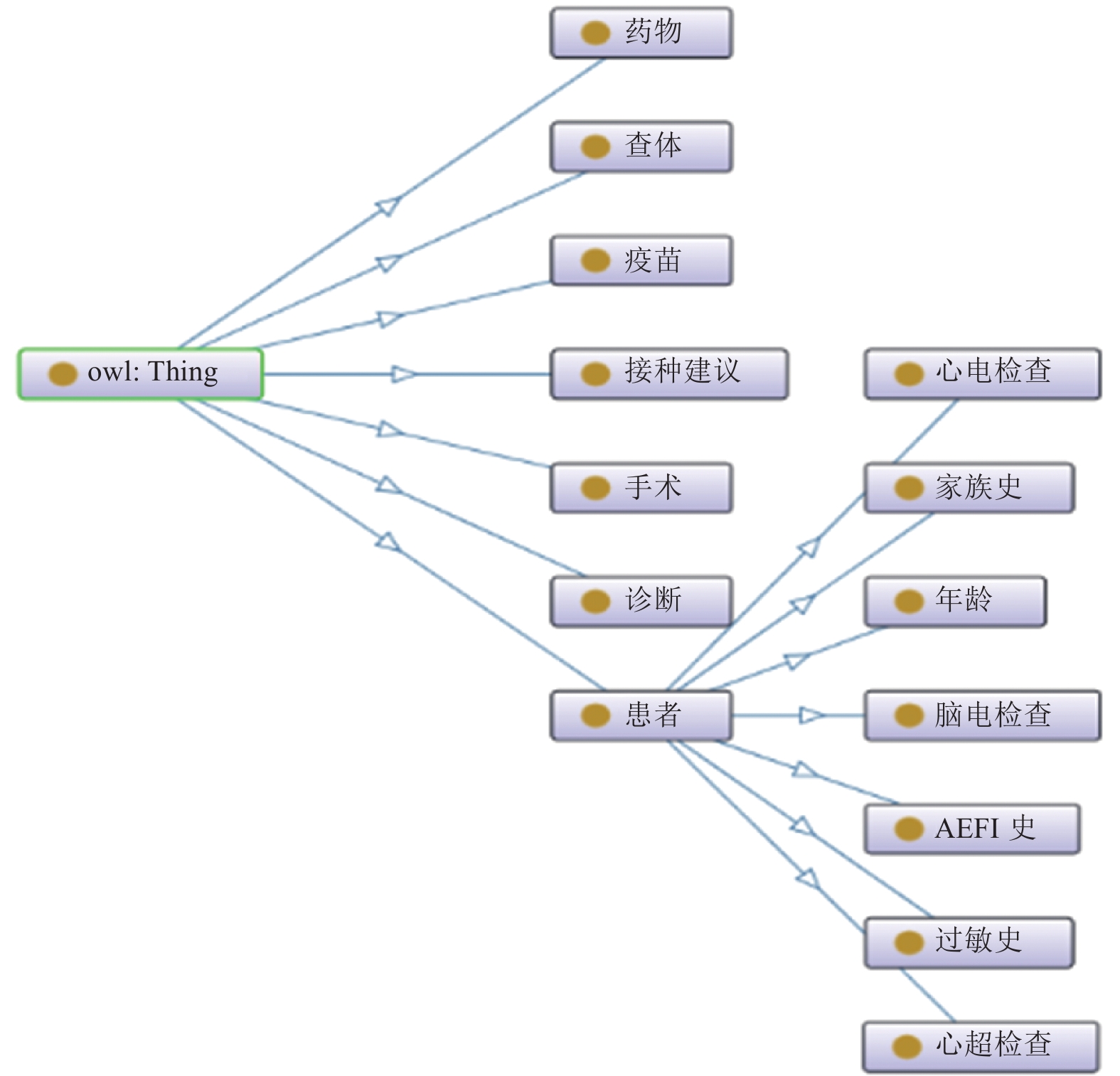
|
图 2 疫苗接种本体 |
广义的知识图谱可表示为
在实际的儿童疫苗接种工作中, 医生需要综合考虑疾病儿童的各项检查、过敏史、手术情况等健康状况, 以确定儿童能否接种疫苗. 因此在疫苗接种推荐过程中, 将患者节点考虑为中心节点, 患者的年龄和过敏史等实体作为节点的属性.
对于第2.2.1节抽取的10类实体, 对其进行后处理, 规定如下: 对于药物实体, 使用药物字典表将药物名称统一规范. 若诊断实体上下文出现亲属称谓词表中的称谓, 则将该诊断实体映射至图谱三元组中的家族史节点. 将过敏症状与过敏源合并为图谱中的过敏史节点. 对于辅助检查实体, 划分为脑电检查、心电检查、心超检查, 分别映射到患者节点的属性中. 在表1中给出这了三元组实体关系的定义, 其中患者拥有姓名编号、脑电检查、心电检查、心超检查、过敏史、家族史、年龄和AEFI史属性.
| 表 1 实体关系定义 |
2.3 基于显著性类别指导的接种建议推荐工作
本文构建了以患者为中心节点的异构知识图谱, 患者间可能存在部分共享实体, 但实体之间并不必然存在联系. 在实际诊疗过程中, 接种建议的推荐并非依赖于某一特定实体进行决策, 而是需要医生综合考虑儿童当前的身体状况. 这也代表着在查询过程中, 需要考虑各个实体对于接种建议的影响. 为了能够综合考虑各类实体, 需要在图谱中查找相似病历的接种建议, 即在图谱中查找最相似的患者子图, 以达到辅助诊断的目的.
通过利用第2.1节的自动化抽取方法, 可以得到医学实体数据, 然后采用三元组的形式进行存储, 进而将患者信息转为患者子图. 尽管这种方法能简明扼要地表示一份电子病历的关键信息, 但也不可避免地导致部分信息的丢失. 原电子病历文本可能包含一些潜在有用信息, 例如主诉文本存在对于病情口语化、细节化的大量描述, 这些信息对医生的诊断决策具有一定的参考价值. 然而, 如果直接将这些信息输入模型进行学习, 会引入过多的噪声, 因此不能将电子病历文本直接送入模型进行处理. 为此, 本文在参考Wang等人[23] 提出的显著性类别指导方法上进行了改进, 来实现接种建议的推荐. 显著性即代表了一个句子对文档中心思想的贡献程度, 其分配意味着显著性在文档中所有句子的分布情况. 本文同时考虑患者子图信息及该患者原本未经过处理的电子病历文本信息, 利用预训练语言模型, 训练了基于显著性类别指导的接种建议分类推荐模型. 该模型的训练过程如图3所示.
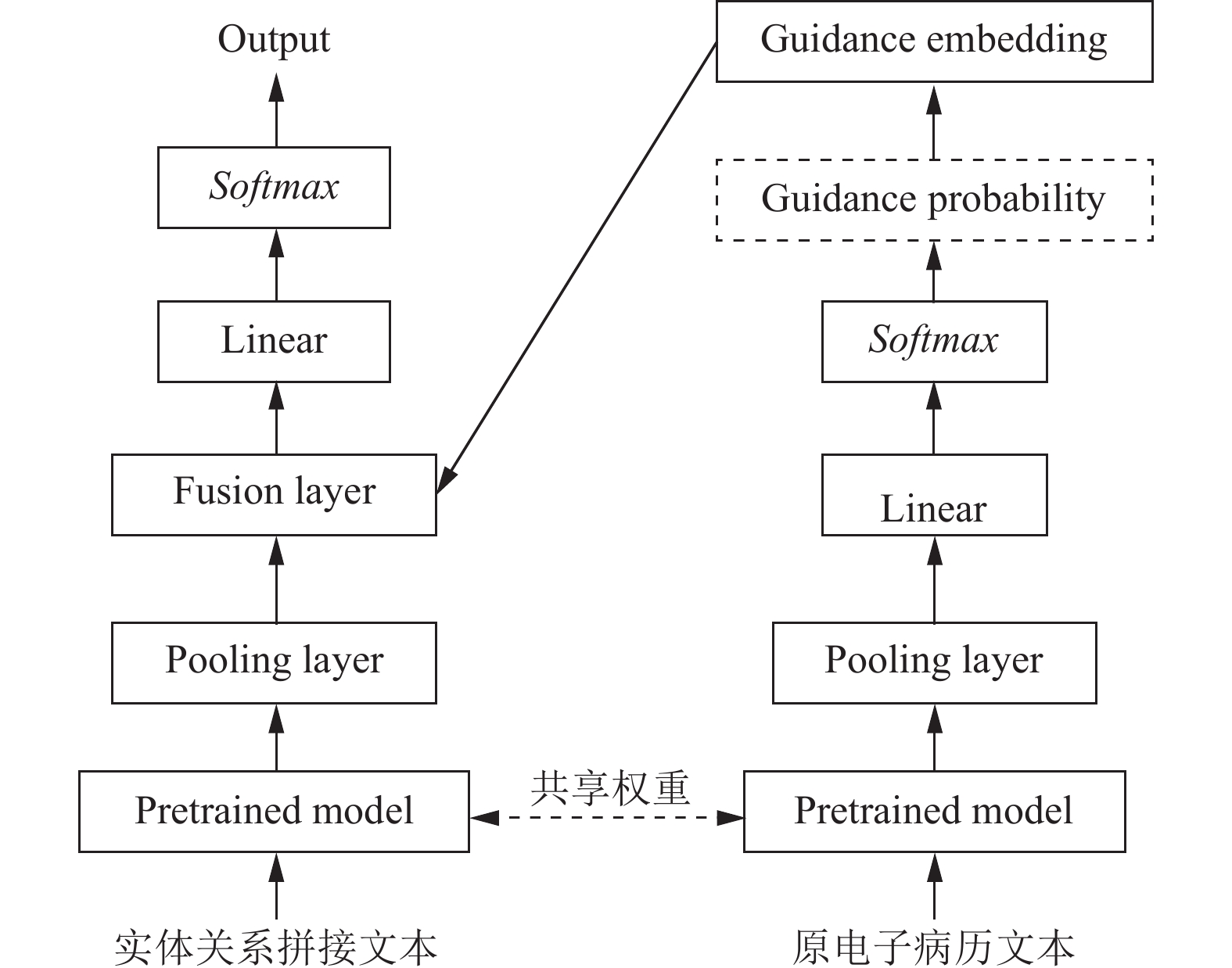
|
图 3 疫苗接种显著性类别指导方法结构图 |
具体实验步骤如下.
1) 在知识图谱中使用Cypher语言的match语句遍历每个患者和其拥有的所有关系及对应实体. 将患者所有的属性值、连接的实体和关系进行拼接, 形成关键信息文本库, 即本文的输入文本库, 输入文本使用式(1)中的
| Eent=Pooling(Pretrained(Textent)) | (1) |
2) 利用预训练语言模型学习输入文本, 得到式(1)中的
| Ecom=Pooling(Pretrained(Textcom)) | (2) |
3) 根据式(3), 通过线性层和Softmax层学习到电子病历文本属于某个类别的概率
| Guidprobability=Softmax(WT⋅Ecom) | (3) |
4) 初始化表中每个类别的编码向量, 并将得到的概率与类别编码向量相乘后, 得到类别指导向量
| Eguid=l∑l=1E(class=l)P(class=l∣x)) | (4) |
5) 将步骤4)的结果与子图查询得到关键信息的文本编码结果通过一个融合层(fusion layer)进行融合, 参照Gong等人[24]提出的融合方法, 具体见式(5). 式(6)则为最终的输出公式.
| Efusion=Concat(Eent,Eguid,Eent⊙Eguid,Eent−Eguid) | (5) |
| Output=Softmax(VT⋅Efusion) | (6) |
本研究使用的数据来源于自东部某三甲医院儿童免疫接种咨询门诊电子病历数据集系统. 采集的免疫接种咨询门诊电子病历数据共
对于模型的评价, 本研究使用准确率(Accuracy)、精确率(Precision)、召回率(Recall)和F1分数(F1-score)作为评价标准. 在式(7)中, Accuracy是指分类正确的样本占总样本个数的比例. 如式(8)所示, Precision指在分类正确的正样本个数占判定为正样本的样本个数的比例. Recall则是指分类正确的正样本个数占真正的正样本个数的比例, 其定义如式(9)所示. 在式(10)中, F1-score通过对Precision和Recall加权计算得出, 用以综合反应模型的性能.
| Accuracy=TP+TNTP+FP+TN+FN | (7) |
| Precision=TPTP+FP | (8) |
| Recall=TPTP+FN | (9) |
| F1-score=2×Precision×RecallPrecision+Recall | (10) |
其中, TP为模型正确地将正类样本预测为正类别的数量, TN为模型正确地将负类样本预测为负类别的数量, FN为模型错误地将正类样本预测为负类别的数量, FP为模型错误地将负类样本预测为正类样本的数量. 此外, 研究的实验环境配置参数如表2所示.
| 表 2 实验环境配置 |
3.3 实体识别实验
实体识别实验选取
实验采用BERT-CRF、BERT-BiLSTM-CRF、RoBERTa-BiGRU-CRF与本文所提出的模型进行对比, 其对比结果如表3所示. 由表3可知, 在模型整体层面, ELECTRA-BiGRU-CRF模型相较于对比模型BERT-CRF的Precison、Recall和F1-score分别提升了9.25%、8.95%和9.13%. 造成这一结果的原因是ELECTRA和BERT的区别, 即在掩码处理方面, BERT从每个样本随机选取15% 的token进行掩盖, 其中80%被真实token替换, 而在预测阶段, 并非对所有被掩盖的token及逆行预测, 而只是针对其中一小部分进行预测. 这么做会造成预训练与微调时的不匹配. 相比之下, ELECTRA在预测任务中, 判别器从所有输入的token中学习, 避免了BERT中的不匹配问题.
| 表 3 模型对比结果表 (%) |
相较于BERT-BiLSTM-CRF的Precision、Recall和F1-score分别提升了5.98%、7.17%和6.55%. 相较于RoBERTa-BiGRU-CRF的Precision、Recall和F1-score分别提升了3.43%、6.21%和4.77%. 由此可见, 本研究所提出模型的学习能力优于对比模型, 具有较优的识别性能. 在10类实体识别层面, 对模型结果进行分析. 如表4所示, 以相对来说表现最好的RoBERTa-BiGRU-CRF为例, 与本文提出的ELECTRA-BiGRU-CRF进行对比, 分析结果发现: 在模型整体层面, ELECTRA-BiGRU-CRF较RoBERTa-BiGRU-CRF的提升幅度并不显著, 但在每一类的实体识别中, RoBERTa-BiGRU-CRF的性能表现得不如ELECTRA-BiGRU-CRF稳定, 尤其在年龄、手术、AEFI史的识别方面表现较差. 总的来说, 在儿童免疫接种咨询门诊电子病历实体识别任务中, ELECTRA-BiGRU-CRF模型更有优势.
| 表 4 RoBERTa-BiGRU-CRF与ELECTRA-BiGRU-CRF模型识别实体效果对比 (%) |
如表5所示, 通过实验发现, 在ELECTRA-CRF模型中引入BiGRU模块, 能够增强序列建模和上下文理解能力, 模型的性能会更为稳定. 进一步分析发现, 在未引入BiGRU模块的情况下, 部分实体识别性能不稳定, 例如“过敏原”实体的识别表现出色, 但“过敏症状” 识别的准确率却不理想.
| 表 5 ELECTRA-CRF与ELECTRA-BiGRU-CRF模型识别实体效果对比 (%) |
总体而言, ELECTRA-BiGRU-CRF在学习电子病历文本数据的特征信息方面是有效的. 具体而言, 对于“诊断”“过敏症状”“过敏原”“查体”“辅助检查”“药物” “疫苗”这8类实体识别的F1-score均在95%以上. 然而“年龄”和“AEFI史”的识别效果欠佳. 分析可能的原因有两点.
1) 在儿童免疫接种咨询门诊中, 儿童的年龄记录精度通常高于成年人, 精确到月份, 例如5月龄、1岁2月龄. 这导致了在病历中, 儿童的年龄会与病历描述中记录的既往病史和部分检查检验的年月产生重叠和歧义, 这会导致模型对实体的边界识别困难, 无法准确区分在当前语境下实体的类型.
2) 对于AEFI史, 由于疫苗接种反应记录大多为医生手动填写, 因此存在相关描述语言风格存在不统一的情况, 且该实体中关于症状实体口语化描述多, 会一定程度上造成“AEFI史”实体识别效果欠佳的问题. 此外, 实验的数据集中, “AEFI史”的样本量相对其他实体较少, 这也会一定程度上对模型学习造成影响.
综上所述, ELECTRA-BiGRU-CRF相较于其他对比模型而言, 无论是在模型整体表面层面, 还是单个实体识别的层面, 都展现出了自身的优势.
3.4 疫苗接种知识图谱存储与可视化本文选用Neo4j图数据库对儿童疫苗接种知识图谱进行存储. 在图数据库存储中, 图的顶点表示知识图谱中的实体, 关系使用顶点与顶点之间的边进行表示.
Neo4j图数据库支持多种方式进行数据导入以满足不同场景和需求. 在本研究中, 对电子病历数据进行处理后, 形成实体和关系的JSON文件, 通过py2neo工具包对实体识别后的数据进行批量导入, 使用Cypher语言进行节点的增、删、改、查等操作.
本研究基于
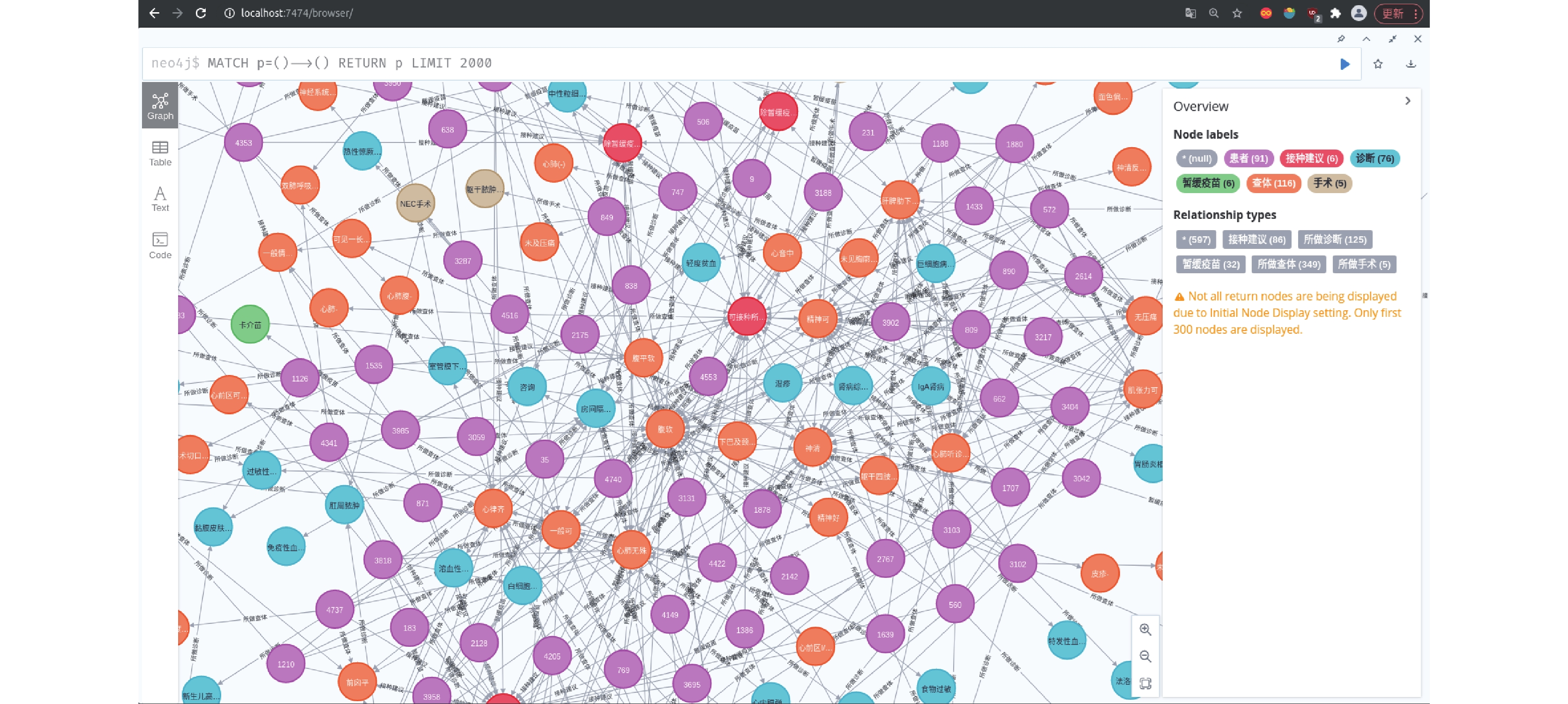
|
图 4 基于Neo4j的图谱可视化 |
图5主要是对患者节点属性进行可视化展示, 其中患者节点包含了8种属性, 使用患者姓名编号作为患者的唯一标识.
3.5 基于显著性类别指导的接种建议推荐实验实验选取
在实验中, 采用了在中文医学语料库上进行预训练的语言模型, 包括BERT[16]、DKPLM[25]、ERNIE[26]进行了分类预测实验. 实验结果如表7和表8所示, 在BERT模型上, 采用显著性类别指导方法, 精确率提高了2.58%. 在DKPLM模型上, 精确率提高了2.3%. 在BERT和DKPLM上, F1-score均有所提升. 而在ERNIE模型上, 采用显著性类别指导方法精确率也有所提升, 表现相对来说较为稳定. 总体而言, 基于显著性类别指导的方法在模型表现结果稳定的情况下, 精确率都得到了提升. 这表明通过引入原始电子病历文本信息, 并使用接种建议显著类别指导的方法帮助模型理解文本信息, 能够让模型分类准确率均得到提升, 从而提高接种建议推荐的准确性.
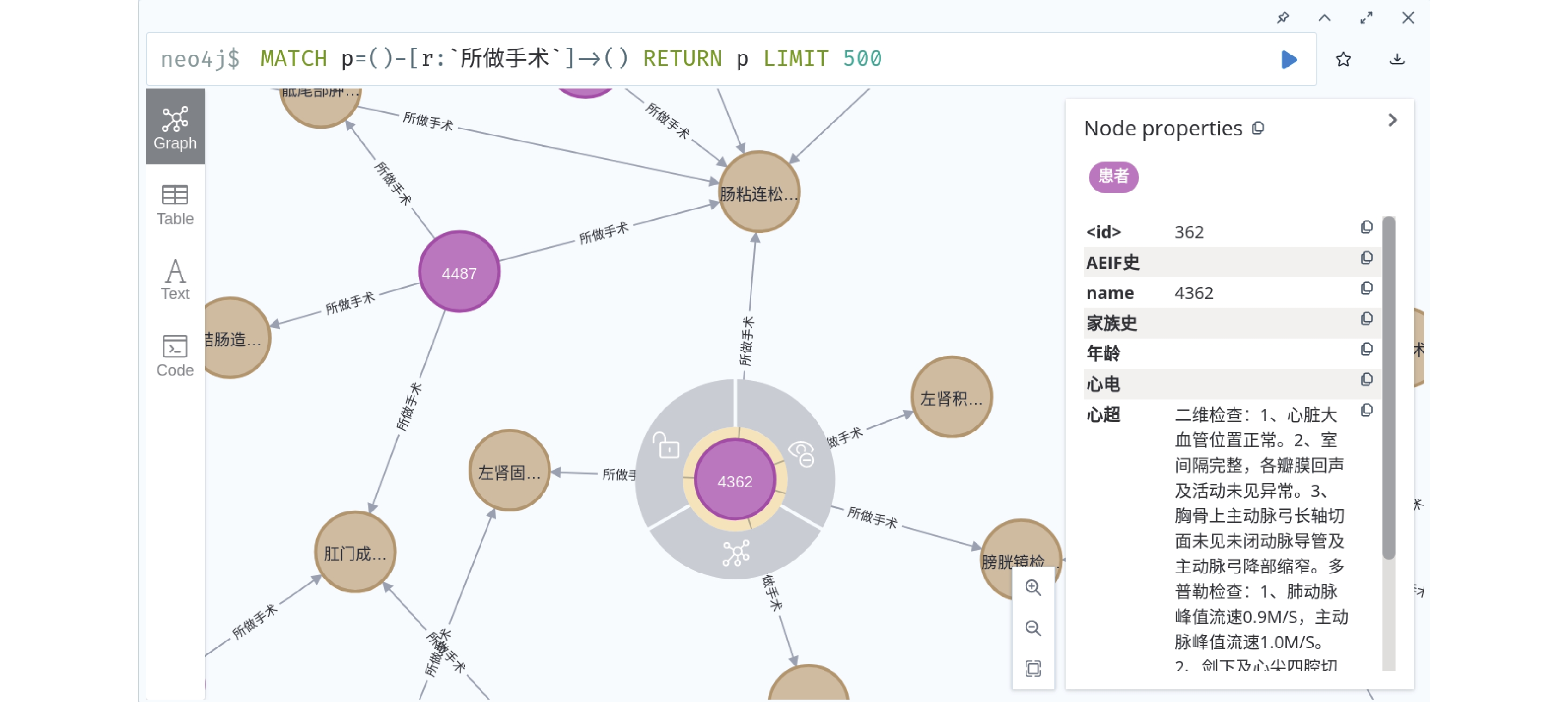
|
图 5 患者节点属性可视化 |
| 表 6 接种建议类型 |
| 表 7 基于预训练语言模型的推荐结果 (%) |
| 表 8 基于显著性类别指导的推荐结果(%) |
4 结论与展望
本文基于儿童疫苗接种咨询门诊电子病历数据构建知识图谱, 实现了儿童疫苗接种建议推荐工作. 为实现知识图谱的构建, 本文提出了ELECTRA-BiGRU-CRF实体抽取方法, 该方法相比于其他基线模型, 在疫苗接种领域具有更好的识别性能, 为后续基于知识图谱的疫苗接种推荐工作奠定基础. 在疫苗接种推荐工作中, 本研究采用接种建议显著性类别指导的思想, 让语言模型在学习图谱存储的患者信息的同时, 考虑原电子病历文本的类别信息, 以提升接种建议推荐的学习效果. 该方法在3种不同的模型上进行实验, 实验结果表明分类推荐准确率均得到了提升. 本文提出的方法能够更有效地完成知识图谱构建以及接种建议分类推荐工作.
医学知识图谱的构建及其下游任务的应用一直以来都是复杂且严谨的工作. 在后续工作中, 将引入更多罕见病患儿的病历数据, 研究数据完整性和规范性方案, 以平衡数据类别分布, 并细化数据类别, 进而提高接种建议分类推荐模型在准确率上的表现. 此外, 本研究未来将探索图神经网络在异构知识图谱查询方法中的应用.
| [1] |
王富云, 刘桂红. 特殊健康儿童预防接种评估及不良反应处理. 广州医药, 2023, 54(10): 46-51. |
| [2] |
陈福星, 潘雪娇, 梁辉, 等. 浙江省2015–2019年儿童型三价灭活流感病毒裂解疫苗预防接种不良反应报告发生率. 中国疫苗和免疫, 2022, 28(4): 436-439. DOI:10.19914/j.CJVI.2022084 |
| [3] |
宋祎凡, 曾淇民, 余文周, 等. 我国儿童监护人免疫规划疫苗犹豫现状及影响因素分析. 中国预防医学杂志, 2023, 24(12): 1303-1308. DOI:10.16506/j.1009-6639.2023.12.006 |
| [4] |
Stassen P, Westerman D. Novice doctors in the emergency department: A scoping review. Cureus, 2022, 14(6): e26245. DOI:10.7759/cureus.26245 |
| [5] |
王彩云, 郑增亮, 蔡晓琼, 等. 知识图谱在医学领域的应用综述. 生物医学工程学杂志, 2023, 40(5): 1040-1044. |
| [6] |
王丹蕾, 邱五七, 都率. 疫苗接种风险沟通中的信任问题与建议. 卫生软科学, 2023, 37(4): 82-86. DOI:10.3969/j.issn.1003-2800.2023.04.020 |
| [7] |
胡丹丹. 神经系统疾病儿童免疫接种策略. 临床儿科杂志, 2022, 40(3): 184-188. DOI:10.12372/jcp.2022.21e0028 |
| [8] |
Wang B, Shao XQ, Wang D, et al. Vaccinations and risk of systemic lupus erythematosus and rheumatoid arthritis: A systematic review and meta-analysis. Autoimmunity Reviews, 2017, 16(7): 756-765. DOI:10.1016/j.autrev.2017.05.012 |
| [9] |
Zhang XM, Meng MM, Sun XL, et al. FactQA: Question answering over domain knowledge graph based on two-level query expansion. Data Technologies and Applications, 2020, 54(1): 34-63. DOI:10.1108/DTA-02-2019-0029 |
| [10] |
Chandak P, Huang KX, Zitnik M. Building a knowledge graph to enable precision medicine. Bioinformatics, 2023, 10(1): 67. DOI:10.1038/s41597-023-01960-3 |
| [11] |
Joshi P, Masilamani V, Mukherjee A. A knowledge graph embedding based approach to predict the adverse drug reactions using a deep neural network. Journal of Biomedical Informatics, 2022, 132: 104122. DOI:10.1016/j.jbi.2022.104122 |
| [12] |
Tao XH, Pham T, Zhang J, et al. Mining health knowledge graph for health risk prediction. World Wide Web, 2020, 23(4): 2341-2362. DOI:10.1007/s11280-020-00810-1 |
| [13] |
An B. Construction and application of Chinese breast cancer knowledge graph based on multi-source heterogeneous data. Mathematical Biosciences and Engineering, 2023, 20(4): 6776-6799. DOI:10.3934/mbe.2023292 |
| [14] |
Liu FF, Liu MT, Li MT, et al. Automatic knowledge extraction from Chinese electronic medical records and rheumatoid arthritis knowledge graph construction. Quantitative Imaging in Medicine and Surgery, 2023, 13(6): 3873-3890. DOI:10.21037/qims-22-1158 |
| [15] |
Floridi L, Chiriatti M. GPT-3: Its nature, scope, limits, and consequences. Minds and Machines, 2020, 30(4): 681-694. DOI:10.1007/s11023-020-09548-1 |
| [16] |
Devlin J, Chang MW, Lee K, et al. BERT: Pre-training of deep bidirectional transformers for language understanding. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. Minneapolis: Association for Computational Linguistics, 2019. 4171–4186.
|
| [17] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 6000–6010.
|
| [18] |
Jagannatha AN, Yu H. Bidirectional RNN for medical event detection in electronic health records. Proceedings of the 2016 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. San Diego: Association for Computational Linguistics, 2016. 473–482. [doi: 10.18653/v1/N16-1056]
|
| [19] |
Wang Q, Zhou YM, Ruan T, et al. Incorporating dictionaries into deep neural networks for the Chinese clinical named entity recognition. Journal of Biomedical Informatics, 2019, 92: 103133. DOI:10.1016/j.jbi.2019.103133 |
| [20] |
Dai ZJ, Wang XT, Ni P, et al. Named entity recognition using BERT BiLSTM CRF for Chinese electronic health records. Proceedings of the 12th International Congress on Image and Signal Processing, BioMedical Engineering and Informatics. Suzhou: IEEE, 2019. 1–5. [doi: 10.1109/CISP-BMEI48845.2019.8965823]
|
| [21] |
Clark K, Luong MT, Le QV, et al. ELECTRA: Pre-training text encoders as discriminators rather than generators. Proceedings of the 8th International Conference on Learning Representations. Addis Ababa: OpenReview.net, 2020.
|
| [22] |
Quan XP, Cai WJ, Xi CH, et al. AIMedGraph: A comprehensive multi-relational knowledge graph for precision medicine. Database, 2023, 2023: baad006. DOI:10.1093/database/baad006 |
| [23] |
Wang F, Song KQ, Zhang HM, et al. Salience allocation as guidance for abstractive summarization. Proceedings of the 2022 Conference on Empirical Methods in Natural Language Processing. Abu Dhabi: Association for Computational Linguistics, 2022. 6094–6106.
|
| [24] |
Gong CG, Yu JF, Xia R. Unified feature and instance based domain adaptation for aspect-based sentiment analysis. Proceedings of the 2020 Conference on Empirical Methods in Natural Language Processing (EMNLP). Association for Computational Linguistics, 2020. 7035–7045. [doi: 10.18653/v1/2020.emnlp-main.572]
|
| [25] |
Zhang TL, Wang CY, Hu N, et al. DKPLM: Decomposable knowledge-enhanced pre-trained language model for natural language understanding. Proceedings of the 36th AAAI Conference on Artificial Intelligence. AAAI, 2022. 11703–11711. [doi: 10.1609/aaai.v36i10.21425]
|
| [26] |
Wang Q, Dai ST, Xu BF, et al. Building chinese biomedical language models via multi-level text discrimination. arXiv:2110.07244, 2022.
|
 2024, Vol. 33
2024, Vol. 33



