2. 成都西图科技有限公司, 成都 610065
2. Chengdu Xitu Technology Co. Ltd., Chengdu 610065, China
岩心颗粒是一种研究颗粒型储层的重要地质资料, 岩心颗粒图像通常包含多种类型的矿物颗粒, 颗粒分割效果对地质专家进行相关地质信息的评估至关重要. 传统的岩心颗粒提取方法主要是由地质研究人员手工分割完成, 需要大量的时间和精力, 容易受到主观因素的影响, 导致颗粒提取准确度不够高.
随着图像处理技术的发展, 越来越多的图像处理算法被运用到图像分割领域. 任大勇等人[1]在分水岭图像分割算法中引入位图切割, 但分割结果仍然存在比较明显的过分割现象. 董领等人[2]提出基于改进的SLIC的岩心颗粒图像边缘分割算法, 该方法能够将颗粒的大致轮廓提取出来, 但存在颗粒的过分割和误分割现象. 郭艳婕等人[3]在分割过程中加入自适应多阈值LBP纹理特征, 并使用DBSCAN算法对超像素块进行聚类合并, 但用于岩心颗粒图像处理时效率低且合并效果并不理想. 高扬等人[4]将模糊距离变换用于岩心图像颗粒的分割, 虽然能够有效地分割出粘连颗粒, 但是分割的颗粒边缘不够平滑. 虽然使用这些方法可以有效地提高颗粒提取的效率, 但仍存在一些问题. 当岩心颗粒边界模糊, 背景区域与颗粒区域相似时, 这些方法难以得到准确的提取结果.
近年来, 随着深度学习的快速发展, 相关的图像处理算法已经显著改善, 并且取得了令人瞩目的效果. Long等人[5]提出全卷积网络FCN, 该网络采用跳跃连接结构, 使分割结果更加准确和精细. Ronneberger等人[6]提出了UNet网络, 该网络通过跳跃连接将特征连接在一起, 形成对称的U型结构. 刘莹等人[7]将迁移学习与UNet网络相结合, 用于室内运动目标阴影分割, 能够基本解决阴影难以识别和分割困难的问题. 郑伟等人[8]在UNet网络中嵌入感受野模块和通道注意力模块, 能够有效提升脑肿瘤分割的精度, 具有良好的分割性能. Oktay等人[9]提出了Attention UNet, 将注意力门加入到UNet的跳跃连接中, 提高了模型对目标区域的灵敏度和准确性. Alom等人[10]提出了R2UNet网络, 该网络在UNet的下采样和上采样过程中增加了循环卷积模块, 使得模型具有较好的分割准确率. Badrinarayanan等人[11]提出了SegNet网络, 该网络将最大池化指数转移至解码器中, 进一步降低了分割的复杂度. Zhou等人[12]提出了UNet++网络, 该网络在编码器和解码器之间加入了密集的短连接, 使得网络更加深层次、更加精细化, 从而提高了分割的效果. Huang等人[13]提出了UNet3+网络, 该网络中提出了全尺度跳跃连接, 将不同尺度特征图中的低级细节与高级语义相结合, 以最大限度地使用全尺度特征图, 提高分割精度.
因此, 本文将UNet3+用于颗粒提取, 并进行深入分析, 针对岩心颗粒图像存在颗粒边缘模糊、背景与颗粒色彩复杂等问题, 设计了一种基于改进UNet3+的岩心图像颗粒提取算法, 在UNet3+网络的各编码层加入感受野模块(receptive field block, RFB)[14], 增大网络的感受野, 提高分割的准确率. 并在RFB模块后嵌入卷积块注意力模块(convolutional block attention module, CBAM)[15]来抑制背景区域的干扰, 提高目标区域的权重.
1 基于改进UNet3+的岩心颗粒提取算法 1.1 改进的UNet3+网络结构UNet3+是一种用于语义分割的卷积神经网络结构, 是对经典的UNet网络的改进和扩展. UNet3+结构在保留UNet的基本框架和思想的同时, 加入了更多的跨层信息交互机制, 提高了网络的性能和泛化能力. UNet3+网络的基本处理过程是在编码过程中使用多个卷积层和池化层, 提取不同尺度的特征表示, 以适应不同大小的物体和场景. 在解码过程中使用跨层连接的方式, 融合下采样过程中提取的特征图与上采样过程中提取的特征图, 以便在分割时更好地保留物体边界和局部细节信息, 增强深层网络的语言信息, 最终实现对逐个像素点分类.
由于岩心颗粒图像的颜色丰富, 形状大小差异较大, 且存在边界模糊等情况, 直接使用UNet3+网络提取岩心颗粒难以提取出比较完整的颗粒, 从而导致颗粒的欠分割或过分割. 鉴于岩心颗粒的特性, 本文提出一种新的岩心颗粒提取模型. 本文在UNet3+的下采样部分加入RFB模块, 增强模型对岩心颗粒的特征表达, 并在RFB模块后加入CBAM模块, 提升模型对多尺度信息的捕获能力.
本文提出的改进的UNet3+网络由编码器, 跳跃连接和解码器3个部分组成, 网络结构如图1所示.
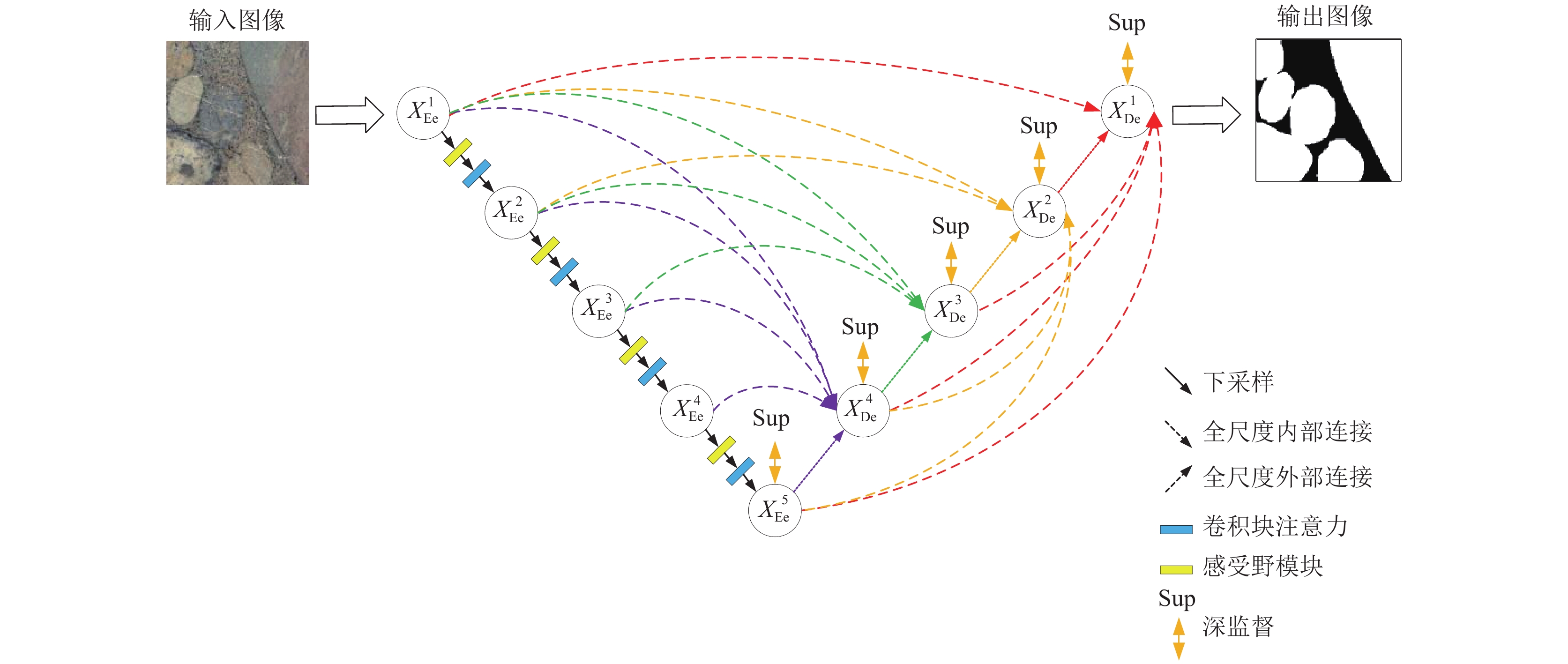
|
图 1 改进的UNet3+网络结构 |
1.2 感受野模块(RFB)
在卷积神经网络中, 每个输出结果中的元素都对应着输入层中的一部分, 这部分被称为感受野. 感受野的大小决定了网络能够看到的信息范围, 因此扩大感受野可以帮助网络更好地理解图像中的上下文信息, 从而提高分割的准确率. RFB模块是一种卷积神经网络模块, 它使用空洞卷积和多尺度卷积核来扩大感受野和获取多尺度信息, 并通过特征融合提高模型性能.
通过引入空洞率(dilation, d)超参数, 空洞卷积可以控制卷积核处理数据时各值的间距, 从而扩大卷积核的感受野. 图2中卷积核尺寸为
| n=k+(k−1)×(d−1) | (1) |
其中,
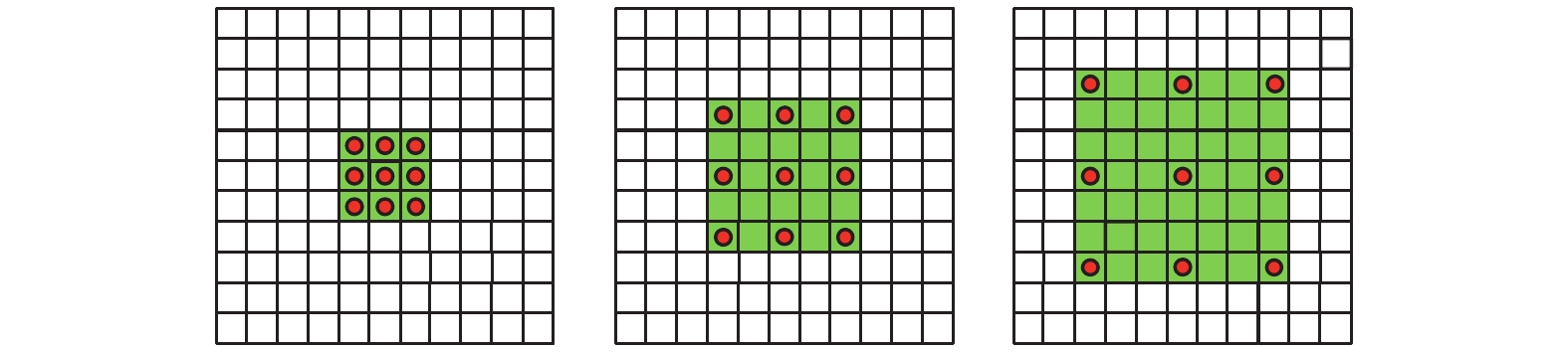
|
图 2 不同空洞率的空洞卷积 |
根据式(1), 我们可以发现, 图2中的感受野分别是3、5和7, 这表明, 通过调整空洞率, 我们可以有效地扩大卷积核的感受野, 从而更好地捕捉图像中的特征信息.
RFB模块的结构如图3所示, 该模块首先使用3个
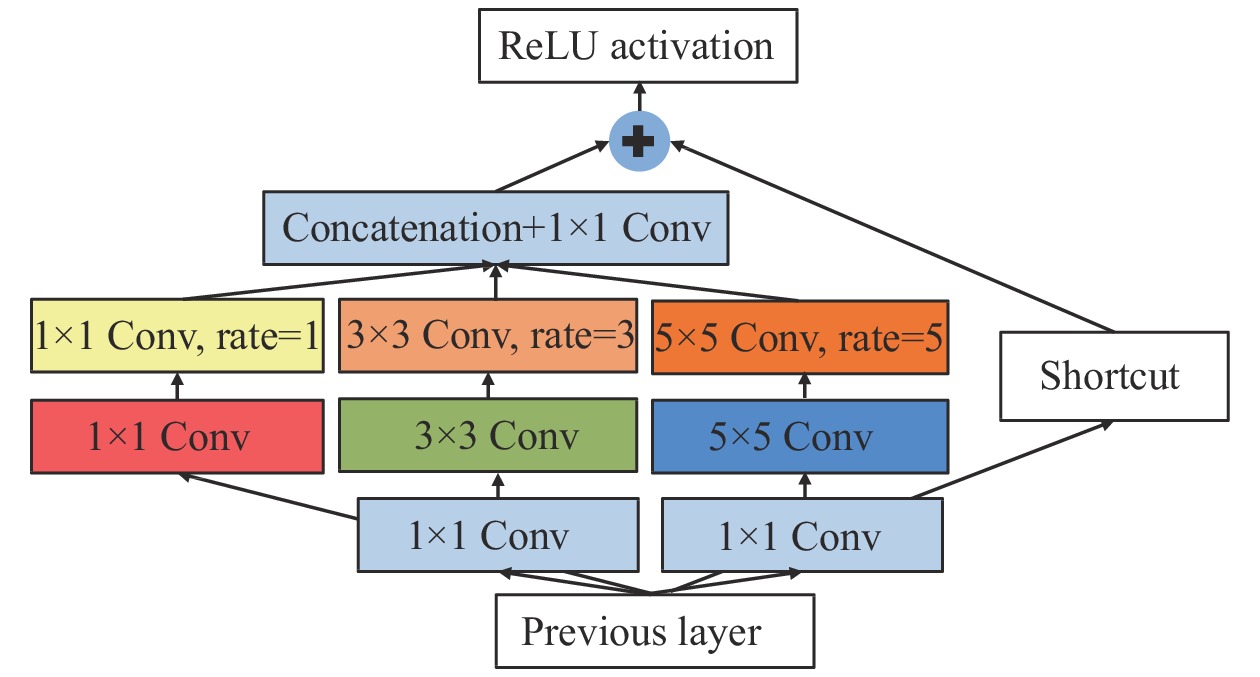
|
图 3 RFB结构 |
通过将RFB模块插入不同的编码层, 可以显著改善岩心颗粒图像的感受野, 从而更好地捕捉到不同形状、颜色的岩心颗粒, 从而极大地提高岩心颗粒分割的精度.
1.3 卷积块注意力模块(CBAM)卷积块注意力是一种用于图像分类和目标检测的神经网络模块, CBAM将通道注意力和空间注意力机制相结合, 以自适应的方式对特征图进行加权, 从而提高了网络的表示和泛化能力. CBAM模块由通道注意力(CAM)和空间注意力(SAM)两部分组成, CBAM的结构如图4所示[16].
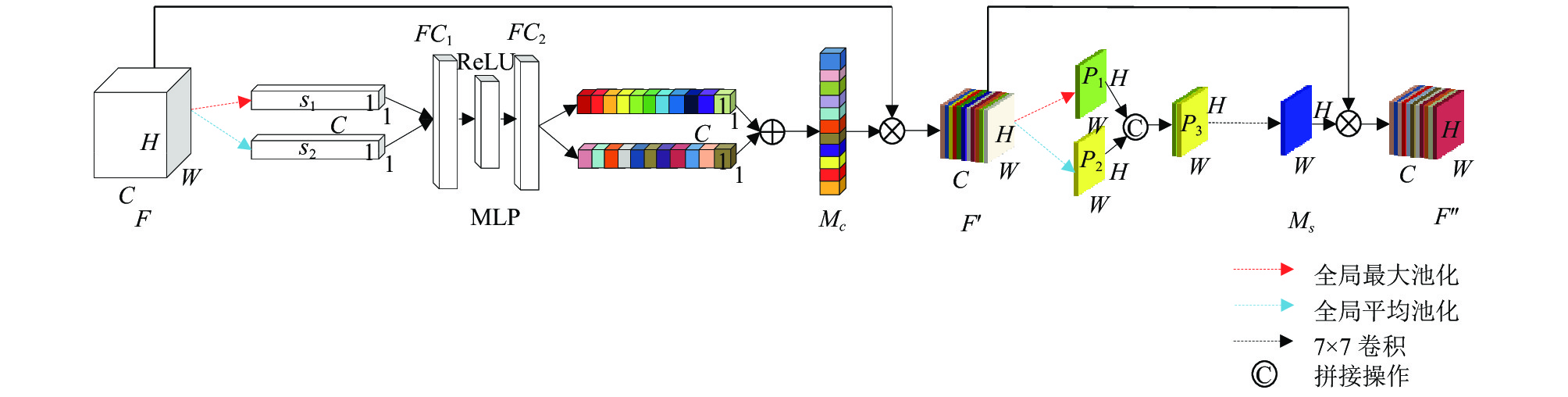
|
图 4 CBAM结构 |
(1)通道注意力模块(CAM)
在这个过程中, 我们需要提取特征图的信息, 记输入特征图为
| Mc(F)=σ{MLP[AvgPool(F)]+MLP[MaxPool(F)])} | (2) |
其中,
(2)空间注意力模块(SAM)
将特征图
| P1=MaxPool(F′) | (3) |
| P2=AvgPool(F′) | (4) |
| Ms=σ{f7×7[P1;P2} | (5) |
其中,
最后, 将
在同样的实验环境下, 本文在岩心颗粒图像数据集上对不同算法进行对比实验来验证所提方法的合理性与有效性, 同时为了评估所提算法的泛化能力, 在两个公开的数据集(EBHI-Seg[17]和EMDS-6[18])上进行了不同算法的对比实验. 另外, 为了验证各个模块的有效性, 本文对它们进行了消融实验.
2.1 数据集介绍岩心颗粒图像是使用高分辨率线阵相机所采集得到的, 所以实际中的岩心颗粒图像分辨率较大, 直接放进网络会使得显存爆炸, 所以先将采集得到的原始岩心图像进行裁剪, 如图5所示, 共得到148张256×256 pixel大小的颗粒图像, 使用LabelMe软件进行标注, 生成JSON文件, 并转换成图像.
EBHI-SEG是一个肠镜病理图像数据集, 该数据集包含6种类型肿瘤分化阶段的图像和相应的标签图像, 可用于结直肠癌医学诊断. 本文选用其中的Polyp类型共474张224×224 pixel图像和相应的标签图像作为对比实验的数据集.
EMDS-6是一个环境微生物数据集, 包含21种环境微生物, 每种环境微生物包含40张原始图像和40张标签图像, 共计1680张图像.

|
图 5 岩心颗粒图片 |
2.2 评价指标
通过使用3个广泛应用于语义分割的衡量指标平均交并比(mIoU)、平均像素准确率(mPA)和频权交并比(FWIoU)来对本文提出的算法进行评估. 上述指标的值都在0和1之间, 越接近1, 模型的效果越好. 平均交并比、平均像素准确率和频权交并比的计算公式如式(6)–式(8)所示:
| mIoU=TPTP+FP+FN | (6) |
| mPA=TP+TNFN+TP+FP+FN | (7) |
| FWIoU=TP+FNTP+FP+TN+FN×TPTP+FP+FN | (8) |
其中,
实验环境为Ubuntu 18.04 LTS系统, 处理器为Intel(R) Core(TM) i7-9700, GPU为NVDIA GeForce RTX2080Ti, 软件环境为CUDA 10.2, 实验中使用的深度学习框架是PyTorch 1.8.0.
训练时使用Adam优化器来优化数据, 学习率调整为0.001, 迭代的批量大小设为2, 最后确定的训练次数为60. 本文需要对前景和背景进行分割, 选择二元交叉熵损失函数(BCEWithLogitsLoss)作为模型训练时的损失函数, 该损失函数的计算公式如式(9)和式(10)所示:
| BCELoss=−1N[N∑i=1(yilnxi+(1−yi)ln(1−xi))] | (9) |
| BCEWithLogitsLoss=Sigmoid+BCELoss | (10) |
其中,
将本文提出的方法与SegNet、R2UNet、UNet、UNet++以及UNet3+进行对比实验来验证本文提出的方法的有效性. 本文提出的方法和各个算法的分割结果和指标结果分别如图6和表1所示. 在这个数据集上, SegNet, R2UNet和UNet++算法提取颗粒的能力较弱, 这与表1中它们对应的指标数据相一致. U2Net和UNet3+算法在提取颗粒边缘表现良好, 但提取的颗粒存在欠分割现象. 本文算法在编码过程中引入RFB模块, 增强了网络提取颗粒的能力, 还在每个RFB模块后嵌入CBAM模块, 提高网络定位颗粒区域的准确性. 与其他算法相比, 本文算法在mIoU, mPA和FWIoU指标上高于其他算法, 并且分割的颗粒更接近于标签图像, 因此本文算法在提取岩心颗粒方面具有最好的性能.
2.4.2 改进的UNet3+泛化性能评估通过对两个公开数据集上的实验进行对比, 我们发现各种算法的分割效果都有明显的差异, 具体情况请参见图7. 在EBHI-Seg数据集上, 其他方法都存在欠分割的现象, 本文方法的结果更接近于标签图像. 在EMDS-6数据集上, 本文方法所受干扰最少, 提取效果最好.
本组实验的指标结果如表2所示, 在EBHI-Seg数据集上, 相比于UNet3+, 本文方法在mIoU, mPA和FWIoU提升了1.24%, 0.66%和1.23%. 在EMDS-6数据集上, 相比于UNet3+, 本文方法在mIoU, mPA和FWIoU提升了6.79%, 8.35%和2.14%. 由于在编码阶段引入CBAM模块, 增强了网络前景与背景的区分能力, 在EBHI-Seg数据集和EMDS-6数据集上提取结果噪声显著降低, 而在CBAM前加入的RFB结构使得本文方法在EBHI-Seg数据集上提取的肿瘤块更加接近于标签图像, 而其他网络则有出现欠分割的情况. 在两个公开数据集上, 本文算法指标高于其他算法, 从而验证了本文提出的方法具有一定的泛化性能.

|
图 6 岩心颗粒图像对比结果可视化 |
| 表 1 岩心颗粒数据集的对比结果 (%) |
2.5 消融实验
本文在实验参数设置相同的情况下做了4组消融实验来验证本文提出的方法所采用模块的有效性, 通过消融实验, 我们发现两个模块都能够显著提升网络的分割精度, 详情请参见表3, 其中N代表不加入模块, Y代表加入模块. CBAM使得模型更加聚焦于颗粒区域, 抑制背景区域的干扰, 提高了分割的准确率, 但是对颗粒边缘的提取效果仍有待提升, 而RFB模块通过增加感受野, 提取不同尺度上的特征, 增强了模型对不同尺度的颗粒的检测能力, 有效改善颗粒粘连而导致分割不准确的情况. 因此本文在UNet3+网络中同时加入CBAM和RFB模块来提高网络对颗粒的分割效果, 不同模块的对比实验如表3所示, 从表3可以看出, 各个模块在改善网络的分割效果方面具有一定的功效.

|
图 7 公共数据集图像对比结果可视化 |
| 表 2 公共数据集的对比结果 (%) |
| 表 3 不同模块的消融实验 |
3 结论
为了解决岩心颗粒提取中感受野受限、分割精度低的问题, 本文提出了一种基于UNet3+网络的改进的岩心颗粒提取算法, 通过引入RFB模块增大网络的感受野, 并使用CBAM模块来增强模型对颗粒的聚焦能力, 有效地提升了模型对岩心颗粒的分割效果. 经过实验证明, 本文提出的算法在提取岩心颗粒方面表现出色.
在EMDS-6数据集和EBHI-Seg数据集上进行的与其他分割模型的比较实验结果表明, 本文方法具有良好的泛化性能, 显示了其有效性, 为岩心颗粒的提取提供了更便捷的解决方案.
| [1] |
任大勇, 贾振红, 杨杰, 等. 结合位图切割和区域合并的彩色图像分割. 计算机工程与应用, 2019, 55(2): 162-167. |
| [2] |
董领, 卿粼波, 何小海, 等. 基于改进的SLIC的岩心颗粒图像边缘分割算法. 智能计算机与应用, 2021, 11(9): 54-58. |
| [3] |
郭艳婕, 杨明, 侯宇超. 改进的SLIC算法在彩色图像分割中的应用. 重庆理工大学学报(自然科学), 2020, 34(2): 158-164. |
| [4] |
高扬, 滕奇志, 熊淑华, 等. 基于模糊距离变换的岩心图像颗粒分割算法. 微型机与应用, 2017, 36(4): 47-50. |
| [5] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015. 3431–3440.
|
| [6] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [7] |
刘莹, 杨硕. 基于改进UNet网络的室内运动目标阴影分割. 计算机系统应用, 2022, 31(12): 412-419. DOI:10.15888/j.cnki.csa.008870 |
| [8] |
郑伟, 赵金芳, 张奕婧, 等. 基于感受野扩增和注意力机制的U-Net脑肿瘤MR图像分割. 河北大学学报(自然科学版), 2022, 42(5): 542-551. |
| [9] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [10] |
Alom MZ, Hasan M, Yakopcic C, et al. Recurrent residual convolutional neural network based on UNet (R2UNet) for medical image segmentation. arXiv:1802.06955, 2018.
|
| [11] |
Badrinarayanan V, Kendall A, Cipolla R. SegNet: A deep convolutional encoder-decoder architecture for image segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(12): 2481-2495. DOI:10.1109/TPAMI.2016.2644615 |
| [12] |
Zhou ZW, Siddiquee MMR, Tajbakhsh N, et al. UNet++: Redesigning skip connections to exploit multiscale features in image segmentation. IEEE Transactions on Medical Imaging, 2020, 39(6): 1856-1867. DOI:10.1109/TMI.2019.2959609 |
| [13] |
Huang HM, Lin LF, Tong RF, et al. UNet3+: A full-scale connected UNet for medical image segmentation. Proceedings of the 2020 IEEE International Conference on Acoustics, Speech and Signal Processing (ICASSP). Barcelona: IEEE, 2020. 1055–1059.
|
| [14] |
Liu ST, Huang D, Wang YH. Receptive field block net for accurate and fast object detection. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 404–419.
|
| [15] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 3–19.
|
| [16] |
胡丹丹, 张忠婷, 牛国臣. 融合CBAM注意力机制与可变形卷积的车道线检测. 北京航空航天大学学报, 1–14. (2022-12-15)[2023-05-08].
|
| [17] |
Shi LY, Li XY, Hua WM, et al. EBHI-Seg: A novel Enteroscope biopsy histopathological hematoxylin and eosin image dataset for image segmentation tasks. Frontiers in Medicine, 2023, 10: 1114673. DOI:10.3389/fmed.2023.1114673 |
| [18] |
Zhao P, Li C, Rahaman MM, et al. EMDS-6: Environmental microorganism image dataset sixth version for image denoising, segmentation, feature extraction, classification, and detection method evaluation. Frontiers in Microbiology, 2022, 13: 829027. DOI:10.3389/fmicb.2022.829027 |
 2024, Vol. 33
2024, Vol. 33



