骨干神经网络特征提取能力是计算机视觉领域的基础. 卷积神经网络自AlexNet[1]取得巨大的进展以来, 卷积神经网络在骨干网络这一领域占据主导地位已经10多年的时间, 其强大而复杂的映射能力, 已经被证实在处理各种视觉任务上是有用模型. 卷积神经网络在医学图像分析领域成为主流, 特别是编码器-解码器结构网络, 在过去的几年里受到了广泛的关注. U-Net网络凭借着跳跃连接方式将编码器提取的低级特征连接到解码器, 以此帮助网络模型获得在下采样过程中模型丢失的上下文信息, 展现了十分优越的性能. 许多U-Net变体也随之产生, 它们通过改进跳跃连接方式或者在跳跃连接之前进行增强编码器特征等方案成功的改进了基线的U-Net.
尽管U-Net及其变体在各种医学图像分割任务上取得了良好的性能, 但是仍然存在性能缺陷. 首先, 卷积更加重视局部信息, 无法获取全局特征之间的长期依赖关系, 这是由于卷积是通过卷积核逐渐获取局部特征, 而不是一次性提取全局特征. 其次, 普通的特征融合没有建模全局多尺度上下文信息的能力, 尽管很多U-Net变体通过卷积层或者残差层来提高这个能力, 并取得了一定的效果, 但是在减小语义差距上仍然还在努力.
在本文中, 我们研究了一种新的3D医学图像分割模型IUINet. 普通的全连接层中的权重对于所有特征来说是共享的, 将全连接层的权重变为每个特征独有的, 这有助于每个特征的表达. 在视频识别任务中Shift的作用起到了显著的效果, 将Shift应用到3D医学CT图像上, 通过切片维度(H)和空间维度(W, D)上的有效位移, 可以有效地提高效益以及精确度, 并且相对于传统的卷积来说, 使用Shift可以使参数和计算量减少, 解决了3D医学CT图像需要大量硬件资源的挑战. 空间可分离主要是处理图像的空间维度, 通过将卷积核划分为两个小的卷积核可以有效地减少计算复杂度, 使得网络模型的运行速度更快. 我们将上述两个方案作为IUINet的编码器, IUINet是双流架构, 必然会带来巨大的参数量和计算量, 使用上述两种方案作为IUINet模型架构的编码器有效地降低了参数量和计算量, 并且整个模型的性能表现仍然可观.
综上所述, 在目前的研究中全连接层仍是共享参数以及模型运行所需要的计算量和参数量大等问题, 为了解决这些问题, 本文贡献如下.
(1)为了应对复杂的特征分布, 增加模型泛化性以及鲁棒性, 我们设计了动态全连接层, 该方法在特征筛选上提供了一个新视角, 从而更好地获取特征上的上下文互信息.
(2)我们设计了一个新的双流映射结构模型IUINet, 在参数量和计算量微增加的情况下, 在CT医学图像器官分割任务上取得了可观的性能.
2 相关工作在本节, 我们主要从医学图像模型主要方法、Shift操作以及动态权重这3个方面进行了相关工作的综述.
2.1 医学图像模型医学图像分析是近几年深度视觉领域的热点, 随之而来的就是一些医学图像处理模型, 其中U-Net被广泛关注. U-Net在医学图像分割中具有开创性和广泛的应用, 在各种医学图像分割任务中取得了先进的结果. 然后, 开发了许多U-Net的变体, 如Unet++、nnUnet、UNet3+和3DUnet, 它们主要基于编码器-解码器范式. 这些方法以CNN为主干网络提取图像特征, 并结合一些精细的技巧如: 跳跃连接、多尺度表示、特征交互、嵌套结构等新的网络结构或机制, 进行特征增强, 提高模型表现能力. 上述模型, 具有不错的性能, 但是在处理3D医学图像分割任务上具有大量的计算量和模型参数. 本文提出的IUINet可以有效地降低网络的计算量以及模型参数量, 并且模型仍然可以保证性能的稳定, 甚至可以超过最新方法.
2.2 Shift操作在2017年Shift操作已经是深度学习中应用的技术, 其思想是通过对输入数据进行像素级的位移操作来实现特征提取和信息交互. 在相关研究中, 出现了一些与Shift操作相关的工作.
ShiftNet[2]方法使用了Shift操作, 以提高模型的性能和计算效率. ShiftNet使用一种无参数、无flop的Shift操作来代替传统的空间卷积操作, 以降低模型的计算复杂度和提高计算效率, 并可以在分类等任务上以较少的参数取得较强的性能. 但是在移动操作的时候参数是固定的, 缺乏了灵活性.
Shift-invariant neural network[3]方法通过使用自适应多相采样, 使得卷积神经网络真正实现位移不变性.
Shift操作在自然图像上的应用已经不是很新鲜的事情了, 但是在医学CT影像上仍然比较新颖. 文献[4]中提出的Shift与VIT的结合用来代替注意力机制的方法, 并表示注意力机制可能不是VIT成功的重要因素. 他们在论文中使用的是一种部分移位操作, 遵循的是TSM中提出的方法[5]. 在通道维度, 只有一部分的通道进行移位操作, 其他的通道保持不变. 上述方法中, Shift操作中的参数是固定的, 无法适应数据集的变化或者处理一些复杂的数据分布. 我们的方法遵循的是文献[6]中提到的可学习的3DShift操作, 在这个方法中操作系数是可以学习的, 可以有效地适应数据集的变化或者处理一些复杂的数据分布从而获得更好的性能.
2.3 动态权重当前深度学习两大领域计算机视觉领域和NLP领域中都有在使用动态卷积核. 在计算机领域中, 通过线性层直接生成卷积核, 但是卷积核具有大量的参数并且在硬件上线性层的效率很低. 在NLP领域, 一些作品也是通过线性层直接生成卷积核的. 在DyNet[7]中解决了上述所描述的问题, 通过预测线性组合固定卷积核的系数, 降低了卷积核之间的相关性解决了这个问题, 并且CNN在硬件上的速度也得到了提升, 这个技术已经在华为得到发展. 尽管动态卷积在减少冗余计算量上表现卓越的乘积, 但是卷积网络仍然是依赖于局部特征的提取, 总会丢失一定的全局特征信息. 在本文中, 我们提出的方法通过基向量注意力重新定义权重和偏置, 对于每个特征来说都有独有的权重以及偏置.
3 方法在本节中, 首先概述了我们提出的模型结构. 然后给出了所提出分层的具体细节.
我们提出的模型的整体流程如图1所示, 遵循了U-Net的设计思想, 由编码层、跳跃层、解码层、映射输出层. 该模型结构中的编码器分为两个骨干网络作为特征提取. 其主要新颖之处: 其一, 在于骨干A运用了shift的思想进行分层特征提取, 骨干B运用了空间可分离的思想进行分层特征提取, 然后将两个骨干网络获取的分层特征进行融合. 其二, 特征融合、残差、输出层, 不再是传统的方法, 而是使用我们设计的动态全连接层进行操作. Conv模块和SAMC模块如图2所示.
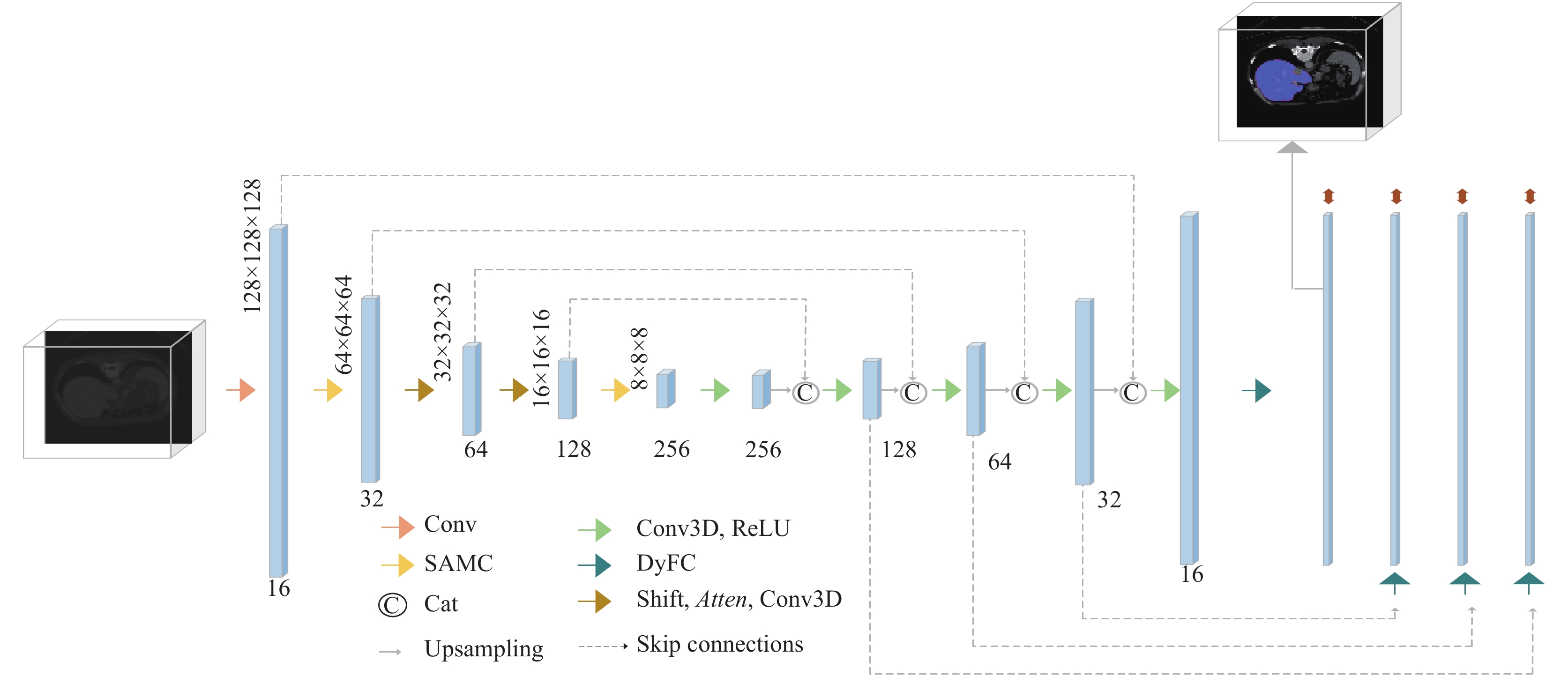
|
图 1 本文模型结构 |
具体来说, 给定输入图像I
近年来, 网络深度不断增加, 梯度爆炸, 梯度消失等问题也随之出现. 直到残差连接被广泛地使用, 卷积融合是进行残差处理的常用方法之一. 由于卷积更专注于局部特征, 对于全局特征的捕获能力仍然存在不足, DyFC可以有效地解决这个问题. 如图3所示.
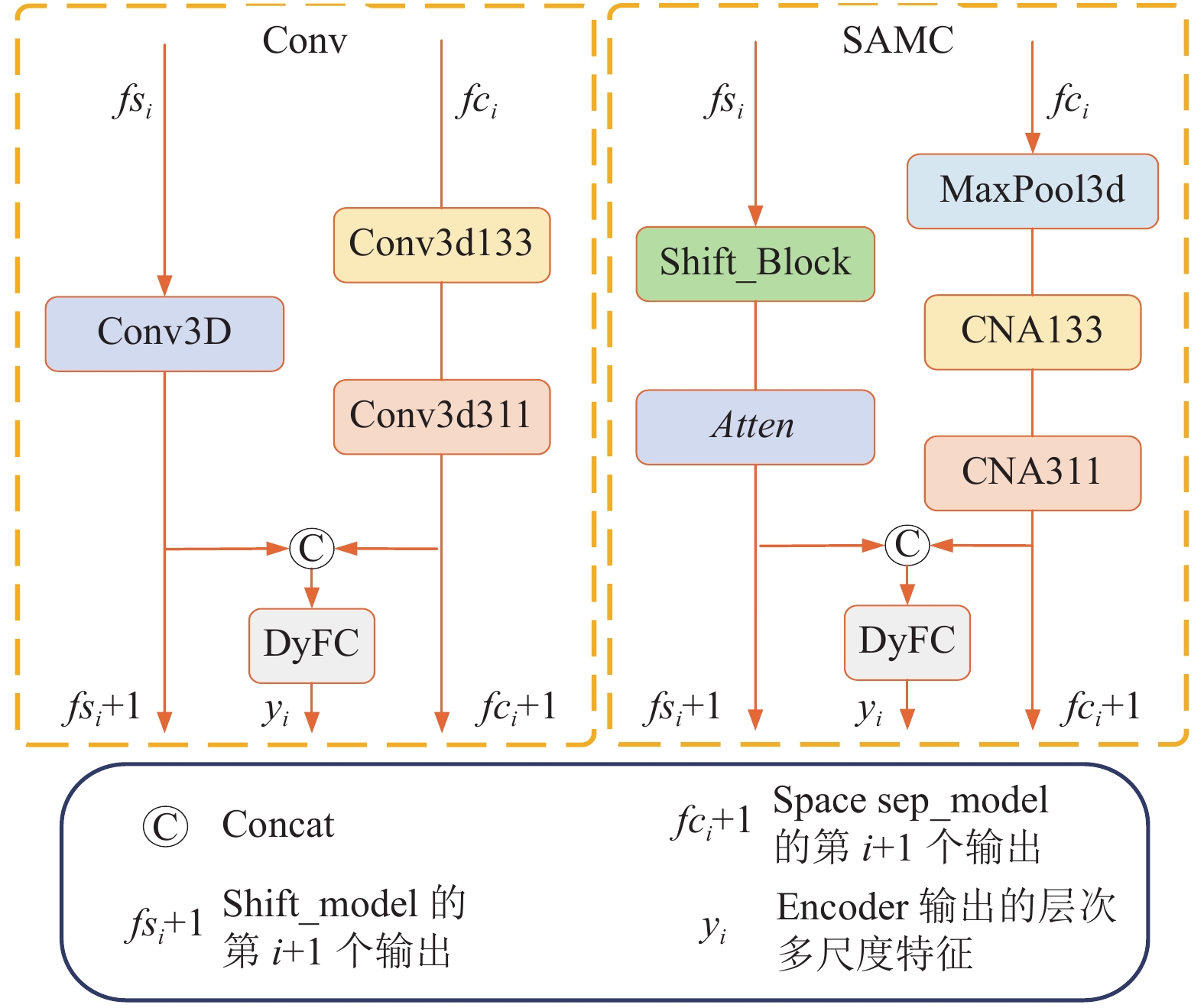
|
图 2 Conv模块和SAMC模块 |
动态全连接层作为残差连接的新方法. 相比较卷积融合操作, 在完成残差操作的同时, 还增强了融合过程中全局特征的捕获能力.
Fully connected layer: 在具有三维图像的传统全连接网络中, 网络的输入是一个四维张量X
| $ Y = X{\text{@}}{\textit{Weight}} + {\textit{bias}} $ | (1) |
其中, Weight是一个(N×M)的矩阵, bias是一个(M×1)的矩阵. 这样的全连接层特征的权重和偏置是共享的, 不具备特殊性, 泛化能力还需要继续努力.
我们提出的动态全连接层与之前的全连接工作有所不同, 即我们的模型可以学习每个特征的权重, 在进行特征融合的时候可以保留更多的上下文联系, 已达到获取更多的全局信息. 这使得在网络参数不过多增加的情况下, 可以更好地表达每个特征.
在先前的全连接工作之后, 我们考虑了注意力机制的方法, 允许我们通过注意力机制来对全连接参数进行优化. 我们动态全连接层的参数可以定义为:
| $ Y = \left( {X{\text{@}}Weigh{t_s} + bia{s_s}} \right){\text{@}}Atten $ | (2) |
其中,
| $ {\textit{Weight}} = {{\textit{Weight}}_s}{\text{@}}Atten $ | (3) |
| $ bias = bia{s_s}{\text{@}}Atten $ | (4) |
其中,
| $ Atten = attention(xlr) $ | (5) |
其中,
| $ xlr = concat(mean - std, mean + std) $ | (6) |
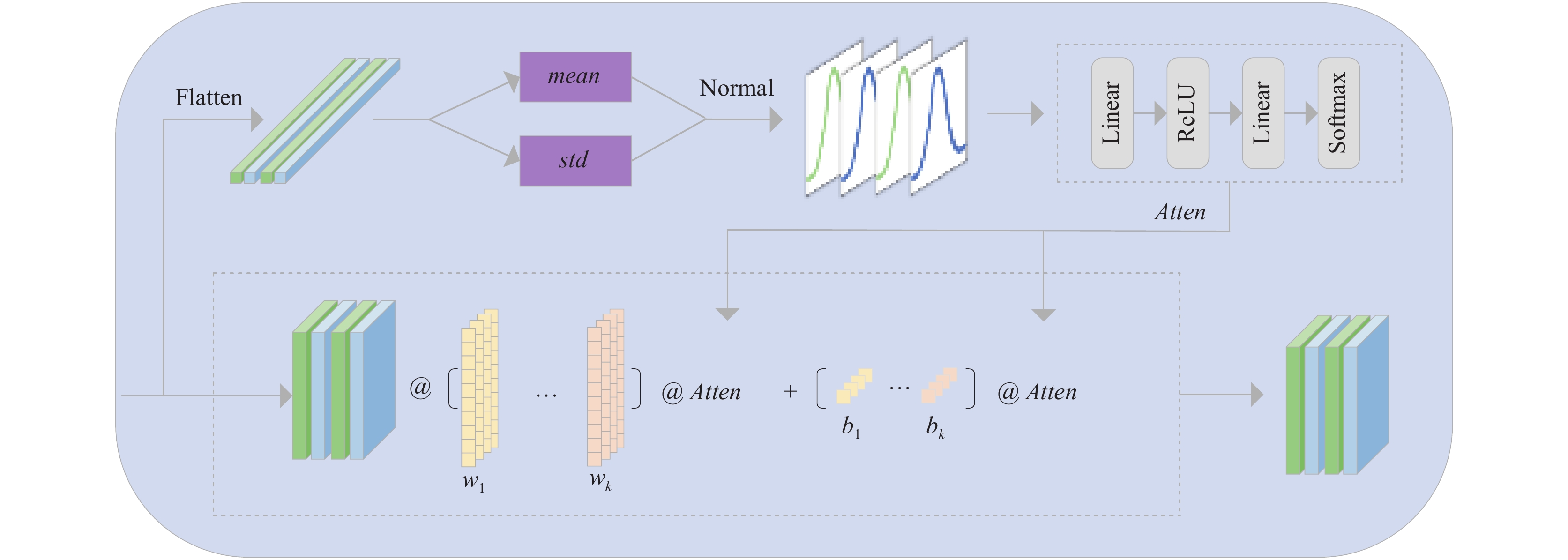
|
图 3 DyFC模块 |
我们设计的DyFC可以用在多个场景: 作为残差连接的新方式、作为输出头. 在我们设计的IUINet中在这两个方法上的改进都进行了实现. 作为残差连接时, 以往的工作是通过直接进行Add、卷积融合等方式实现的, DyFC可以作为一个全新的连接方式, 不仅可以在通道维度进行操作, 还可以指定维度进行操作. 原始的全连接层特征的权重和偏置是共享的, 不具备特殊性, 通过基向量重新设计权重和偏置可以有效地解决共享的问题, 增加权重和偏置的特殊性, 这种基向量的选择可以方便地提取、表示图像的特征, 选择合适的基向量, 可以将高维度特征空间中的冗余特征去除, 从而提高模型的泛化能力和精度.
3.2 Shift_Encoder: Model Architecture Design该模型整体架构反映了ResNet架构中的残差块的设计. 每一个Shift_Block包含Shift操作以及逐点Conv1×1操作, 用来促进跨通道的信息交换. 这里的残差块的设计是使用了我们的动态全连接层(DyFC)进行特征融合的, 这样可以更好地融合信息, 使得信息具有更多的全局特征.
我们的Shift_Block具体来说, 该块是由3个顺序堆叠的组件组成: 卷积、Shift和动态全连接层(DyFC). Shift运算在CNN中已经得到了很好的研究. 它有很多方向, 例如主动位移[8]、稀疏位移[9]和部分位移操作[5]等. 在这项研究中, 我们遵循的是可学习的3D位移操作[6], 如图4所示. 给定一个输入张量, 在H, W, D方向, 分别沿着通道维度进行移动. 在这里使用了传统离散位移操作的连续形式, 允许位移操作直接通过反向传播来优化位移参数. 这种可学习3D移动操作能够使整体架构以高效的方式学习一个联合的3D位移核, 该核以有效的方式聚合激活中的判别特征, 并且可以学习切片维度(H)位移原语的能力, 在联动切片(H)和空间(W, D)上下文上共同移动, 这使得网络能够以更少的总体参数有效地参考重要的切片(H)和空间(W, D)上下文跨度.
3.3 空间可分离网络: Model Architecture Design该模型架构主要是运用空间可分离方法, 将传统的卷积替换为参数量更少的两个小核卷积. 3D医学CT图像在训练时, 参数大, 计算量大是其面临的重大挑战之一, 将空间可分离卷积应用到分析3D医学CT图像上可以有效地改善这个问题. 空间可分离在参数方面, 由于使用两个更小的卷积核, 所以空间可分离卷积的参数量比传统卷积要少很多; 在计算量方面, 计算量比传统卷积也要小很多.
我们这个IUINet模型的设计采用了双流模型的思想. 在编码器阶段, 我们使用Shift网络和空间可分离网络进行特征提取, 这两个模型都在效率上有着一定的优势, 通过动态全连接层将两个网络的特征进行融合作为对方的信息补丁, 然后进入解码器阶段, 对解码器输出的特征进行映射, 这使得模型在具有精度不丢失甚至有所提升的情况下, 整体参数和计算并不会明显提升.
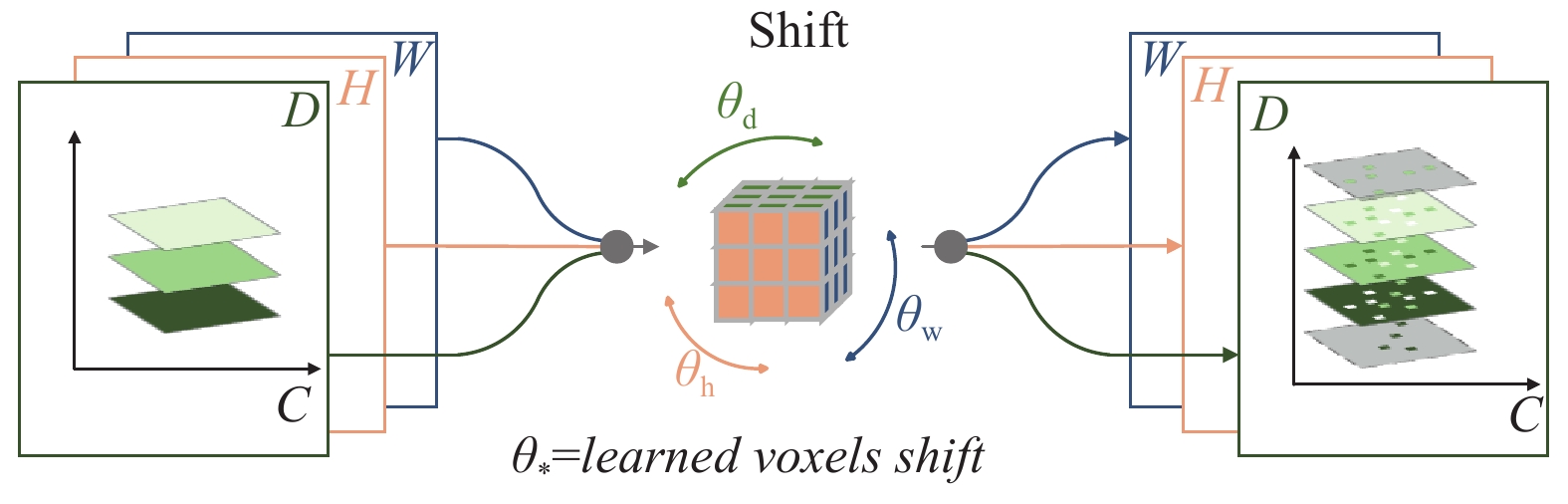
|
图 4 Shift模块 |
4 实验分析 4.1 数据集描述
本文使用的数据集来自Synapse的多器官腹部分割数据集. 该数据集是从正在进行的结直肠癌化疗试验和回顾性腹壁疝病例研究中随机选取了50例腹部CT扫描, 该过程是在机构审查委员会(IRB)的监督下进行的. 这50个扫描是在门静脉造影期间捕获的, 具有不同的体积尺寸(512×512×85–512×512×198)和视野范围(约280×280×280 mm3–500×500×650 mm3). 平面分辨率从0.54×0.54 mm2到0.98×0.98 mm2不等, 切片厚度范围为2.5–5.0 mm. 我们先使用Training-Testing进行预训练然后利用TransUNet[10]中提供的数据分割在18个训练样本上训练我们的模型, 并在12个验证案例上进行评估.
4.2 实验详情本文的方法均使用Torch实现的, 所有的实验均在一台配置为Intel(R) Core(TM) i9-10900K CPU @ 3.70 GHz 处理器、24 GB NVIDIA GeForce RTX 3090, 操作系统为Linux系统上进行的.
模型训练和超参数设置: 为了防止过拟合, 减小模型参数, 加快模型训练速度, 我们将图像和标签的大小调整为128×128×128, 批次大小为1, Epoch设置为2000, 但当模型在验证集中的损失值不降低的轮次大于等于100的时候结束训练. 使用Adam优化器, 学习率初始设置为0.001, 训练过程中最优DSC超过20次训练后不变化, 学习率就减小为0.2倍.
在本文中, 为了定量评估方法的准确性, 本文使用了Dice similarity coefficient (DSC)作为评判标准.
4.3 模型复杂度我们模型按照输入尺寸(1, 1, 128, 128, 128)得到计算量为153.8G, 参数量为49.0M, 本文使用了ptflops中的get_model_complexity_info方法在NVIDIA GeForce RTX 3090对分割方法进行了计算量和参数的计算.
4.4 对比实验为了证明我们提出的方法的有效性, 我们将在Synapse数据集上与其他14种方法进行比较得到定量结果. 表1中提供了在Synapse数据集中的3个器官上我们方法与其他14种方法的分割统计结果, 在肝脏、脾脏、胃上的平均DSC结果我们提出的方法为第1名, 3个器官的DSC结果分别为0.9546、0.9135、0.9063. 肝脏分割结果与第1名相差0.0138个精度, 但是这明显高于基线方法. 胃部的分割精度达到了0.9063的高度, 比第1名高出0.038个精度. 3个器官的平均得分为0.9248.
| 表 1 实验结果对比 |
图5是IUINet和UNETR++Learnable Weight的胃部对比实验图, 其中红色细线是真实标签, 蓝色区域是预测结果, 如黄色正方形所在区域显示, 本文的方法是有效的. 这些结果表明, 我们方法的性能比第一名的方法具有一定的优势, 尤其是分割胃部的能力.
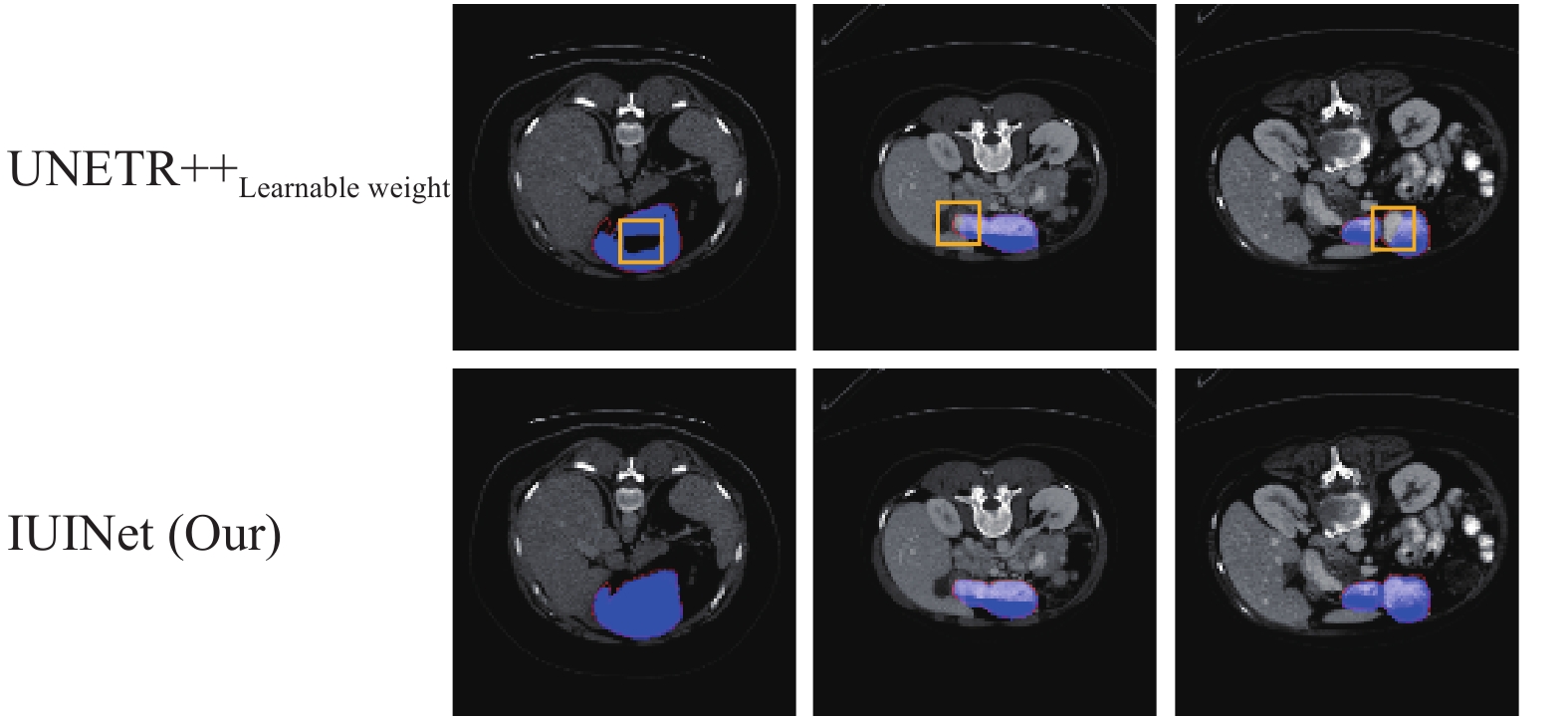
|
图 5 对比实验图 |
4.5 消融实验
本节对我们提出的DyFC以及Shift进行消融实验, 在表2中我们展示了基线效果、DyFC作为输出头和不作为输出头的对比结果以及使用Shift和不使用Shift的对比结果.
| 表 2 基线实验结果对照 |
在表3中展示了使用DyFC作为输出头和不使用DyFC作为输出头的结果. 在实验中, 通过比较两种模型的参数量(Params)、计算量(FLOPs)、DSC等指标, 评估了DyFC对模型性能的影响. 表3中的√代表使用DyFC作为输出头, ×代表不使用DyFC作为输出头. 结果表明使用DyFC作为输出头比不使用的性能要好. 而且参数量仅增加了0.37, 计算量并没有提升反而降低了0.09. 综上所示, DyFC对模型是具有积极作用的.
| 表 3 DyFC消融实验对照 |
在Shift模块消融实验中, 针对使用和不使用两种情况对模型性能进行了评估. 表4中展示了实验结果, 实验表明Shift模块不影响模型的参数以及计算量, 但是在精度上具有略微的提升. 3个器官分别提升了0.0024、0.0067和0.0118. 在各项指标上都具有更好的性能. 因此使用Shift模块可以有效地提升模型的性能.
| 表 4 Shift消融实验对照 |
5 结论与展望
我们提出了一个用于3D医学分割的双流映射结构的模型, 命名为IUINet. 在模型中我们有效地使用了DyFC和可学习的Shift模块, 通过注意力机制来应对复杂的特征分布, 增加模型的泛化性以及鲁棒性. 其中DyFC改变了传统的全连接层的权重和偏置的设置, 使它们变得更加灵活. 与现有的方法相比, 我们的IUINet在Synapse数据集上具有良好的分割性能, 同时相比传统的3D网络也减小了参数量和计算量.
| [1] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe: Curran Associates Inc., 2012. 1097–1105.
|
| [2] |
Wu BC, Wan A, Yue XY, et al. Shift: A zero flop, zero parameter alternative to spatial convolutions. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 9127–9135.
|
| [3] |
Chaman A, Dokmanić I. Truly shift-invariant convolutional neural networks. Proceedings of the 2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Nashville: IEEE, 2021. 3772–3782.
|
| [4] |
Wang GT, Zhao YC, Tang CX, et al. When shift operation meets vision transformer: An extremely simple alternative to attention mechanism. Proceedings of the 36th AAAI Conference on Artificial Intelligence. Palo Alto: AAAI Press, 2022.
|
| [5] |
Lin J, Gan C, Han, S. TSM: Temporal shift module for efficient video understanding. Proceedings of the 2019 IEEE/CVF International Conference on Computer Vision. Seoul: IEEE, 2019. 7082–7092.
|
| [6] |
Fan LX, Buch S, Wang GZ, et al. RubiksNet: Learnable 3D-shift for efficient video action recognition. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 505–521.
|
| [7] |
Zhang YK, Zhang J, Wang Q, et al. DyNet: Dynamic convolution for accelerating convolutional neural networks. arXiv:2004.10694, 2020.
|
| [8] |
Jeon Y, Kim J. Constructing fast network through deconstruction of convolution. Proceedings of the 32nd International Conference on Neural Information Processing Systems. Montréal: Curran Associates Inc., 2018. 5955–5965.
|
| [9] |
Chen WJ, Xie D, Zhang Y, et al. All you need is a few shifts: Designing efficient convolutional neural networks for image classification. Proceedings of the 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019. 7234–7243.
|
| [10] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [11] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [12] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [13] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [14] |
Fu SH, Lu YY, Wang Y, et al. Domain adaptive relational reasoning for 3D multi-organ segmentation. Proceedings of the 23rd International Conference on Medical Image Computing and Computer-assisted Intervention. Lima: Springer, 2020. 656–666.
|
| [15] |
Huang XH, Deng ZF, Li DD, et al. MISSFormer: An effective medical image segmentation transformer. arXiv:2109.07162, 2021.
|
| [16] |
Cao H, Wang YY, Chen J, et al. Swin-Unet: Unet-like pure transformer for medical image segmentation. Proceeding of the 2022 European Conference on Computer Vision. Tel Aviv: Springer, 2022. 205–218.
|
| [17] |
Yao C, Hu MH, Li QL, et al. TransClaw U-Net: Claw U-Net with Transformers for medical image segmentation. Proceedings of the 5th International Conference on Information Communication and Signal Processing (ICICSP). Shenzhen: IEEE, 2022. 280–284.
|
| [18] |
Xu GP, Wu XR, Zhang X, et al. LeViT-UNet: Make faster encoders with transformer for medical image segmentation. arXiv:2107.08623, 2021.
|
| [19] |
Li YJ, Cai WT, Gao Y, et al. More than encoder: Introducing transformer decoder to upsample. Proceedings of the 2022 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). Las Vegas: IEEE, 2022. 1597–1602.
|
| [20] |
Zhou HY, Guo JS, Zhang YH, et al. nnFormer: Interleaved transformer for volumetric segmentation. arXiv:2109.03201, 2021.
|
| [21] |
Kunhimon S, Shaker A, Naseer M, et al. Learnable weight initialization for volumetric medical image segmentation. arXiv:2306.09320, 2023.
|
 2024, Vol. 33
2024, Vol. 33


