近些年, 腹部各类器官病变(肝肿瘤、结肠肿瘤、肾结石、胰腺炎等)一直在发生, 每年的病死率也是在增加. 病死率居高不下的原因就是患者病变早期医生难以通过外在表现进行检查判断, 病变明显时已经达到中晚期, 错过最佳治疗时间. 腹部器官众多且脆弱, 形态结构较为复杂, 各类器官发病率较高. 如果在发病早期及时发现就能得到有效的治疗, 就能极大减少病死率; 针对某些轻微病症甚至可以达到根治的效果. 腹部器官数量多, 疾病往往发生在体内, 这就需要进行手术穿刺, 对医生的操作技术要求高且需要患者花费更多时间和精力等待.如果检查每个器官是否发生病变, 这十分的耗时耗力且有害患者健康. 目前, 常用的诊治辅助手段就是计算机断层扫描.
在治疗疾病之前, 需要专业医师参考医学影像结果作出判断并标注病变部位, 然后制定手术方案. 腹部器官结构复杂且重叠, 标注和识别过程是耗时耗力的. 对于解决该问题, 可以使用医学图像分割技术. 医学图像分割[1]在临床诊断和疾病治疗中起着至关重要的作用, 是计算机视觉研究的重要课题. 全自动分割系统在医学图像分析领域得到了广泛的研究. 然而, 由于复杂的组织关系和结构, 目标组织与相邻器官之间的对比度低、边界模糊[2], 现有技术在分割精度上仍然存在一些限制. 例如, 许多方法未能改善器官边界的轮廓分割效果. 原因是组织间对比度差, 器官间灰度强度不均匀, 以及数据集中目标器官和背景不平衡.
目前, 有些方法试图解决分割任务中的边界模糊问题, 提高分割腹部器官病灶的准确位置. 很多专家研发新的模型[3], 设计新的预处理和后处理方法, 但是利用好形状先验信息[4]和网络分割结果也很重要, 将网络改进并再次利用分割结果, 通过闭环结构促进训练, 可以更好地提高分割精度.
1.2 相关工作已有方法包括基于形状边界和基于Transformer的分割方法.
医学图像分割任务[5], 其中使用形状轮廓完成分割的方法值得研究. Oktay等人基于CNN提出Attention U-Net[6], 设计包含空间和通道注意力的注意门(AG), 学习跳层连接中不同形状的目标区域. Meng等人基于GNN设计用于提取并融合区域和边界特征的注意力模块(AEM)[7], 在息肉分割任务上效果较好. Lin等人基于CNN、ViT和边界检测算子构建CTO[8], 它使用边界掩码作为显式监督, 指导解码学习过程. Liu等人提出了一种感知形状和边界的多分支模型[9], 利用一个V型模型来进行像素分割图(PSM)预测和有符号距离图(SDM)回归. 通过SDM和特征融合模块(FFM), 进一步提取边界增强特征, 并在BraTS2019取得较好的结果.
与以往形状特征提取和融合的方法不同: DBLNet的BSSN模块有效利用轮廓先验; 设计SFF模块处理跳层连接, 通过注意力机制逐渐锁定待分割目标区域, 使用GCL提取边界特征, 再通过残差结构融合区域和边界形状等特征.
Vision Transformer: 将Transformer[10]用于医学图像分割任务[11], 弥补CNN学习全局上下文信息和长程空间依赖性等方面的缺失信息. ViT模块可用于编码层、瓶颈层或者解码层[12]等, 主要为混合架构或者单一Transformer架构. Chen等人首次将Transformer和CNN串联构建出强大编码器的TransUNet[13], 在BTCV数据集效果不错. Hatamizadeh等人为改善卷积的局限性, 将3D图像分割任务转变为序列的预测问题并设计UNETR[14]. 该网络的编码器由多层Transformer构成, 提取不同尺度的特征图并通过跳层连接输出到解码器, 该网络在BTCV数据集效果显著. Hatamizadeh等人[15]设计SwinUNETR[16], 它使用了Swin Transformer的跨局部和全局特征交互能力以及UNet的上采样和下采样结构, 语义和空间感知能力更强. 该网络在BTCV数据集测试排行榜较优秀.
DBLNet与以前方法的区别: DBLNet使用并联形式的双分支闭环结构, 其中CEE分支使用结合卷积优势的CvT和GCL构成层级编码器, 主要学习轮廓细节和形状等特征; 并针对双分支结构的分割结果首次设计闭环结构, 实现分支间信息流动.
1.3 本文贡献我们提出了一种基于CvT和CNN构建的双分支结构网络, 首次将CvT[17–19]和GCL由2D形式改造3D形式用于腹部器官分割. 创新点如下: (1)提出一个形状特征融合模块SFF并构建BSSN分支, SFF采用3D GCL提取边界特征, 并使用注意力提取区域特征, 再通过残差结构将两种特征融合. (2)提出利用3D CvT构建多层级的CEE分支网络, 克服了ViT的缺点, 可以充分提取局部和全局特征中的轮廓形状特征. (3)提出基于双分支的DBLNet模型, 设计闭环结构充分利用自身分割结果和形状先验信息. 相互促进对方网络的优化收敛, 对最终双分支分割结果采用加权融合方式获得最精确的分割结果.
2 双分支编码结构的闭环分割算法 2.1 DBLNet算法本算法网络结构如图1所示, 首先需要利用分割掩码提取到形状先验信息, 并将其附加到ROI图像中.两者分别输入CEE和BSSN分支模块, 经过训练并输出分割结果, 分别使用Canny和DPA算法提取形状轮廓和关键轮廓点. 将两者分别输入对方网络, 以此实现间接训练自身网络并帮助直接训练对方网络. 双分支模型拟合后或达到设定训练次数, 即可停止训练, 将两者的分割结果进行加权融合获得最优分割结果.
设计如算法1的算法步骤.
算法1. 双分支编码的闭环分割算法
1) 输入原图和掩码, 获取ROI图像.
2) 根据掩码获取目标器官坐标, 根据位置坐标裁剪提取ROI图像
3) 根据掩码图像
4) while t < maxIteration do:
5) DPA: 使用DPA关键点检测算法从分割掩码
6) 提供关键轮廓点图像
7) Canny: CEE输出分割掩码
8) 提供融合后的ROI和分割轮廓图像
9) 双分支模型通过闭环结构促进对方训练, 通过复合损失函数(Dice+CE Loss)拟合模型, 直到收敛.
10)通过计算合适的比例系数结合BSSN和CEE的分割结果, 生成最终预测分割结果.
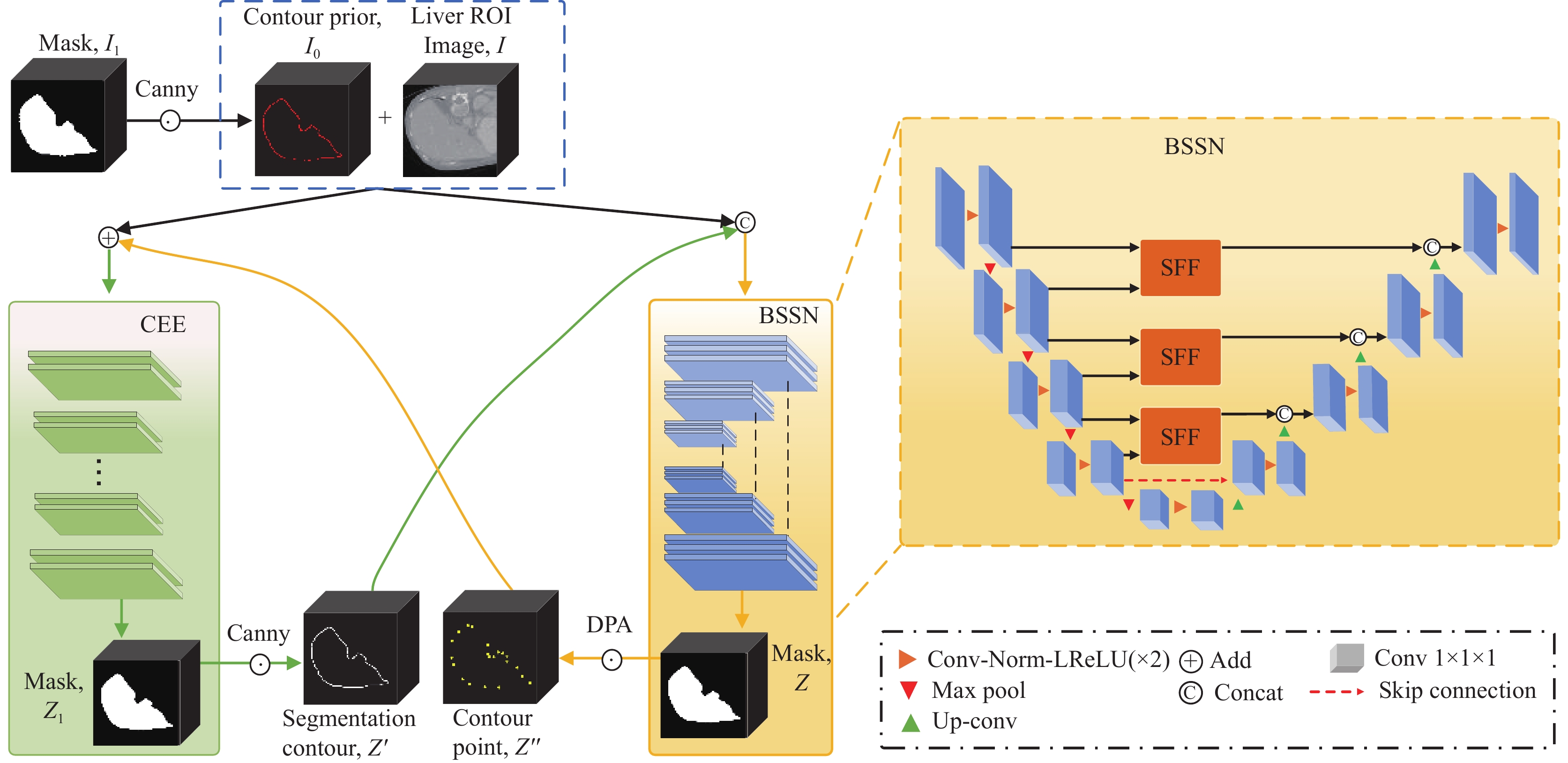
|
图 1 DBLNet网络结构图 |
2.2 提取形状先验和ROI图像
根据掩码坐标位置裁剪输入图像, 裁剪掉部分背景区域, 保留掩码中目标区域. 对应到输入图像坐标, 裁剪后就能获取到224×224×32的ROI图像
获取轮廓先验
| Gx=I2×Hx,Gy=I2×Hy,Gz=I2×HzG=√G2x+G2y+G2z,θ=arctan(Gz√G2x+G2y) | (1) |
其中,
轮廓提取编码模块(CEE module): 由3D CvT、3D GCL和反卷积构成, 结构如图2所示. CvT Encoder有4层, 注重提取图像轮廓特征信息. 算法描述: ROI图像
边界形状分割网络(BSSN): 网络各层结构如图1右侧所示, 在UNet基础上设计SFF模块提取特征图中边界形状和区域特征, 使分割掩码更精确. 算法描述: 轮廓先验
形状特征融合模块(SFF): SFF如图3所示, 主要由AGs[7]和3D GCL组成, 用于提取内部区域和边界形状等特征并融合输出. 具体实现: 首先将相邻编码层尺度为(N, C, H, W, D)的特征图

|
图 2 CEE分支网络结构 |
| {S3=Attention(S3,S4)S′4=Upsample(S4,scale=2)S′3=CovBlock(concatenate(S3,S′4)) | (2) |
提取大致区域特征, 再将提取的区域特征图
| αt=σ(C1×1×1(S′3||S′4)) | (3) |
其中, ||表示特征图的连接. 给定注意力图
| S″3=(S″4×wt)(i,j,k)=((S″4(i,j,k)×αt(i,j,k))+S″4(i,j,k))Twt | (4) |

|
图 3 SFF模块的结构流程图 |
最终通过残差结构将区域特征融合边界轮廓信息后输出到对应解码层.
SFF各阶段可视化热力图, 如图4所示: 对于某种医学图像数据(以BTCV中的Liver为例), 图4(a) AGs热力图: 特征图输入AGs中, 重点关注Liver的区域. 图4(b) GCL热力图: 从AGs获取得到区域经过3D GCL处理后重点关注边界特征. 图4(c) SFF热力图: AGs和GCL提取的特征通过残差结构实现特征融合.

|
图 4 各阶段热力图 |
2.5 输出结果
DBLNet考虑到各自模块具有的分割特点和优势, 最终输出分割掩码采用两种模型输出结果的加权融合. 计算式(5)如下:
| ZDBLNet=λZ+(1−λ)Z1 | (5) |
其中,
为了验证DBLNet的有效性, 我们使用BTCV (CT)和医院临床数据集进行实验.
3.1 实验环境硬件配置: Intel(R) Core(TM) i9-10900K CPU @ 3.70 GHz; 128 GB 内存; GeForce RTX 3090 24 GB 显卡. 软件环境: Ubuntu 18.04 LTS 操作系统; Python 3.8 编程语言; PyTorch 1.7 深度学习框架. 算法训练epoch数量为2000, batch_size为1, 初始学习率为0.01, 动量为0.99的SGD优化器. BSSN的损失函数[22]采用Dice loss 和Cross entropy loss的复合形式, CEE使用Boundary loss训练.
3.2 数据集BTCV (CT)[16]: 对50名患者进行腹部CT扫描获得, 腹部13个器官在范德比尔特大学医学中心放射科医生的监督下进行注释. 将30个CT图像划分为训练集24个和验证集6个, 测试集20个病例. 每张CT面内分辨率为0.54 mm×0.54 mm至 0.98 mm× 0.98 mm, 而切片厚度范围为2.5–5.0 mm. 主要分割Spleen、Right Kidney、Esophagus和Liver等13个器官, 器官种类较多且形态各异.
临床医院的腹部数据集: 来源于大型合作医疗机构, 通过专业设备扫描得到并由多位专家手工标注并矫正. 每张CT由120–210个切片组成, 面内分辨率为0.63 mm×0.63 mm至 0.80 mm×0.80 mm, 切片厚度范围为1.0–1.5 mm, 尺寸大小为512×512像素. 其中数据集共50个病例, 划分为训练集30个和验证集10个, 测试集 10个病例. 本次主要分割食管、肾上腺、静脉、胰腺和肝脏等一些体积小、低对比度等特点的器官, 评估本方法的适应性.
3.3 评价指标本次实验使用常见的医学图像分割指标来评估分割结果的精度, 主要有Dice score和95% Hausdorff distance (HD). Dice系数是一种相似性度量指标, 范围在[0, 1], 分割最好结果为1, 最差为0. HD95也叫豪斯多夫距离, 相比于Dice来说, 它对分割出的边界更敏感, HD95使用真实标签和预测标签表面点集之间的距离的第95百分位. 因此, 在计算HD95时, 可使得离群值中非常小的子集的影响最小化. Dice, HD以及HD95公式如下:
| {Dice(G,P)=2∑Ii=1GiPi∑Ii=1Gi+∑Ii=1PiHD(G′,P′)=max{maxg′∈G′minp′∈P′||g′−p′||, maxp′∈P′ming′∈G′||p′−g′||}HD95(G′,P′)=max{HD(G′,P′),HD95(G′,P′0.95)} | (6) |
其中,
ASD通常用于衡量分割结果与真实分割之间的距离. 它衡量的是两个表面之间的平均距离, ASD数值越小表示两者越相似. Average surface distance (ASD)公式如下:
| ASD=1NG′+NP′(NG′∑i=1Np′minj=1||G′i−P′j|| + NP′∑i=1NG′minj=1||P′i−G′j||) | (7) |
其中,
BTCV的实验结果: DBLNet对比其他模型, 可视化结果如图5所示, 结果表明模型具有强大的分割能力. 对大器官较好, 对小目标器官的边缘和形状分割比其他模型更加细致精确, 使得各个器官分割准确率有所提高. 各器官Dice得分如表1所示, DBLNet的平均Dice为0.878, 比PaNN[23]、CoTr和TransUNet的方法高出2.4%、3.4%和4.0%. 在小器官(胆囊、胰腺)的分割中, 我们的方法显著优于PaNN, Dice要高出4.2%和4.6%.
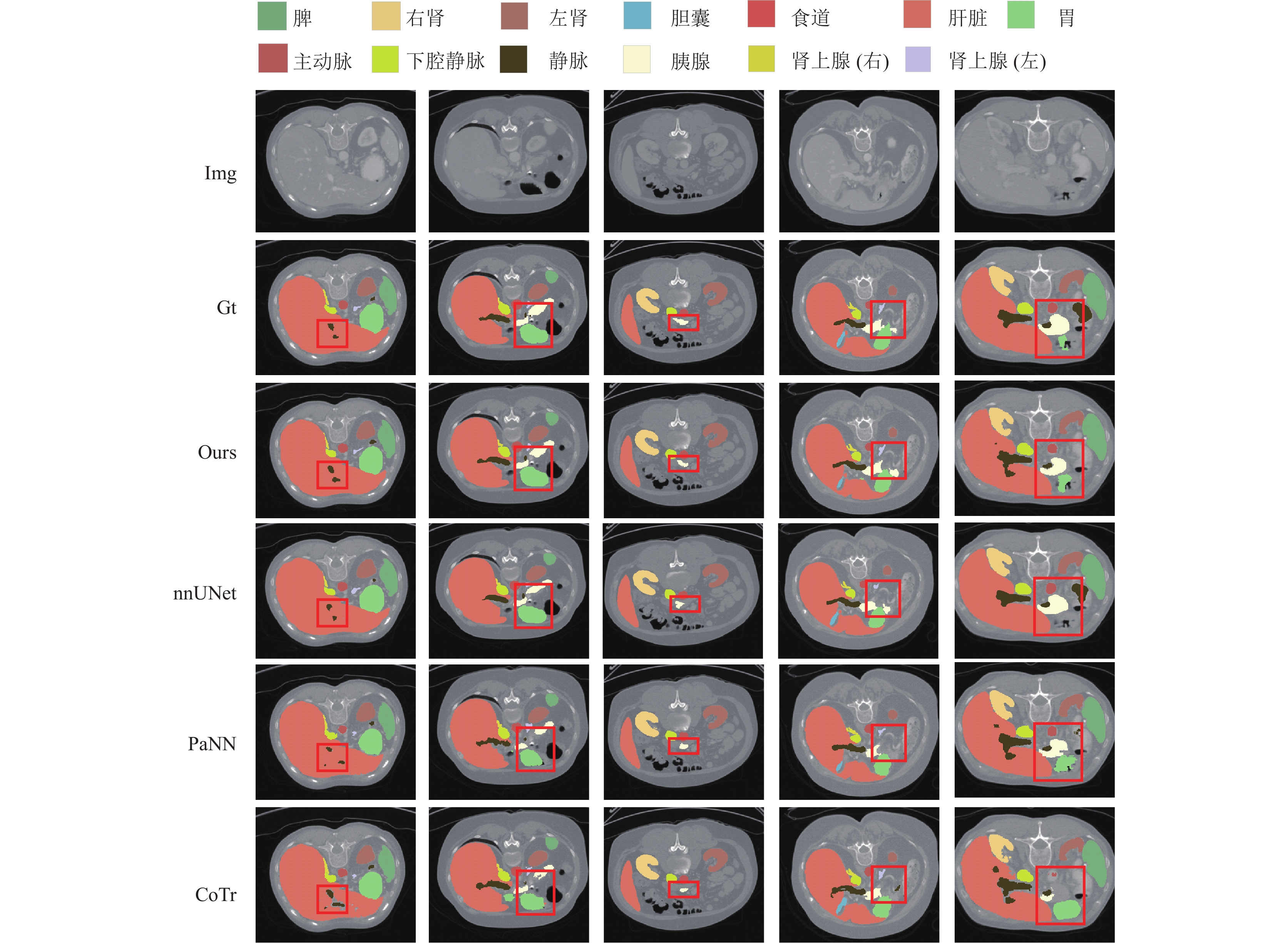
|
图 5 在BTCV数据集上可视化对比分割结果 |
| 表 1 BTCV标准挑战赛的测试集的Dice得分 |
根据实验结果测试可知, 本文提出的方法总体平均Dice为0.878, 排名第13位, ASD得分为1.329, 排名第11位, HD得分为18.276, 排名第10位. 总体来看, 超越大多数方法, 这些数据证实了本文提出的SFF和双分支闭环结构是有效的. 图5为腹部多器官的分割结果, 可以说明本文的方法占有优势. 与表1结果一致, 本文分割方法在提取各器官形状边界方面效果更好. 与现存方法相比, 本文方法甚至可以更完整的提取到小器官和其他器官的形状. 根据测试结果, 仔细观察图5可知, 除了分割结果外, 我们还提供了真实标签方便更好的比较. 随机选5个样本数据, 前4列表明: 对小目标区域的分割完整性和精度较高且形状更规则, 同时改善了CoTr具有残缺孔洞的缺点, 并且边缘形状比其他模型更好. 第5列中PaNN预测的胰腺和胃出现边缘像素残缺, CoTr和nnUNet中甚至丢失预测目标, 而DBLNet分割的目标区域相对准确且完整.
临床医院数据的实验结果: Dice和HD95计算结果如表2所示. 其中肾上腺的Dice为0.847, 胰腺的Dice为0.886, 肝脏的Dice为0.986. 除肝脏外, 其他器官Dice比Swin UNETR均提高了约12%以上, 并且HD95大幅降低, DBLNet的平均HD减小到3.208, 超过其他众多方法. 观察个体病例可视化结果如图6, 可知DBLNet具备分割细小或狭长物体的能力; 其他方法分割的食道、静脉和胰腺存在边缘轮廓多余像素粘连的问题, 肾上腺出现边角像素残缺问题. 而DBLNet分割的目标区域和边缘轮廓相对完整, 克服了上述问题.
| 表 2 腹部器官平均Dice和平均HD95得分 |

|
图 6 个体病例可视化各器官三维分割结果和Dice |
3.5 消融实验
为研究不同因素对模型的影响, 我们使用BTCV数据集进行消融研究. 具体研究分支CEE module和BSSN中SFF module对最终分割结果的影响. 我们考虑以下3种情况, 研发并试验以下模型: BL: 基线UNet+批规范化. DBLNet*: UNet+CEE. DBLNet: UNet+CEE+SFF.
CEE module: UNet增加CEE分支改造为并联的双分支结构, 并将CEE的分割轮廓输入BSSN模块. 与BL比较, 结果如表3所示, 多器官分割准确率提高, 证明提取的轮廓特征发挥了作用. 多器官平均Dice比BL提高0.403, 平均HD减少了4.5556.
BSSN module: BSSN中增加SFF模块处理相邻编码层的跳层连接, 提取掩码的轮廓点输入CEE模块. 分割结果如表3所示, SFF模块也提高了多器官的Dice, 使得模型分割精度平均Dice得分比BL提高0.753, 平均HD减少4.9834, 故BSSN中的SFF模块是有效的, 具有重要的作用.
| 表 3 对CEE和BSSN模块进行的消融实验 |
实验结果表明, DBLNet在分割精度方面优于单分支. CEE和SFF进一步提高了模型分割的精度. DBLNet比DBLNet*的Dice提高了0.35, ASD和HD分别降低0.1093和0.4278.
4 结论与展望本文介绍了一种基于CvT和CNN的双分支编码结构的闭环网络, 命名为DBLNet. 我们在BTCV数据集进行实验, 结果表明该方法对多器分割官的有效性; 并在临床医院数据进行应用, 发挥了模型的应用价值. 总之, DBLNet为提取目标轮廓特征以及区域特征而设计并联的双分支结构, 并为实现信息的流动而首次设计闭环结构. 虽然本算法精度不是最高, 但有效解决了边界轮廓模糊不清问题, 下一步需要进行的研究工作是: (1)精简优化模型, 减小计算量; (2)进一步提高具有复杂结构的器官的预测精度.
| [1] |
黄晓鸣, 何富运, 唐晓虎, 等. U-Net及其变体在医学图像分割中的应用研究综述. 中国生物医学工程学报, 2022, 41(5): 567-576. DOI:10.3969/j.issn.0258-8021.2022.05.006 |
| [2] |
He AL, Wang K, Li T, et al. H2Former: An efficient hierarchical hybrid Transformer for medical image segmentation. IEEE Transactions on Medical Imaging, 2023, 42(9): 2763-2775. DOI:10.1109/TMI.2023.3264513 |
| [3] |
Wu HP, Xiao B, Codella N, et al. CvT: Introducing convolutions to vision Transformers. Proceedings of 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 22–31.
|
| [4] |
Liu TR, Huang JJ, Dai TH, et al. Gated multi-layer convolutional feature extraction network for robust pedestrian detection. Proceedings of the 2020 IEEE International Conference on Acoustics, Speech and Signal Processing. Barcelona: IEEE, 2020. 3867–3871.
|
| [5] |
徐光宪, 冯春, 马飞. 基于UNet的医学图像分割综述. 计算机科学与探索, 2023, 17(8): 1776-1792. DOI:10.3778/j.issn.1673-9418.2301044 |
| [6] |
Oktay O, Schlemper J, Folgoc LL, et al. Attention U-Net: Learning where to look for the pancreas. arXiv:1804.03999, 2018.
|
| [7] |
Meng YD, Zhang HR, Zhao YT, et al. Graph-based region and boundary aggregation for biomedical image segmentation. IEEE Transactions on Medical Imaging, 2022, 41(3): 690-701. DOI:10.1109/TMI.2021.3123567 |
| [8] |
Lin Y, Zhang D, Fang X, et al. Rethinking boundary detection in deep learning models for medical image segmentation. Proceedings of the 28th International Conference on Information Processing in Medical Imaging. San Carlos de Bariloche: Springer, 2023. 730–742.
|
| [9] |
Liu XW, Hu YK, Chen JG, et al. Shape and boundary-aware multi-branch model for semi-supervised medical image segmentation. Computers in Biology and Medicine, 2022, 143: 105252. DOI:10.1016/j.compbiomed.2022.105252 |
| [10] |
Raffel C, Shazeer N, Roberts A, et al. Exploring the limits of transfer learning with a unified text-to-text Transformer. The Journal of Machine Learning Research, 2020, 21(1): 140. |
| [11] |
Xu GP, Wu XR, Zhang X, et al. LeViT-UNet: Make faster encoders with transformer for medical image segmentation. arXiv:2107.08623, 2021.
|
| [12] |
Zhang JP, Xie YT, Wang Y, et al. Inter-slice context residual learning for 3D medical image segmentation. IEEE Transactions on Medical Imaging, 2021, 40(2): 661-672. DOI:10.1109/TMI.2020.3034995 |
| [13] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [14] |
Hatamizadeh A, Tang YC, Nath V, et al. UNETR: Transformers for 3D medical image segmentation. Proceedings of the 2022 IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2022. 1748–1758.
|
| [15] |
Hatamizadeh A, Nath V, Tang YC, et al. Swin UNETR: Swin transformers for semantic segmentation of brain tumors in MRI images. Proceedings of the 7th International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Philadelphia: Springer, 2021. 272–284.
|
| [16] |
Larsson M, Zhang YH, Kahl F, et al. Robust abdominal organ segmentation using regional convolutional neural networks. Applied Soft Computing, 2018, 70: 465-471. DOI:10.1016/j.asoc.2018.05.038 |
| [17] |
Liu Z, Lin YT, Cao Y, et al. Swin Transformer: Hierarchical vision transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 9992–10002.
|
| [18] |
Yuan L, Chen YP, Wang T, et al. Tokens-to-token ViT: Training vision transformers from scratch on ImageNet. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021. 538–547.
|
| [19] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 6000–6010.
|
| [20] |
李祥健, 朱家明, 徐婷宜. 基于改进Canny算子的双水平集医学图像分割. 无线电通信技术, 2021, 47(2): 226-231. DOI:10.3969/j.issn.1003-3114.2021.02.015 |
| [21] |
Xie Z, Wang HM, Wu L. The improved Douglas-Peucker algorithm based on the contour character. Proceedings of the 19th International Conference on Geoinformatics. Shanghai: IEEE, 2011. 1–5.
|
| [22] |
陈英, 张伟, 林洪平, 等. 医学图像分割算法的损失函数综述. 生物医学工程学杂志, 2023, 40(2): 392-400. DOI:10.7507/1001-5515.202206038 |
| [23] |
Zhou YY, Li Z, Bai S, et al. Prior-aware neural network for partially-supervised multi-organ segmentation. Proceedings of the 2019 IEEE/CVF International Conference on Computer Vision. Seoul: IEEE, 2019. 10671–10680.
|
 2024, Vol. 33
2024, Vol. 33



