2. 上海浦东复旦大学张江科技研究院, 上海 200120;
3. 上海交通大学附属第一人民医院, 上海 200080;
4. 上海市眼病防治中心, 上海 200041;
5. 上海市眼科疾病精准诊疗工程技术研究中心, 上海 200080
2. Fudan Zhangjiang Institute, Shanghai 200120, China;
3. Shanghai General Hospital, Shanghai 200080, China;
4. Shanghai Eye Disease Prevention Center, Shanghai 200041, China;
5. Shanghai Engineering Research Center of Precise Diagnosis and Treatment of Eye Diseases, Shanghai 200080, China
近年来, 人工智能得到了飞速的发展, 在诸如交通、物流、医疗、金融等行业, 发挥着越来越重要的作用[1]. 在医疗行业的不同领域, 人工智能也取得了丰富的成果, 如疾病健康管理、疾病风险预测、电子病例、药物开发等领域[1]. 人工智能的研究人员研发出了诸如手术机器人、智能专家系统、计算机辅助诊断等多种产品[2].
深度学习是人工智能取得突破性发展的重要技术. 其中, 又以监督学习为落地应用中的主要学习模式, 监督学习需要使用带有标签的数据集进行训练[3]. 随着深度学习在医疗行业的进一步发展, 其对医学图像数据的需求量也以惊人的速度增长. 医院每天都会产生大量的医学图像, 但这些医学图像却很少被用于计算机辅助诊断(CAD)的应用, 也没有被公开[4]. 并且这些数量庞大的医学图像是未经过标注的, 即不带标签的原始图像. 因此, 对这些医学图像数据进行标注成为一项重要的工作, 由此也出现了众多的医学图像标注系统.
然而目前大多数的标注系统在医学图像的实际标注工作中还存在不少问题, 许多标注系统还是停留在纯手工标注的阶段, 并且在标注过程中并未很好地考虑到团队标注的需求, 系统界面与人机交互不够友好. 其中有部分医学图像标注系统内置了算法模型, 因此具有一定程度的自动化标注能力. 然而由于医学领域疾病种类繁多, 不同医学图像数据成像差异极大, 面向不同疾病、不同数据的标注任务往往存在其特殊的需求, 因此针对特定器官或者特定疾病的标注系统应运而生. 眼睛作为人体最重要的器官之一, 相关的科学研究也受到了充分的重视.
根据《中国眼健康白皮书》显示, 中国儿童青少年的近视发生率为53.6%, 大学生中的近视发生率为90%以上. 在糖尿病患者中的成年人里, 糖尿病视网膜病变的患者数量达到了3200万–4800万; 而在60–89岁人群中, 患白内障的人数达到了2亿之多. 根据《爱尔眼科中国国人眼底病变状况大数据报告》的数据表明, 在60岁以上的人群中, 每5个人中就有一个眼底异常的人, 而在70岁以上的人群中, 至少有65%的老年人正在遭受眼科疾病的影响[5]. 老年相关的眼底病变正在成为老年人群中首位致盲原因, 并且因为眼底病变致盲具有不可逆性, 所以对于眼底病变需要早发现、早治疗.
在不同的视网膜成像方式中, 眼底图像被广泛应用[6]. 通过眼底图像能够观测出许多疾病, 其中包括大部分的眼科类疾病, 如: 白内障、青光眼、老年性黄斑病变[7]. 除此之外, 还有许多诸如高血压、糖尿病的非眼科疾病也能通过眼底图像观测出[7]. 许多科研人员也在积极推进基于眼底图像的辅助诊断研究[8-12]. 然而支持这些研究持续进行的必要条件是大量有价值的眼底图像数据. 根据调查显示, 眼底疾病的特征在初期的时候表现出的特征并不明显, 加上眼底疾病的种类繁多[13], 对于眼底疾病的判断以及标注, 需要具有专业知识医生来进行. 相较于庞大的眼底图像数据需求, 具有专业知识的医生显得非常稀缺, 这就使得眼底图像的标注工作异常艰巨. 并且, 当前大多数的眼底图像数据集都只对单病种进行标注, 如糖尿病视网膜病变、青光眼. 但是就如文献[14]所认为的, 在实际的临床案例中, 一个人的眼睛可能同时被几种疾病征兆所困扰, 而一些工作往往只涉及对单病种的预测[15, 16]. 所以, 为了推进多疾病检测的相关研究, 建立一个具有多病种的数据集是非常有意义的. 然而, 现有的眼底图像标注系统并不能很高效地建立起眼底图像多病种的数据集, 如何高效地建立一个眼底图像多病种的数据集, 是一个值得研究的内容.
基于此, 本文提出了眼底图像半自动标注系统, 该系统旨在为眼底图像多病种的标注工作提升效率, 能够同时对眼底图像中的多种疾病进行预测并进行半自动标注. 针对在实际的标注过程中会遇到的问题, 本系统也实现了一些辅助功能, 比如任务分配等功能. 针对计算资源和操作逻辑等的方面, 本系统也经过一定的优化, 从各方面为眼底图像的标注工作提供了一个优秀的标注体验.
1 眼底图像标注系统现状目前针对眼底图像设计的公开标注系统较少, 其中大多都只支持手工标注. 虽然如何利用算法进行自动化标注早已是一个重要的研究[17], 但针对眼底图像的半自动标注系统还是寥寥可数, 并且这一类半自动标注系统都只支持眼底图像的单病种标注, 无法同时对多病种进行半自动标注.
ORIGA-light 是一个用于青光眼分析和研究的在线视网膜数据库, 其标注工具为 ORIGA-GT, 这款标注工具需要标注人员对眼底图像进行手工进行分割和分级[18]. iMED-Origa650 同样是一个用于青光眼自动诊断和视杯视盘分割的权威数据集[19], 该数据集的标注工作是由标注人员在标注工具上用椭圆来对视盘视杯进行标注[20], 整个标注也是手工完成的[21].
在 REFUGE 眼底青光眼挑战赛中公开的数据集, 是由7位青光眼专家标注人员独立标注, 最终再合并到一起得到的[22]. 类似这样的标注工作虽然能得到高精度的标签, 但其标注工作是由多名专家纯手工标注, 往往标注成本非常大, 并且在合并结果的这一过程也会非常费时费力[19]. 针对大量需要标注的眼底图像, 该方案并不是一个很好的选择.
以上的眼底图像标注工具, 都是由手工完成的, 这样的标注方式需要消耗大量的人力和时间. 目前也有一些研究项目是结合了深度学习的半自动的眼底图像标注系统[19, 23-25], 这一类系统在和一定程度上提升了标注效率, 但是只能针对单种眼病, 而面向多病种的眼底图像标注需求, 这些标注系统还是无法满足. 并且以上提到的眼底图像标注工具都需要在本地进行, 这也就意味着标注人员只能独立进行标注任务. 而对于需要团队协作的标注任务, 其中涉及任务分配, 任务变更等功能, 以上标注工具均无法满足.
2 系统功能介绍在进行半自动标注的过程中, 本系统先通过“多眼病检测算法”对眼底图像的各种疾病的存在性进行检测, 针对存在的疾病, 再用对应的疾病预测模型对存在的疾病进行进一步的预测, 包括疾病分级, 病灶分割. 标注人员从原先的手工标注工作, 变成了审核并修改工作.
本系统是基于 B/S 结构的网络结构模式, 核心服务在服务器端运行, 标注人员可以使用带有浏览器的终端设备对本系统进行访问. 本节将详细介绍眼底图像半自动标注系统的总体架构以及核心模块.
2.1 系统总体架构设计眼底图像半自动标注系统的架构设计图如图1所示, 总体上分为表现层、数据处理层、基础设施层. 表现层提供标注人员在该系统上进行注册登录等基本操作, 数据的可视化, 以及完成图像标注的人机交互等工作. 数据处理层主要包含对图像的信息入库、标注、获取, 对标注人员的身份验证、添加、修改、任务分配, 对算法模型的调取并录入结果等任务. 基础设施层主要包含 MySQL 数据库与 Nginx 图像服务器, Tomcat 应用服务器, Nginx 前端应用服务器等服务器软件.

|
图 1 系统总体架构设计图 |
2.2 系统功能模块划分
眼底图像半自动标注系统的功能模块图如图2所示, 主要分为用户管理、项目管理、图像管理、算法模型管理4个模块. 其中用户管理模块实现标注人员的注册与登录, 当前标注人员标注进度的展示等功能; 项目管理模块实现对项目创建与修改、结果导出以及任务分配等功能; 图像管理模块实现对于图像上传、展示、标注等功能; 算法模型管理实现算法模型的调用与模型预测结果的保存.

|
图 2 功能模块图 |
用户管理模块可以实现标注人员的注册登录, 对于未登录的访问请求或者登录过期的访问请求, 系统会进行拦截, 并返回到登录页面, 以保证标注数据只对标注人员可见.
项目管理模块包含4个子模块, 分别为项目的创建与修改子模块、项目标注结果导出子模块、项目人员任务分配与变更子模块、项目标注进度展示子模块. 管理员在创建项目时可以编辑项目名称, 项目描述, 参与项目的标注人员. 在选定项目标注人员后, 在该项目上传的数据, 都会以病例为单位(一个病例中会有多张图像)将数据平均分给参与该项目的标注人员. 标注人员在文件列表中只能看见自己所负责的病例, 其他病例默认被隐藏. 在项目中的部分图像被标注之后, 若需要对项目中的标注人员进行变更, 该子模块会自动对剩下的图像重新进行分配. 项目标注结果导出子模块可将该项目的标注结果生成为 CSV 文件, 并供标注人员下载. 标注人员可以在项目列表界面看到当前所有项目标注进度.
图像管理模块包含3个子模块, 分别为压缩包上传子模块、图像信息获取与展示子模块、标注子模块. 压缩包上传子模块支持标注人员在前端页面将打包好的压缩包进行上传, 并在后端对压缩包进行解压. 之后由图像信息录入子模块对图像进行基本信息入库, 如, 图像的所属病例、保存路径等基本信息. 图像信息展示子模块可以读取图像的信息, 并在前端对图像以及图像所属病例、疾病等相关信息进行展示. 图像标注模块可以支持标注人员对图像进行手工标注, 或者是对算法预测结果进行修改.
算法模型管理模块是该系统中的半自动标注功能能够实现的关键模块. 该模块包含4个子模块. 主要分为算法调用与结果保存子模块, 多病种检测子模块, 单病种等级预测子模块, 单病种病灶分割子模块. 算法调用与结果保存子模块将需要预测的图像相关信息发送给模型程序, 在得到模型预测的结果后, 该子模块再将结果存入数据库汇总, 而模型预测这一过程对于该子模块来说是透明的. 模型预测的过程需要其他3个子模块协作完成. 模型首先会对目标图像进行多病种进行检测, 用来判断各个疾病的存在情况, 针对存在的疾病, 将进一步调取单病种等级预测模型和单病种病灶分割模型进行预测. 最终将预测的结果返回给调用接口.
3 系统实现 3.1 关键技术考虑到系统的可维护性以及开发的高效性, 该系统采用前后端分离的开发模式. 该系统的前端框架使用的是 Vue, 方便系统的快速开发, 前端状态管理使用的是 Vuex 库, 前后端通信库使用的是 axios, 前端样式库使用的是element ui. 后端主要采用 SpringCloud 进行微服务开发, 数据库使用 MySQL, 消息队列使用 Redis, 图像服务器以及前端服务器使用 Nginx 来实现.
以下从该系统的4个模块来详细介绍该系统的实现方式.
3.2 用户管理模块实现用户管理模块主要是对为登录的访问进行拦截、实现标注人员的注册登录.
除注册页面和登录页面以外, 在未登录的情况下, 游客对系统进行访问, 会被系统拦截, 并返回登录页面. 这样可以保证医疗数据只对注册过的标注人员进行公开, 保证数据的安全性. 实现方式如下: 在后端接收到访问请求后, 检查所有对系统的请求的请求头, 并检查请求头中是否有带有 token, 如果没有 token 字段, 或者 token 字段失效, 则请求被拦截. 只有 token 字段有效的情况下, 请求才可正常进行.
用户注册功能, 会对标注人员的用户名和密码做出一定的限制, 如: 用户名不能为空、用户名不能出现特殊符号、密码长度在6–20位之间等. 这些验证操作在前端实现. 注册成功后, 标注人员的信息会被写入数据库. 用户登录功能, 在后端验证用户名和密码都正确之后, 会为该用户生成一个 token, 并传回给前端, 前端使用 Vuex 对 token 进行保存, 在发送访问请求的时候将 token 放到请求头中, 以用来保证一定时间内的用户免登录.
3.3 项目管理模块实现项目管理模块实现了项目的新建和修改功能、项目标注结果导出、项目人员的任务分配与变更、项目进度展示.
项目的新建和修改都是对 Vue 前端组件 EditForm 中的项目名、参与者、简介等字段进行编辑. 其中项目名和参与者是不可缺省的两条字段. “项目名”这一字段用来区分不同项目, 而“参与者”这一字段将用于该项目平均分配标注任务.
新建与修改项目调用的是同一个后端接口 public Boolean addOrModifyProject, 该接口通过检查当前工程 ID 是否在数据库中已存在, 来决定是修改项目 modifyProject 还是添加项目 addProject.
添加项目功能, 除了需要将管理员指定的项目名称、参与人员、简介写入到数据库, 还会自动生成项目创建时间, 并在磁盘上新建一个项目文件夹, 该文件夹用于保存之后标注人员上传的眼底图像, 并将项目创建时间与项目的文件夹的路径一同写入数据库. 其中, 参与人员字段是以逗号分割的用户名字符串来进行存储, 如 “user1, user2, user3”. 并在需要对标注人员进行遍历的时候, 可以将该字符串以逗号进行分割, 以此得到标注人员用户名的列表. 这种方式的存储可以减少数据库对用户名的遍历.
任务分配功能的触发条件有两种.
(1)首次分配: 在项目中上传压缩包并解压之后, 系统会对新上传的图片以病例文件夹为基本单位进行任务分配. 这种情况下, 需要对解压后的病例文件夹进行遍历, 并且将每一个病例内的图像对应一个用户名存入数据库. 例如: 当前项目的标注人员为 user1, user2, user3, 那么在遍历过程中就会循环依次将文件夹指定给 user1, user2, user3, 并把这个关系写入数据库. 由此任务分配完成.
(2)任务变更: 在执行modifyProject 方法, 即修改项目的过程中, 需要判断该项目的“参与者”字段是否有变化, 参与者的增加, 减少, 变更. 如果项目的参与者发生了变化, 系统会对该项目中未被完整标注的病例文件夹进行任务重新分配——完整标注意味着该病例文件夹中所有图像都被标注——已经完整标注过的病例不受影响. 具体分配过程还是对未完整标注的病例文件夹进行遍历, 并依次循环分配给新的标注人员.
标注结果导出, 在项目标注的过程中或者标注完成之后, 可以对标注结果进行导出. 在项目编辑界面点击下载按钮, 即可对项目的结果进行导出下载. 当点击下载按钮, 前端发送项目的 ID 到后端, 后端根据项目 ID 从数据库中取出该项目的标注结果, 编码成 JSON 数据并返回给前端. 前端得到 JSON 数据后将其解析为 CSV 文件, 并下载到本地. 保存着标注结果的 CSV 文件, 每一行记录一张图像的标注信息, 其中分别为病例名, 图像名, 以及各种疾病等级.
管理员和标注人员可以在系统主页看见各个项目的基本信息, 以及各个项目的标注进度, 便于实时掌握各个项目的完成情况. 标注进度展示方式有两种: 已标注的百分比, 已标注的图像数量/图像总数量.
3.4 图像管理模块实现图像管理模块是项目中的重要模块, 压缩包的上传、图像的展示以及图像的标注等任务都由该模块来完成.
为了方便图像的管理, 系统对图像文件夹的层级分为3级: 项目文件夹, 批次文件夹, 病例文件夹. 其中, 不同的项目有各自的项目文件夹. 而一个项目内可以有多个批次文件夹, 每次压缩包的解压都会形成一个新的 批次文件夹, 每个批次文件夹都包含该次上传的所有病例文件夹. 设置的批次文件夹的目的: 防止新上传的病例与之前标注的病例混在同一个文件夹中无法区分. 每个病例文件夹下包含该病例的所有图像. 项目文件夹、批次文件夹、病例文件夹在后端都以对象的形式定义和操作.
3.4.1 压缩包上传压缩包上传的前端页面使用 element ui 前端库中的 el-upload 组件, 该组件可以大大降低这一步骤的代码量, 方便修改关键参数. 在 action 参数中指定后端的 API, 如: action=" http://localhost/api/upLoad", 为了防止标注人员上传的文件大小和格式不规范, 可以在 accept 参数中指定接收格式为 zip 格式.
后端接收到压缩包之后, 需要完成解压压缩包、录入数据库、调用模型对图像进行预测这3个操作. 调用模型对图像进行预测的步骤将在第3.5节进行说明.
在解压完成之后, 系统通过对该批次文件夹进行遍历的方式, 将图像基本信息录入数据库中的 image 表中, 该表的主要字段为 project_id、part、case_name、image_name、username、labeled. 这些字段分别代表的含义依次为: 项目ID, 文件的批次, 病例文件夹名, 图像文件名, 对该病例进行标注的标注人员, 是否已经标注(默认为0, 即, 未标注)进行录入.
3.4.2 图像信息的获取与展示在对眼底图像进行标注之前, 在进入标注页面后, 前端会获取并展示一些基本信息: 该项目的文件夹列表, 当前图像路径, 以及当前图像基本标注信息.
标注页面的前端界面的左侧是树状文件夹列表, 右侧是标注工具, 中间是主要部分, 即为眼底图像的展示.
标注界面的左侧是树状文件夹列表, 为了减轻服务器的压力, 在获取文件夹列表时, 只会获取第一个批次 文件夹中的病例文件夹列表, 而每个病例文件夹中图像列表以及其他批次文件夹中的病例文件夹列表可以暂时不用获取. 在标注人员点击某个病例名的时候, 前端才会向后端发起请求, 并获得该病例下的图像文件名列表, 再在前端详细显示当前病例下有哪些图像. 这样的加载方式可以减少不必要的数据库搜索, 降低了服务器的压力, 也可以更快地获得图像列表. 为了方便标注人员对当前标注图像的位置进行定位, 左侧列表有两个筛选按钮, 分别是筛选自己需要标注的图像, 筛选还未标注过的图像. 两个筛选按钮默认同时打开, 即只显示自己需要标注但还未标注的图像.
3.4.3 图像标注模块每张眼底图像的标注状态都分为“未标注”“AI标注”“AI标注已审核”“手工标注”这4种状态. 图像标注状态会显示在右上角, “未标注”代表当前眼底图像未经过算法或者人工的标注; “AI标注”代表当前眼底图像虽然已经经过标注, 但是该标注结果是由算法预测得到且未经过审核的; “AI标注已审核”代表当前眼底图像是算法预测得到并且通过了标注人员的审核, “手工标注”代表当前眼底图像还未经过算法预测就由标注人员标注完成了. 这4种状态可以作为标注人员是否需要进一步审核的参考.
标注界面的标注工具实现了多种眼病的标注, 分别为: 糖尿病视网膜病变、青光眼、白内障、年龄相关性黄斑变性、高血压性视网膜病变、病理性近视和其他眼部疾病. 标注工具的标注类型分别为: 疾病存在性标注、疾病分级标注、病灶分割标注. “疾病存在性标注”是最基础的标注方式, 即对以上各个疾病进行“有”或者“无”的标注. “疾病分级标注”是对病变的严重程度进行分级. 其中, 糖尿病视网膜病变分为详细的4个等级, 分别为“轻度非增殖期”“中度非增殖期”“重度非增殖期”“增殖期”. “病灶分割标注”是最精细的标注方式, 标注人员通过画笔工具将疾病病灶的边缘进行勾勒. 例如, 糖尿病视网膜病变中的微动脉瘤、出血、硬性渗出物等.
在页面刷新的时候, 前端向后端发起当前图像所有疾病的标注信息的请求, 后端从数据库获取相关数据并返回给前端后, 各种眼病的存在性信息和等级信息会在右侧标注工具栏进行展示. 在对当前图像进行标注或修改的时候, 点击工具栏中的等级之后, 前端将会自动将标注的结果发送给后端, 并在数据库进行保存, 无需手动点击保存. 分割标注的工具栏在顶端, 在选择对应的病灶之后, 即可进行病灶的分割标注或者修改. 该系统还实现了快捷键操作, 标注人员通过键盘的上下键可以切换图像的上一张和下一张. 快捷键的实现不仅在一定程度上提升了标注效率, 也提升了标注的用户体验.
3.5 算法模型管理模块实现算法模型管理模块的主要功能是对目标图像进行多病种预测. 由于当前研究领域主要聚焦于眼底图像单疾病分割算法, 缺乏多疾病分割算法. 若要直接实现多疾病分割, 通常需要单独运行所有的单疾病分割算法. 当眼底图像中存在的疾病较少时, 该方法效率低下, 且产生极大的资源浪费. 因此本系统使用工程的思维来解决该算法问题. 该模块提出了一种两阶段眼病诊断框架, 第1阶段采用“眼底图像多病种检测模型”进行眼病存在性识别. 第2阶段对于存在的眼病, 进一步使用分级算法或分割算法进行等级或者病变点的预测. 通过这种方式可以减少大量无效的计算.
本系统第1, 2阶段的算法使用的均为现有研究中公开的检测算法和分割算法, 并不是本系统设计的重点, 可以根据算法表现效果来替换成其他性能更高的算法. 本系统经过改造之后具有一定的泛化能力, 可以用于交通、安防等图像标注场景, 尤其是当图像在符合两阶段分割的场景下——即缺少现成的多实例分割算法, 但是存在多目标存在性检测以及单实例分割算法的情况——会让本系统发挥更明显的优势.
经过统计, 在 OIA-ODIR 数据集中[26], 有42.86%的图像是无疾病的正常眼底图像. 而在有疾病的眼底图像中, 有80%的眼底图像只有一种疾病, 所以直接对一张图像使用多个“眼底图像单病种预测模型”是效率低下的做法. 该模块首先使用第1类模型检测出疾病的存在性, 针对存在的疾病, 再调取第2类模型中对应的预测模型进行进一步的等级或者分割预测, 得到疾病的等级, 病灶分割图像. 相较于将所有疾病对应的模型都对眼底图像进行一次预测, 两阶段眼病诊断框架的方式大大缩短了预测疾病的时间. 前者平均预测一张眼底图像需要8.5 s, 而两阶段眼病诊断框架预测一张眼底图像只需2.1 s. 由于本系统的主要性能瓶颈并不在网络或者磁盘, 而是在图像预测上, 因此提升图像预测环节的性能就是对整个系统性能的最大提升.
算法调取模块是基于 Python 的调用逻辑. 该模块具体实现方式如下, 图像预测请求是来自于后端的 Java 程序, 接下来要通过 Java 对 Python 的业务逻辑发起请求. 这里通过 Redis 的消息队列能力进行实现. 该模块需要在 Java 与 Python 程序中进行两次消息的传递, 第1次, 是 Java 向 Python 程序发送目标图像的路径, Python 程序负责预测图像. Java 程序是消息的生产者, Python 程序是消息的消费者. 第2次, 是 Python 程序向 Java 程序返回预测结果, Java 程序负责将预测结果存入数据库. Python 程序是消息的生产者, Java 程序是消息的消费者. 这两个步骤需要两个单独的消息队列频道, 否则会导致消息的混乱. Java 程序接收到预测的结果之后, 将预测结果写入数据库, 标注的 username 字段使用 algorithm 关键字, 以表示该结果是由算法预测的. 前端获得由算法预测得到的结果时, 会在页面上提示该结果是由算法预测所得, 并且未经标注人员审核. 在经过标注人员审核确认或者修改之后, 提示会修改为“已审核”.
4 系统对比与功能测试以上就是对各个模块的实现过程的介绍. 为了验证该半自动标注系统的有效性, 让两组资历相当的标注人员对100张眼底图像分别进行纯手工标注以及对AI标注的结果进行审核修改标注, 并对比两种标注方式消耗的时间. 统计结果显示, 纯手工标注一张眼底图像平均需要4 min, 而对AI标注的结果进行审核修改标注平均只需要42 s. 可以看出, 该系统在标注时间上有很大提升. 相较于ORIGA-GT等眼底图像标注系统, 本系统支持自动保存、团队标注、团队任务自动分配等功能, 这些功能都可以让标注人员更加专注于标注任务上.
接下来对系统的各个功能进行测试. 如图3所示, 该界面为注册界面, 对用户名字符串和密码长度做出了一些限制, 如果不满足限制就会注册登录失败, 并给出提示.

|
图 3 用户注册界面 |
在注册登录成功之后, 就进入了图4, 即为项目仓库界面, 在该界面可以看见各个项目的基本信息, 如项目名, 参与人员, 标注进度等信息.

|
图 4 项目仓库界面 |
点击界面中的加号, 将如图5所示, 得到一个项目编辑界面, 在该界面填写好必填信息, 即可添加新项目.
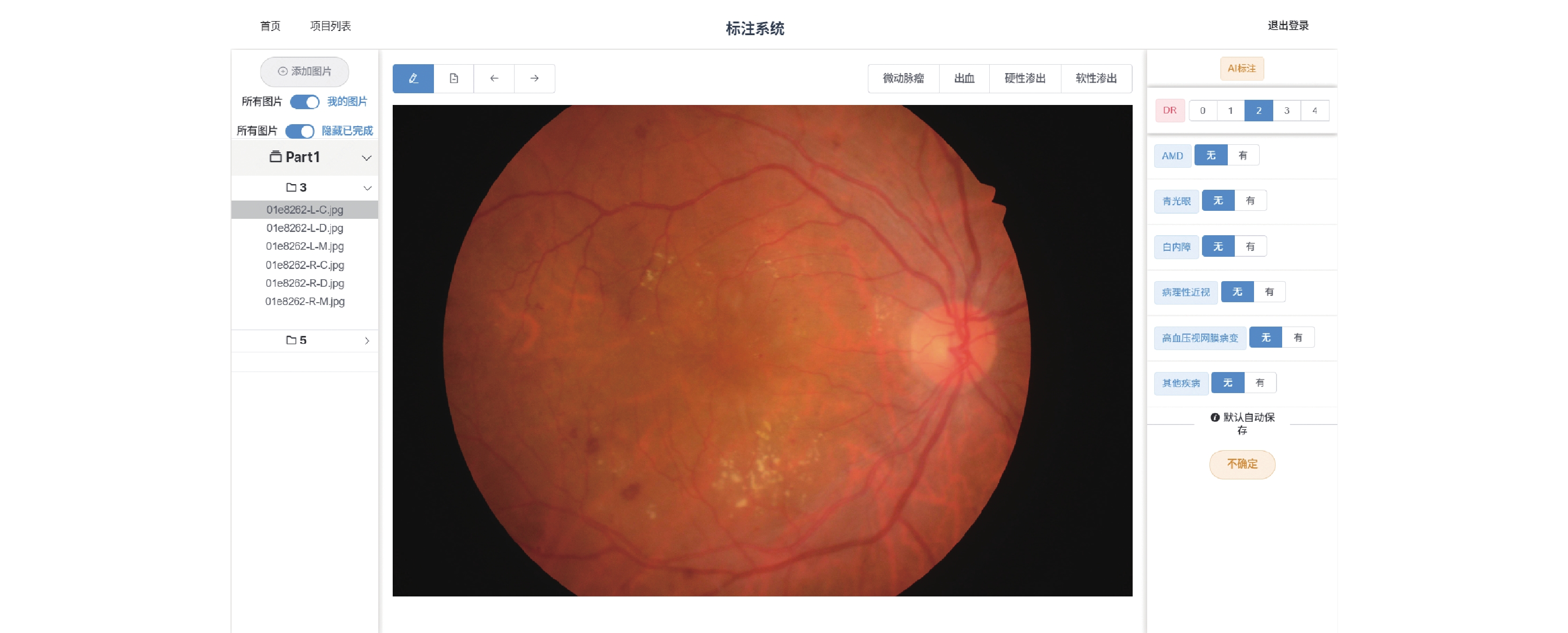
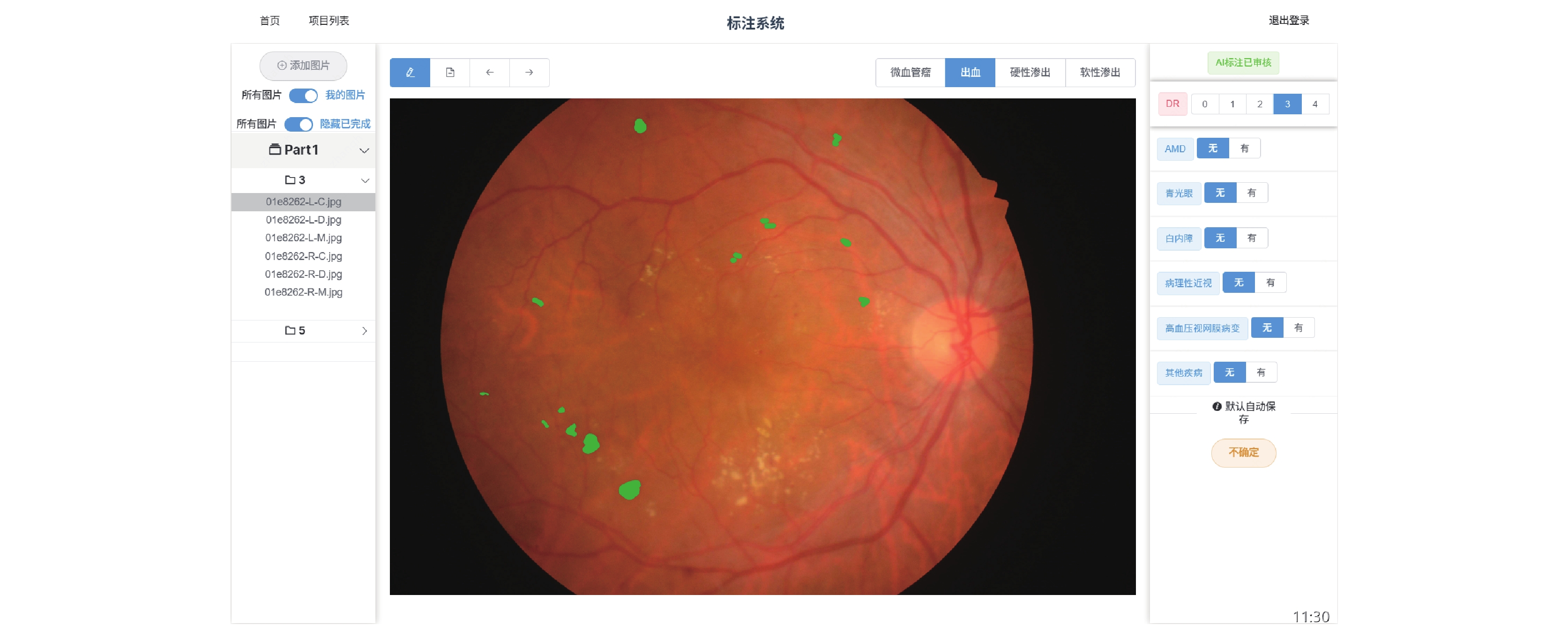
点击进入项目页面, 如图6和图7, 中间为选中的眼底图像, 左侧为图像列表, 右侧为标注工具栏. 图6为算法预测的结果, 糖尿病视网膜病变为2级. 图7由人工进行修改, 改为3级, 并在选中糖尿病视网膜病变的版块后, 便能进入分割标注, 选中“微动脉瘤”按钮就能看到微动脉瘤分割结果.
左侧图像列表的上方有个“添加图像”的按钮, 点击之后会出现如图8所示的图像上传界面, 在该处上传的图像会保存在一个新的批次文件夹中. 在“添加图像”按钮的下方, 有图像筛选按钮, 标注人员可以点击对图像进行筛选: 只显示自己任务的图像或者显示所有图像, 显示已经标注的图像或者显示所有图像, 两个筛选条件默认同时开启.

|
图 5 项目添加/编辑界面 |
|
图 6 标注系统界面(AI预测结果) |
|
图 7 标注系统界面(人工审核结果) |

|
图 8 文件上传界面 |
右侧是等级标注工具栏, 如图9所示, 该工具栏包含了6种疾病的标注. 在标注工具栏中点击数字就完成了对这张图像的等级标注, 系统会自动保存. 当图片的结果是由算法预测得到的时候, 标注工具栏的最上方会提示该图片是“AI标注”, 如图9所示. 只有当标注人员审核通过之后, 才会提示“AI标注已审核”.

|
图 9 标注工具栏 |
5 结束语
本文分析了当前眼底图像标注系统的优点和不足, 并对眼底图像标注系统进行了重新设计和实现. 该系统通过“两阶段眼病诊断框架”实现了眼底图像标注系统的多病种半自动化标注, 能够对多种眼病进行预测, 降低标注人员的标注压力, 并使得系统的运作更加合理且节省计算资源. 同时更加针对真实的标注场景进行设计, 解决了真实场景的标注过程中所遇到的标注人员更替、系统响应速度慢、操作逻辑复杂等问题. 该系统实现了图像管理、项目管理、用户管理、算法模型管理4个模块. 本系统在各个方面的改进和设计. 当然本系统还存在一些不足, 比如在面对庞大的数据量时, 标注人员依然需要对大量的图像进行审核, 这也是一大工作量, 本系统将在下一步工作中加入主动学习算法, 从而解决庞大数据量的半自动标注的审核压力问题.
| [1] |
袁波, 代华, 伍佳, 等. 人工智能在全科医学领域的应用. 中华全科医学, 2021, 19(9): 1433-1436, 1572. DOI:10.16766/j.cnki.issn.1674-4152.002079 |
| [2] |
韩冬, 李其花, 蔡巍, 等. 人工智能在医学影像中的研究与应用. 大数据, 2019, 5(1): 39-67. |
| [3] |
陆萌. 面向深度学习的医学图像标注系统的研究与实现[硕士学位论文]. 北京: 北京邮电大学, 2020.
|
| [4] |
Leopold HA, Zelek JS, Lakshminarayanan V. Deep learning for retinal analysis. In: Sejdic E, Falk TH, eds. Signal Processing and Machine Learning for Biomedical Big Data. Boca Raton: CRC Press, 2018. 329–365.
|
| [5] |
张子振. 基于深度学习的糖尿病视网膜图像的分析与研究[硕士学位论文]. 保定: 河北大学, 2020.
|
| [6] |
Sengupta S, Singh A, Leopold HA, et al. Ophthalmic diagnosis using deep learning with fundus images—A critical review. Artificial Intelligence in Medicine, 2020, 102: 101758. DOI:10.1016/j.artmed.2019.101758 |
| [7] |
曹颖. 基于半监督学习的白内障眼底图像自动标注方法研究[硕士学位论文]. 太原: 中北大学, 2019.
|
| [8] |
Lim G, Cheng Y, Hsu W, et al. Integrated optic disc and cup segmentation with deep learning. Proceedings of the 27th IEEE International Conference on Tools with Artificial Intelligence (ICTAI). Vietri sul Mare: IEEE, 2015. 162–169.
|
| [9] |
Mitra A, Banerjee PS, Roy S, et al. The region of interest localization for glaucoma analysis from retinal fundus image using deep learning. Computer Methods and Programs in Biomedicine, 2018, 165: 25-35. DOI:10.1016/j.cmpb.2018.08.003 |
| [10] |
Liu SD, Graham SL, Schulz A, et al. A deep learning-based algorithm identifies glaucomatous discs using monoscopic fundus photographs. Ophthalmology Glaucoma, 2018, 1(1): 15-22. DOI:10.1016/j.ogla.2018.04.002 |
| [11] |
Edupuganti VG, Chawla A, Kale A. Automatic optic disk and cup segmentation of fundus images using deep learning. Proceedings of the 25th IEEE International Conference on Image Processing (ICIP). Athens: IEEE, 2018. 2227–2231.
|
| [12] |
Shan J, Li L. A deep learning method for microaneurysm detection in fundus images. Proceedings of the 1st IEEE International Conference on Connected Health: Applications, Systems and Engineering Technologies (CHASE). Washington: IEEE, 2016. 357–358.
|
| [13] |
丁廷波. 基于体检大数据眼底图像快速筛选方法的研究[硕士学位论文]. 重庆: 重庆理工大学, 2021.
|
| [14] |
Islam T, Imran SA, Arefeen A, et al. Source and camera independent ophthalmic disease recognition from fundus image using neural network. Proceedings of the 2019 IEEE International Conference on Signal Processing, Information, Communication & Systems (SPICSCON). Dhaka: IEEE, 2019. 59–63.
|
| [15] |
Pratt H, Coenen F, Broadbent DM, et al. Convolutional neural networks for diabetic retinopathy. Procedia Computer Science, 2016, 90: 200-205. DOI:10.1016/j.procs.2016.07.014 |
| [16] |
Burlina PM, Joshi N, Pekala M, et al. Automated grading of age-related macular degeneration from color fundus images using deep convolutional neural networks. JAMA Ophthalmology, 2017, 135(11): 1170-1176. DOI:10.1001/jamaophthalmol.2017.3782 |
| [17] |
陈思坤. 医学图像的自动标注[硕士学位论文]. 成都: 电子科技大学, 2011.
|
| [18] |
Zhang Z, Yin FS, Liu J, et al. ORIGA-light: An online retinal fundus image database for glaucoma analysis and research. Proceedings of the 2010 Annual International Conference of the IEEE Engineering in Medicine and Biology. Buenos Aires: IEEE, 2010. 3065–3068.
|
| [19] |
中国科学院宁波材料技术与工程研究所. 宁波材料所所属慈溪医工所智能医疗影像团队iMED-Origa650数据集向学术界公开发布. https://cibe.nimte.ac.cn/view-13671.html. (2017-09-28)[2022-05-19].
|
| [20] |
廖晶安. 基于深度学习的互动式眼底图标注系统设计与实现[硕士学位论文]. 广州: 华南理工大学, 2019.
|
| [21] |
Cheng J, Zhang Z, Tao DC, et al. Similarity regularized sparse group lasso for cup to disc ratio computation. Biomedical Optics Express, 2017, 8(8): 3763-3777. DOI:10.1364/BOE.8.003763 |
| [22] |
Fu HZ, Li F, Orlando JI, et al. REFUGE: Retinal fundus glaucoma challenge. https://refuge.grand-challenge.org/home/. (2019-09-07)[2022-05-19].
|
| [23] |
Zou BJ, Guo YD, Chen ZL, et al. BGIDB: A fundus ground truth building tool with automatic DDLS classification for glaucoma research. Journal of Central South University, 2018, 25(9): 2058-2068. DOI:10.1007/s11771-018-3895-4 |
| [24] |
Antal B, Hajdu A. An ensemble-based system for microaneurysm detection and diabetic retinopathy grading. IEEE Transactions on Biomedical Engineering, 2012, 59(6): 1720-1726. DOI:10.1109/TBME.2012.2193126 |
| [25] |
Shao JH, Li J, Kong WZ, et al. A crowdsourcing framework for retinal image semantic annotation and report documentation with deep learning enhancement. Journal of Physics: Conference Series, 2021, 1955: 012037. DOI:10.1088/1742-6596/1955/1/012037 |
| [26] |
Peking University. Peking University international competition on ocular disease intelligent recognition (ODIR-2019). https://odir2019.grand-challenge.org/. [2022-05-19].
|
 2023, Vol. 32
2023, Vol. 32


