近10年, 全球204个国家的癌症负担和趋势呈上升趋势, 2019年全球癌症死亡人数上升至1 000万, 与2010年相比死亡人数增加了20.9%. 其中肝癌的伤残调整生命年(disability adjusted life years, DALYs)约为5%, 死亡人数约为48万人, 是癌症死亡的主要原因之一[1]. 肝癌主要分为3种, 肝细胞癌(hepatocellular carcinoma, HCC)、胆管细胞癌以及肝细胞癌和胆管细胞癌混合型的肝癌, 常见的是HCC. HCC治疗同时是临床的重大难题, 手术治疗是早期HCC患者最佳的治疗方式, 但是仍有一半左右患者会在两年内复发. 并且不少患者已经处于癌症晚期阶段, 失去了手术治疗的机会, 这就需要新型治疗方法. 血管介入治疗因为具有微创、精准以及并发症少等优势, 目前成为晚期HCC患者的重要治疗方式, 可显著提高患者生存率, 延长生存期. HCC血管介入治疗是通过肝脏供血系统对肿瘤组织进行药物灌注化疗或者供血血管栓塞治疗. 在治疗之前, 需要医生参考医学影像检查结果来标注血管, 通过标注好的血管结构来制定手术方法. 但由于肝脏血管结构复杂, 这个过程对于医生来讲, 是一项耗时且复杂的工作. 对于该问题, 如今已经可以通过自动化和半自动化的图像处理工具来完成血管分割, 这一定程度上也能够大大减轻医生的负担, 让医生更加专注在治疗上[2, 3]. 然而目前的分割精度经过系统研究发现还有很大的提升空间, 通过深入研究应从网络模型、数据预处理、后处理等阶段下手进行下一步研究. 尽管目前已经有很多杰出学者已经着手研究, 但是目前研究人员对于如何提高分割准确度上, 更多关注在网络结构上的改进和微调, 但是数据的预处理和后处理工作同样重要, 将网络进行的改进与数据处理相结合势必可以取得更好的分割准确度.
1.2 相关工作经过深入学习研究, 目前肝脏血管分割研究主要以机器学习为主并向深度学习领域发展. 研究方法可分为无监督学习和监督学习. 对于采用无监督学习方法的研究方向上, Goceri等人[4]通过K-means聚类的方法对肝脏血管完成初始分割, 然后通过形态学的方法来细化分割结果, 从而达到不错的分割效果; Oliveira等人[5]提出了一种区域生长的方法, 通过与高斯函数匹配并使用高斯混合模型获得所确定区间的极值来进行血管分割. Zeng等人[6]将4种3D血管过滤器进行组合来分割血管, 过滤器包括Sato、Frangi、offset medialness和strain energy[7-10], 然后使用极限学习机的方法将血管从背景中进行分离, 但是无监督学习方法量化分割效果困难, 相对于监督学习没有明确的学习目的.
监督学习是从训练集中提取特定的特征, 然后使用这些特征与相应标签共同来训练机器学习模型, 再用训练好的模型来完成分割任务. 卷积神经网络(convolutional neural networks, CNN)就是其主要代表网络模型之一, 它是一种具有前馈机制的神经网络, 能够实现端到端的训练, 当然也可以直接用于训练血管. Ibragimov等人[11]使用CNN在肝脏CT影像中分割门静脉, 在后处理阶段采用马尔可夫随机场来优化结果, 并去除由分割错误引起的孤立区域, 没有对数据预处理过多研究. 此外, 医学图像数据集的获取难度较大, 3D U-Net[12]网络的提出改善了这一现状, 其不仅将2D网络改为了3D网络, 还可以用少量的训练集和不完整的标注来进行提取分割, 但在测试上受到数据集差异影响, 对每个测试用例不能保证准确性. Isensee等人[13]在U-Net基础上又提出了nnU-Net, 在不对基础网络进行改动的基础上, 分别关注了预训练、训练阶段、推理策略及后处理部分, 在各大分割任务上绝大多数都取得了当时最优的效果, 可见对于提升分割效果上不能只关注网络改进. Hu等人[14]在ImageNet2017比赛中以SENet取得冠军, 该模型的核心思想是对每个输出通道, 预测一个常数权重, 随后在对每个通道进行加权, 通过该方法可以使网络有更好的学习能力, 受其启发, 可以在通道维度上进行特征重新校准来提高网络的泛化能力. 近两年, Transformer[15]从自然语言处理领域引入到计算机视觉领域并取得了突破性成果, 其将注意力机制作为网络中的一层, 使用自注意力机制和前馈网络进行编码和解码. 之后越来越多基于Transformer的模型相继被提出, 例如Dosovitskiy等人提出ViT[16]来做图像分类任务, 在大规模数据集上取得比CNN更好的效果, 但在中小型数据集上不如CNN效果好. Cao等人[17]提出了基于U-Net结构的Transformer分割网络, 在医学图像分割任务中成为最优模型, 表明基于Transformer的网络在分割任务上是非常可行的.
1.3 本文贡献本文基于以注意力机制为主的UNetR网络[18]进行改进, 用于分割肝脏血管. 创新点如下: (1)提出一个注意力门控单元, 其采用分块及注意力机制, 并结合通道注意力思想, 用来提高提取以及保留特征的能力. (2)提出UNetR-AGM模型, 该模型在UNetR网络模型的基础上引入注意力门控单元, 通过该单元提高网络模型的学习能力. (3)提出一种均衡过滤策略, 利用不同组织HU值范围不同的特点, 对数据集进行血管粗提取, 再通过归一化处理来简化分割任务难度, 使网络更好的收敛.
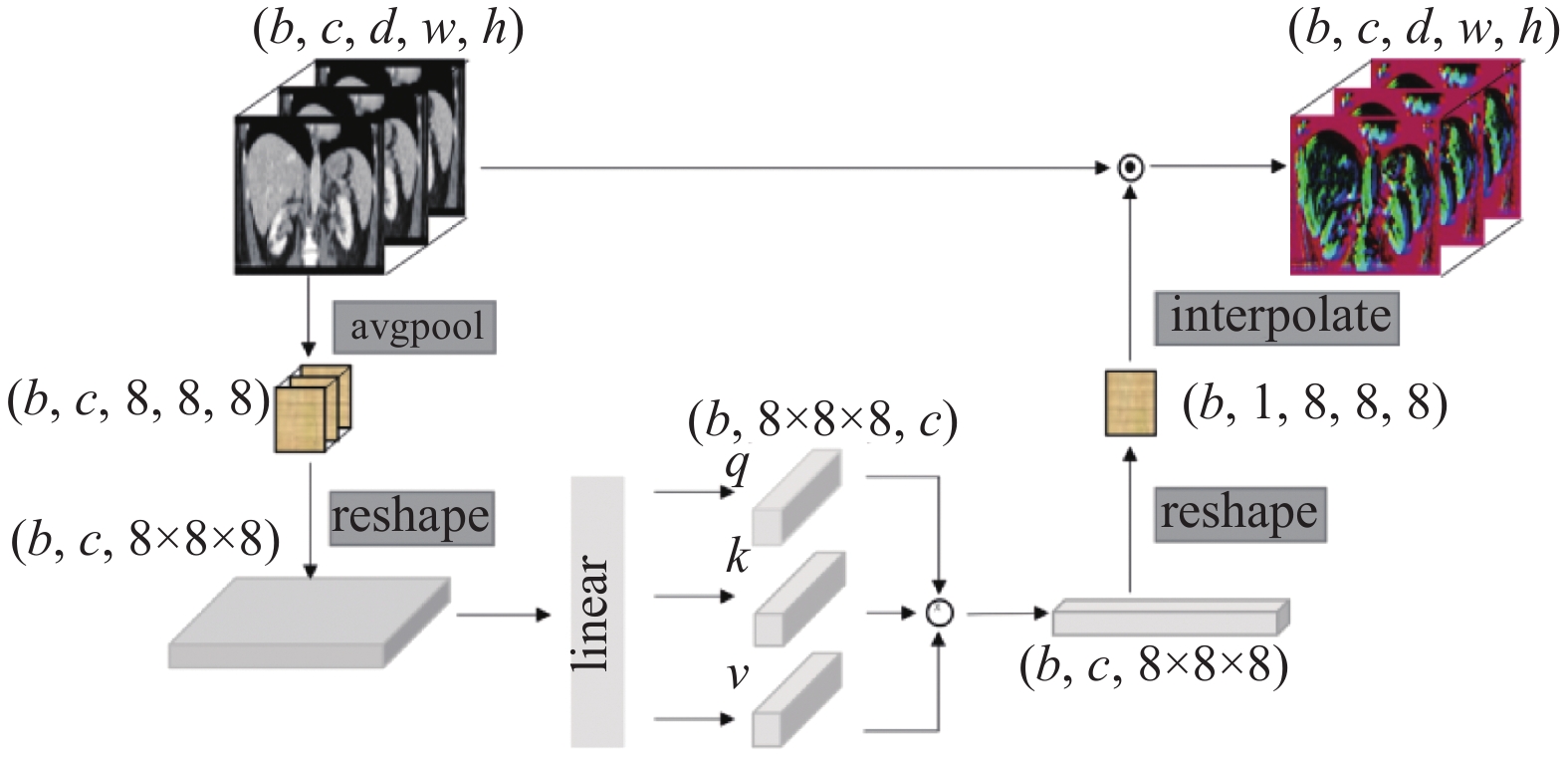
2 方法 2.1 注意力门控单元本文提出了一个注意力门控单元, 此单元是为了弥补卷积操作在全局信息提取上的不足. 该单元能获取输入的每个像素的注意力信息, 随后将获取到的像素注意力信息作为门控单元输入, 再进行注意力程度的门控过滤, 从而完成对输入的全局注意力提取. 注意力门控单元的结构如图1.
|
图 1 全局注意力门控单元 |
具体实现方案: 首先将输入为D×W×H的三维特征进行分块压缩, 在本文中我们通过均值池化的方法进行压缩, 压缩后的特征定义为8×8×8结构大小的分块特征图, 该特征图的每个数值分别代表了输入中对应区块的特征. 随后将8×8×8的特征图压缩为一维特征. 接下来结合注意力机制的设计思路, 通过全连接层来计算出q、k、v:
| q=WqX | (1) |
| k=WkX | (2) |
| v=WvX | (3) |
其中, X为输入, W为权重矩阵, 通道数为c. 再将其进行矩阵乘法并通过Sigmoid函数得到注意力门控矩阵 (atten-tion gating matrix, AGM):
| AGM=Sigmoid(WqXXTWTkWvX) | (4) |
接下来经过全连接层在通道维度进行压缩, 将c个通道进行压缩整合, 给出图像中每个区块的注意力门控值. 最后使用最近邻线性插值方法, 重采样注意力门控矩阵为输入特征的大小, 并采用按位乘法的操作对输入特征进行门控过滤.
2.2 UNetR-AGM本研究在UNetR的基础上进行改进, 在网络的跳跃连接和解码部分加入本文提出的AGM模块, 在全局信息的提取和保留方面对网络进行优化, 以此来提高网络的泛化能力和鲁棒能力. 在该网络中, 编码器通过ViT实现, 该阶段的Trans-former共有12层, 分别取其3、6、9、12层的编码信息与解码器通过跳跃连接合并. 在合并之前, 考虑到卷积操作对全局信息保留不足, 因此需要先通过AGM block率先对编码器的信息进行提取, 将编码阶段特征信息的局部和全局特征进行保留, 对于上采样的过程采取相同操作. 然后通过跳跃连接操作将编码器和解码器的特征信息合并. 最后, 将最终的输出送入到带有Softmax激活函数的1×1×1的卷积层中, 以生成语义预测. 具体的网络结构如图2所示.
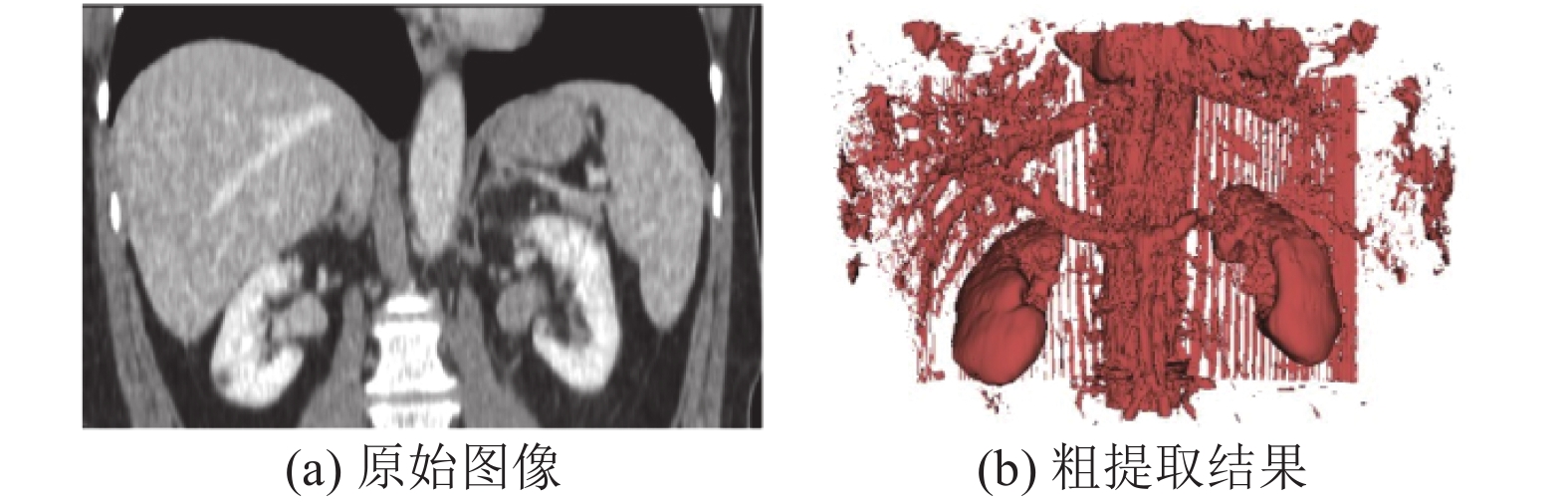
2.3 均衡过滤策略在腹部器官的分割任务中, 一般需要在整个CT图像中完成特定器官的分割任务, 而特定器官在整个CT图像中只占小部分, 大量的无关信息不仅对模型训练效率有所影响, 还会增加模型的负担损耗性能. 据了解, CT图像中不同的器官Hu值有特定的取值范围, 在腹部的CT图像中, 软组织的CT值一般在+20 Hu至+1000 Hu之间. 利用这一特征可以对原始图像进行目标器官的粗提取, 进而再送入后续网络进行模型训练, 以此来降低网络训练的复杂度. 本文对数据集进行了如下预处理: 首先, 将标签与原始CT图像进行叠加运算, 以此来计算出标签覆盖区域即血管组织的上下取值边界, 以该范围作为血管粗提取的依据, 以此完成对数据集的血管粗提取. 最后, 将粗提取之后的CT图像按照正态分布进行归一化处理. 粗提取后的结果如图3所示.
2.4 loss损失函数本论文的loss损失结合了预测分割掩码常用的soft-Dice loss[19]和像素级别的交叉熵loss, 前者以整体的方式看待预测输出, 而后者将每个像素看作独立样本来预测. 本文分别使用Gi和Pi来表示真实值和预测值, 其中I表示像素总量, 其中J表示分类数目. 具体的公式表达如式(5)所示:

|
图 2 网络结构图 |
|
图 3 肝脏血管粗提取 |
| L(G,P)=1−2JJ∑j=1∑Ii=1Gi,jPi,j∑Ii=1G2i,j+∑Ii=1P2i,j−1II∑i=1J∑j=1Gi,jlogPi,j | (5) |
硬件配置: Intel(R) Core(TM) i9-10900K CPU @ 3.70 GHz; 128 GB内存; NVIDIA GeForce RTX 3090 24 GB显卡.
软件环境: Ubuntu 16.04.7 LTS 操作系统; Python 3.6.13编程语言; PyTorch 1.8深度学习框架.
3.2 数据集Hepatic vessels dataset: 该数据集是2018年西班牙格拉纳达医学图像计算和计算机辅助干预会议期间举行的医学分割十项全能挑战(MSD)提供的数据集[20, 21]. 它由Sloan Kettering癌症中心提供的多种原发性和转移性肝癌(胆管癌、肝细胞癌、转移瘤等)患者病例组成. 包括443次门静脉期CT扫描. 对于肝脏血管的真实标签, 通过Scout半自动分割肝血管应用程序 [22]来处理. 该程序会首先在感兴趣的区域中绘制种子点, 然后使用基于水平集的方法基于种子点进行生长. 最后的结果再由腹部放射科医生对其边界轮廓进行手动调整. 该数据集包含了303份训练集和140份测试集, 以NIFTI格式来存储. 本实验对训练集按照7:3的比例划分为242份训练集和61份验证集, 以便于训练时验证模型的好坏.
对于肝脏血管分割任务, 在预处理阶段, 所有CT在各个方向维度上都按照像素间距1.0 mm进行重采样. 根据CT数据中Hu值, 将肝脏血管进行粗提取, 再将CT的Hu值归一化到范围[0, 1]. 随后随机采样体积大小为[64, 64, 64]的图像块, 作为后续网络模型的输入.
3.3 评价标准本论文采用DSC (Dice similarity coefficient)和ASD (average surface distance)来评价网络模型的分割效果. 在Dice使用过程中, 本文定义Gi和Pi来表示真实值和预测值, I表示像素总量. Dice相似系数的数学定义如式(6)所示:
| DSC(G,P)=2∑Ii=1GiPi∑Ii=1Gi+∑Ii=1Pi | (6) |
在ASD的使用过程中, 先定义Aseg来代表预测Pi中的边界的像素, 定义Agt代表真实Gi中的边界的像素, 然后通过计算Aseg中每个像素到Agt的最近距离来获得Bseg, 数学公式表达如式(7):
| Bseg={∀p1∈Aseg,closet_distance(p1,p2)|∃p2∈Agt} | (7) |
同理可以得到Bgt, 通过Bseg和Bgt两者的平均值可以得到ASD, 方法如式(8):
| ASD=mean({Bseg,Bgt}) | (8) |
本文将提出的算法与nnU-Net、nnFormer、UNetR进行了对比实验, 所有对比的实验方法均为3D分割网络. 在训练轮次达到500个epoch后, nnU-Net、nnFormer、UNetR的平均DSC精度分别为0.6624、0.6823、0.7753, 本文的算法平均DSC精度为0.7978, 均比其他算法的精度高. 其中, nnU-Net分割结果DSC准确率最高为0.7225, 最低为0.5001; nnFormer分割结果DSC准确率最高为0.7264, 最低为0.6292; UNetR分割结果DSC准确率最高为0.8839, 最低为0.653 4.
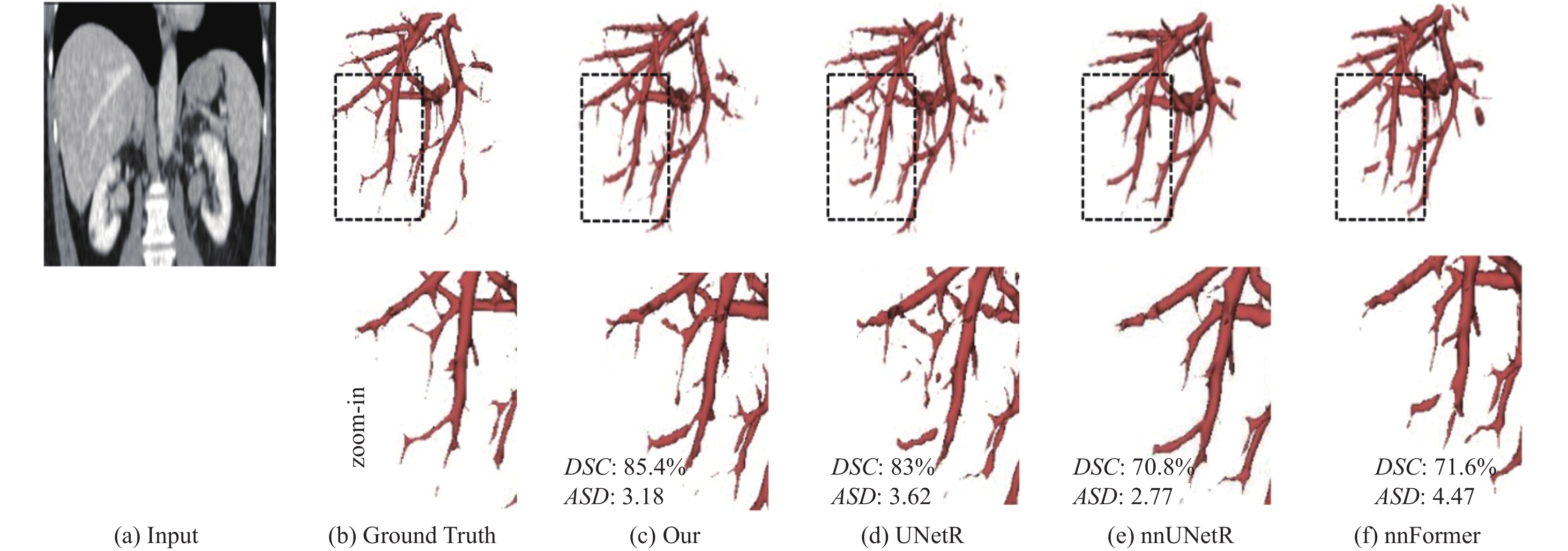
根据实验结果, 本文提出的方法与其他方法相比在全局信息的提取和保留上更具有优势, 在结构复杂关联性强的血管结构上表现也更为突出, 这也证实了本文提出的AGM和数据预处理方法在肝脏血管分割任务上是有效的. 在UNetR的基础上其分割效果提升了2%左右. 图4为肝脏血管分割的三维结果, 可以说明本文的分割方法在肝脏血管分割上是有优势的. 与表1结果一致, 本文的分割方法在提取肝脏血管方面表现更好. 与现有方法相比, 在保证分割性能的基础上, 本文的方法提取了更多了细小分支. 在成功分割到大量肝脏血管后, 我们仔细检查了错误预测. 如图5显示, 可以注意到本文的方法可能无法正确地对一些孤立的血管片段进行预测. 在血管末端的预测有所欠佳, 还有待下一步对网络进行改进来解决这一问题.
|
图 4 对比实验图 |
3.5 消融实验
我们需要研究肝脏血管粗提取方案和注意力门控单元在改善分割性能方面的有效性.
① 均衡过滤策略
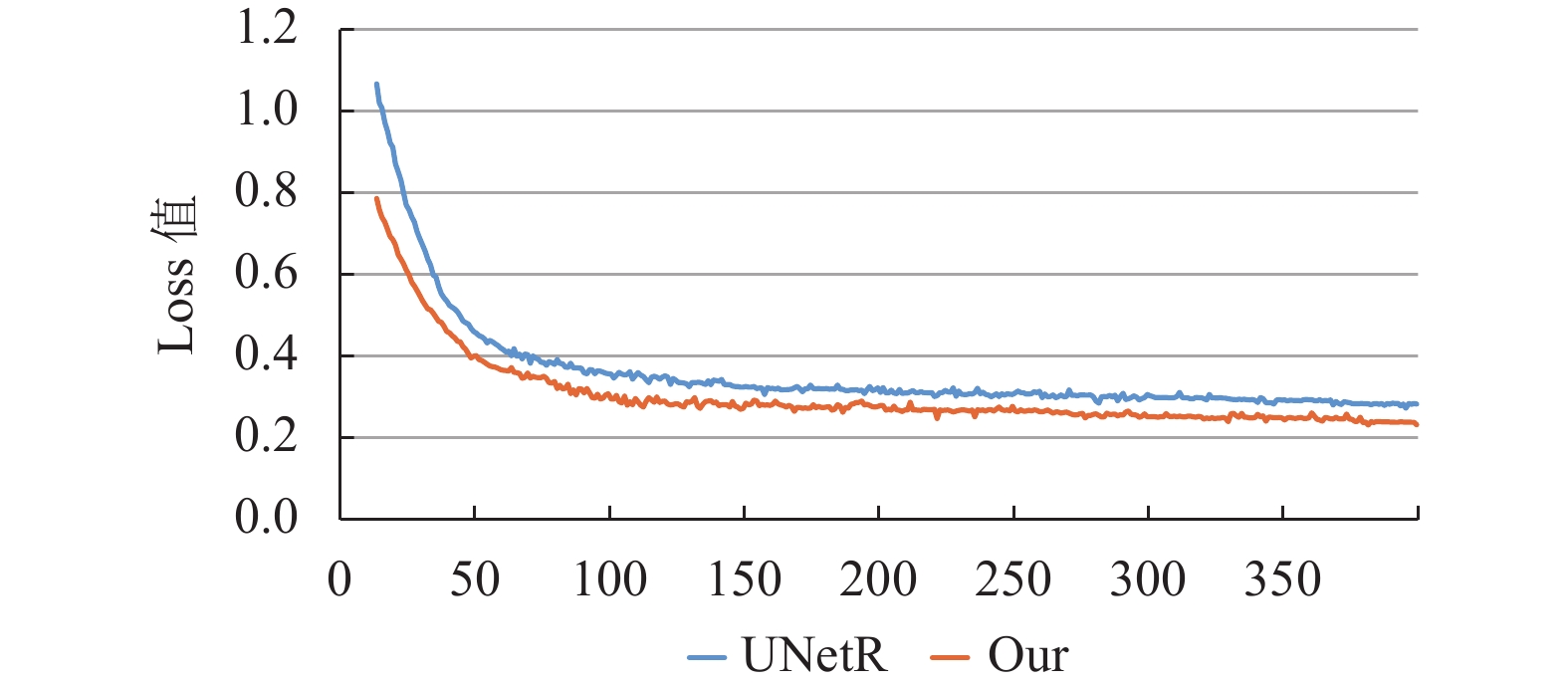
以往的分割方法通常直接将整个图像输入网络进行预测, 但是在肝脏数据集中, 血管占比总体较小, 且集中于某个特定部位. 因此, 考虑到这一问题, 本文并不直接将整个原始CT图像直接输入网络中进行训练, 而是首先利用Hu值对血管进行粗提取, 这样就可以很大程度上剔除无关区域, 然后将粗提取结果进行归一化处理. 实验结果表明, 通过粗分割步骤缩小分割区域, 可以更好地帮助网络区分分割边界, 减少运算量, 加快网络的收敛速度. 和没有血管粗提取步骤的UNetR相比, 在epoch为50时的本文模型loss值降到了0.4, 而UNetR在epoch为70时才达到该损失值, 并且在之后的训练中, 本文的模型一直有更好的收敛效果, 可视化结果如图6所示.
| 表 1 对比实验表 |

|
图 5 预测与标注差异图 |
|
图 6 Loss收敛图 |
② 注意力门控单元
医学分割对边界轮廓分割要求较高, 而以往的网络通过不断地卷积和池化操作后会丢失边界细节信息, 导致网络最终的边界分割效果不够理想. 为了更好地保留细节信息, 我们提出了门控单元. 该单元的作用类似于跳跃连接, 将encoder信息与decoder信息进行融合, 但不同于普通的跳跃连接, 而是加入了AGM模块后再进行融合. 我们的AGM模块将更多的注意力放到了重要的细节信息上, 有效保留了细节信息. 表2结果表明, AGM模块对肝脏血管的细节信息可以很好地保留, 分割准确率也有了进一步的提高.
| 表 2 消融实验表 |
4 结论
在本研究中, 我们对UNetR网络模型进行了改进, 将改进后的网络用来分割肝脏血管. 首先对于卷积层提取全局特征能力弱的问题, 引入了本文提出的AGM模块, 改善卷积层在网络结构中丢失全局信息的问题, 以提高网络对肝脏血管的分割能力. 在数据集预处理阶段, 在本文提出了一种均衡过滤策略, 一方面可以对原始数据进行粗提取, 另一方面还可以使网络更好的收敛. 实验结果表明, 引入AGM模块和血管均衡策略能够加快模型收敛的同时还提高了肝脏血管的分割准确度, 比先前的网络提升了2%左右.
| [1] |
Kocarnik JM, Compton K, Dean FE, et al. Cancer incidence, mortality, years of life lost, years lived with disability, and disability-adjusted life years for 29 cancer groups from 2010 to 2019: A systematic analysis for the global burden of disease study 2019. JAMA Oncology, 2022, 8(3): 420-444. DOI:10.1001/jamaoncol.2021.6987 |
| [2] |
陈小彬, 郑凡, 黎成金, 等. 肝细胞癌相关独立危险因素研究进展. 临床普外科电子杂志, 2021, 9(4): 59-63. DOI:10.3969/j.issn.2095-5308.2021.04.011 |
| [3] |
文良志, 肖潇, 颜綦先, 等. 肝细胞癌血管介入治疗临床研究新认知. 临床肝胆病杂志, 2021, 37(8): 1761-1764. DOI:10.3969/j.issn.1001-5256.2021.08.005 |
| [4] |
Goceri E, Shah ZK, Gurcan MN. Vessel segmentation from abdominal magnetic resonance images: Adaptive and reconstructive approach. International Journal for Numerical Methods in Biomedical Engineering, 2017, 33(4): e2811. DOI:10.1002/cnm.2811 |
| [5] |
Oliveira DA, Feitosa RQ, Correia MM. Segmentation of liver, its vessels and lesions from CT images for surgical planning. BioMedical Engineering Online, 2011, 10: 30. DOI:10.1186/1475-925X-10-30 |
| [6] |
Zeng YZ, Zhao YQ, Liao M, et al. Liver vessel segmentation based on extreme learning machine. Physica Medica, 2016, 32(5): 709-716. DOI:10.1016/j.ejmp.2016.04.003 |
| [7] |
Sato Y, Nakajima S, Shiraga N, et al. Three-dimensional multi-scale line filter for segmentation and visualization of curvilinear structures in medical images. Medical Image Analysis, 1998, 2(2): 143-168. DOI:10.1016/S1361-8415(98)80009-1 |
| [8] |
Frangi AF, Niessen WJ, Vincken KL, et al. Multiscale vessel enhancement filtering. Proceedings of the 1st International Conference on Medical Image Computing and Computer-assisted Intervention. Cambridge: Springer, 1998. 130–137.
|
| [9] |
Krissian K, Malandain G, Ayache N, et al. Model-based detection of tubular structures in 3D images. Computer Vision and Image Understanding, 2000, 80(2): 130-171. DOI:10.1006/cviu.2000.0866 |
| [10] |
Xiao CY, Staring M, Shamonin D, et al. A strain energy filter for 3D vessel enhancement with application to pulmonary CT images. Medical Image Analysis, 2011, 15(1): 112-124. DOI:10.1016/j.media.2010.08.003 |
| [11] |
Ibragimov B, Toesca D, Chang D, et al. Combining deep learning with anatomical analysis for segmentation of the portal vein for liver SBRT planning. Physics in Medicine & Biology, 2017, 62(23): 8943-8958. |
| [12] |
Çiçek Ö, Abdulkadir A, Lienkamp SS, et al. 3D U-Net: Learning dense volumetric segmentation from sparse annotation. Proceedings of the 19th International Conference on Medical Image Computing and Computer-assisted Intervention. Athens: Springer, 2016. 424–432.
|
| [13] |
Isensee F, Jaeger PF, Kohl SAA, et al. nnU-Net: A self-configuring method for deep learning-based biomedical image segmentation. Nature Methods, 2021, 18(2): 203-211. DOI:10.1038/s41592-020-01008-z |
| [14] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [15] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 6000–6010.
|
| [16] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16×16 words: Transformers for image recognition at scale. Proceedings of the 9th International Conference on Learning Representations. ICLR, 2021.
|
| [17] |
Cao H, Wang YY, Chen J, et al. Swin-UNet: Unet-like pure transformer for medical image segmentation. Proceedings of the 2022 European Conference on Computer Vision. Tel Aviv: Springer, 2022. 205–218.
|
| [18] |
Hatamizadeh A, Tang YC, Nath V, et al. UNetR: Transformers for 3D medical image segmentation. Proceedings of 2022 IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2022. 1748–1758.
|
| [19] |
Milletari F, Navab N, Ahmadi SA. V-net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [20] |
Simpson AL, Antonelli M, Bakas S, et al. A large annotated medical image dataset for the development and evaluation of segmentation algorithms. arXiv:1902.09063, 2019.
|
| [21] |
Antonelli M, Reinke A, Bakas S, et al. The medical segmentation decathlon. arXiv:2106.05735, 2021.
|
| [22] |
Dawant BM, Li R, Lennon B, et al. Semi-automatic segmentation of the liver and its evaluation on the MICCAI 2007 grand challenge data set. Proceedings of the 2007 MICCAI Workshop on 3D Segmentation. Clinic: A Grand Challenge, 2007. 215–221.
|
 2023, Vol. 32
2023, Vol. 32



