2. 中国科学院大学, 北京 100049;
3. 山东大学 大数据技术与认知智能实验室, 济南 250100;
4. 中国人民解放军总医院, 北京 100039;
5. 中科智禾数字科技(北京)有限公司, 北京 101499
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Laboratory of Big Data and Artificial Intelligence Technology, Shandong University, Jinan 250100, China;
4. Chinese PLA General Hospital, Beijing 100039, China;
5. Zhongke Zhihe Digital Technology (Beijing) Co. Ltd., Beijing 101499, China
随着机器学习与深度学习的发展, 计算机视觉技术在医学图像分析中得到了广泛的应用. 图像分割是医学图像分析的重要组成部分, 特别是在计算机辅助诊断与图像引导的临床手术中发挥着重要作用[1].
现有的医学图像分割方法主要基于U形结构的全卷积神经网络(FCNN). 独特的U型结构U-Net[2], 由一个带有跳跃连接的对称编码器-解码器组成. 在编码器中, 采用一系列卷积层和连续下采样层来提取具有较大感受野的深度特征. 然后, 解码器将提取的深度特征上采样进行像素级语义预测, 在此过程中将不同尺度的高分辨率特征图信息通过跳跃连接融合, 来减少下采样造成的空间信息损失. U-Net凭借其优秀的结构设计, 在各种医学影像的任务中取得了优异的成绩. 跟随着这一技术路线, 许多算法如3D U-Net[3]、Res-UNet[4]、U-Net++[5]和UNet3+[6]已被创造出来, 用于各种医学格式的图像分割. 这些基于FCNN的方法在心脏分割、器官分割和病变部位分割中的出色表现证明了CNN具有较强的提取特征的学习能力.
目前, 尽管基于CNN的方法在医学图像分割领域取得了优秀的表现, 但仍然不能完全满足医学实际应用中对分割精度的严格要求. 在医学图像分析中, 图像分割仍然是一个具有挑战性的任务. 由于卷积运算的固有局域性, 基于CNN的方法很难学习到全局语义信息和像素相对距离较远的语义信息[1]. 一些研究试图通过使用空洞卷积、自注意力机制[7]和图像金字塔[8]来解决这个问题. 然而, 这些方法在建立较远的依赖关系方面仍然有局限性. 最近, 受到Transformer在自然语言处理(NLP)领域取得巨大成功的启发[9], 研究人员试图将Transformer引入视觉领域[10]. 文献[11]提出了使用Vision Transformer (ViT)来进行图像识别任务. 用带有位置信息的二维图像块序列作为输入, 在大数据集上进行预训练, ViT的性能与基于CNN的方法相当. 此外, 文献[12]还提出了data-efficient image Transformer (DeiT), 使得Transformer可以在中等规模的数据集上训练, 并且结合知识蒸馏方法可以得到一个更稳健的Transformer. 文献[13]设计了一个分层的Swin Transformer, 将Swin Transformer作为视觉特征提取骨干网络, 在图像分类、目标检测和语义分割方面取得了明显的进步. ViT、DeiT和Swin Transformer在图像识别方面的成功应用表现出了Transformer在视觉领域的应用潜力.
本文利用Transformer的优势进行2D医学图像分割, 模型是将Transformer、ResNet和U-Net++思想于一体的语义分割网络. 与其他网络结构相似, 网络由编码器、解码器和跳跃连接组成. 编码器基于ResNet-50和Vision Transformer构建的, 输入的医学图像经过多层CNN特征提取结构后, 被分割成不重叠的图像块, 将分割的图像块组合成图像块序列输入到基于Trans-former的编码器学习全局特征. 然后进入基于CNN的解码层对提取的多层次特征进行上采样, 并通过跳跃连接与下采样过程中的多尺度特征融合, 恢复特征图到近似输入图像的分辨率尺寸, 最后进行像素级的分割预测. 在UW胃肠道图像分割数据集上的实验表明, 该方法具有优秀的分割精度和泛化能力. 具体而言, 模型的贡献可以概括为: (1)基于CNN和Transformer, 构建了一个具有多层级跳跃连接的非对称编码器-解码器体系结构. 在编码器中实现了从局部到全局的自注意力机制. 在解码器中, 将全局特征上采样到输入分辨率, 进行像素级的分割预测; (2)充分结合卷积神经网络CNN和Transformer结构的优势对医学图像进行分割预测; (3)利用多层级跳跃连接结构, 充分利用各层级特征信息, 最终构造了一个基于CNN和Transformer的带有跳跃连接的U型编码器-解码器网络结构.
2 相关工作早期的医学图像分割方法主要是基于轮廓和传统的基于机器学习的算法. 随着深度学习卷积神经网络的发展, U-Net神经网络被提出用于医学图像分割. 由于其简单的U形结构和优越的性能, 各种类似U-Net的方法不断涌现, 如Res-UNet、Dense-UNet[14]、U-Net++和UNet3+. 并将其引入3D医学图像分割领域, 如3D-UNet和V-Net[15]. 目前, 基于CNN的方法凭借其强大的表现能力在医学图像分割领域取得了巨大成功.
众所周知, 上下文信息是提升语义分割性能的关键因素, 而感受野的大小就基本决定了网络可以利用多少有用的信息. 理论上, 通过堆叠足够深的卷积层, 网络的感受野便能够覆盖到输入图像的全局领域, 然而He等人[16]发现, 随着模型层数的增加, 识别精度不会提升反而会下降. Transformer最初是为机器翻译任务而提出的[9]. 在自然语言处理领域, 基于Transformer的方法在各种任务中均取得了最优秀的成绩[17]. 在Transformer的成功引领下, 研究人员引入了一种开创性的方法Vision Transformer (ViT) , 它实现了惊人的速度与精度均衡的图像识别任务. Transformer不像卷积操作那样有固定且有限的感受野, 它的核心self-attention可以直接提取整体的感受野. 也正是这样丰富的感受野, 以及它在各项挑战赛中优异的性能, 吸引了很多图像领域的研究学者. 值得一提的是Swin-UNet[18], 使用纯Vision Transformer结构来进行提取特征. 基于U-Net网络结构, Swin-UNet实现了医学图像分割的优秀性能, 但Swin-UNet网络的分割结果相较于CNN方法的分割结果表现在分割的边缘较为粗糙.
近年来, 研究人员试图在CNN中引入自注意力机制来提高网络模型的性能. 文献[7]在U型结构中加入了带有附加注意门的跳跃连接, 进行医学图像分割. 然而, 这仍然是基于CNN的方法. 目前, 人们正在努力将CNN和Transformer结合起来, 打破CNN在医学图像分割中的主导地位[19]. 文献[1]将Transformer与CNN相结合, 构成一个用于二维医学图像分割的强编码器. 与文献[1]的工作相似, 文献[20]和文献[21]利用Transformer和CNN的互补性来提高模型的分割性能. 在本文中, 尝试进一步融合CNN与Vision Transformer模块, 构建一个具有多层级跳跃连接的U型编码器-解码器架构, 用于医学图像分割, 从而为Transformer在医学图像领域的发展进一步做出探索.
3 医学图像分割网络模型给定一个分辨率为
对于图像分割, 最直接的上采样解决方法是简单地将编码的特征表示
网络模型的整体结构如图1所示. 网络并没有使用纯CNN或者纯Transformer作为特征提取器, 而是采用了CNN-Transformer混合网络模型. 网络由编码器、解码器和跳跃连接组成. 对于编码器, 首先将图片输入CNN结构进行局部特征提取, CNN特征提取部分使用ResNet-50网络结构. 为了将输入信息转化为序列编码, Transformer部分首先将CNN下采样部分的特征图像块进行编码转换. 转换后的图像块序列经过12个Transformer模块, 来学习全局特征. 受到U-Net++网络模型的启发, 设计了一个多层级的跳跃连接结构. 解码器部分由图像块序列扩展层、CNN上采样层和跳跃连接模块组成. 通过跳跃连接将提取的上下文特征与编码器的多尺度特征相融合, 来弥补下采样造成的信息损失. 解码器部分首先将Transformer结构输出的结果进行位置恢复, 之后利用级联的上采样器将相邻维度的特征图多次进行2倍分辨率上采样. 最后利用一个扩展层进行4倍上采样, 将特征图恢复到与输入相近的分辨率
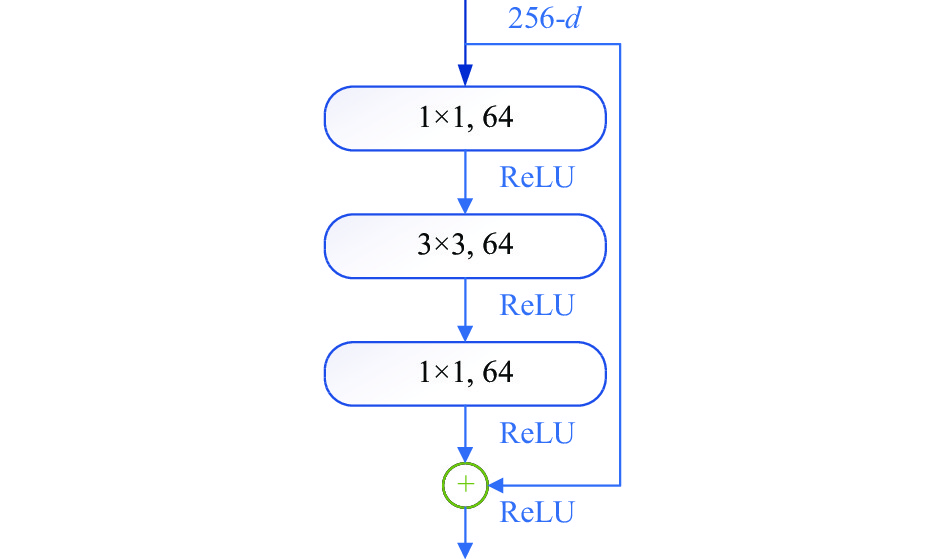
首先对输入的图片使用CNN卷积神经网络进行下采样局部特征提取, 这部分使用残差网络ResNet-50作为特征编码, 共分为3个下采样阶段, 详细CNN下采样模块参数见表1. 其中每一个模块由多个bottleneck结构重复叠加组成. Bottleneck部分先通过一个1×1的卷积来减少通道数, 使得中间卷积的通道数缩小4倍, 中间的普通3×3卷积做完卷积后输出的通道数等于输入通道数, 第3个1×1的卷积用于恢复通道数, 使得输出通道数等于输入通道数. 这两个1×1的卷积有效减少了卷积的参数个数和计算量, bottleneck的结构如图2.

|
图 1 网络基本架构 |
| 表 1 ResNet-50结构 |
|
图 2 Bottleneck结构 |
图2中的符号d表示特征图的通道数, 256-d即表示输入的特征图通道数有256个.
3.3 Transformer编码器首先对输入的特征图
使用可训练的线性映射将向量化的图像块
| z0=[x1pE;x2pE;⋯;xNpE]+Epos | (1) |
其中,
Transformer编码结构由L层多头自注意力机制(MSA)和多层感知机(MLP)组成. 其中, 多头自注意力机制即含有多个分支的self-attention模块, 每个分支即表示一个头, 而多层感知机即隐藏层和输出层都是全连接层的神经网络. 编码结构中第
| zℓ′=MSA(LN(zℓ−1))+zℓ−1 | (2) |
| zℓ=MLP(LN(z′ℓ))+zℓ′ | (3) |
其中,
解码部分引入了一种级联上采样器(CUP), 它由多个上采样及多层跳跃连接组合来解码隐藏特征, 来得到最终的分割掩码图. 在将隐藏特征
与U-Net相似, 跳跃式连接用于将编码器的多尺度特征与上采样特征进行融合. 将浅层特征和深层特征拼接在一起, 来减少下采样造成的空间信息损失. 但是第1个阶段下采样仅提取了图像的浅层特征, 直接与上采样的最后一层特征融合会丢失很多信息, 所以加入了密集的跳跃连接, 将U-Net网络架构的中心填满, 使水平方向上的每一尺度之间都有连接. 上采样的方法有很多, 本文中使用双线性插值上采样方法. 将两个特征图进行拼接后使用1×1卷积进行通道数减半操作, 这个操作的目的是使得特征图的通道数与拼接前的通道数相同, 具体操作如图4所示.

|
图 3 Transformer编码结构 |

|
图 4 特征拼接结构 |
4 实验分析
实验中使用UW-Madison GI Tract Image Segmentation胃肠道医学图像分割数据集(https ://www.kaggle.com/competitions/uw-madison-gi-tract-image-segmentation/data)对本文算法进行详细的评估, 并且和近几年优秀的医学图像分割模型进行比较, 最后对所提出的模型的多个模块部分进行消融实验.
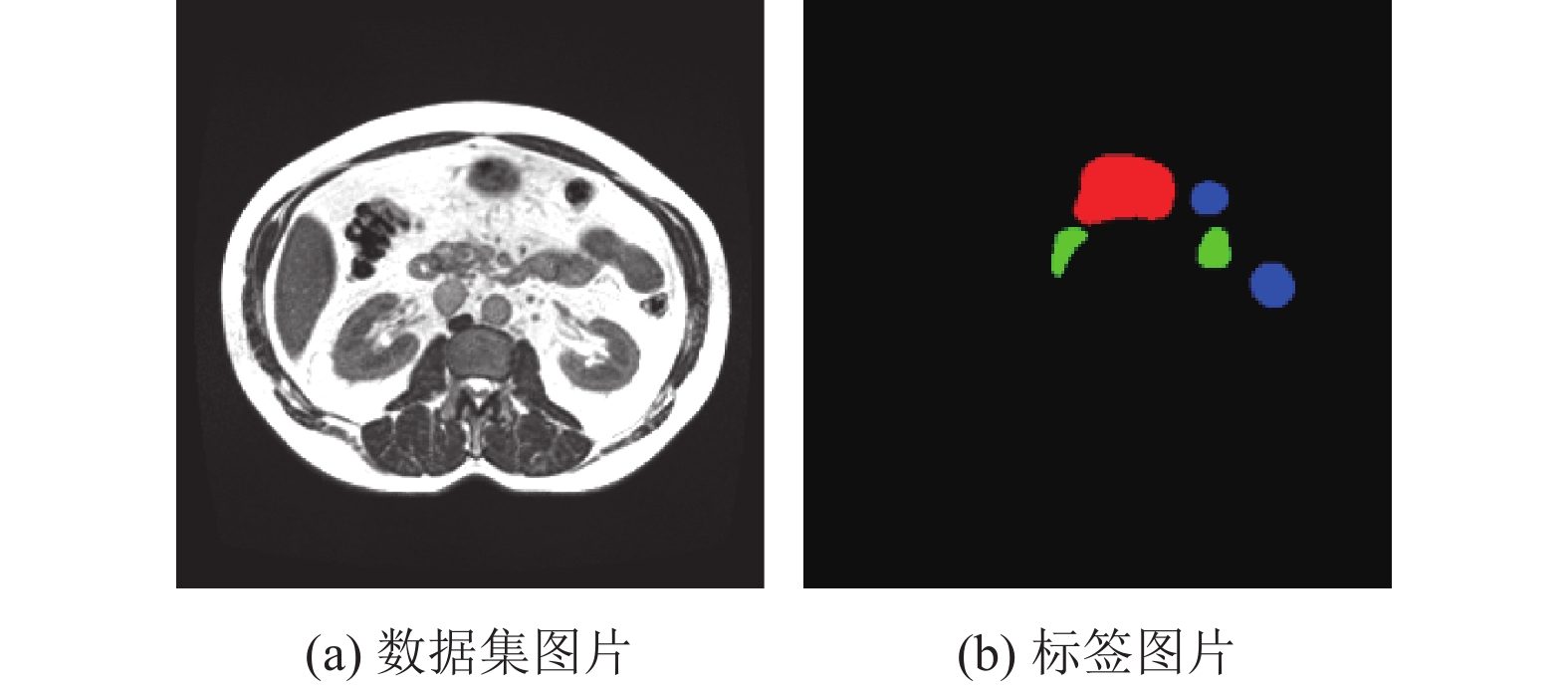
4.1 数据集介绍实验使用的UW-Madison GI Tract Image Segmentation胃肠道医学图像数据集来源于kaggle的胃肠道图像分割比赛. 如图5(a)所示, 原始数据集的图片存在肉眼完全看不到的情况, 肉眼看不到并不是图片中不存在器官, 肉眼是否可见与图片中有无器官并无直接关联, 通过将图像的全局亮度调高, 可以看到图像的各个部位. 数据集共包含85个病例的CT扫描图片, 每个病人包含多个时期阶段的扫描结果, 每个时期阶段包含144张图片, 图片尺寸不统一, 大多为
|
图 5 数据集样本 |
此数据集官方所提供的图像为16位灰度的PNG格式, 标签以RLE编码的掩码形式提供, 标签文件不可将其直接作为网络模型的输入. 因此, 首先需要将标签文件转换为适合网络模型输入的格式, 实验中将标签文件转换为与图像格式相同的PNG格式. 如图5(b), 此为转换为PNG格式后的标签图片.
4.2 实验环境及参数设置实验环境使用的深度学习框架为PyTorch 1.8.2, 开发环境为Python 3.8.13, 操作系统为Ubuntu 18.04.3 LTS, GPU选用的是一张GeForce RTX 3090, 显存为24 GB. 以下所有的实验均使用此实验环境进行.
实验详细参数设置如下: 首先将原始图片转换为同一尺寸, 使网络模型的输入大小为
| W←W−η∂L∂W | (4) |
其中,
为了评估图像语义分割方法的性能, 需要从语义分割的准确性来进行衡量算法模型的性能, 性能评价指标使用Dice相似系数(Dice similarity coefficient, DSC)和Hausdorff_95 (HD95). 其中, Dice系数对分割的内部填充比较敏感, 而Hausdorff_95对分割出的边界比较敏感.
Dice相似系数(DSC)是一种集合相似度度量函数, 通常用于计算两个样本的相似度, 取值范围为[0, 1], 分割结果最好时值为1, 最差时值为0, 如式(5)所示:
| S=2|X∩Y||X|+|Y| | (5) |
其中,
HD95计算两个集合之间的距离, 值越小, 代表两个集合的相似度越高, 故数值越小越好, 如式(6)所示. 最后的值乘以95%, 目的是为了消除离群值的一个非常小的子集的影响.
| dH(X,Y)=max{maxx∈Xminy∈Y(x,y),maxy∈Yminx∈X(x,y)} | (6) |
其中,
表2和表3展示了所提出的网络模型在UW-Madison GI Tract Image Segmentation胃肠道医学图像分割数据集上与目前各类型优秀方法的比较. 所比较的方法包括R50 UNet、R50 Att-UNet 、U-Net++、TransUNet和Swin-UNet. 实验结果表明, 所提出的方法获得了最佳的分割精度, 其分割精度DSC达到84.54%.
在分割结果的DSC上可以看出U-Net++网络的分割结果准确率高于除本文模型之外的其他对比方法, R50 UNet的分割结果准确率低于其他对比方法. 但是, 通过观察图6可以发现, U-Net++的分割结果似乎看起来是最差的, 而R50 UNet的结果看起来似乎比其他对比方法要好. 根据上述评价指标DSC可以推断, 这是因为U-Net++产生了欠分割, 导致预测的结果与真实值的和变小, 导致DSC分母偏小, 进而导致准确率相对高一些. 同理, R50 UNet是产生了过分割, 导致预测的结果与真实值的和偏大, 分母偏大, 而分子中的交集并未增大, 所以DSC准确率偏低. 而本文的方法的分割效果则处于二者之间, 所以准确率与分割效果均优于上述两个网络. 同时, 本文模型的参数量与TransUNet网络参数量相当, 但网络DSC准确率远高于TransUNet, 且在HD95评价指标中此方法的数值均小于对比模型, 进一步证明了本文模型的有效性.
| 表 2 测试集对比结果(DSC) (%) |
| 表 3 测试集对比结果(HD95) (mm) |
4.5 各个模块的消融实验
为了探究不同因素对模型性能的影响, 又继续在UW-Madison GI Tract Image Segmentation胃肠道医学图像分割数据集上进行了各个模块的消融实验. 分别对模型的输入图像分辨率、模型中不同数量的Transformer层以及Transformer层中图像块的不同尺寸和序列长度进行详细实验对比.
网络模型的默认输入分辨率为
| 表 4 不同输入分辨率的消融实验 |
为了验证模型的有效性, 继续对不同数量的Transformer层进行了消融实验, 共进行了3种不同数量Transformer层的影响, 分别为2层、8层、和12层, 详细结果见表5. 实验过程中除Transformer层的数量变化外, 其他模型参数均保持不变. 实验结果表明, 随着Transformer层数的增加, 模型的准确率也随之增加, 进一步验证了Transformer结构的有效性.
| 表 5 Transformer层数量的消融实验 |
实验中又进一步研究了Transformer层中的图像块尺寸(patch size)对模型效果的影响. 结果汇总在表6中. 从实验结果可以观察到, 图像块的尺寸越小, 分割性能越好. 可以注意到, Transformer的序列长度与图像块大小成反比, 比如图像块大小为16的序列长度为64, 而图像块大小为32的序列长度仅为16. 为了更长的序列长度, 通过减小图像块的大小, 模型表现出了更强大的分割准确率, 这是因为Transformer对每个元素之间的更复杂的依赖关系进行了编码学习, 使得分割准确度更高. 参考ViT中的设置, 本文中的其他相关实验均使用16×16的默认图像块尺寸.
如图6所示, 在UW-Madison GI Tract Image Segmentation胃肠道医学图像分割数据集上进行了定性比较. 可以看出: (1)基于纯CNN的方法U-Net++、R50 UNet和R50 Att-UNet更容易对器官进行过分割或欠分割. 而基于纯Transformer的Swin-UNet的分割结果相比于其他网络模型的分割结果相对边缘粗糙. 这表明结合CNN与Transformer的模型, 如TransUNet和本文的模型对全局上下文进行特征学习和语义分割的能力更强. (2)第1行的结果表明, 与其他方法相比, 本文提出的模型预测的假阳性更少, 这表明在抑制噪声方面, 此方法比其他方法更有优势. (3)在第3行中, 正确预测了大肠和小肠的位置, 而TransUNet则出现了过分割现象, U-Net++则出现了欠分割现象. 这些对比结果表明, 本文所提出的网络模型能够进行更精细的分割各器官, 并保留详细的边缘形状信息. 这进一步证明了本文模型方法的有效性.
| 表 6 图像块尺寸及序列长度消融实验 |

|
图 6 对不同方法进行定性比较 |
5 结论与展望
本文提出将CNN与Transformer并结合多层级跳跃连接结构应用于医学图像分割. 不仅通过将图像特征作为序列来编码学习全局信息, 而且结合U形结构和多层级跳跃连接结构来融合多尺度特征, 这种设计充分利用了CNN各层次的特征信息. 本文网络模型通过ResNet-50结构对图像进行CNN提取特征, 然后利用Vision Transformer结构提取全局空间特征, 加强器官各部分内在的关联性信息提取. 在解码器中通过融合不同尺度的器官特征, 使网络模型保留更多的细节和边缘信息. 下一步的研究工作将进一步改进Transformer模块, 进一步提升医学图像分割的准确率.
| [1] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [2] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [3] |
Çiçek Ö, Abdulkadir A, Lienkamp SS, et al. 3D U-Net: Learning dense volumetric segmentation from sparse annotation. Proceedings of the 19th International Conference on Medical Image Computing and Computer-assisted Intervention. Athens: Springer, 2016. 424–432.
|
| [4] |
Xiao X, Lian S, Luo ZM, et al. Weighted Res-UNet for high-quality retina vessel segmentation. Proceedings of the 9th International Conference on Information Technology in Medicine and Education. Hangzhou: IEEE, 2019. 327–331.
|
| [5] |
Zhou ZW, Rahman Siddiquee MM, Tajbakhsh N, et al. U-Net++: A nested U-Net architecture for medical image segmentation. Proceedings of the 4th International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Granada: Springer, 2018. 3–11.
|
| [6] |
Huang HM, Lin LF, Tong RF, et al. UNet3+: A full-scale connected UNet for medical image segmentation. Proceedings of the 2020 International Conference on Acoustics, Speech and Signal Processing. Barcelona: IEEE, 2020. 1055–1059.
|
| [7] |
Schlemper J, Oktay O, Schaap M, et al. Attention gated networks: Learning to leverage salient regions in medical images. Medical Image Analysis, 2019, 53: 197-207. DOI:10.1016/j.media.2019.01.012 |
| [8] |
Zhao HS, Shi JP, Qi XJ, et al. Pyramid scene parsing network. Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2019. 6230–6239.
|
| [9] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: Curran Associates Inc., 2017. 6000–6010.
|
| [10] |
Carion N, Massa F, Synnaeve G, et al. End-to-end object detection with transformers. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 213–229.
|
| [11] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16×16 words: Transformers for image recognition at scale. Proceeding of the 9th International Conference on Learning Representations. OpenReview.net, 2021. 1–21.
|
| [12] |
Touvron H, Cord M, Douze M, et al. Training data-efficient image transformers & distillation through attention. Proceedings of the 38th International Conference on Machine Learning. PMLR, 2021. 10347–10357.
|
| [13] |
Liu Z, Lin YT, Cao Y, et al. Swin transformer: Hierarchical vision transformer using shifted windows. Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision (ICCV). Montreal: IEEE, 2021. 9992–10002.
|
| [14] |
Li XM, Chen H, Qi XJ, et al. H-DenseUNet: Hybrid densely connected UNet for liver and tumor segmentation from CT volumes. IEEE Transactions on Medical Imaging, 2018, 37(12): 2663-2674. DOI:10.1109/TMI.2018.2845918 |
| [15] |
Milletari F, Navab N, Ahmadi SA. V-Net: Fully convolutional neural networks for volumetric medical image segmentation. Proceedings of the 4th International Conference on 3D Vision (3DV). Stanford: IEEE, 2016. 565–571.
|
| [16] |
He K, Zhang X, Ren S, et al. Deep residual learning for image recognition. Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2016. 770–778.
|
| [17] |
Devlin J, Chang MW, Lee K, et al. BERT: Pre-training of deep bidirectional transformers for language understanding. Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. Minneapolis: Association for Computational Linguistics, 2019. 4171–4186.
|
| [18] |
Cao H, Wang YY, Chen J, et al. Swin-UNet: UNet-like pure transformer for medical image segmentation. arXiv:2105.05537, 2021.
|
| [19] |
Hatamizadeh A, Tang YC, Nath V, et al. UNETR: Transformers for 3D medical image segmentation. Proceedings of the IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2022. 1748–1758.
|
| [20] |
Valanarasu JMJ, Oza P, Hacihaliloglu I, et al. Medical transformer: Gated axial-attention for medical image segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-assisted Intervention. Strasbourg: Springer, 2021. 36–46.
|
| [21] |
Zhang YD, Liu HY, Hu Q. TransFuse: Fusing transformers and CNNs for medical image segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-assisted Intervention. Strasbourg: Springer, 2021. 14–24.
|
 2023, Vol. 32
2023, Vol. 32



