2. 上海浦东复旦大学张江科技研究院, 上海 200120;
3. 复旦大学附属儿科医院, 上海 201102
2. Fudan Zhangjiang Institute, Shanghai 200120, China;
3. Children’s Hospital of Fudan University, Shanghai 201102, China
近年来, 越来越多的研究致力于将人工智能技术用于改善人类的生存现状, 而医疗领域无疑是关乎人类的重要一环. 深度学习[1]的提出使得医学图像的处理、分析以及诊断技术得到了快速进步. 研究表明[2, 3], 深度学习在医学图像分析领域可以达到甚至超越专家水平. 然而开发由深度学习支持的医学图像分析系统面临一个很大的阻碍: 即训练和测试模型需要庞大的, 高质量的带标注的数据集.
当今医学图像领域缺少类似ImageNet[4]这样大规模且经过广泛验证的可靠基准数据集[5]. 而如果使用自构的数据集又面临几个困难: 一是医学图像的获取途径少, 医院的医疗影像往往私密性强, 这类影像中会包含病人的个人信息, 直接使用的话会存在隐私和伦理问题; 二是医学图像由于其专业性和复杂性, 标注工作往往需要医生的经验积累和专业知识, 并且图像的标注主观性强, 不同经验的医生可能会给出不同的结果; 三是标注工作本身需要耗费相当大的人力成本, 而医生的精力有限, 不应该将大量时间投入到这种重复而低效的劳动中.
因此本文设计并实现了一个半自动化的医学图像标注系统, 旨在解决获取高质量带标注的医学图像数据集困难的问题. 系统的核心在于通过深度主动学习和人机协作, 优化人工标注的工作流程和深度学习模型的训练流程, 以较小的人力成本训练得到一个相对高精度的深度学习模型. 模型训练完成后用户可以用其对医学图像进行标注, 进而通过AI模型获取自动标注的医学图像数据集.
1 基于深度主动学习的半自动标注方法 1.1 深度学习与主动学习简介深度学习与主动学习都是机器学习的一个子领域. 深度学习起源于对人工神经网络的研究, 实现了对数据特征的自动提取. 由于网络结构复杂、参数众多, 深度学习具有很强的学习能力, 但这也意味着深度学习需要大量的标注样本来完成相应的训练. 主动学习专注于数据集的研究, 也被称为查询学习[6]. 主动学习假设在机器学习模型的更新过程中, 同一数据集中的不同样本对当前模型的更新有不同的价值, 因此主动学习会试图选择具有最高价值的样本来构建训练集, 而医生只需对这些高价值的样本进行标注, 而无需标注所有数据, 因此可以用最小的人力成本完成标注任务.
深度学习和主动学习在机器学习领域都有重要的应用, 具体来说, 深度学习在多种任务[7, 8]中取得了优异的表现, 但是取得良好效果的前提很大一部分归因于大规模带标注数据集[9]. 因此在医学图像分析领域, 深度学习受到了样本标注成本高的限制. 相比之下, 一个有效的主动学习算法在理论上可以实现标注效率的指数级提升[10]. 传统的主动学习往往用于简单的机器学习模型, 因为这些模型的特征往往比较简单, 然而, 传统的主动学习算法难以处理像医学图像这类的高维数据. 因此, 将深度学习的提取特征的能力与主动学习降低标注成本的能力结合是解决获取医学图像高精度自动标注困难问题的一个可行方案, 也就是所谓的深度主动学习.
1.2 主动学习查询策略样本查询策略在主动学习的流程中占据重要地位, 有价值的样本通过查询策略选择出来, 因此它是影响最终任务模型效果的最关键的部分. 传统的主动学习对样本逐一进行查询, 并且每次获得样本的标注后都对模型进行更新. 这导致了在训练数据变化微小的同时任务模型被频繁的训练, 此方法在使用深度学习模型作为任务模型时不仅效率十分低下, 还容易造成模型过拟合. 因此在深度主动学习的背景下, 本文使用基于批量的查询策略(BMDAL), 在每次查询样本时, 一次选择一批新的数据.
查询批量数据的方式有很多, 大致可以分为3类, 分别是深度贝叶斯主动学习(DBAL)、基于不确定性以及多样性的查询策略和基于密度的方法. 其中基于样本不确定性的查询策略形式简单, 计算复杂度低, 而在本文所研究的医学图像标注场景下设备往往计算能力不强, 主动学习的查询过程不应耗费过多时间, 因此是一个理想的选择. 在传统的应用于浅层模型的主动学习中, 计算不确定性的方法有边缘采样或者信息熵计算等, 但是在深度学习背景下, 查询策略和当前模型参数紧密相关, 模型自身的不确定性同样会影响样本总的不确定性, 而简单的不确定性采样并不能衡量深度模型的不确定性.
为解决此问题, 本文采用了基于委员会(ensemble)的查询策略, 通过训练多个模型, 评估各个模型之间的结果差异性来衡量模型的不确定性, 以得到较于单个模型更准确的不确定性衡量指标.
1.3 基于深度主动学习的医学图像分割算法鉴于深度主动学习在医学图像分析领域的良好前景, 本文拟探究在医学图像分割任务中使用深度主动学习技术进行半自动化标注的方法.
1.3.1 方法思路本文使用的深度主动学习算法, 其基本思想是利用少量经过人工标注的样本数据集, 针对医学图像分割模型进行训练或调优, 再使用训练和优化后的模型来对大规模未被标注的医学图像进行标注, 进而解决大规模医学图像标注时人力成本过高的问题[11].
算法的主要思路是使用针对于医学图像的高价值样本的查询策略, 在用于图像分割的深度学习模型的更新过程中不断筛选对于当前模型高价值的样本, 并且将这些样本提供给医生进行手工标注, 在分割模型达到精度要求或者标注的数量达到预设的阈值后停止训练模型.
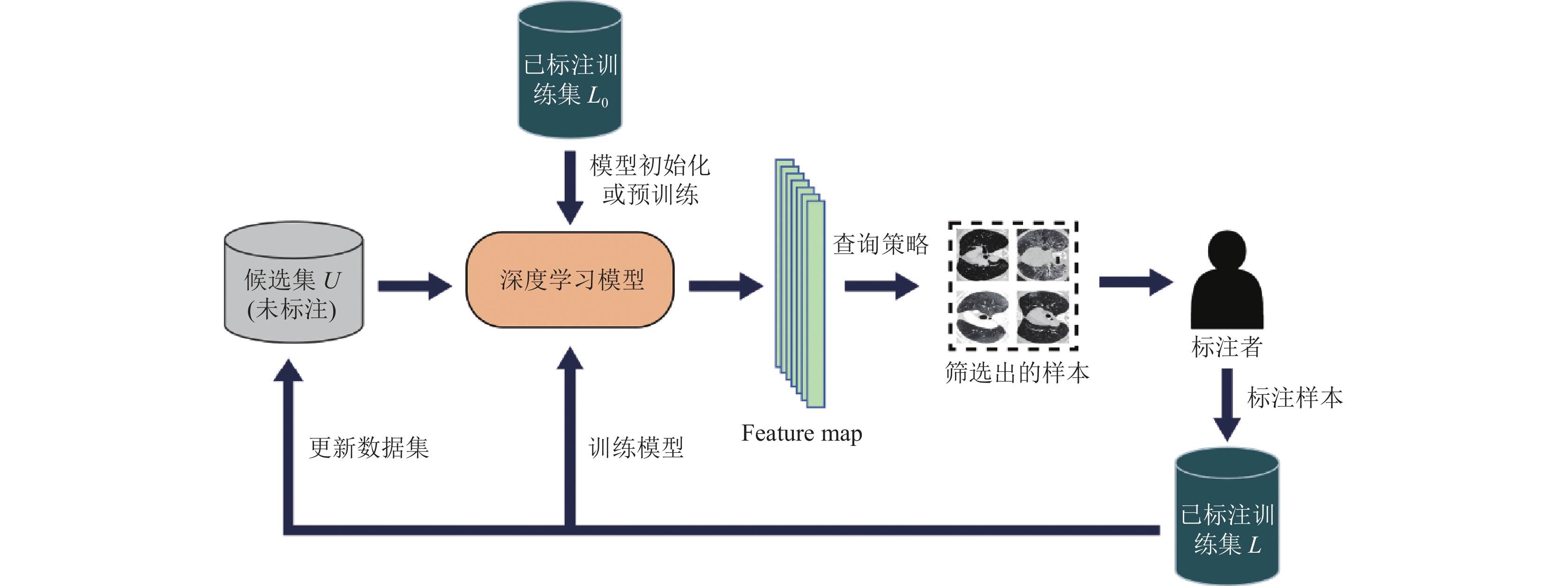
1.3.2 流程设计算法的大致流程如图1所示.
|
图 1 算法流程 |
(1)初始标注: 首先, 在所有原始无标注的医学图像集合U中手工标注少量的样本, 标注的样本组成集合L0.
(2)模型初始化: 根据具体任务选择合适的深度学习模型, 并在L0上训练此任务模型, 使得深度学习模型具有初始的特征提取能力.
(3)全样本特征提取: 将更新的未标注图像集合U中的样本送入当前的任务模型进行特征提取, 每张图片都对应其特有的feature map.
(4)高价值样本筛选: 将步骤(3)中提取到的feature map通过特定的主动学习查询策略进行筛选, 得到一批对应当前任务模型和查询策略的高价值样本.
(5)人工标注: 将步骤(4)中得到的高价值样本展示给用户, 由用户对这些样本进行手工标注, 标注后的样本送入已标注的训练集L供模型使用, 同时在无标注集合U中去除这些已经标注好的样本.
(6)模型更新: 使用所有可用的已标注样本对任务模型进行更新, 使用重新训练的方式优化当前模型.
(7)循环执行步骤(3)–(6), 直到任务模型的效果达到要求或者手工标注的样本达到预设的数量, 则结束算法的流程.
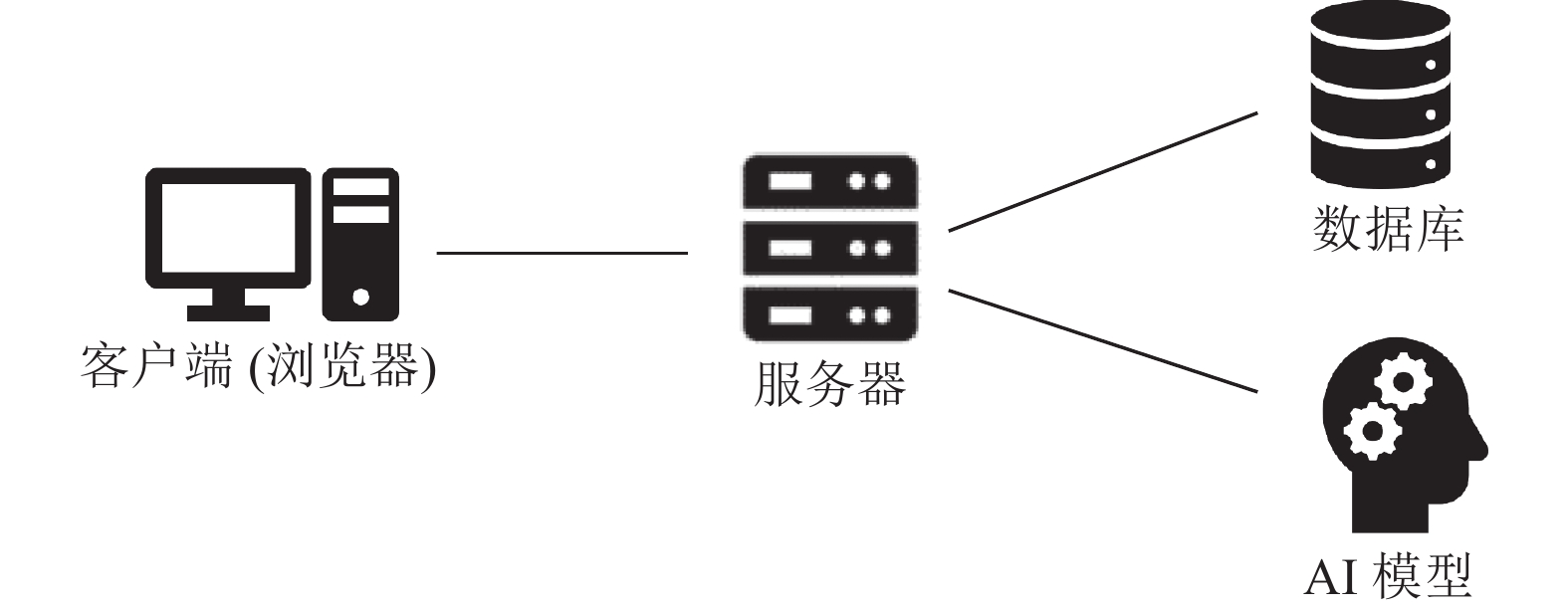
2 系统设计 2.1 架构设计系统基于Web应用构建, 整体使用B/S (浏览器/服务器)架构, 如图2所示. 客户端为Web浏览器, 用于向服务器发起Http请求. 服务器中的Web服务层能够处理客户浏览器发来的Http请求; 服务器中的应用层部分会根据Http请求调用业务逻辑上的各种方法, 并最终将结果返回给客户端浏览器. 如有必要的话, 服务器会和数据库进行数据交换, 并提供基于AI模型的半自动标注服务. B/S架构的优点在于其客户端无需安装, 只需要使用Web浏览器就可以访问系统的人机交互界面, 并且有很强的可扩展性, 系统升级时无需升级多个客户端, 只需要升级服务器即可, 且可以随时更新版本而用户无需重新下载.
|
图 2 系统架构 |
2.2 系统功能模块划分
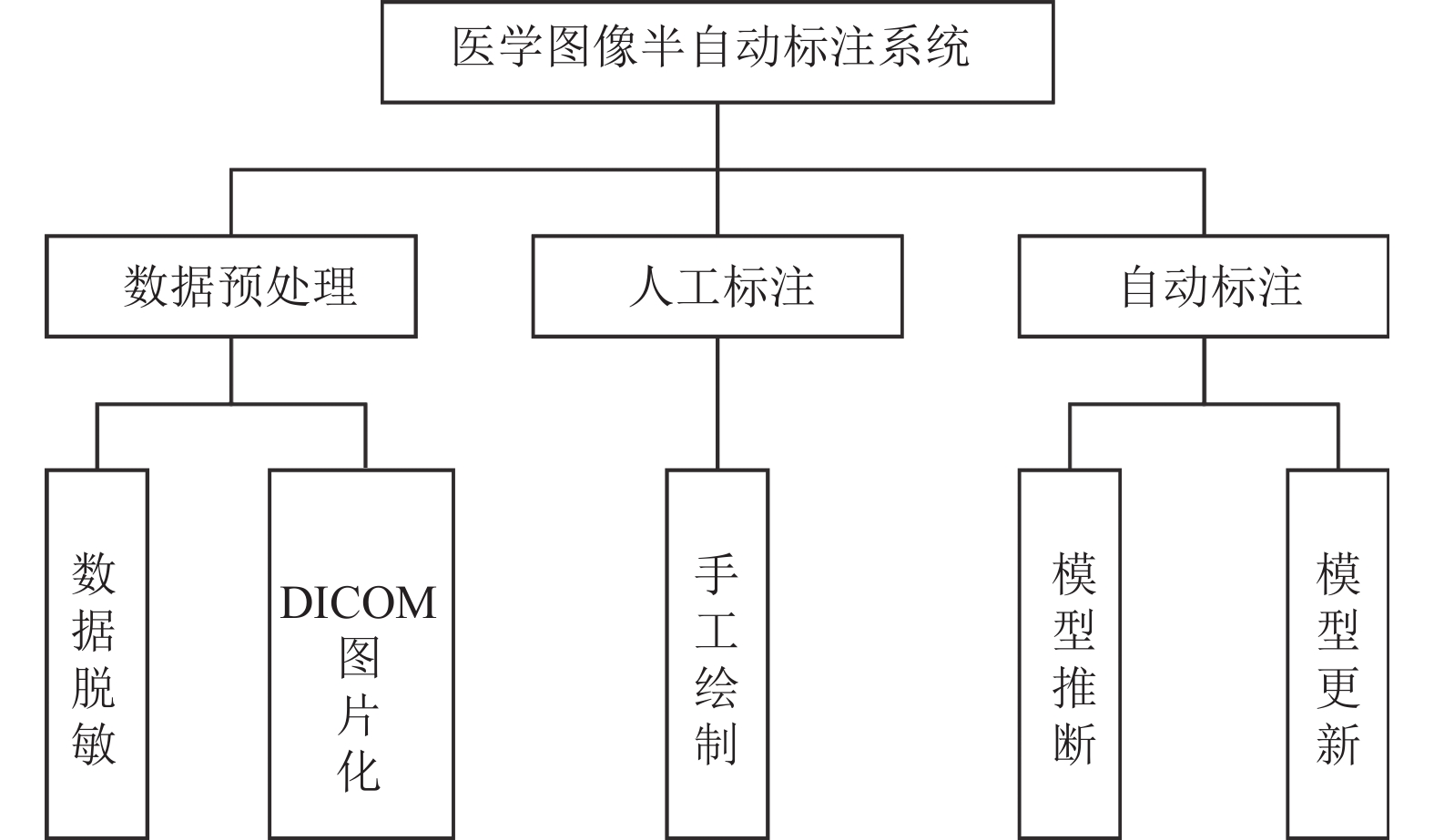
基于深度主动学习的医学图像半自动化标注系统包含数据预处理、人工标注和自动标注3部分. 数据预处理模块主要负责对输入的医学图像进行一些预处理, 便于后续系统的运作. 人工标注模块负责向用户派发标注任务, 用户对未标注的医学图像进行手工绘制标注. 自动标注模块对应系统中的深度主动学习模型, 负责对应任务模型的推断以及更新. 系统功能模块的划分如图3所示.
|
图 3 系统功能模块 |
数据预处理模块包括数据脱敏和DICOM格式图像图片化的功能. DICOM (digital imaging and communi-cations in medicine), 即医学数字成像和通信, 是医学图像和相关信息的国际标准(ISO 12052). 从医院获取的医疗影像往往是DICOM格式的, 并且包含部分病人的隐私信息. 数据脱敏主要负责将输入的原始医学图像中的隐私信息去除. DICOM图片化是将DICOM格式的文件中的影像提取并转化为jpg或者png类型的图片格式, 这样可以使得输入的医学图像以更加直观的方式展现给用户.
人工标注模块的主要功能是收集深度主动学习模型提出的标注需求, 并提供给用户, 由用户对这些未标注的样本进行手工标注绘制, 最后再对用户已经标注完成的样本进行收集, 后续提供给自动标注模块进行算法模型的更新. 此模块和系统中的深度主动学习模型联系紧密, 是主动学习算法与人进行交互的接口.
自动标注模块是系统的核心, 主要包含模型推断和模型更新两部分功能. 此模块与系统中的深度主动学习模型进行交互. 在系统的后台任务模型已经训练完成后, 模型推断功能便可以将经过系统预处理的大批医学图像输入此任务模型, 得到模型给出的自动标注结果, 实现整个数据集的标注. 模型更新功能对应系统主动学习的部分, 在每一轮主动学习给出需要标注的高价值样本, 并且这些样本经过用户的手工标注后, 系统会启动模型更新功能, 根据新的标注数据对当前的任务模型进行更新, 实现闭环反馈优化.
2.3 深度主动学习模型设计系统深度主动学习模型的设计使用了文献[12]中提出的用于医学图像分割的通用框架, 通过结合经过优化的全卷积网络(full convolutional network, FCN)任务模型和混合式主动学习价值查询策略, 有效减少了训练任务模型所需要的标注样本的数量.
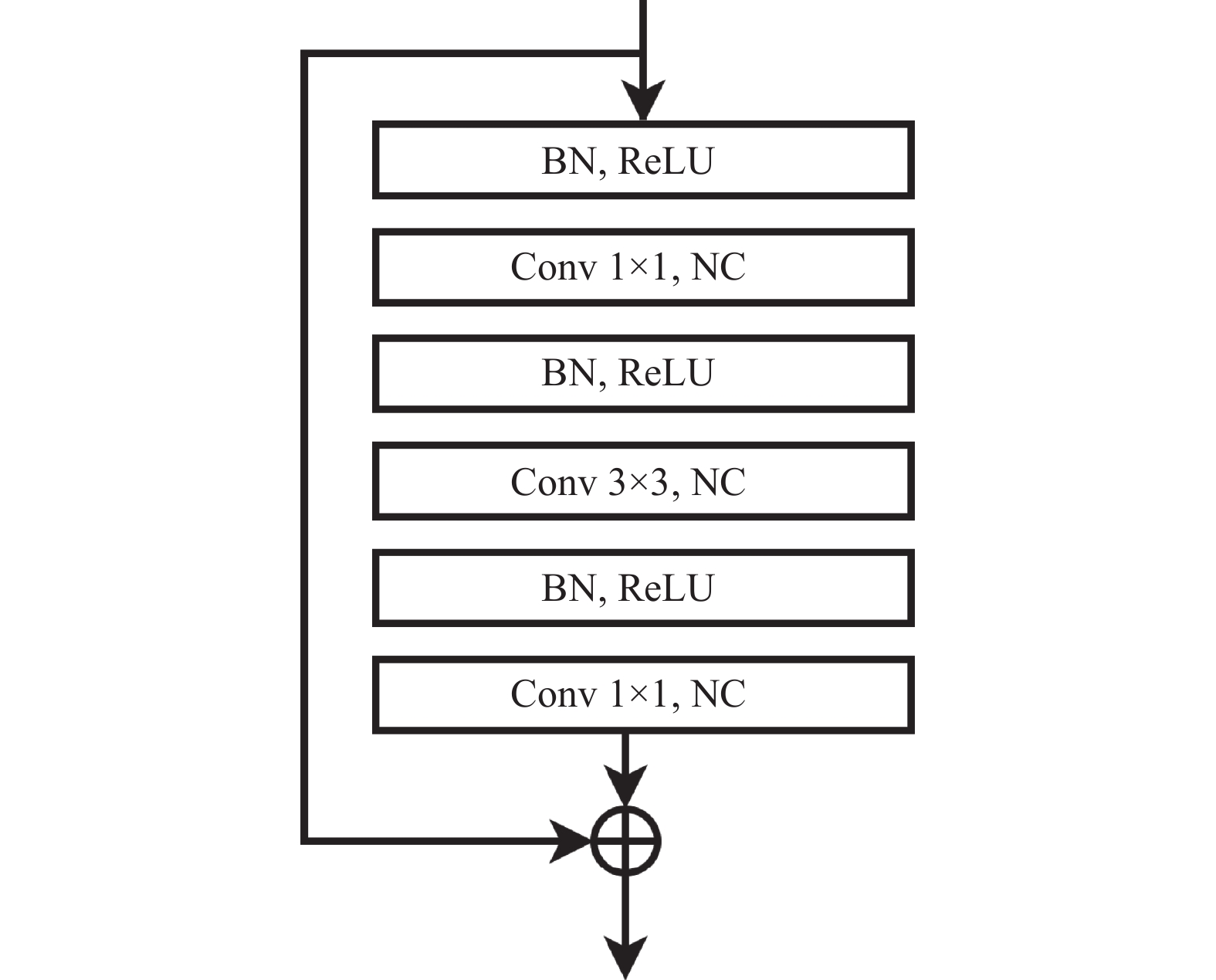
由于训练常规的FCN通常需要几小时才能达到一个较为合理的性能表现, 在主动学习每一轮更新模型的过程中这个时间过于漫长, 因此本文中所使用的任务模型FCN把原来的卷积层改成了带有批归一化的残差模块, 这样可以使用更少的训练步骤达到和原始FCN相同的精度[12]. 此外由于在主动学习的开始阶段可用的标注样本稀缺, FCN如果有过多的自由参数会导致模型难以训练, 因此引入了瓶颈(bottleneck)设计来减少参数的数量, 同时在每个残差模块的末端保持相等的特征通道数, 具体结构如图4所示. 在网络的解码部分, 逐步扩大特征图的大小, 以保证结果的稳定. 最后应用一个3×3的卷积层和一个1×1卷积层将不同尺度的经过上采样得到的特征图结合在一起, 所使用的FCN结构细节如图5.
|
图 4 Bottleneck结构 |
寻找价值量最高样本的较为直接的方法是使用不确定性抽样, 将样本价值量与当前任务模型对样本预测的不确定度相等同. 这种方法的弊端在于筛选出的高不确定样本往往会有很多相似的, 即选出的样本不能很好地代表整个样本空间的分布, 这不利于提升最终模型的分割效果和泛化性. 因此本文使用了基于不确定性和多样性的混合式查询策略. 具体来说, 本文中使用的方法既要考虑当前任务模型对所有图像的不确定性, 还有挑选更具代表性的那些图像. 整个深度主动学习的框架如图6所示.
3 系统实现为优化系统的可维护性和实现开发的高效性, 系统整体使用前后端分离的开发模式, 其中前端的开发使用Vue.js, 后端使用纯Python的后端框架Django, 数据库方面则选择了关系型数据库MySQL, 深度学习模型的搭建使用了Facebook公司推出的开源Python机器学习库PyTorch.
3.1 客户端实现客户端采用前端框架Vue加Element组件库进行开发. Vue是一套用于构建用户界面的渐进式框架. 与其他大型框架不同的是, Vue 被设计为可以自底向上逐层应用. Vue 的核心库只关注视图层, 不仅易于上手, 还便于与第三方库或既有项目整合. 另一方面, 当与现代化的工具链以及各种支持类库结合使用时, Vue 也完全能够为复杂的单页应用提供驱动. Element则提供了很多功能丰富且主题鲜明的组件供用户直接引入并使用, 而无需关注其底层实现.
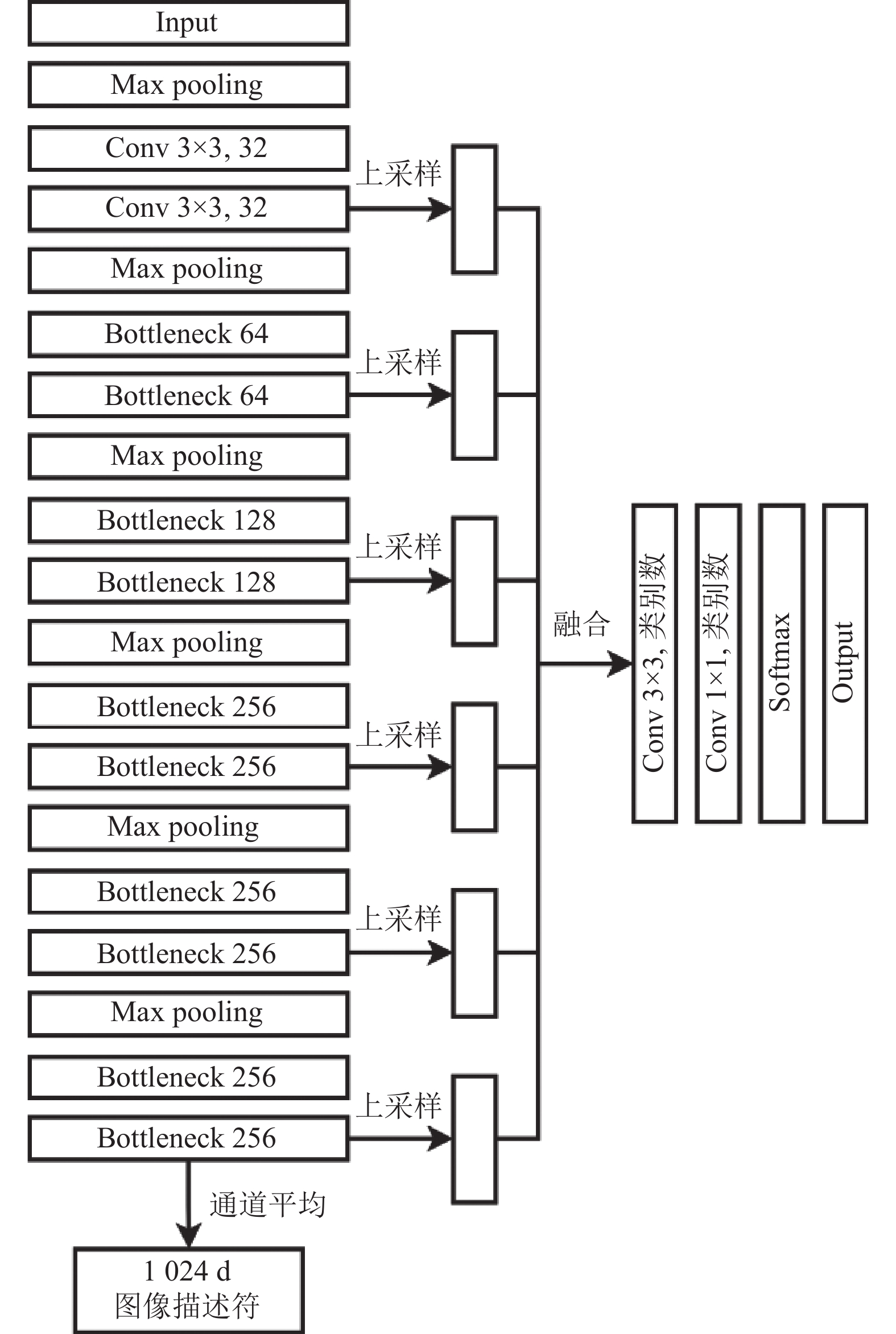
|
图 5 FCN结构 |

|
图 6 深度学习总体框架 |
由于使用单页应用程序(single page application, SPA)的设计, 实现不同组件的加载需要设置不同的路由规则, 主要包括表1中的几个路由.
前端页面通过axios向后台服务器发送Ajax请求, 进行数据的交互以及业务的处理, 并通过浏览器的localstorage暂存部分状态, 以便进行持久化的访问.
3.2 服务器实现Web服务器使用Django进行开发. Django是一个开源的Web应用框架, 由Python写成, 采用了MTV的框架模式, 即模型model, 视图view和模版template. 使用这种架构, 程序员可以方便、快捷地创建高品质、易维护、数据库驱动的应用程序. 另外, 在Django框架中, 还包含许多功能强大的第三方插件, 使得Django具有较强的可扩展性.
| 表 1 前端路由表 |
系统的页面展示交由Vue渲染, 因此不需要在后端处理template部分, 只需关心数据库交互和后台具体业务逻辑, 即model和view部分. 服务器的应用程序接口(application programming interface, API)见表2.
| 表 2 服务器API表 |
数据库使用MySQL搭建. MySQL是一个开源的关系型数据库管理系统, 其优点在于数据库体积小、速度快, 在中小型网站的开发中有着广泛应用. 其默认的InnoDB引擎对事务有良好支持和优化. 由Django通过model抽象出的数据类与底层数据库的表进行交互.
在用户完成模型提出的标注任务后, 服务器将启动模型的更新策略, 具体表现为运行特定的更新脚本, 在模型更新完成后将服务器中调用的模型权重文件替换成新的权重文件, 后续对图像的自动标注将使用优化更新后的模型参数.
3.3 深度主动学习模型实现为实现前文提到的不确定性和多样性相结合的高价值样本查询策略, 需要对样本的不确定性和在数据集中的代表性进行衡量. 比较简单的做法是计算FCN网络最后一层的Softmax输出的所有像素的预测值, 并用其计算样本的信息熵. 实际上, 使用Softmax的输出作为衡量不确定性的方式在深度学习的背景下被证明是不可靠的[13]. 虽然DL模型可以通过Softmax获得标签的概率分布, 但是由于深度模型自身的不确定性, 此结果并不能作为置信度指标. 因此本文对样本的不确定性评估使用了基于委员会的查询策略. 委员会模型的具体实现使用Bootstrapping方法[14], 这是一种评估学习模型不确定性的标准方法, 其基本思想是对整个训练集进行放回式抽样, 产生一系列训练数据的子集, 并用这些子数据集训练一组FCN. 得到这样一组FCN后将判断某样本输入各个FCN后产生的特征图的差异性, 每个样本的不确定性定义为其特征图各个像素在不同FCN上差异性的平均值, 像素差异性越大的样本其不确定性越高. 虽然各个FCN内部的方差会导致对最终方差的预估高于实际, 但是在实践中已经证明, 这种方法仍然可以对不确定性进行良好的估计[12]. 通过这种委员会方法计算的不确定性虽然引入了额外的计算成本, 但是能更好地对样本价值进行表征. 对样本的多样性评估同样利用了FCN的骨干网络部分提取出的特征图. 为了消除图像的旋转和平移的等变化, 对特征图的每个通道计算均值, 得到图像的一个高维描述符, 即图5中的图像描述符, 它是一个1 024维的向量. 输入的图像可以用
| $ sim({I_i}, {I_j}) = cos ine\_similarity(I_i^c, I_j^c) $ | (1) |
| $ f(S, {I_x}) = {\max _{{I_i} \in S}}sim({I_i}, {I_x}) $ | (2) |
由于医学图像往往各个像素之间差异不大, 本身就比较相似, 因此图像的不确定性相较于筛选出的图像的多样性往往更为关键. 本文的做法是使用二阶段的查询策略, 先在所有未标注样本中挑选K个不确定性最高的样本, 再从这些样本中选取k个相互之间最不相似的图像, 其中k<K. 最终这k张图像会由系统交给用户进行标注, 用于改善任务模型的效果.
模型搭建采用开源的Python机器学习库PyTorch. PyTorch是一个简洁且高效快速的框架, 计算图的搭建使用动态图机制, 设计符合人类思维. 模型开发完成后, 使用互联网上公开的新冠肺炎分割数据集COVID-19 CT segmentation dataset进行训练和测试. 数据集中包含来自超过40名新冠肺炎患者的100张轴向CT图像, 每个图像有3类标注, 分别是毛玻璃(ground-glass)、实变(consolidation)和胸腔积液(pleural effusion). 实验主要的评估指标是平均交并比(mean IoU)和F1分数. 平均交并比是各个类别预测掩膜和真实掩膜之间交集和并集的比值的平均值, 而F1分数则被定义为精确率和召回率的调和平均数.
4 实验与测试 4.1 任务模型与样本查询策略本文首先对系统使用的FCN医学图像分割任务模型进行了实验, 在使用完整的标注数据集的情况下, 实验结果如表3所示.
| 表 3 任务模型性能对比 |
可见本文中的FCN任务模型在全标注的数据集上能够取得更好的效果. 除此之外, 本文还对使用的二阶段主动学习查询策略进行了实验, 以验证主动学习算法是否有效降低了训练医学图像标注模型所需的标注图像数量, 查询的具体参数为K=16, k=8, 实验结果见表4.
| 表 4 查询策略测试结果 |
可见本文中主动学习算法使用基于不确定性和多样性的二阶段混合查询策略在仅标注50%的图像数量时就基本达到了全量标注的效果, 这证明了本文使用的主动学习算法可以有效降低训练模型所需的标注图像数量. 而与单一的不确定性查询策略进行比较, 二阶段的查询策略取得了更好的效果, 说明了样本多样性的选择过程是必要的, 因为它可以有效降低主动学习查询样本分布的冗余. 另外, 本文还与其他的经典查询策略在仅使用50%标注图像数量时的效果进行了对比, 实验结果如表5所示.
通过与其他经典的方法进行对比可以发现, 本文使用的FCN任务模型与基于委员会的二阶段混合查询策略结合, 在使用少量标注样本训练模型时取得了更好的效果. 并且在使用本系统时, 用户能够以较少的标注工作量达到全量标注的效果, 实现了本系统降低标注成本的目标.
| 表 5 与其他查询策略方法的对比 |
4.2 系统运行测试
在将本文中的深度学习模型与主动学习算法部署之后, 本文对系统的运行情况进行了测试. 模型的训练和推理使用单张Nvidia GTX 1080Ti GPU进行计算加速, 主要对模型进行样本查询的速度以及模型迭代更新的速度进行评估. 随着已标注图像数量的增加, 模型训练的速度会降低, 因此本文对所有训练轮次花费的时间取平均值, 最终得到系统标注模型的平均训练时长为1 547 s, 样本查询的平均时长为83 s.
通过对系统运行的评估结果进行分析可以得出, 模型进行一次迭代更新大约需要25 min, 两次样本查询大约间隔27 min. 在本文使用的数据集中, 如果用户标注一张图像用时5 min, 在假设用户可以一直提供标注的情况下, 整个标注模型完成训练总共需要约7 h. 而仅对整个数据集进行全量标注就需要8.3 h, 并且标注的工作量提高了一倍. 由此可以证明, 本文设计的系统能够使得用户所需标注图像的数量更少, 并且整个标注流程用时更短, 有效减轻了用户的标注负担.
5 结论与展望本文针对构建大规模带标注医学图像数据集时, 手工标注专业性强、需要耗费大量人力和时间的难点, 设计了通过训练深度学习模型实现AI自动标注的方法, 并在此基础上通过主动学习策略减少训练模型所需要的标注样本数量, 最终实现了一个基于深度主动学习的人机交互系统, 可以用于多种医学图像的分割标注数据集构建. 在医疗行业与人工智能技术联系日益紧密的今天, 该系统为推动医学图像分析与疾病诊断的智能化助力.
| [1] |
LeCun Y, Bengio Y, Hinton G. Deep learning. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 |
| [2] |
Greenspan H, van Ginneken B, Summers RM. Guest editorial deep learning in medical imaging: Overview and future promise of an exciting new technique. IEEE Transactions on Medical Imaging, 2016, 35(5): 1153-1159. DOI:10.1109/TMI.2016.2553401 |
| [3] |
Rajpurkar P, Irvin J, Ball RL, et al. Deep learning for chest radiograph diagnosis: A retrospective comparison of the CheXNeXt algorithm to practicing radiologists. PLoS Medicine, 2018, 15(11): e1002686. DOI:10.1371/journal.pmed.1002686 |
| [4] |
Deng J, Dong W, Socher R, et al. ImageNet: A large-scale hierarchical image database. Proceedings of 2009 IEEE Conference on Computer Vision and Pattern Recognition. Miami: IEEE, 2009. 248–255.
|
| [5] |
陈哲, 黄巍, 陈昭. 基于Web应用的医学图像半自动标注系统. 计算机应用与软件, 2019, 36(10): 1-5. DOI:10.3969/j.issn.1000-386x.2019.10.001 |
| [6] |
Settles B. Active Learning Literature Survey. Madison: University of Wisconsin-Madison, 2009.
|
| [7] |
He T, Zhang Z, Zhang H, et al. Bag of tricks for image classification with convolutional neural networks. Proceedings of 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019. 558–567.
|
| [8] |
Paoletti ME, Haut JM, Fernandez-Beltran R, et al. Capsule networks for hyperspectral image classification. IEEE Transactions on Geoscience and Remote Sensing, 2019, 57(4): 2145-2160. DOI:10.1109/TGRS.2018.2871782 |
| [9] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 |
| [10] |
Balcan MF, Beygelzimer A, Langford J. Agnostic active learning. Proceedings of the 23rd International Conference on Machine Learning. Pittsburgh: ACM, 2006. 65–72.
|
| [11] |
白雪冰, 韩志峰, 蒋龙泉, 等. 音视频数据半自动化标注方法. 微型电脑应用, 2021, 37(8): 9-13, 17. DOI:10.3969/j.issn.1007-757X.2021.08.004 |
| [12] |
Yang L, Zhang YZ, Chen JX, et al. Suggestive annotation: A deep active learning framework for biomedical image segmentation. Proceedings of the 20th International Conference on Medical Image Computing and Computer-assisted Intervention. Quebec City: Springer, 2017. 399–407.
|
| [13] |
Ren PZ, Xiao Y, Chang XJ, et al. A survey of deep active learning. ACM Computing Surveys, 2022, 54(9): 180. |
| [14] |
Efron B, Tibshirani RJ. An Introduction to the Bootstrap. New York: CRC Press, 1994.
|
| [15] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [16] |
Chen H, Qi XJ, Cheng JZ, et al. Deep contextual networks for neuronal structure segmentation. Proceedings of the 30th AAAI Conference on Artificial Intelligence. Phoenix: AAAI Press, 2016. 1167–1173.
|
| [17] |
Gal Y, Islam R, Ghahramani Z. Deep Bayesian active learning with image data. Proceedings of the 34th International Conference on Machine Learning. Sydney: PMLR, 2017. 1183–1192.
|
 2023, Vol. 32
2023, Vol. 32


