随着人工智能与目标检测应用技术的发展, 使用计算机视觉辅助检测胃部息肉成为一种趋势. 利用内窥胃镜采集的图像进行息肉的检测与识别是计算机视觉辅助的重要内容. 息肉检测过程中由于光照变化、息肉边缘不明显、视角遮挡、分辨率等问题使得准确识别困难, 因此对于计算机视觉辅助检测的准确性和稳定性有很高的需求. 根据不同的检测手段, 将计算机视觉辅助检测分为传统方法[1, 2]和基于深度学习的目标检测方法. 在传统方法中, 医生会通过仪器捕捉处理过的画面来快速判断息肉是否存在[3]. 如果画面中息肉特征不明显或有许多干扰, 传统方法则无法快速准确判断息肉. 基于深度学习的目标检测方法分为一阶段检测算法和二阶段检测算法. 一阶段检测算法代表有SSD系列[4]和YOLO系列[5]等. 二阶段检测算法代表有R-CNN系列[6]和SPPNet[7]等.
一阶段目标检测算法着重于实时检测方面进行优化. 如马燕[8]基于SSD进行改进, 在动态肠镜息肉图像实时定位算法中, 使用多种inception模块代替SSD基础特征提取网络结构中的传统卷积层来增加神经网络深度提取能力, 最终平均检测精度(mAP)达到96%; 王博等人[9]在M2det基础上进行改进, 在提出的基于M2det内窥镜息肉检测方法中, 他们通过FFMs模块融合主干网络特征, 使图像特征得到了充分利用, 以及在SFAM模块中增加SENet注意力机制来保留有效特征, 最终mAP达到了98.25%; Laddha等人[10]在针对息肉大小不一的基础上, 使用YOLOv3和YOLOv3-tiny两种不同规模大小的模型, 最终实验得到了91%和82%的准确率; Cao等人[11]在YOLOv4的基础上, 设计出一个特征提取和融合模块, 并将其与YOLOv4网络相结合, 最终在准确率方面达到了91.6%. Yu等人[12]在Scaled-YOLOv4的基础上, 设计出ITH模块, ITH模块与YOLO检测头共享权重以进行快速特征提取, 实验结果表明, 该模块对比原先提升了30%的识别速度. 二阶段目标检测算法注重于目标检测精度方面进行提升. 如Taş等人[13]基于Faster R-CNN进行改进, 首先使用ResNet-101替代VGG16, 然后使用基于卷积神经网络的超分辨率(SRCNN)方法提高结肠镜图像的分辨率, 最终提升模型的准确率; Cheng等人[14]基于Mask R-CNN进行改进. 首先提出了一种可学习的定向导数网络(LOD-Net), 计算息肉每个像素的8个定向导数, 然后用这些导数值构成息肉的候选框, 最终提高息肉分割边界预测的准确性. 不过二阶段目标检测算法在实时性方面比较差. 在实例分割领域, 任莉莉等人提出了基于U-Net网络改进的GLIA-NET网络[15], 主要特点是在双层卷积后加入交互式注意力卷积模块, 引入后检测精度提升6.7%.
这些方法在针对常规息肉检测准确率或实时性上有良好的改进. 由于胃部环境复杂, 小型息肉具有分辨率低、特征信息不足问题, 常规特征提取到的小型息肉特征不足, 导致漏检现象发生. 针对小型息肉漏检问题, 本文在YOLOv4的基础上进行了以下改进: (1)对CSPDarknet-53基础网络上进行改进, 设计出一种精简的CSPDarknet-49网络, 减少网络深度, 使得网络能够保留更多的小目标特征信息, 同时增加准确率; (2)利用息肉数据集的特点, 使用K-means++聚类算法对数据集进行聚类分析, 得到9组不同比例大小的锚框, 提升模型对小目标的检测精度; (3)在YOLOv4的骨干网络中引入卷积注意力模块, 使得网络能够在空间和通道两个维度进行特征提取, 从而学习到更深层次的特征信息, 从而提高小目标的检测能力.
1 YOLOv4网络介绍YOLOv4网络[16]结构如图1所示. YOLOv4由CSPDarknet53主干特征提取网络、SPP、PANet特征融合模块和检测头YOLO head组成. CSPDarknet53由CBM和CSP模块组成, CSP模块内部残差网络可以降低网络复杂结构, 同时对残差边的信息进行少量处理后与原先提取特征进行堆叠, 保留深层次特征同时过滤背景信息. 经过主干网络处理后得到输出特征. 经过SPP最大池化操作后, PANet模块的上采样, 卷积堆叠等操作对于不同层次的特征信息进行特征融合, 最后将特征信息传给YOLO Head进行预测判断, 从而实现目标检测.
2 YOLOv4改进相关工作前面介绍了在YOLOv4网络模型. 由于息肉检测环境复杂, 小型息肉分辨率不足和特征信息过少, YOLOv4模型在检测小型息肉时容易发生漏检现象. 本文针对上述问题, 改进出YOLOv4-polyp网络模型. 本文从锚框优化, 主干网络结构和注意力机制3方面对YOLOv4模型[16]进行改进, 改进后的YOLOv4-polyp模型如图2所示. 其中橙色部分为对比图1中YOLOv4模型改进点. 首先结合息肉数据集特点, 使用K-means++聚类算法对息肉数据集得到合适的锚选框. 其次为了提高小型息肉的检测能力, 在主干网络中引入CBAM卷积注意力模块, 使得YOLOv4-polyp模型能够从通道和空间两个注意力机制增强小型息肉特征提取能力, 从而避免发生漏检. 为了降低CBAM卷积注意力模块的增加导致检测速率的降低, 对主干网络Res模块层数进行精简, 将部分CBM替换为CBL简化模型复杂度. 在保证准确率的同时降低模型复杂度, 从而提升模型的检测速率.
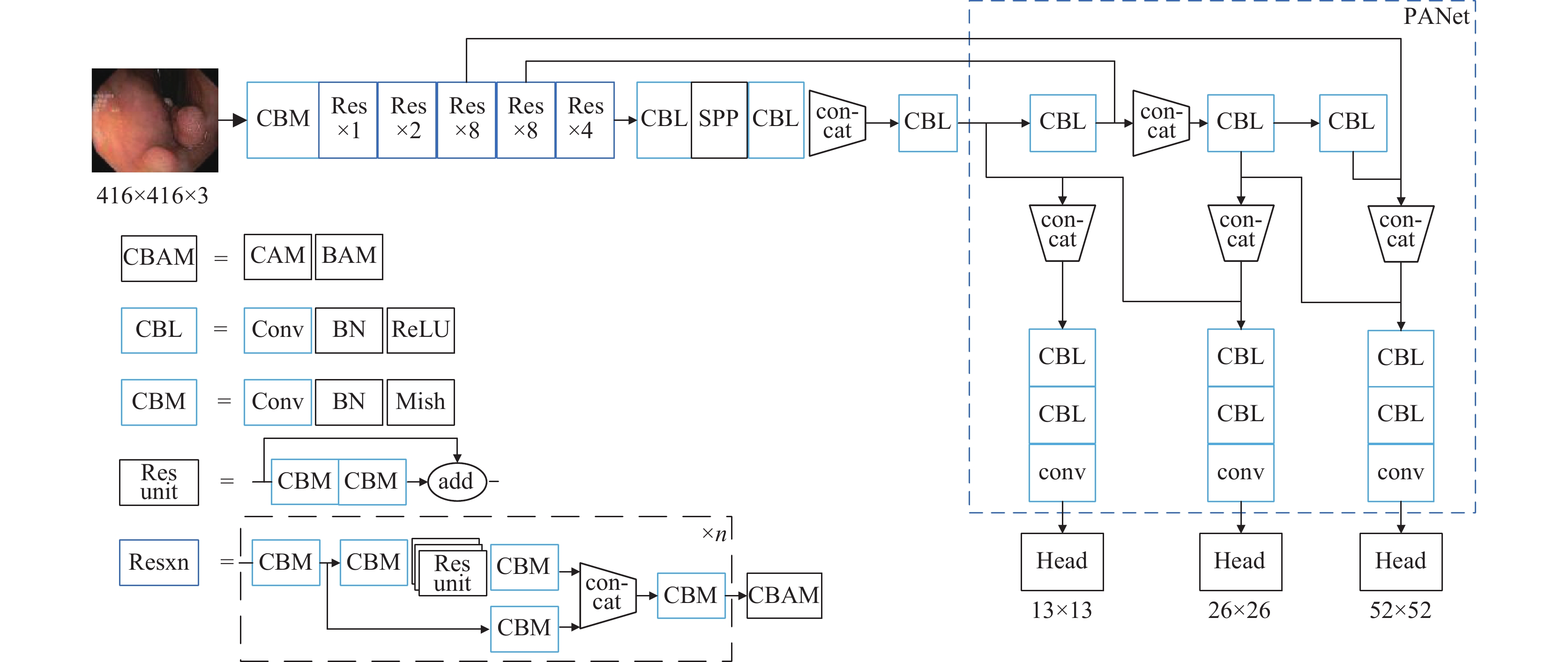
|
图 1 YOLOv4网络结构 |
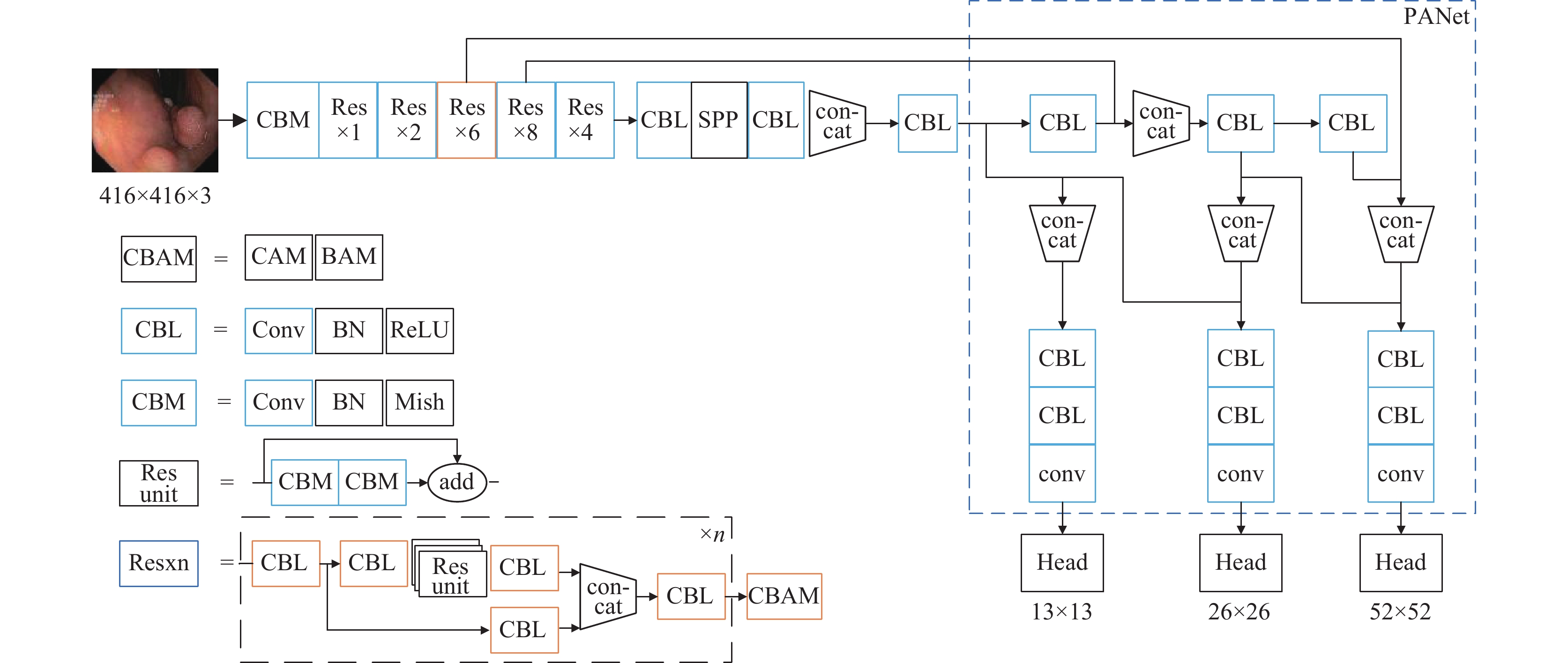
|
图 2 YOLOv4-polyp网络结构 |
YOLOv4-polyp网络结构处理流程如图2所示, 首先网络输入图片大小为416×416×3. 该网络主要由CBAM, CBL, CBM, Res unit和Res组成, CBAM卷积注意力模块由CAM和BAM组成, CBL代表卷积层, 正则化层和LeakyReLU激活函数组成, CBM与CBL不同的是激活函数使用了Mish激活函数. Res层是由残差网络, 卷积层和CBAM模块组成. Res层在经过特定次数卷积后, 会在尾部经过一次CBAM模块, 即经过Res层处理后的特征图会依次与通道注意图和空间注意图进行特征相乘, 抑制无关背景信息干扰, 提升模型的自适应特征提取能力, 最终提升胃息肉检测精度. 由于降低了模型深度以及加入了CBAM卷积注意力机制, 在经过多次卷积操作后依旧保留小型目标的特征信息, 避免了深层次卷积网络在卷积过程中损失过多小型息肉特征信息, 从而加强网络的小型息肉特征提取能力.
2.1 CSPDarknet-49网络YOLOv4通过CSPDarknet-53网络结构提取目标特征, 输出3种特征图进行多尺度检测. 3种特征图的大小分别为13×13, 26×26, 52×52. 其中52×52的尺度用于小型目标检测. 由于模型深度问题, 小型息肉在特征提取过程中会丢失部分特征信息, 最终导致检测效果不佳. 为了提高YOLOv4小型息肉检测能力, 通过实验得到一种简化的CSPDarknet-49网络结构, 在能够提高小型息肉检测能力情况下保持检测精度. 如表1所示, CSPDarknet-49网络在CSPDarknet-53网络基础上, 缩减CSPnet层数数量达到降低模型深度效果, 同时将部分Mish激活函数替换成LeakyReLU激活函数, 提升模型对小型目标检测精度并提升检测速度. 尺寸为416×416的图像在经过CSPDarknet-49网络处理后, 最终得到52×52, 26×26, 13×13三种不同尺度的特征图.
| 表 1 CSPDarknet-49网络结构 |
2.2 K-means++与锚框优化
在实时目标检测网络中, 多数目标检测算法是根据实验来预设锚框尺寸和宽高比, 如SSD, YOLO系列模型. 但根据实验得到的锚框尺寸, 只适用于常规情况下的目标尺寸. 息肉数据集与常规数据集不同, 大多真实框与预设的锚框差距过大, 从而容易发生错检, 漏检现象. 因此本文采用K-means++[17]聚类算法对数据集生成合适的锚框, 从而提升检测精度.
传统的K-means++聚类算法在计算每个样本与聚类中心的最短距离是指欧式距离, 然而这种距离计算方式不利于区分大型边框与真实框和小型边框与真实框的误差, 因此在计算距离方面替换成利用预测的边框与真实框的交并比IOU (intersection over union)来解决误差问题, 如式(1)所示:
| $ Distance(box, cluster) = 1 - IOU(box, cluster) $ | (1) |
其中, box表示真实框的集合, cluster表示各聚类中心集合, IOU(box, cluster)表示真实框与聚类中心交集与并集的比例. IOU值越高代表真实框与聚类中心越接近, 即两者相关度越高. 假设聚类框与box具有相同坐标系, 则计算交集如式(2)所示:
| $ intersection = {\rm{min}}(cla, boxa) \times {\rm{min}}(clb, boxb) $ | (2) |
聚类框与box框面积计算方式为:
| $ area1 = cla \times clb $ | (3) |
| $ area1 = cla \times clb $ | (4) |
| $ area2 = boxa \times boxb $ | (5) |
因此IOU计算公式如式(5)所示:
| $ IOU = \frac{{intersection}}{{area1 + area2 - intersection}} $ | (6) |
利用K-means++聚类算法对数据集进行聚类分析, 最终得到9个不同比例的锚框参数, 将这些锚框参数按照从小到大分成3组, 不同尺度的使用对应的锚框来进行预测, 最终结果如表2所示.
| 表 2 使用K-means++聚类分析优化的锚框尺寸 |
2.3 卷积注意力模块机制
针对解决息肉数据集中息肉大多偏小、边缘不明显导致漏检问题, 本文在YOLOv4网络模型的主干网络中引入CBAM注意力模块. CBAM注意力模块[18]是一种用于前馈卷积神经网络的简单而有效的注意力模块. 与SENet (squeeze-and-excitation networks)[19]注意力模块不同在于, SENet模块仅在通道上进行特征提取, 而CBAM卷积注意力模块结合通道和空间两个方向进行特征提取, 使得模型能更有效提取息肉边缘特征, 抑制背景信息干扰并丰富高层次特征信息, 从而减少胃息肉漏检现象.
如图3所示, 设输入特征F大小为H×W×C, 在通道注意力模块(CAM)中, 输入特征F分别进行最大池化操作和平均池化操作得到两个大小为1×1×C特征图. 接着两个特征图分别经过共享MLP层卷积处理后得到两个大小为1×1×C特征图. MLP层包含2层卷积层和一个ReLU激活函数, 第1个卷积层的输出通道数为C/r, 其中r为缩减系数, 第2个卷积层的输出通道数为C. 经过MLP层卷积处理后的两个特征层进行按位相加操作后, 经过Sigmoid激活函数最终得到权重系数Mc. Mc与输入特征F进行按位相乘后得到通道注意力特征. 在空间注意力模块中, 首先对通道注意力特征分别进行最大池化和平均池化操作后得到两个大小为H×W×1的特征图, 并对这两个特征图按通道进行拼接后得到大小为H×W×2的特征图. 然后经过大小为7×7卷积层进行卷积操作后, 再经过Sigmoid激活函数后得到权重系数Ms. Ms与通道注意力特征图进行按位相乘操作得到输出特征F'.
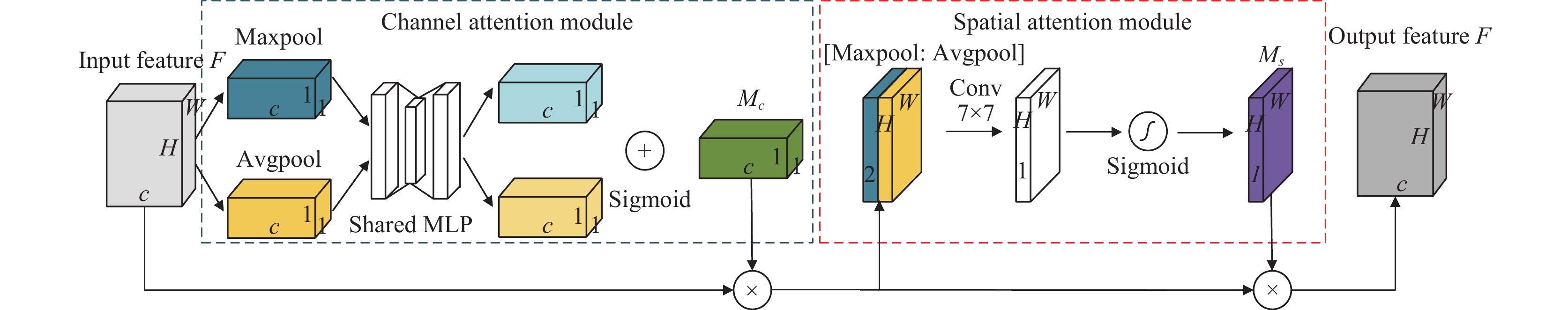
|
图 3 CBAM模块结构 |
3 实验和结果分析
本文的实验环境为OS: Ubuntu 12.04; CPU: Xeon (R) CPU E5-2678 v3; RAM: 16 GB; GPU: NVIDIA GeForce RTX 2080 Ti, GPU加速软件CUDA 11.1, 使用PyTorch 1.8版本作为深度学习框架.
3.1 数据集和训练设置本次实验使用两个数据集, 分别是Hyper-Kvasir[20]和CVC-ClincDB[21]数据集. Hyper-Kvasir数据集是一个肠胃道图像和视频数据集, 总共包含了11万张图像和374个视频. 这些数据是在挪威的Baerum医院进行的胃镜和结肠镜检查期间收集的, 其中部分图像由经验丰富的肠胃内镜医师标记. 本次实验将采用1000张带“polyp”标签的胃息肉图像做实验. 该数据集息肉图片尺寸较大, 通常在622×530像素之间. 息肉目标尺寸差别较大且不均匀, 其中多为中等大小目标. 小型息肉的图片数量偏少.
数据集中部分息肉标注丢失导致部分数据无法使用, 对数据集进行数据清洗后得到994张标注准确的图片用于数据集训练. 随机抽取636张作为训练集, 剩下的200张图片和158张图片分别作为测试集和验证集. 训练验证集和测试集的比例为8:2.
CVC-ClinicDB数据集是一个开源的数据集, 包含来自 29 个结肠镜序列的 612 张静态图像. 每张图片都带有手动注释息肉位置, 可用于医学目标图像检测. 该数据集图片尺寸较小, 通常在388×288像素之间, 相比Hyper-Kvasir数据集每张图片带有更多的息肉目标.
对数据集进行数据清洗后得到610张标注准确的图片用于训练. 随机抽取390张图片作为训练集, 剩下的122张图像和98张图像分别作为测试集和验证集. 训练验证集和测试集的比例为8:2.
3.2 模型训练与参数设置实验采用PyTorch深度学习框架搭建网络. 在训练图像送入模型前, 需要将图像缩放至416×416像素大小. 在训练时采用迁移学习方法, 使用在Imagenet数据集中训练好的CSPDarknet-53预训练权重. 因为YOLOv4-polyp模型更改了CSPDarknet-53主干网络结构, 在导入前需要手动调整预训练权重参数以便预训练权重导入. 由于Hyper-Kvasir数据集和CVC-ClincDB数据集属于小型数据集, 在数据增广上采用常规的旋转, 缩放, 对称等常见数据增强操作. 同时图像预处理采用了Mosaic数据增强, 即随机抽取4张图像, 进行随机裁剪缩放后在拼接到一张图像上. 这样不仅对于单次训练能够增加模型训练量, 在随机裁剪过程中也会随机出现小型目标, 进而增强模型的小型息肉特征提取能力. 训练参数设置如表3所示. 训练至模型停止收敛时结束训练.
3.3 评价指标实验采用准确率(precision, P), 召回率(recall, R), 平均AP值(mAP)和F值作为评价评价指标. 在目标检测中, 根据交并比IOU来判断目标是否被成功检测到. 对于数据集中的某类目标, 假设阈值为α, 当预测框和真实框的IOU大于α时, 表示模型正确预测, 相反则表示模型错误预测. 混淆矩阵如表4所示.
| 表 3 模型训练参数 |
| 表 4 混淆矩阵定义 |
在表4中, TP表示正确预测的阳性样本数, FP表示错误预测的阴性样本数, FN表示错误预测的阳性样本数, TN表示正确预测的阴性样本数. 准确率和召回率的计算公式如下:
| $ P = \frac{{TP}}{{TP + FP}} $ | (7) |
| $ {R} {{ = }}\frac{{{{TP}}}}{{{{TP + FN}}}} $ | (8) |
AP值通常作为目标检测性能的评价指标. AP值是P-R曲线下的面积, 其中召回率取X轴, 精度取Y轴, N表示类别的个数. AP代表特定类别中模型的准确性. mAP表示所有类别的平均精度, 并可测量所有类别中网络模型的性能. mAP50表示预测框和真实框的IOU大于0.5. mAP的计算公式如下:
| $ {{m}}AP = \frac{1}{N}\sum\limits_{i = 1}^N {\int_0^1 {PdR} } $ | (9) |
Precision和Recall的指标大多数时候是互相排斥的, 为了能够综合考虑这两个指标, 常见的处理方法是使用F值指标. F值的计算公式如下:
| $ {{F = }}\frac{{({a^2} + 1)P \times R}}{{{a^2}(P + R)}} $ | (10) |
当a=1时, F1的计算公式为:
| $ {{F}}1 = \frac{{2PR}}{{P + R}} $ | (11) |
为了验证YOLOv4-polyp算法的合理性, 本文将与现有的目标检测模型SSD, RetinaNet, YOLOv3, 及YOLOv4分别在Hyper-Kvasir数据集和CVC-ClincDB数据集进行实验对比, 通过对比数据集中的mAP指标, F值和平均检测速率(time)来进行对比, 由表5可见, YOLOv4-polyp与原YOLOv4相比, 平均检测速率增加了1 ms, 但是mAP提高了5.21%, 在所有模型中YOLOv4-polyp的检测精度是最高的. 由表6可见, YOLOv4-polyp与原YOLOv4相比, 平均检测速率增加1 ms, 但是mAP提高了2.05%, 在所有模型中YOLOv4-polyp依然是所有模型中检测精度最高的. 结合表5、表6可知, YOLOv4相比较于YOLOv3, 平均检测速度增加了2 ms, 但是在检测精度上均优于YOLOv3. SSD和RetinaNet在检测精度和平均检测速率均表现较差, SSD由于先验框固定大小, 导致在检测小息肉方面会存在漏检现象, 导致检测精度下降. RetinaNet在检测精度方面优于SSD, 但是平均检测速率远不如SSD, 无法满足实时检测需求, 因此YOLOv4-polyp在保证平均检测速率相较于YOLOv4几乎不变的情况下, 在检测精度达到了更好的效果, 最终检测性能更好.
| 表 5 不同模型在Hyper-Kvasir数据集下的表现 |
| 表 6 不同模型在CVC-ClincDB数据集下的表现 |
图4为在Hyper-Kvasir数据集中针对常规和小型息肉检测试验对比, 图4(a)为YOLOv4算法检测结果图像, 图4(b)为YOLOv4-polyp检测结果图像.
可以看出, 当目标尺寸较小、特征不明显时, YOLOv4模型难以检测出目标位置, 从而发生漏检现象. YOLOv4-polyp通过改进模型深度, 引入CBAM模块来一定程度上缓解模型过深导致小型目标特征信息丢失, 提高模型特征提取能力, 从而可以提升模型在小型息肉和常规息肉检测能力.
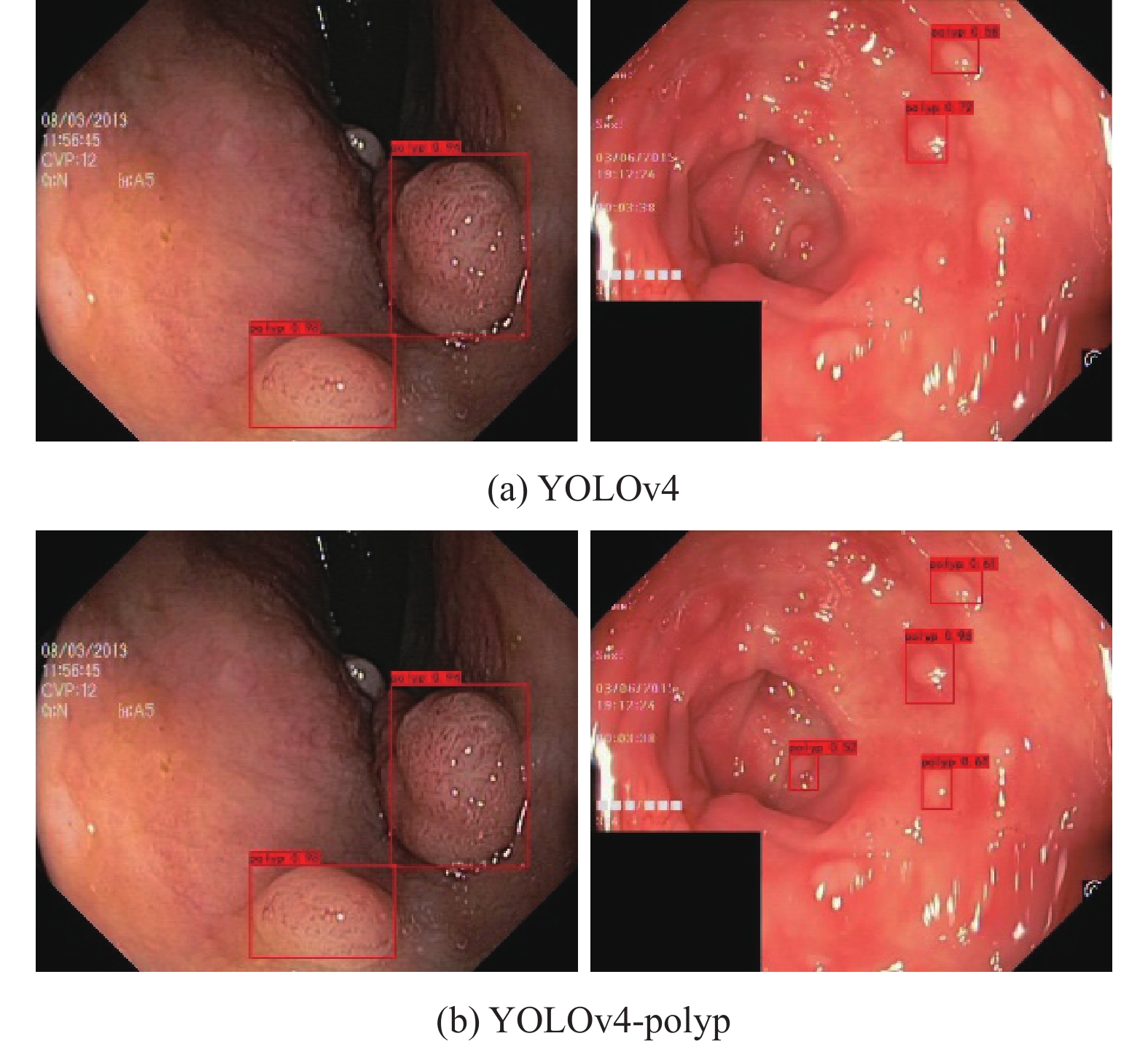
|
图 4 不同模型在Hyper-Kvasir数据集下检测对比 |
图5为在CVC-ClincDB数据集中小型息肉检测试验对比, 图5(a)为YOLOv4算法检测结果图像, 图5(b)为YOLOv4-polyp检测结果图像. 可以看出YOLOv4在针对形状缺失, 轮廓不完整的情况下容易丢失目标, 容易发生漏检现象. YOLOv4-polyp通过引入CBAM注意力机制, 提升模型的特征提取能力, 可以准确检测在轮廓缺失或形状不规则下的小型息肉.
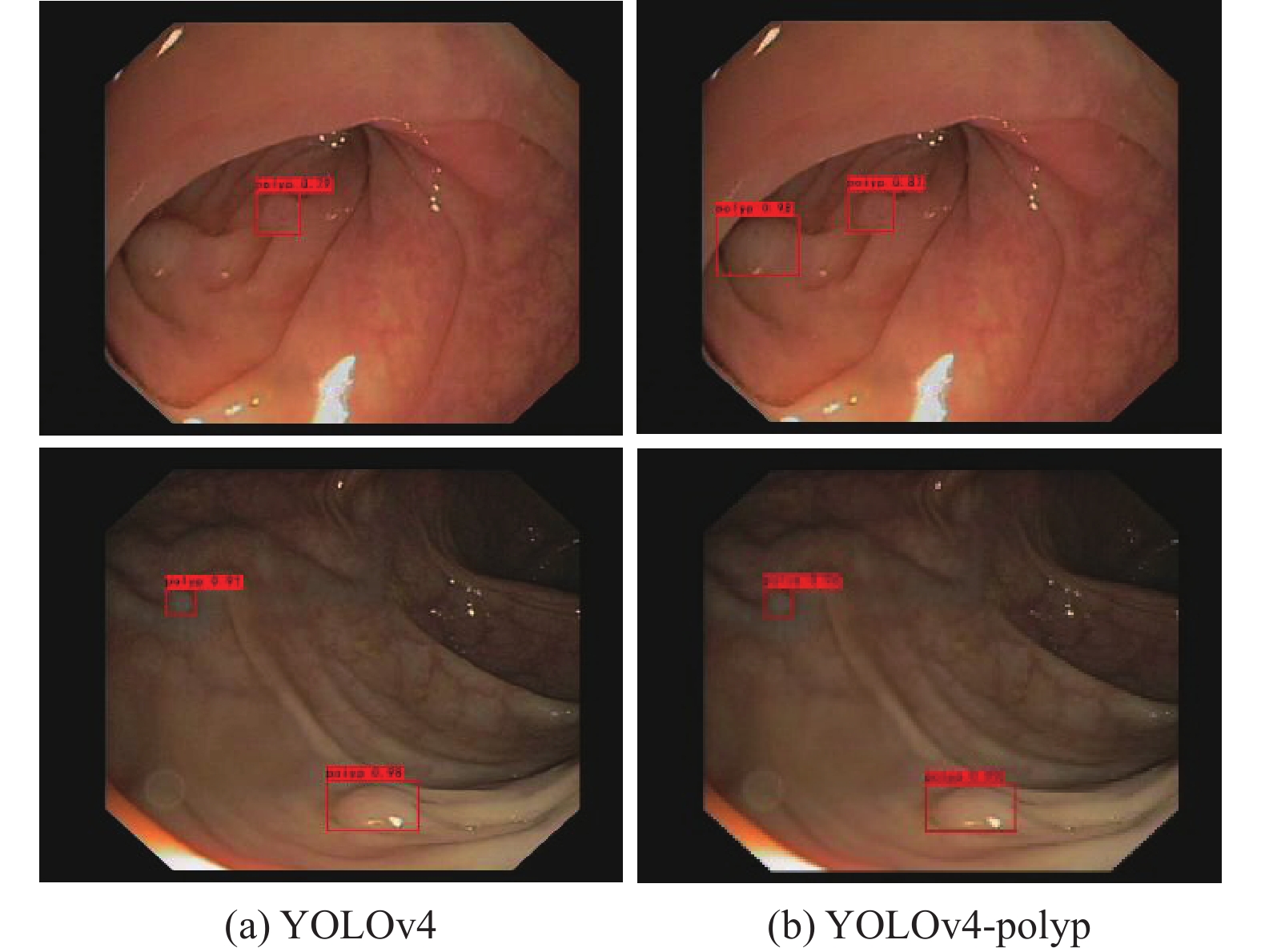
|
图 5 不同模型在CVC-ClincDB数据集下检测对比 |
3.5 消融实验
表7、表8记录了不同数据集在不同改进下算法的预测率, 召回率, mAP和平均检测时间. 对YOLOv4使用CSPDarknet-49, CBAM模块进行优化. 从表7得知, YOLOv4模型使用改进的CSPDarknet-49在准确度上分别提升了1.3%, 平均检测时间减少2 ms. 这是因为减少了CSPDarknet的卷积层数可以缓解小目标在多次卷积后边缘信息的丢失, 同时降低了网络的参数量和训练量, 加快模型的收敛速度, 并提升模型的准确精度. 对比表7, 表8可知, 在YOLOv4模型嵌入CBAM模块可以使准确率分别提升4.48%和1.11%. 这是因为YOLOv4加入了注意力模块后, 可以更丰富的提取特征层的特征, 增强特征融合并提高检测精度. 综合上述改进点后, 相比于原YOLOv4模型, 本文提出的YOLOv4-polyp模型在保持检测速度的同时分别提高了5.21%和2.05%, 说明了改进的YOLOv4-polyp模型的有效性.
| 表 7 在Hyper-Kvasir数据集下的消融实验 |
| 表 8 在CVC-ClincDB数据集下的消融实验 |
4 结论与展望
针对息肉实时检测在面对小型息肉检测困难、检测精度低等问题, 本文对此进行了研究, 综述了目前息肉检测的方法, 提出了YOLOv4-polyp胃息肉检测算法, 并将其应用到息肉检测中. 本文算法使用YOLOv4作为基础结构, 在主干网络上使用CSPDarknet-49作为特征提取网络, 在提高检测速度的同时降低网络模型参数; 结合了CBAM卷积注意力模块, 使网络可以在空间和通道两方面更加注意目标特征信息; 根据息肉数据集特点, 使用K-means++聚类算法对息肉数据集进行聚类分析得到合适的锚框, 使网络不仅适合检测常规大小胃息肉, 也适合检测小型胃息肉. 相对原YOLOv4模型, 在保持检测速率的同时提高了对小型息肉的准确率. 在之后的工作中, 将进一步改善检测效果, 优化网络模型检测速度, 在保证精度的同时提高检测速度.
| [1] |
Bernal J, Tajkbaksh N, Sánchez FJ, et al. Comparative validation of polyp detection methods in video colonoscopy: Results from the MICCAI 2015 endoscopic vision challenge. IEEE Transactions on Medical Imaging, 2017, 36(6): 1231-1249. DOI:10.1109/TMI.2017.2664042 |
| [2] |
Felzenszwalb PF, Girshick RB, Mcallester D, et al. Object detection with discriminatively trained part-based models. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2010, 32(9): 1627-1645. DOI:10.1109/TPAMI.2009.167 |
| [3] |
van der Post RS, van Dieren J, Grelack A, et al. Outcomes of screening gastroscopy in first-degree relatives of patients fulfilling hereditary diffuse gastric cancer criteria. Gastrointestinal Endoscopy, 2018, 87(2): 397-404.e2. DOI:10.1016/j.gie.2017.04.016 |
| [4] |
Liu W, Anguelov D, Erhan D, et al. SSD: Single shot multibox detector. Proceedings of the 14th European Conference on Computer Vision. Amsterdam: Springer, 2016. 21–37.
|
| [5] |
Redmon J, Divvala S, Girshick R, et al. You only look once: Unified, real-time object detection. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 779–788.
|
| [6] |
Girshick R, Donahue J, Darrell T, et al. Rich feature hierarchies for accurate object detection and semantic segmentation. Proceedings of 2014 IEEE Conference on Computer Vision and Pattern Recognition. Columbus: IEEE, 2014. 580–587.
|
| [7] |
He KM, Zhang XY, Ren SQ, et al. Spatial pyramid pooling in deep convolutional networks for visual recognition. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015, 37(9): 1904-1916. DOI:10.1109/TPAMI.2015.2389824 |
| [8] |
马燕. 动态肠镜息肉图像实时定位算法研究与实现[硕士学位论文]. 郑州: 郑州大学, 2020.
|
| [9] |
王博, 张丽媛, 师为礼, 等. 改进的M2det内窥镜息肉检测方法. 计算机工程与应用, 2022, 58(2): 193-200. DOI:10.3778/j.issn.1002-8331.2008-0084 |
| [10] |
Laddha M, Jindal S, Wojciechowski J. Gastric polyp detection using deep convolutional neural network. Proceedings of the 2019 4th International Conference on Biomedical Imaging, Signal Processing. Nagoya: ACM, 2019. 55–59.
|
| [11] |
Cao CT, Wang RL, Yu Y, et al. Gastric polyp detection in gastroscopic images using deep neural network. PLoS One, 2021, 16(4): e0250632. DOI:10.1371/journal.pone.0250632 |
| [12] |
Yu T, Lin N, Zhang X, et al. An end-to-end tracking method for polyp detectors in colonoscopy videos. Artificial Intelligence in Medicine, 2022, 131: 102363. DOI:10.1016/j.artmed.2022.102363 |
| [13] |
Taş M, Yılmaz B. Super resolution convolutional neural network based pre-processing for automatic polyp detection in colonoscopy images. Computers & Electrical Engineering, 2021, 90: 106959. |
| [14] |
Cheng MJ, Kong ZS, Song GL, et al. Learnable oriented-derivative network for polyp segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer-Assisted Intervention. Strasbourg: Springer, 2021, 720-730. |
| [15] |
任莉莉, 边璇, 王光磊, 等. GLIA-NET: 基于深度学习的息肉分割网络. 计算机工程, 2022, 1-12. DOI:10.19678/j.issn.1000-3428.0062860 |
| [16] |
Bochkovskiy A, Wang CY, Liao HYM. YOLOv4: Optimal speed and accuracy of object detection. arXiv:2004.10934, 2020.
|
| [17] |
Arthur D, Vassilvitskii S. K-means++: The advantages of careful seeding. Proceedings of the 18th Annual ACM-SIAM Symposium on Discrete Algorithms. New Orleans: Society for Industrial and Applied Mathematics, 2007. 1027–1035.
|
| [18] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 3–19.
|
| [19] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [20] |
Borgli H, Thambawita V, Smedsrud PH, et al. HyperKvasir, a comprehensive multi-class image and video dataset for gastrointestinal endoscopy. Scientific Data, 2020, 7(1): 283. DOI:10.1038/s41597-020-00622-y |
| [21] |
Bernal J, Sánchez FJ, Fernández-Esparrach G, et al. WM-DOVA maps for accurate polyp highlighting in colonoscopy: Validation vs. saliency maps from physicians. Computerized Medical Imaging and Graphics, 2015, 43: 99-111. DOI:10.1016/j.compmedimag.2015.02.007 |
 2023, Vol. 32
2023, Vol. 32


