2. 中北大学 软件学院, 太原 030051
2. School of Software, North University of China, Taiyuan 030051, China
根据世界卫生组织2019年10月发布的《世界展望报告》, 全球有4.18亿人患有糖尿病视网膜病变、青光眼、年龄相关性黄斑病变或者其他可能导致失明的眼部疾病. 眼病患者往往不知道无症状病情的恶化, 因此早期筛查和治疗眼病尤为重要.
随着近几年患病人数的增多, 医生的工作负担加重, 繁重的阅片任务导致医生无法给予患者及时的反馈, 从而错过最佳治疗时机, 同时由于病灶在眼底图像中占比较小, 医生极易出现误诊、漏诊的情况[1]. 因此亟需开发计算机辅助系统来提高医生的筛查率, 从而降低患者的风险.
近年来, 以深度学习方法为基础的系统在DR疾病检测中取得显著成果[2], 尤其是卷积神经网络及其相关算法的改进[3]. 但传统的DR筛查系统通常首先识别感兴趣的区域进行特征提取, 然后再使用提取的特征进行分类[4]. 但这种深度学习系统在DR筛查中缺乏可解释性, 这导致了语义分割的研究, 该研究将像素分为各种视网膜特征, 即视盘、微动脉瘤、出血、软硬渗出物等[5].
用于DR病灶分割的方法主要有两种: 第1种是编码器-解码器架构, 该类架构中最具代表的网络是全卷积网络[6]、U-Net[7]等; 第2种是基于空洞卷积的模型, 该类架构中最具代表性的是DeepLab[8]系列模型.
本文用滑动窗口切割的方法, 将大尺寸的图像切割成小尺寸的子图, 并将不存在病灶的子图从子图数据集中剔除, 只剩下含有病灶的子图, 以保证每张图片的学习都能学到至少一种病灶. 多任务学习可以多个任务同时学习, 在一定程度上可以缓解模型的过拟合, 提高模型的泛化能力. 结合以上思路, 本文提出了一种利用UNet++多任务学习属性结合滑动窗口切割处理数据集的方法. 分别在两个公开数据集DDR和IDRiD进行实验验证.
1 病灶图像分割 1.1 图片预处理通过观察数据集中的图像可知, 眼底图像的边界处有面积较大的黑色部分, 其中并不含有任何眼部图像相关的信息, 所以首先需要做的就是将这部分冗余信息去除. 在这里, 选用Otsu阈值算法. Otsu阈值算法会根据图像的灰度值, 计算出一个阈值, 将图像分为前景和背景, 然后获取前景边界的坐标, 在原图上进行背景的切割. 效果如图1所示, 白色部分为前景, 黑色部分为背景, 最后一张为将部分背景切除后的效果.

|
图 1 Otsu算法预处理图片 |
1.2 子图切割
由于医疗图像自身的高分辨率特性, 以及病灶在整张图像中占比较小, 对实验环境有较高要求, 同时注意力网络聚集到大病灶导致局部细小的特征会被忽略[9]; 通用的做法是对图像做出等比例的缩放, 但这个过程会导致小病灶的模糊甚至丢失, 故使用滑动窗口切割图像的方法, 将图像切割成一个个子图. 由于小病灶的存在, 所以切割以后, 大量的子图中不含有任意一种病灶, 我们需要将这部分子图剔除, 以提高含病灶子图的占比. 子图切割如图2所示.
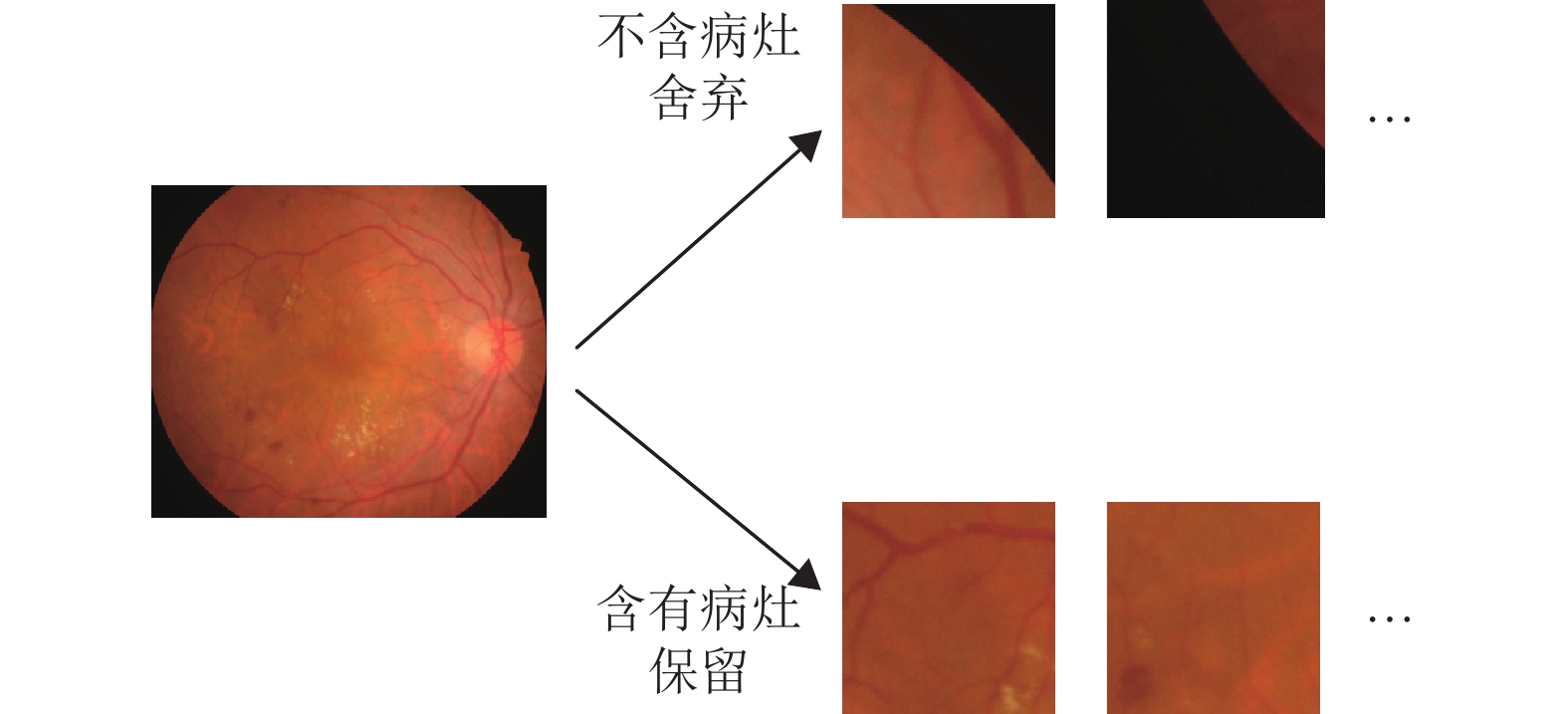
|
图 2 子图切割 |
1.3 多任务学习 1.3.1 多任务学习形式
多任务学习的一种具体形式, 主要包含两部分: 一部分为共享层, 另一部分为每个任务的特殊层, 共享层是多个任务之间共用的一部分网络, 特殊层是每个任务本身拥有的特性. 多任务学习有效的原因主要是: 多个任务一起学习时, 会互相增加噪声从而提高模型的泛化能力, 多个任务相互作用, 逃离局部最优解, 降低过拟合风险[10].
1.3.2 UNet++多层次的特征提取网络U-Net作为经典的医学图像骨干网络, 在医学图像方向广泛使用. 但其固定深度的下采样并不一定是最佳的下采样深度, 而UNet++不倾向于选择网络深度, 因为它在其体系结构中嵌入了不同深度的U-Net网络[11]. 这样就可以一次训练多个层次不同深度的U-Net, 然后从中选出最佳的深度.
UNet++中所有的U-Net共用一个编码器层, 而解码器层相互交织, 所有的U-Net会被同时训练, 这种设计提高了整体的分割性能[11]. 如图3所示, 橙色的为共享层, 其余为特殊层.
1.4 转置卷积代替传统上采样为了将低分辨率的图像还原成高分辨率图像, 会进行多次上采样, 常用的上采样方式包括: 最近邻插值、双线性插值等. 转置卷积也可以作为一种上采样方式, 因为其包含可学习的参数, 所以经过训练的转置卷积可以更好地实现上采样. 首先引入卷积输入和输出关系的计算公式[12]:
| $ {W_{{\text{out}}}} = \frac{{{W_{{\rm{in}}}} + 2P - F}}{S} + 1 $ | (1) |
其中, Win为输入的特征图宽度或高度, Wout为输出的特征图宽度或高度, P为padding的取值, F为卷积核的大小(一般卷积核形状为方形), S为卷积核移动步幅. 将Win和Wout交换位置公式就成为转置卷积的计算公式. 假设我们需要完成n倍的上采样操作, 设Wout=nWin, 则这些参数应该满足如下关系:
| $ W = \frac{{nW + 2P - F}}{S} + 1 $ | (2) |

|
图 3 UNet++多任务学习形式 |
然后可以推算出:
| $ \left\{ {\begin{array}{*{20}{l}} {S = n} \\ {F - S - 2P = 0} \end{array}} \right. $ | (3) |
上采样倍数一般是2的倍数, 于是有:
| $ \left\{\begin{array}{l} S=2 k \\ P=t \\ F=2(k+t) \end{array}\right. $ | (4) |
其中, k、t取1到正无穷的整数.
当卷积核大小F无法被步长S整除时, 容易出现棋盘状伪影, 所以需要保证F/S为整数.
从上述推导可以找到满足条件的一组解为: F=2, P=0, S=2.
1.5 损失函数图像分割任务输入尺寸为: h×w×c, 预测输出S的尺寸为: h×w×n, 其中h和w为长和宽, c为通道数, n为分割的种类. 然后将向量S和真值分割掩膜Y进行比较. 为此我们使用多分类的交叉熵损失函数, 如式(5)所示:
| $ L(S, Y) = - \sum\limits_{i = 1}^h {\sum\limits_{j = 1}^w {\sum\limits_{k = 1}^n {{Y_{i, j, k}}} } } \log {S_{i, j, k}} $ | (5) |
由于多任务学习可以进行多输出, 而且每个输出都进行相同的多分类任务, 所以赋予每个输出相同的损失函数, 最终的损失函数为所有的损失函数和求平均, 如式(6)所示:
| $ L = \frac{1}{N}\sum\limits_{i = 1}^N {{L_i}(S, Y)} $ | (6) |
如图4所示为网络的具体结构, 用VGG16替换传统网络的编码器部分, 解码器部分为VGG16的镜像, 然后输出4个结果. 其中K表示卷积核的个数.
2 实验验证和结果分析 2.1 数据集和评价指标糖尿病视网膜病变图像数据集来自于IDRiD和DDR两个公开数据集. IDRiD即印度糖尿病视网膜病变图像数据集, 是印度人口眼底图像数据集[13]; IDRiD是一个高质量的数据集, 其中的81幅图像提供了典型的DR相关病变和视盘的像素级标注. DDR为来自中国的公开数据集, 其包含数据较多, 采用国际标准标注, 所有图像都有像素级的病变标注, 包括了所有的病灶种类[14]. 两个数据集标注情况见表1.
从表1可以看出DDR相对于IDRiD有更多的标注图像. 在实际训练过程中, IDRiD数据集特征更为明显, 同时所有图像的分辨率均为4288×2848; 而DDR数据集的图像分辨率大小不固定, 但DDR包含更多不同严重程度的病灶图像, 多样性更好. 同时这也是先切割成子图的原因之一, 为了统一训练图像的分辨率.
2.2 多病灶图像分割图像分割关注4种病灶, 分别是: 微动脉瘤(micro-aneurysm, MA)、出血(hemorrhage, HE)、硬性渗出(hard exudate, EX)和软性渗出(soft exudate, SE). 病灶图像分割在训练时可以采取多分类或者多个二分类两种方法.
多分类就是将多张标签最终合并成一张标签, 里面包含背景、4种病灶等多个类别; 多个二分类是指打算分几个类别就有几张标签, 每张标签由病灶和背景组成. 本文选用多分类的方法, 因为通过初始实验对实际模型的训练比较, 这种方法分割效果更好, 各病灶的特征更加明显. 其中IDRiD数据集类别为: MA、HE、EX、SE以及黑色背景, 同时将除前面5类以外的像素都归为其他类, DDR数据集与IDRiD一致. 这样做的原因是在实验过程中观察到有将别的类识别成这4类的情况出现, 为了尽可能地减少这种情况, 同时为了提升分割效果, 所以在这里分割了多于4类. 虽然有多个类别, 但最终结果只计算MA、HE、EX以及SE这4类的指标.
在本文中, 我们使用精确召回曲线下的面积(area under precision recall curve, AUPR)作为我们的评估指标, 这与IDRiD挑战中使用的指标相同. 阳性预测值(positive predictive value, PPV)和真阳性率(true positive rate, TPR)定义如下:
| $ PPV = \frac{{TP}}{{TP + FP}} $ | (7) |
| $ TPR = \frac{{TP}}{{TP + FN}} $ | (8) |
其中, 真阳性(true positives, TP)是正确分类的病变像素, 假阳性(false positives, FP)是错误分类为病变像素的非病变像素, 假阴性(false negatives, FN)是错误分类为非病变的病变像素. 通过绘制给定不同阈值的精确召回对, 获得精确召回曲线, 然后计算出面积.
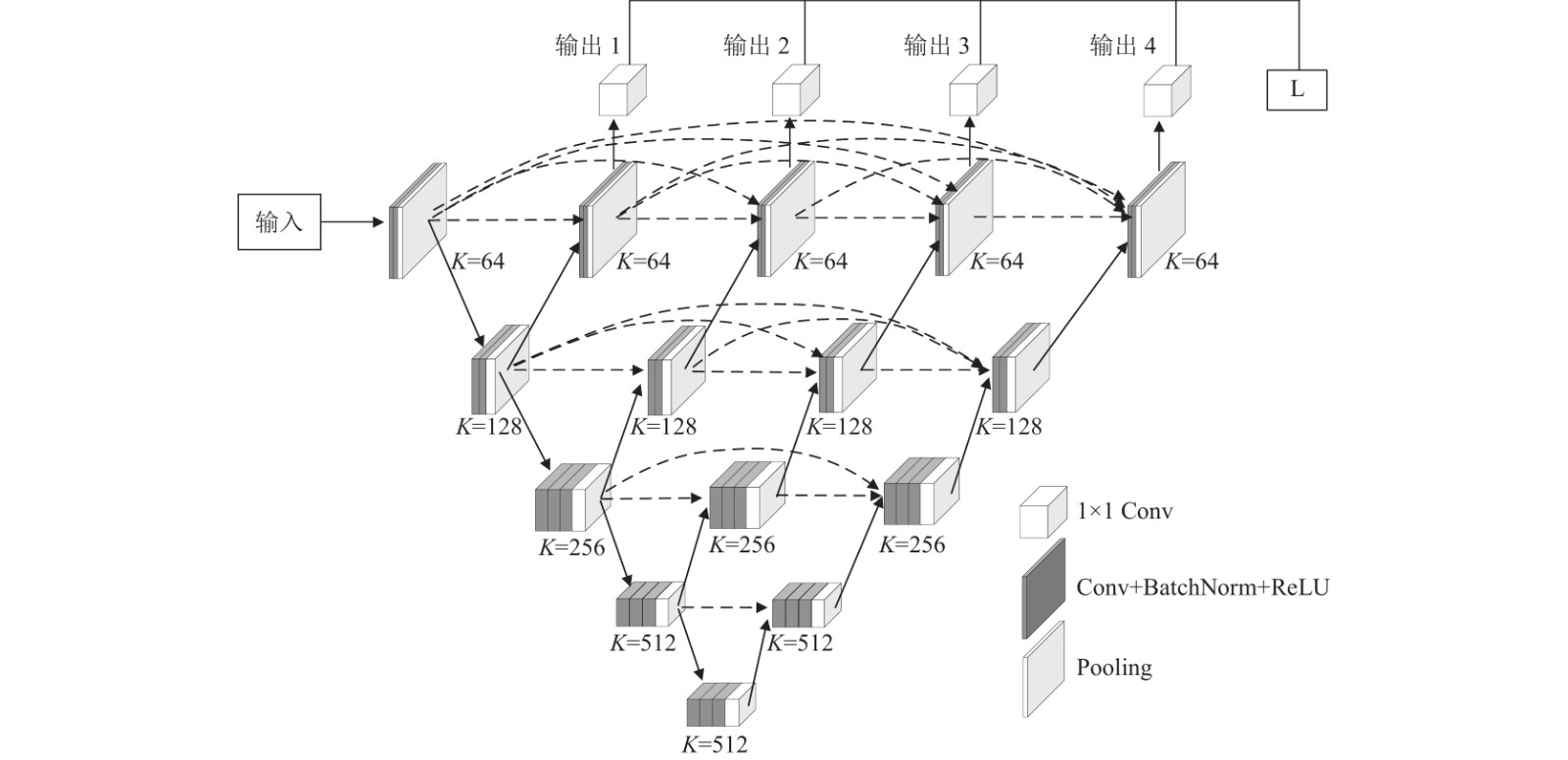
|
图 4 本文方法 |
| 表 1 糖网数据集分布 |
此外, 我们还利用不同类型病变的平均精确召回曲线下的面积(mean area under precision recall curve, mAUPR)来评估该模型在多病变分割中的整体性能.
2.3 结果分析为了探究不同分辨率对最终分割效果会产生什么影响, 同时满足网络所要求的子图分辨率为32的倍数, 分别使用多个32整数倍的尺寸进行对比实验, 实验结果如表2、表3所示.
| 表 2 IDRiD数据集不同分辨率AUPR对比 |
| 表 3 DDR数据集不同分辨率AUPR对比 |
表2、表3中的子图尺寸都为32的倍数, 为了使子图的分辨率变化更为明显, 从64开始每次增加一倍, 同时为了尽可能地提高病灶在图像中的占比, 最终将256作为最后一个分辨率. 从表中可以看出, 在两个数据集中, 64×64是使得AUPR指标综合表现最好的分辨率, 虽然64×64分辨率没有在所有类别上做到最好, 但其综合效果最佳, 所以选择64×64作为子图切割的分辨率. 从上述分析可知不同的分辨率大小确实对图像分割的结果产生了影响.
本文利用了UNet++的多任务学习属性, 实质上是多个完全相同的任务, 不同于一般定义中的多个相关任务, 或者说是多个不完全相同的任务. 为了验证该方法能否像一般多任务学习一样缓解学习中的过拟合, 从损失函数曲线变化角度进行分析, 如图5所示.
训练集损失不断下降, 验证集损失趋于不变或者上升, 说明网络开始进入过拟合. 从图5(a)和图5(b)可以看出在IDRiD数据集上, U-Net与本文方法经过一段时间的学习以后训练集和验证集的损失开始较为明显的差值, 说明网络进入过拟合, 同时两种方法进入过拟合的训练周期(epoch)也较为相近; 图5(c)和图5(d)为DDR数据集上的损失曲线, 两种方法损失曲线趋势相近, 看不到较为明显的过拟合点, 到训练的后半段学习速度也明显放缓. 抑制过拟合的一种常用方式为减少模型参数, 本文方法参数远多于U-Net, 但在学习趋势上与U-Net相近, 说明本文方法有更强的抑制过拟合的能力, 能够缓解在网络规模更大的情况下的过拟合.
为了证明多任务学习在糖尿病视网膜病变中的有效性, 设计了一组实验, 将本文方法多个输出的最好结果和经过相同改进的U-Net网络进行对比, 表4、表5为实验结果.
表4和表5中的输出整合为将各个输出最好值整合到一起. 从表4可以看出, 在IDRiD数据集上, 输出3在MA上, 输出4在EX、HA以及mAUPR上效果比U-Net效果更好, 在SE上与U-Net相差3.9%; 从表5可以看出, 在DDR数据集上, 输出4在EX、MA、SE上比U-Net效果更好, 在HE上与U-Net相差4.6%, 最终的mAUPR比U-Net表现更好. 从上述分析可知, 多任务学习可以取得更好的多分类效果, 即使在一个输出表现不佳的情况下, 也可由其他输出来补足. 在IDRiD数据集上相比于单任务的U-Net提升明显; 在DDR数据集上有提升, 但提升不是很大. 图6、图7为部分子图分割结果.

|
图 5 Loss曲线 |
| 表 4 多任务学习在IDRiD数据集效果对比 |
表6、表7中将本文方法和不同模型之间的AUPR值对比, 可以看出本文的方法, 在多个病灶上都取得较好的效果.
| 表 5 多任务学习在DDR数据集效果对比 |

|
图 6 IDRID部分子图分割结果 |
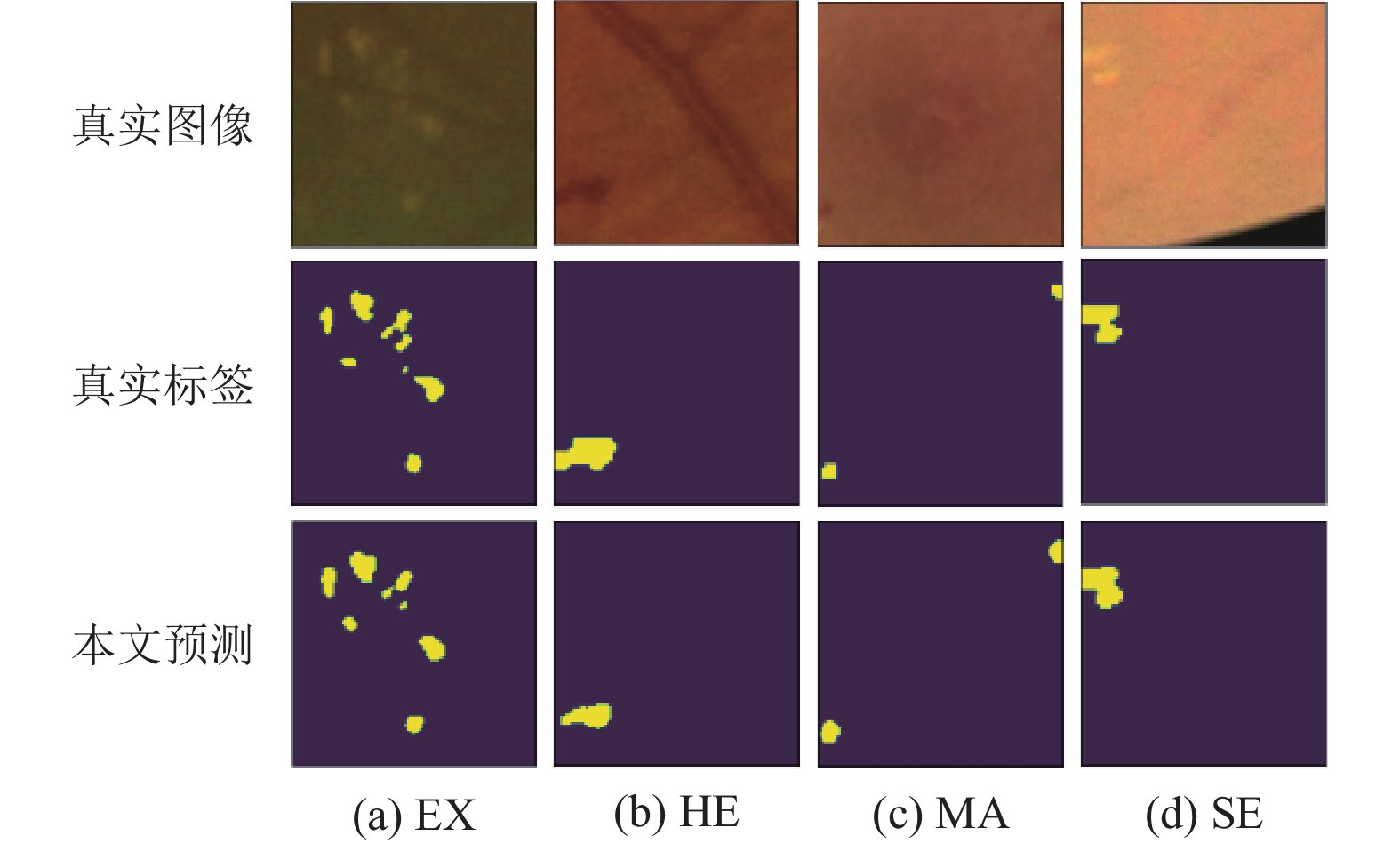
|
图 7 DDR部分子图分割结果 |
| 表 6 IDRID数据集上不同模型结果比较 |
3 结语
针对糖尿病视网膜病变的图像分割问题进行了研究, 本文提出了基于多任务学习结合滑窗切割的方法来进行多病灶图像分割. 该方法一方面考虑到医学图像高分辨率导致的病灶占比较小对分割结果产生的影响, 同时也考虑到样本不均衡导致学习效果不好的问题, 并通过增加非病灶种类以及提升含病灶子图占比的方法来解决. 实验结果表明了本文方法能够提升多种病灶的分割效果.
| 表 7 DDR数据集上不同模型结果比较 |
| [1] |
Bellemo V, Lim G, Rim TH, et al. Artificial intelligence screening for diabetic retinopathy: The real-world emerging application. Current Diabetes Reports, 2019, 19(9): 72. DOI:10.1007/s11892-019-1189-3 |
| [2] |
Asiri N, Hussain M, Al Adel F, et al. Deep learning based computer-aided diagnosis systems for diabetic retinopathy: A survey. Artificial Intelligence in Medicine, 2019, 99: 101701. DOI:10.1016/j.artmed.2019.07.009 |
| [3] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 |
| [4] |
Lim G, Lee ML, Hsu W, et al. Transformed representations for convolutional neural networks in diabetic retinopathy screening. Proceedings of the Workshops at the 28th AAAI Conference on Artificial Intelligence. Québec City: AAAI, 2014.
|
| [5] |
Saha O, Sathish R, Sheet D. Fully convolutional neural network for semantic segmentation of anatomical structure and pathologies in colour fundus images associated with diabetic retinopathy. arXiv:1902.03122, 2019.
|
| [6] |
Shelhamer E, Long J, Darrell T. Fully convolutional networks for semantic segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [7] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [8] |
Chen LC, Zhu YK, Papandreou G, et al. Encoder-decoder with atrous separable convolution for semantic image segmentation. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 833–851.
|
| [9] |
Wang Z, Yin YX, Shi JP, et al. Zoom-in-Net: Deep mining lesions for diabetic retinopathy detection. Proceedings of the 20th International Conference on Medical Image Computing and Computer-assisted Intervention. Québec City: Springer, 2017. 267–275.
|
| [10] |
Ruder S. An overview of multi-task learning in deep neural networks. arXiv:1706.05098, 2017.
|
| [11] |
Zhou ZW, Siddiquee MR, Tajbakhsh N, et al. UNet++: Redesigning skip connections to exploit multiscale features in image segmentation. IEEE Transactions on Medical Imaging, 2020, 39(6): 1856-1867. DOI:10.1109/TMI.2019.2959609 |
| [12] |
Dumoulin V, Visin F. A guide to convolution arithmetic for deep learning. arXiv:1603.07285, 2016.
|
| [13] |
Porwal P, Pachade S, Kokare M, et al. IDRiD: Diabetic retinopathy—Segmentation and grading challenge. Medical Image Analysis, 2020, 59: 101561. DOI:10.1016/j.media.2019.101561 |
| [14] |
Li T, Gao YQ, Wang K, et al. Diagnostic assessment of deep learning algorithms for diabetic retinopathy screening. Information Sciences, 2019, 501: 511-522. DOI:10.1016/j.ins.2019.06.011 |
| [15] |
Xie SN, Tu ZW. Holistically-nested edge detection. Proceedings of 2015 IEEE International Conference on Computer Vision. Santiago: IEEE, 2015. 1395–1403.
|
| [16] |
Mo J, Zhang L, Feng YQ. Exudate-based diabetic macular edema recognition in retinal images using cascaded deep residual networks. Neurocomputing, 2018, 290: 161-171. DOI:10.1016/j.neucom.2018.02.035 |
| [17] |
Yu ZD, Feng C, Liu MY, et al. CASENet: Deep category-aware semantic edge detection. Proceedings of 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017. 1761–1770.
|
| [18] |
Guo S, Li T, Kang H, et al. L-Seg: An end-to-end unified framework for multi-lesion segmentation of fundus images. Neurocomputing, 2019, 349: 52-63. DOI:10.1016/j.neucom.2019.04.019 |
 2023, Vol. 32
2023, Vol. 32


