胶质瘤是最常见的原发性颅脑恶性肿瘤, 发病率约占脑部疾病的1.3%, 约为颅内肿瘤的35.2%–61.0%, 具有发病率高、复发率高、死亡率高以及治愈率低的特点[1]. 在5–9岁的儿童和中老年人中比较常见, 是一种难以根治, 容易反复, 对人体危害极大的疾病. 根据GLOBOCAN数据集表明, 全球脑瘤新发病例25.6万例, 发病率为34%, 占恶性肿瘤的1.8%, 位居肿瘤发病数的第17位, 死亡18.9万例, 占总死亡人数的25%, 占恶性肿瘤死亡的2.3%, 位居恶性肿瘤死亡人数的第12位. 2011年, 全国脑瘤病例新发8.7万例, 发病率64.7%, 占恶性肿瘤发病率的2.59%, 位居恶性肿瘤发病的9位[2], 胶质瘤作为脑瘤中最常见的病症之一, 近30年来, 胶质瘤的发病率逐渐递增, 目前, 我国的胶质瘤年发病率逐渐增加, 经诊断治疗后的5年内, 其死亡率仍在全身肿瘤中仅次于胰腺癌和肺癌[3], 情况非常严峻, 目前胶质瘤主要采用手术切除为主, 放化疗为辅的常规综合性标准治疗, 但常规治疗患者预后较差. 由于胶质瘤浸润性生长模式, 手术切除只能在肉眼可见的范围内进行切除[4], 且因其所在位置的敏感, 难以被完全切除, 所以一直是术后康复较差的几种肿瘤之一, 每年病发致死的患者约在3万人以上. 因此, 计算机辅助诊断胶质瘤, 帮助医生提升其诊断效率是一项很有必要, 也同样迫在眉睫的任务.
攻克胶质瘤的任务虽然极其紧急, 但是也存在着一些不容忽视的障碍. 首先, 现有的胶质瘤数据集大部分存放在欧美, 数据集量小且很难反映患病特征, 其次, 现有的数据库缺乏复发或复发配对患者的详细信息, 无法评估治疗对胶质瘤在基因组水平的影响, 因此, 目前医学界对于胶质瘤的研究进展处于一个较为缓慢的状态. 本文便基于当前形势下, 通过与辽宁省肿瘤医院交流合作, 对患者的实际胶质瘤图像和病灶展开具体详细的分类分析, 旨在如何将医院的实际图像和患者的原始数据, 转化为可以被进一步应用和实验的数据集, 对胶质瘤数据集缺少这一难题, 提出可行的解决方案, 使胶质瘤的研究更进一步, 也可使医生对于患者的情况有一个更好的了解, 通过计算机的辅助诊断来帮助医生, 提高临床的判断能力.
1 数据集建立 1.1 研究背景随着医学技术的发展, 许多医学成像方式都可以通过不同的角度观测到胶质瘤的形态, 对胶质瘤原始数据集的建立提供了很大的帮助. 本文主要通过分析患者的核磁共振成像(magnetic resonance imaging, MRI)来进行原始数据集的建立. MRI是一种非侵入性且效果良好的软组织对比成像方式, 可以提供类似于脑肿瘤形状, 大小和空间位置描述等信息, 并为胶质瘤的分类和分割提供详细而有价值的信息. 与其他成像方式相比, MRI提供的信息量大, 参数多, 可以从多角度多模态进行分析, 对疾病的诊断具有很大的优越性. 因此, MRI通常被用于脑部疾病的诊断, 进而制定后续治疗方案, 还可以持续观察脑部肿瘤的发展情况[5, 6].
在分析患者的MRI图像时, 通常从横断面、矢状面和冠状面3个方位入手. 横断面(transverse plane)又称水平面, 即以横切面为基准构成的水平切割面, 从上至下, 将人体横切为上下两部分; 矢状面(sagittal plane)是以正中面为基准把人体分成左右两面的解剖面; 而冠状面(coronal plane)是从左右方向, 沿着人体的额状轴将人体纵切为前后两部分的切面, 具体如图1所示.

|
图 1 人体矢状、冠状、横断面示意图 |
1.2 数据来源
本文的原始数据来源于辽宁省肿瘤医院, 该数据包括了从2017–2021年医院就诊期间着重选取的300名胶质瘤患者的多序列MRI检查图像. 每个案例中均包含患者横断面、矢状面和冠状面的图像, 以及每种图像T1加权图像(T1WI)、T2加权图像(T2WI)、磁共振成像液体衰减反转序列(FLAIR)等不同的模态. 使用NEUSOFT PACS软件打开原始数据, 在观察过程中可以清楚地看到不同患者不同级别的肿瘤分布, 每名患者的症状不尽相同, 大大地提高了数据集的广泛度和包含度. 如图2, 便是随机选取一名胶质瘤患者的横断面、矢状面、冠状面的图像. 图像质量清晰, 纹理清楚, 肿瘤的位置可以很容易地分辨出来. 图像内容完整详细, 每种模态的图像序列分别包含18–170个切片不等, 对于本文进行原始数据集的建立提供了良好的资源基础和极大的帮助. 除此之外, 该胶质瘤分类的过程也是在辽宁省肿瘤医院医生的协助下进行, 保证了原始数据集分类的效率和真实性.
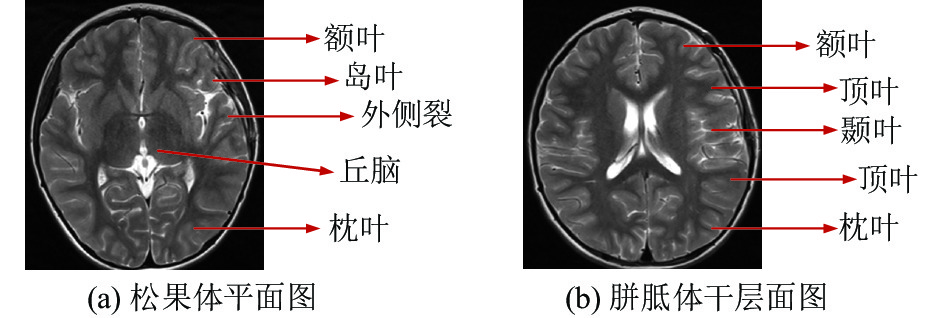
大脑是人体最复杂的器官, 掌握着人类的一切动作和功能, 其内部结构非常复杂, 胶质瘤的病变范围是人体大脑内部, 因此想要充分了解胶质瘤, 必须对人体大脑结构也要有着一个初步的了解. 图3简单地描述了大脑内部横断面的具体结构.

|
图 2 人体大脑三维切面图 |
1.3 数据分类
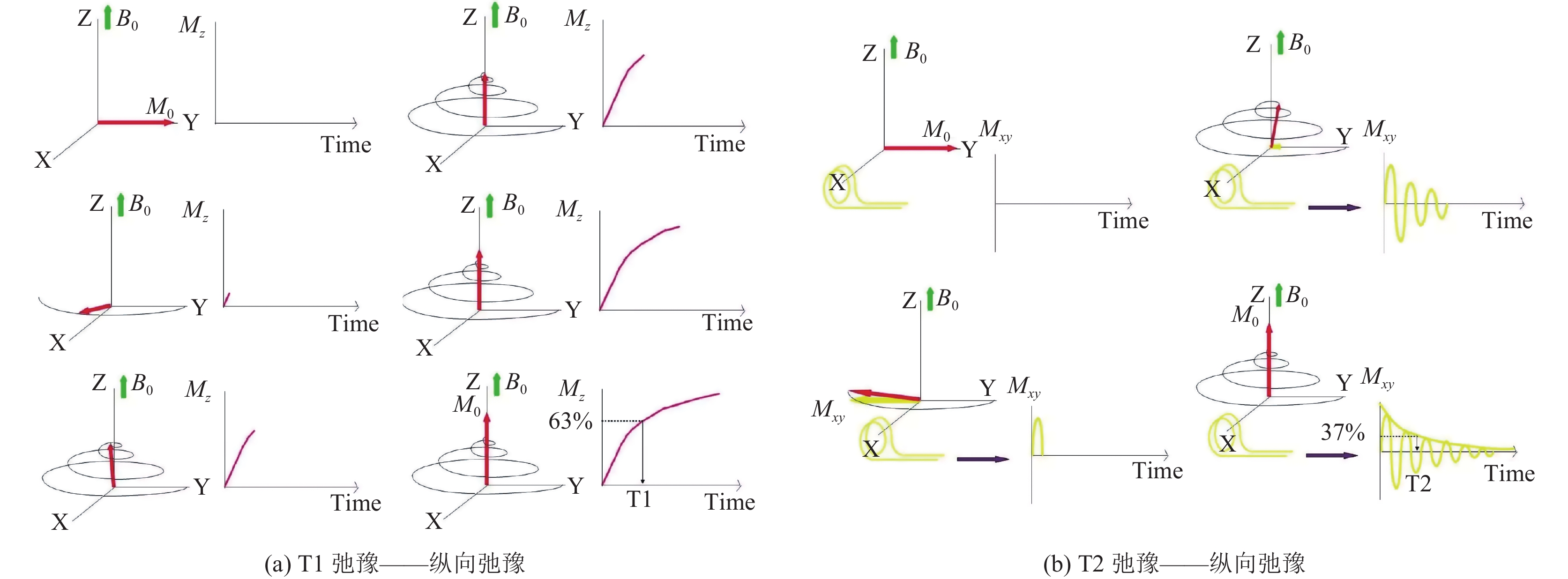
在原始数据集的建立过程中, 数据分类是至关重要的. 在临床医学中, 不同序列的常规MRI检查图像, 如T1WI、T2WI、FLAIR等, 都可以从不同方面来判断出肿瘤的位置、范围、形态和信号特点. 其中T1WI主要作用于突出组织T1弛豫(纵向弛豫)的差别, T1弛豫又被称作自旋-晶格弛豫, 是代表宏观磁化矢量恢复快慢的, 其具体表现为吸收过射频能量后跃迁到高能级的质子释放能量的过程. 能量释放得越快, T1弛豫所需的时间就越短, 恢复稳态的过程就越快. 在图4(a)中表示出T1弛豫的数学定义和运动规律: T1弛豫是宏观纵向磁化矢量恢复到最大值的63%所需要的时间. 在液体中, 特别是纯水, 同样由于热运动频率比质子的进动频率高, 所以同样纵向弛豫过程满, T1时间长, 质子能量释放得越快, 纵向弛豫过程越短, 也就是T1越短.
|
图 3 大脑内部结构图 |
|
图 4 T1、T2弛豫运动规律图 |
而T2WI主要作用于突出组织T2弛豫(横向弛豫)的差别. T2弛豫又被称为自旋-自旋弛豫. 它产生的原因是每个质子在射频脉冲能量撤销以后, 由于化学位移效应, 每个质子周围感生的-磁场都不同, 这样便导致了不同质子的进动频率出现差别, 在水平面上逐渐失相位, 产生信号衰减. 由图4(b)中可看出T2弛豫代表的是横向磁化矢量的衰减, 其数学定义是横向磁化矢量衰减到最大值37%所需要的时间. 而FLAIR图像与T2相类似, 但TE和TR时间非常长, TE是指回波时间, 也就是第一个射频脉冲到回波信号产生所需要的时间; 而TR是指重复时间, 即脉冲序列执行一次需要的时间, 因此, 原本的异常处仍然很明亮, 但正常的脑脊液会减弱并变黑[7]. 这几种图像都能够多方位、无骨质伪影地显示解剖结构, 极大地方便了胶质瘤的分类过程. 而在分类过程中, 主要分为病变判定、病变定位和病变定性3个步骤.
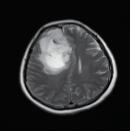
1.3.1 病变判定病变判定, 主要就是通过观察患者不同序列的MRI图像, 来判断患者脑部是否存在病变的过程. 在患者的T2WI图像中, 发生病变的区域会呈高亮显示, 因此在判断的过程中, 针对T2WI图像, 分析患者一整个周期的图像变化, 如果患者的胶质瘤较为明显, 那么经过仔细观察便可以初步判断出病变的存在. 图5是抽取辽宁省肿瘤医院某患者的脑部MRI的T2WI图像的周期图, 在图5(a)–图5(c)中无法判断出患者是否存在胶质瘤, 但在图5(d)中, 高亮部分非常明显, 因此可以很容易对患者是否存在胶质瘤进行初步的判定.

|
图 5 患者MRI T2WI横断面流程图 |
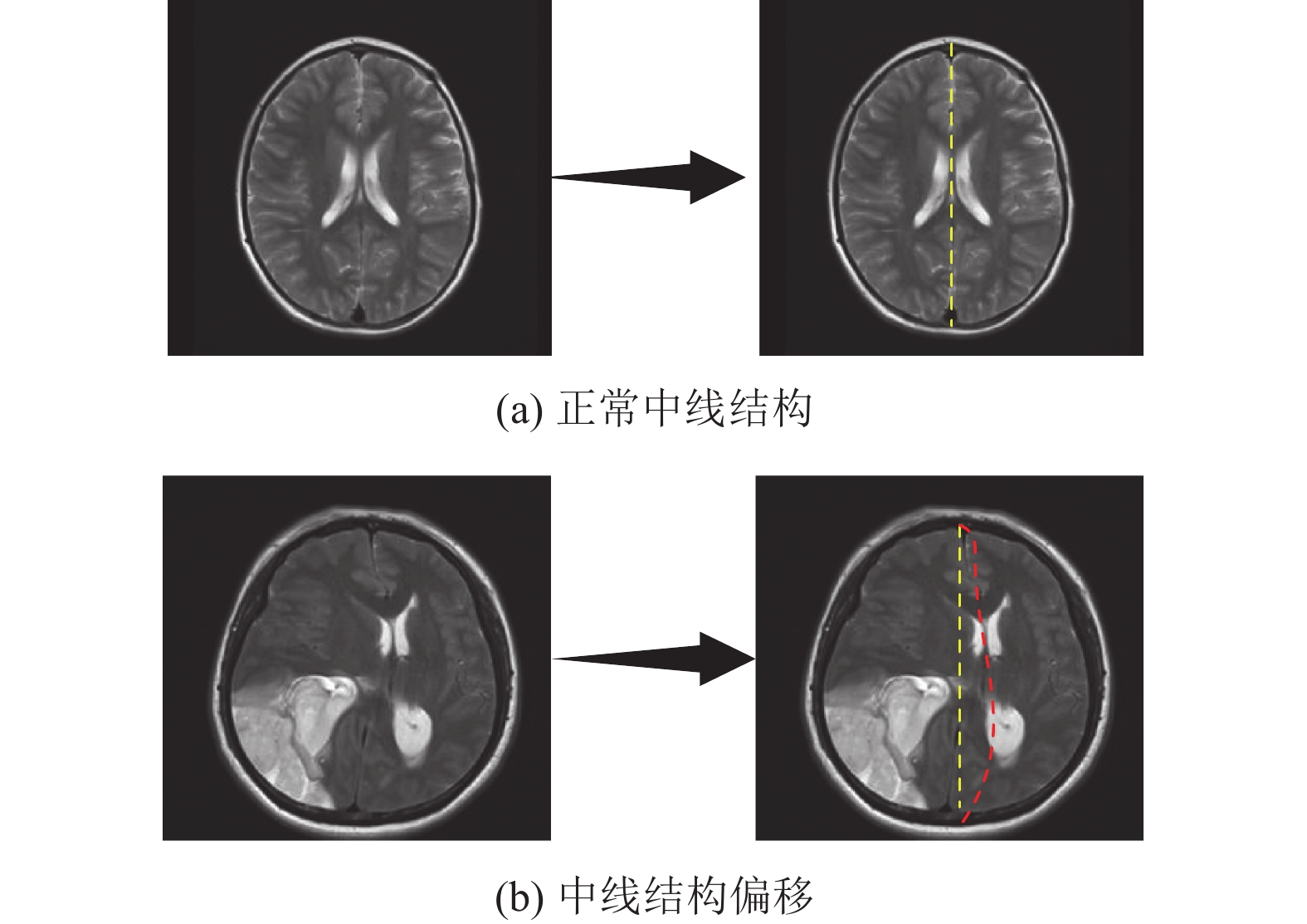
在此基础上, 对于一些比较轻微或不太明显的肿瘤, 占位效应, 是一个很重要的能够辨别患者大脑是否存在病变的方法. 具体来说, 因为正常人的大脑的左右结构基本上是完全对称的, 如果患者脑中存在肿瘤, 肿瘤会占据一定的空间且对其所在周围区域进行挤压, 进而导致中线的偏移和左右结构的不对称. 大脑中线, 又称为大脑镰, 是将大脑均匀分成左半球和右半球的结构, 简而言之, 通过观察MRI图像中大脑中线是否偏移, 是判断有无病变的重要标志. 在图6中, 选取了一位正常人和一位患者的MRI横断面的T2WI图像, 图6(a)是正常情况下的大脑中线情况, 图6(b)是患者大脑中线被挤压的情况, 可以明显看出患者的大脑中线与正常情况下相比, 因为肿瘤的挤压而产生了明显偏移, 因此可以通过大脑中线是否偏移, 来准确地判断出患者脑部是否存在病变.
1.3.2 病变定位病变定位, 即已经初步判定胶质瘤的存在后, 对其所在位置进行判断和确认. 在计算机辅助诊断中, 最常见的MRI序列是T1WI和T2WI. 常规的T1WI和T2WI图像, 可以帮助快速判断出病变的位置. T1WI和T2WI有着明显的区别, 通过使用较短的TE和TR来生成T1加权, 图像的对比度和亮度由T1WI决定. 相反, 通过使用更长的TE和TR来生成T2加权[8], 则由T2WI来决定图像的对比度和亮度. 在分析图像时, 可以通过脑脊液的信号来对T1WI和T2WI进行区分, 在T1W1图像中, 水呈黑色, 脑脊液呈现为低信号, 表现为比较暗的形式, 而在T2WI中恰恰相反, 水呈白色, 脑脊液呈现为高信号, 表现为比较亮的形式, 因此通过判断脑脊液信号的高低, 可以很轻易地分辨出T1WI和T2WI图像. 如图7(a)中的整体图像偏暗, 脑脊液呈现为较暗的低信号, 为T1WI图像; 图7(b)中的整体图像偏亮, 脑脊液呈现为较亮的高信号, 为T2WI图像.
|
图 6 占位效应对比图 |

|
图 7 T1WI 、T2WI对比图 |
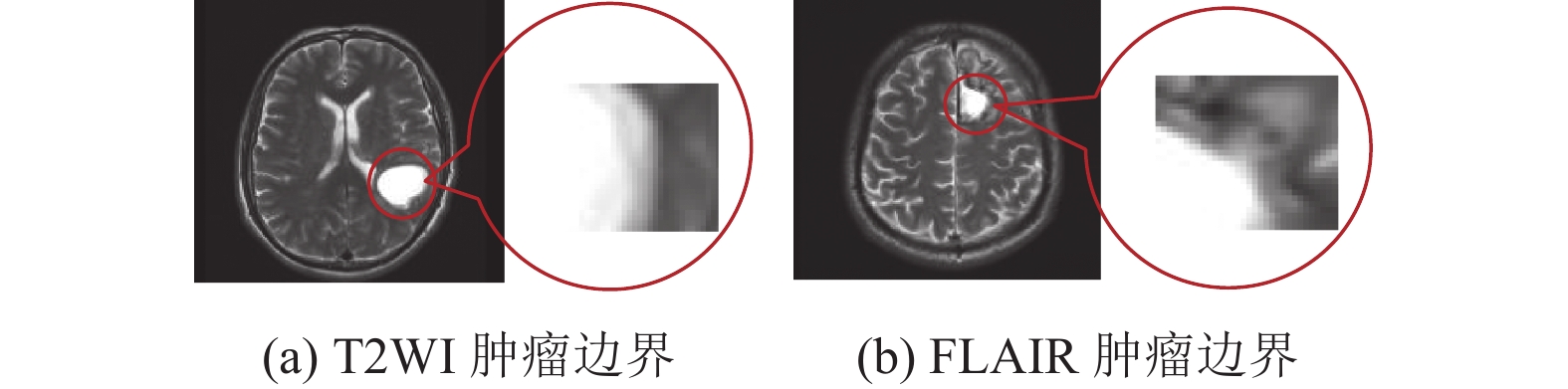
胶质瘤病灶通常与正常脑组织混合在一起, 不易分辨出边界, MRI不同序列的图像则对其边界的分辨提供了很大帮助. T2WI信号与水含量有关, 而且许多病灶的T2信号要明显强于周围的正常组织, 常呈现出高亮状态, 除此之外, 与T2相类似的FLAIR序列, 同样对肿瘤位置非常敏感, FLAIR序列异常处同样高亮, 而正常的脑脊液颜色会变黑. 因此在目前的临床医学中, 常用T2WI图像和FLAIR图像来对病变进行定位. 在图8中, 可以清楚地看出肿瘤病灶高亮区域和周围较暗组织, 有着明显的边界. 因此通过分析MRI中的T1、T2、FLAIR图像, 可以帮助定位病变.
|
图 8 患者肿瘤边界图 |
1.3.3 病变定性
在确定是否具有病变和病变的大体位置后, 接下来需要通过观察MRI图像将病变定性, 判断其严重程度. 世界卫生组织(WHO)已经将其分为WHO-I至WHO-IV四个等级, 其中WHO-I级、WHO-II级被称为低级别胶质瘤(low grade glioma, LGG), WHO-III级、WHO-IV级被称为高级别胶质瘤(high grade glioma, HGG)[9], 每级的表现形式均不同. 对于WHO-I级, 其大多数为“良性”肿瘤, 位于脑白质, 以儿童和20–40岁成人发病居多, 常发生于双侧大脑半球、额叶及双侧颞叶和脑干的位置, 主要影像学表现为病变位置局限, 边界清楚, 出血少见, 几乎无占位效应和瘤周水肿. 图9为WHO-I级影像, 可以明显看出WHO-I级肿瘤边界清晰, 无瘤周水肿; 而中间标注的虚线是大脑的中线位置, 可看出标注的中线与大脑本身的中线基本重合, 几乎无占位效应, 符合WHO-I级细胞瘤特征.

|
图 9 WHO-I级细胞瘤图 |
而对于WHO-II–III级细胞瘤来说, II–III级胶质细胞瘤总体特征相似, 细胞瘤强化方式多样, 主要表现为浸润性肿块和局灶结节状, 并且在增强扫描下, 不强化或者均匀/斑片状强化均有可能发生. 此外, T2WI不均匀高信号和有可能蔓延到皮质也是其特征之一. 图10为WHO-II–III级胶质细胞瘤T1WI、T2WI影像, 上面为T1WI图像, 下面为T2WI图像, 其中图10(a)为WHO-II级细胞瘤图像, 从中可看出扫描结果为均匀长T1、T2信号, 存在浸润性肿块且增强扫描强化不明显, 图10(b)为WHO-III级细胞瘤图像, 从图可看出呈现为不均匀长T1、T2信号, 增强扫描局部或斑片状强化, 均符合WHO-II–III级细胞瘤特征.
对于WHO-IV级细胞瘤, 主要表现为T1WI信号稍低、T2WI信号稍高的形态, 多发生于胼胝体附近的肿瘤常累及中线两侧呈蝶翼状生长, 病变呈明显的不均质信号, 瘤体内出血、坏死常见, 进而导致肿瘤边界不清晰, 占位效应和瘤周水肿非常明显, 增强后多呈花环状强化提示胶质母细胞瘤. WHO-IV级细胞瘤和低级别胶质瘤主要是病理学上的恶性程度划分, MRI中差异显示中主要体现在T2WI图像和增强扫描上[10]. 图11是一位WHO-IV级细胞瘤患者的MRI图像, 图11(a)为T1WI图像, 而图11(b)为T2WI图像, 从中可以很明显看出该肿瘤生长于胼胝体附近, 而该图像T1大体较暗, 而T2更亮一些, 因此主要表现为T1WI信号稍低、T2WI信号稍高的形态, 肿瘤边界不够清晰, 而且体内肿瘤颜色偏黑, 判断瘤有可能出血或坏死, 符合WHO-IV级细胞瘤的特征.

|
图 10 WHO-II–III级细胞瘤图 |

|
图 11 WHO-IV级细胞瘤图 |
将原始数据进行详细准确的分类是建立数据集的基础, 也是建立过程中非常重要的步骤之一. 在庞大繁杂的原始数据中, 如何快速精确的分类无疑是很有必要的. 本文希望通过对MRI不同序列图像的分析, 确认肿瘤的存在及病灶区域, 为后续的诊断和治疗提供客观信息.
本文基于胶质瘤图像构建原始数据集的基本流程图如图12所示, 将通过辽宁省肿瘤医院提供的300名患者原始数据进行预处理, 然后进行病变判定、病变定位和病变定性操作, 将分类后的图像记录后进行分割操作, 得出的结果验证了本文的实验流程可以把胶质瘤图像的原始数据, 转换成可以被应用的原始数据集.
2 应用 2.1 图像分类胶质瘤是致死率排名第二高的恶性肿瘤, 被诊断为恶性肿瘤的患者, 在被诊断5年后依然可以存活的概率不足5%[11], 因此根治胶质瘤是非常有必要的. 但由于脑部是人体最为重要的绝对控制性器官[12], 操作起来必须小心谨慎, 因此如何彻底的根治胶质瘤, 一直是医学界中比较棘手的问题之一. 近年来, 从影像大数据集中提取肉眼难以有效识别的图像信息, 通过分析这些信息来建立诊断疾病的预测模型, 已成为具有广阔应用前景的技术手段[13]. 随着医学技术的发展, 借助计算机辅助实现对疾病的诊断成为流行趋势[14], 目前已逐渐应用于脑肿瘤的MRI诊断和鉴别[15]. 而如今现代医疗领域已经配备医疗数据的采集及存储系统, 并且能够在大容量的信息系统中实现集成和分享. 用机器学习和深度学习对医疗大数据进行挖掘, 以总结、获取新的知识, 通过深度学习高效进行数据集的挖掘整合, 从而实现精确诊断、鉴别诊断的数字医学技术迅猛发展[16], 相比常规的临床研究和传统统计学, 具有更强的数据处理和知识获取能力[17, 18]. 本数据集在基于学习的领域, 其应用范围非常广阔, 在数据集建立过程中, 已经在数据集中做出了是否发生病变的标记以及病灶对应的分级, 有助于基于学习的模型和算法的优化, 并在此基础上, 来实现对胶质瘤的进一步研究, 最终达到在临床治疗中, 使医生对患者的病变情况有一个准确的分析. 而在分类的特征提取方面, 本文将手工提取与机器提取相结合, 把通过各种机器学习和深度学习神经网络对图像提取的特征与手工提取特征结合起来, 可以对胶质瘤图像的分类有更深入的作用, 也有助于进行后续的分类和分级的研究. 图13是本文对原始数据进行分类的结果, 图13(a)是正常情况下的图像, 图13(b)是胶质瘤病变的图像. 图14是对原始数据进行分级的结果, 图14(a)–图14(d)分别是从建立完成的数据集中挑出的WHO-I至WHO-IV四个等级的胶质瘤图片, 图片清晰全面, 可见在此研究下, 能够有效缓解当前胶质瘤原始数据集不足的难题, 对胶质瘤的更深层次的剖析和研究, 提供根基上的帮助.
2.2 图像分割本数据集的建立, 在图像分割方面也起到重要应用. 图像分割是图像处理中的重要技术, 在临床诊治中, 为了准确地对患者的病情进行诊断, 减少判断误差, 常常使用图像分割技术来辅助医生确定病灶区域的位置和大小[14], 肿瘤及周边病灶组织的分割与测量可以给医生提供重要的定量指标, 为后续治疗方案提供客观依据[19]. 同时, 通过对原始数据集的分割, 可以帮助确定数据集的可用性, 其分割效果直接证明了数据集可进行后续研究的应用性.

|
图 12 原始数据集建立流程图 |

|
图 13 胶质瘤分类图 |

|
图 14 胶质瘤分级图 |
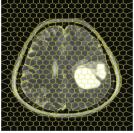
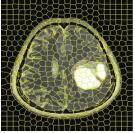
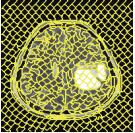
本文通过几个简单的超像素分割算法, 验证数据集的分割结果. SLIC算法[20]最早由Achanta等人提出, 它是一种采用距离属性来划分聚类的优化算法, 使生成的超像素形状较为规则, 并且在保证分割质量的同时一定程度上降低了图像的计算成本. LSC算法[21]最早由Li等人提出, 它是一种非常成功的融合算法, 既能考虑图像中的全局范围的信息, 又可以实现可观的处理效率, 呈现出了高质量的超像素分割结果并且保持了线性的时间复杂. DBSCAN算法[22]由Shen等人提出, 是近年来新兴的一种独特的超像素算法, 它可以构建任意形状的聚类, 在一定程度上可以对复杂图形进行精准分割. 且为了产生较为规则的超像素轮廓, 在算法的构建中还增添了几何图形的规制.
在表1中, 选取原始数据集中的A、B、C、D四组二维脑肿瘤MRI切片数据进行实验. 第1行代表MRI切片原影像, 第2行代表以SLIC超像素算法生成的600块的超像素结果, 第4行代表以LSC超像素算法生成的600块的超像素结果, 第6行代表以DBSCAN超像素算法生成的600块的超像素结果, 第3、5、7行代表二维图像结果. 最后一行为肿瘤医院医师手绘勾勒的肿瘤真实值辅助验证分割的准确性. 由此可以看出, 本文建立的原始数据集可以被分割且分割效果良好, 具有可分割的实施性和准确性, 后续可以被用于胶质瘤的研究, 为攻克胶质瘤提供医学上的帮助.
| 表 1 图像分割处理结果 |
2.3 肿瘤生长与重建
除了在图像处理上最常见的分类分割之外, 本数据集在未来肿瘤的生长模型和三维重建上, 也可以提供原始的图像数据帮助. 目前来看, 基于图像的肿瘤生长模型主要有3类: 扩散模型、力学模型[23]以及两种模型的结合. 肿瘤生长模型应用于早期诊断, 能够帮助医生预测肿瘤的生长变化, 产生的参数反映肿瘤近一段时间的形态变化及周围边缘变化, 因此, 本文为肿瘤的生长模型提供原始的图像数据这一应用, 有助于方便医生预测患者可能会发生的问题并及时调整治疗方案, 选择更适宜的治疗方式和药物, 确定患者的最佳治疗时间, 实现更加精准的定性和定量的评估. 甚至, 该模型可以加入基因信息, 预测未来肿瘤会发生的结果. 此外, 该肿瘤生长模型还应用于外科肿瘤切除手术以及放射治疗前肿瘤生长对周围血管神经产生形变的预测. 基于ADRE的肿瘤生长模型除了用于肿瘤进化预测外, 还用于定义肿瘤侵袭边缘, 并将生长模型纳入到患者大脑的解剖中进行胶质瘤的分割等, 对胶质瘤的研究和治疗都有着很大的帮助.
而在未来肿瘤的三维重建的应用方面, 本文的研究也可以起到作用. 肿瘤MRI图像的三维重建就是对获得的一系列的医学图像进行处理, 从而构造出三维的几何模型, 将看不见的组织或器官通过三维的方式显示出来. 而本文可以为肿瘤的三维重建提供原始的图像数据, 如图15是与本文有关的肿瘤三维重建切片图像, 图15(a)为肿瘤三维等高线切片, 而图15(b)是肿瘤三维重建的可视图像, 由此可见本文提供的原始图像数据, 有助于推动肿瘤重建的发展, 使未来肿瘤图像的建模的显示效果更加直观, 便于医生在观察病变切片时, 可以更加仔细和准确.
3 结束语胶质瘤对人类的生命健康威胁极大, 而胶质瘤逐年递增的发病率, 也表明了加深胶质瘤研究的紧迫性和必要性. 基于此条件下, 本文提供了一种可行的基于胶质瘤图像的原始数据集建立过程和方法, 通过观察和提取不同序列的胶质瘤MRI图像的影像学表现, 按照病变判定、病变定位和病变定性3个步骤, 分析和分类了大量真实二维脑肿瘤MRI切片数据, 标记其是否有病变, 并对胶质瘤进行WHO-I至WHO-IV四个等级的分类, 建立了原始数据集. 然后针对数据集进行了其应用的可行性分析并实验, 可以证明本文所建立的原始数据集具有较高的分类性能和分割性能且可提供肿瘤生长与重建的图像数据, 可以用于后续胶质瘤的实验和研究, 同时通过计算机辅助诊断, 帮助医生在临床治疗上获得更多有效信息.

|
图 15 脑肿瘤重建图 |
| [1] |
干秀灵. 基于注意力机制的轻量化U-Net脑部胶质瘤图像分割研究[硕士学位论文]. 重庆: 西南大学, 2021.
|
| [2] |
毕旭. 基于影像组学的脑部胶质瘤高低级别分类模型研究与实现[硕士学位论文]. 武汉: 武汉理工大学, 2019.
|
| [3] |
国家卫生健康委员会医政医管局. 脑胶质瘤诊疗规范(2018年版). 中华神经外科杂志, 2019, 35(3): 217-239. DOI:10.3760/cma.j.issn.1001-2346.2019.03.001 |
| [4] |
姚婷婷, 刘梦昱, 赵鹏翔, 等. 替莫唑胺治疗胶质母细胞瘤的耐药性产生机制研究进展. 生物技术进展, 2021, 11(6): 705-710. DOI:10.19586/j.2095-2341.2021.0022 |
| [5] |
赵俊庭. 脑部MRI图像中胶质瘤的检测技术[硕士学位论文]. 天津: 天津大学, 2019.
|
| [6] |
张坤毅. 核磁共振(MRI)的成像原理与临床应用. 中国医疗设备, 2008, 23(5): 101-103. DOI:10.3969/j.issn.1674-1633.2008.05.040 |
| [7] |
崔晓萌. 基于磁共振影像的脑部疾病分类方法研究[硕士学位论文]. 济南: 山东大学, 2020.
|
| [8] |
王朔. 张量分解模型在脑部磁共振影像诊断中的应用[硕士学位论文]. 济南: 山东大学, 2018.
|
| [9] |
戴宏, 符冉迪, 金炜. 基于影像组学与集成学习的脑胶质瘤分级预测. 宁波大学学报(理工版), 2021, 34(4): 28-34. |
| [10] |
张洪, 曾文兵. MRI影像分析在高级别脑胶质瘤鉴别诊断中的应用. 中国CT和MRI杂志, 2017, 15(8): 37-39, 72. DOI:10.3969/j.issn.1672-5131.2017.08.011 |
| [11] |
马其鹏. 基于深度学习的MRI脑肿瘤分割算法研究[硕士学位论文]. 无锡: 江南大学, 2021.
|
| [12] |
倪鹏程. 基于脑部胶质瘤的医学影像配准与融合研究[硕士学位论文]. 成都: 电子科技大学, 2018.
|
| [13] |
Thust SC, Heiland S, Falini A, et al. Glioma imaging in Europe: A survey of 220 centres and recommendations for best clinical practice. European Radiology, 2018, 28(8): 3306-3317. DOI:10.1007/s00330-018-5314-5 |
| [14] |
庞恩帅. 基于多编码结构与Cox分析的脑部影像分析方法研究与实现[硕士学位论文]. 济南: 山东大学, 2021.
|
| [15] |
Booth TC, Williams M, Luis A, et al. Machine learning and glioma imaging biomarkers. Clinical Radiology, 2020, 75(1): 20-32. DOI:10.1016/j.crad.2019.07.001 |
| [16] |
赵维维, 孙静, 诸静其. 人工智能在脑胶质瘤MRI诊断中的研究进展. 磁共振成像, 2021, 12(8): 88-90. DOI:10.12015/issn.1674-8034.2021.08.019 |
| [17] |
Altman RB. Artificial intelligence (AI) systems for interpreting complex medical datasets. Clinical Pharmacology & Therapeutics, 2017, 101(5): 585-586. |
| [18] |
Sun YJ, Todorovic S, Goodison S. Local-learning-based feature selection for high-dimensional data analysis. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2010, 32(9): 1610-1626. DOI:10.1109/TPAMI.2009.190 |
| [19] |
吴强, 何泽鲲, 刘琚, 等. 基于机器学习的脑胶质瘤多模态影像分析. 山东大学学报(医学版), 2020, 58(8): 81-87. |
| [20] |
Achanta R, Shaji A, Smith K, et al. SLIC superpixels compared to state-of-the-art superpixel methods. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2012, 34(11): 2274-2282. DOI:10.1109/TPAMI.2012.120 |
| [21] |
Li ZQ, Chen JS. Superpixel segmentation using linear spectral clustering. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Boston: IEEE, 2015. 1356–1363.
|
| [22] |
Shen JB, Hao XP, Liang ZY, et al. Real-time superpixel segmentation by DBSCAN clustering algorithm. IEEE Transactions on Image Processing, 2016, 25(12): 5933-5942. DOI:10.1109/TIP.2016.2616302 |
| [23] |
Konukoglu E. Modeling glioma growth and personalizing growth models in medical images [Ph.D. thesis]. Nice: Université Nice Sophia Antipolis, 2009.
|
 2023, Vol. 32
2023, Vol. 32