近年来, 人工智能技术的飞速发展在医疗领域受到广泛关注. 与此同时, 随着医学数据集的不断扩增、硬件计算能力的快速提升以及应用算法的突破性进展, 人工智能在医疗场景中的技术积累日渐成熟, 应用范围逐步拓宽. 2019年1月, 由上海交通大学人工智能研究院领衔发布的《人工智能医疗白皮书》[1]中指出, 人工智能在医疗领域应用最广的场景就是医学影像分析, 该领域更是被业内人士认为是最有可能率先实现商业化的人工智能医疗领域.
医学影像可以直观地呈现人体内部的解剖结构与病灶信息, 通过医学图像便可观察病人身体内部器官、组织的变化情况, 是医生在临床诊断和手术规划时的重要辅助手段. 在临床实践中, 往往首先需要根据人体骨骼结构对医学影像中的解剖标志点进行标记, 再通过这些标志点和参考线计算相关的线段长度或测量角度, 进而完成疾病的诊断[2]. 由此可见, 解剖标志点定位的准确性对于临床影像分析具有重要意义. 深度学习为利用人工智能技术进行医学影像标志点定位开辟了新的视角: 深度神经网络通过学习大规模的医学影像数据, 对原始输入图片进行多层非线性变换, 进而实现从数据中归纳出从低级到高级的特征[3], 摒弃了传统方法中根据领域知识手动提取特征的过程, 进而使计算机自动完成解剖标志点定位的影像分析任务.
由此可见, 标准规范的影像数据及数据标注是利用人工智能算法进行医学影像分析发展的基础. 然而, 与自然图像的获取不同, 医学影像的获取十分艰难: 一方面, 训练样本的获取需要成本高昂的专业影像采集设备; 另一方面, 影像数据的标注需要受过专门培训的专业医生参与, 而由于临床、科研任务重, 医疗专家往往没有时间进行大量的数据标注工作. 因此, 医学影像领域本身面临着高质量标注数据匮乏的现实. 在这种情况下, 如何利用医学影像本身所固有的特点, 进一步提升网络的特征提取能力, 更好地挖掘有限数据中更多的特征, 进而实现自动化提取、测量和分析影像学表型, 对于辅助医生快速诊断疾病具有重要的理论价值和现实意义, 不仅可以有效提升医学影像自动读片效率、降低患者就医成本, 还可以在一定程度上缓解地区医疗水平差距大、医疗资源分布不均等社会问题.
本文提出了一种基于拓扑结构约束和特征增强的医学影像标志点定位算法, 使用多任务U-Net网络[4]作为骨干网络, 利用医学影像上各个标志点之间形成的拓扑结构具有变换不变性这一特点, 为网络添加额外的约束; 通过为网络引入多分辨率注意力模块[5], 为不同分辨率下的特征图生成不同分辨率的注意力信息, 使网络加强对重要特征信息的关注, 以避免图像中其他冗余特征的干扰; 此外, 受启发于空洞空间金字塔池化结构(atrous spatial pyramid pooling, ASPP)[6], 本文通过使用多分支空洞卷积来实现增大感受野的目的, 多个分支并行操作可以同时捕获不同尺度的上下文信息, 且空洞卷积的使用也避免了参数量的增加.
本文的贡献可总结为以下4个方面:
(1)考虑到各个解剖标志点之间的拓扑关系存在结构不变性的特点, 本文为深度神经网络加入拓扑结构约束, 并提出结构不变性损失来增强网络的特征提取能力.
(2)通过在网络内部嵌入多分辨率注意力机制, 为不同分辨率的特征图生成不同分辨率的注意力系数图, 帮助网络提取与标志点更相关的特征.
(3)通过在低层特征和高层特征的融合阶段引入多分支空洞卷积层, 在不增加网络参数量的情况下实现增大卷积层感受野的作用范围, 使网络更好地感知上下文信息.
(4)本文在公开数据集上验证了提出算法的有效性. 实验表明, 本文算法在各个指标上超过了当前主流的解剖标志点定位算法.
1 相关工作医学影像解剖标志点定位是临床诊断中非常重要的预处理步骤, 是制定医学治疗计划的基础环节. 在实践中, 往往需要首先对重要的解剖标志点进行准确定位、测量, 进而才能完成如骨龄预测[7]、膝关节手术[8]、骨盆外伤手术[9]及颌面部手术[10]等医学诊疗任务.
传统的解剖标志点定位方法大致可分为两类: 基于模型的方法和基于回归的方法[11]. 基于模型的方法根据预定义的模板迭代地找到最佳的标志点位置, 并通过形状模型调节全局空间形状[12, 13]; 基于回归的方法则直接从图像特征回归标志点坐标[14]. 除此之外, 还有部分学者提出了基于先验知识的边缘检测方法[15, 16], 然而此类方法只能成功检测位于清晰边缘的标志点, 且先验知识的引入使得算法对解剖结构变化较为敏感, 降低了算法的泛化能力.
随着人工智能技术的不断发展, 学术界已经提出了一系列基于深度学习的医学影像解剖标志点定位方法, 此类方法利用深度神经网络自动地学习图像特征并进行标志点定位, 避免了传统方法依赖手工定义特征的弊端, 不仅提升了标志点定位的准确程度, 且减少了定义特征的人力和时间成本.
基于深度学习的解剖标志点定位算法的主要思路是输入图像和对应的标志点坐标, 深度神经网络自动挖掘图像特征并选出图像上概率最大的像素点作为预测坐标, 通过不断降低预测坐标和真实坐标之间的损失以不断优化网络参数, 进而实现标志点自动定位. Zhang等人[17]提出了一种两阶段的深度学习方法, 在第1阶段利用基于回归模型的卷积神经网络学习局部图像和目标解剖标志点之间的内在关联, 在第2阶段进一步建模各个局部图像之间的关联, 进而实现在有限的训练数据上实时联合检测多个解剖标志点. Oh等人[18]提出了一个新的头影测量标志点检测框架, 该框架通过引入局部特征扰动器(local feature perturbator, LFP)和解剖上下文损失(anatomical context loss, AC loss), 使得卷积神经网络在训练期间可以学习更丰富的解剖上下文特征. Chen等人[10]提出了基于自注意力模块的方法, 该方法可以提取语义增强的多层次融合特征, 以提升标志点定位的精度. Payer等人[19]将空间结构信息引入深度神经网络来定位标志点, 从而利用标志点之间的结构关系预测标志点在图片各个像素上出现的概率. 此外, 对于3D影像数据, Yang等人[8]提出可以将3D空间视为2D正交平面的组合, 使用常规卷积神经网络处理3组独立的2D磁共振图像(magnetic resonance imaging, MRI)切片, 并将3D位置定义为3个2D切片的交点.
然而, 现有方法大多具有以下3个主要问题:
(1)标志点定位过程中未能挖掘医学影像本身固有的特点. 医学影像中的解剖结构往往相对固定, 而标志点位置通常与解剖结构密切相关. 以上方法大多利用深度神经网络强大的非线性拟合能力, 未能针对医学影像固有的结构特点进行优化.
(2)特征提取过程中缺乏对显著特征的关注. 深度神经网络提取的特征图中不同位置的特征往往对于预测结果有着不同的重要程度, 有的特征对于预测标志点位置至关重要, 而有的特征对于预测结果作用较小, 属于冗余特征. 冗余特征的存在为实现精准的标志点定位带来额外开销.
(3)特征融合的过程中缺乏对上下文信息的利用. 医学影像中的器官位置及骨骼结构等信息通常有助于判断解剖标志点的位置, 而这些信息则蕴含在更大的感受野之中. 但传统的卷积操作只能对局部特征进行处理, 往往忽略了上下文特征所带来的额外信息.
因此, 本文立足于医学影像本身固有的结构特点, 提出基于拓扑结构约束和特征增强的医学影像标志点定位算法, 利用标志点之间拓扑关系的结构不变性构建额外约束, 通过多分辨率注意力机制和多分支空洞卷积结构提升网络对精细特征的提取能力以及上下文信息的感知能力, 使得网络可以更好地挖掘医学影像的结构和语义, 并提取鲁棒性更强的特征, 进而取得更好的标志点定位效果.
2 网络结构与算法原理考虑到医学影像数据一般较少, 且图像中的语义和结构都对预测标志点坐标具有重要意义, 所以网络预测时不仅需要利用高层语义特征, 还需要同时考虑低层结构特征. 因此, 本文使用可以同时结合低层特征和高层特征的U-Net[20]作为主干网络, 提出一种新的解剖标志点定位模型, 模型整体流程如图1所示.
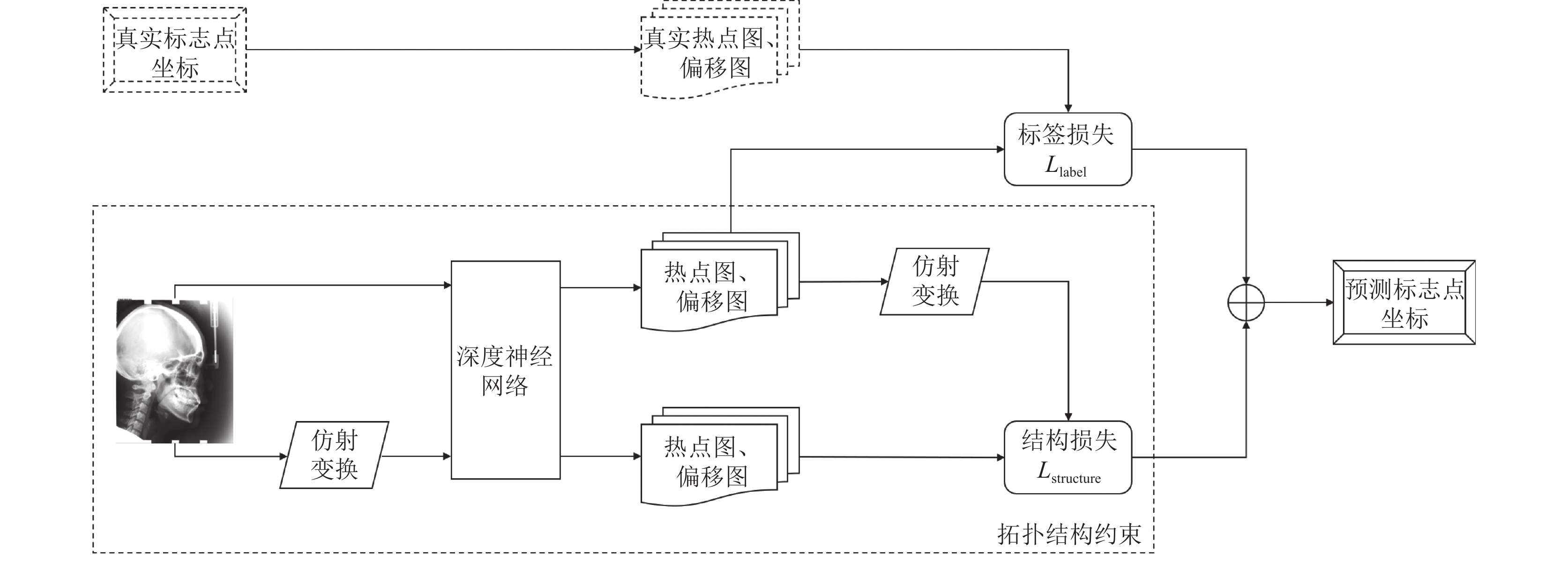
|
图 1 标志点定位整体流程 |
为避免网络直接预测坐标具有较大的偏差, 本文使网络同时预测热点图(heatmap)和偏移图(offset map), 其中热点图表示标志点在图中每个像素位置出现的概率, 偏移图表示标志点与真实标志点之间的坐标偏移, 分为x轴和y轴两个方向. 将输入图片和经仿射变换后的图片输入到深度神经网络, 对于原图使用根据真实坐标计算的热点图和偏移图构建损失函数; 对于仿射变换的图片, 由于标志点之间形成的拓扑结构也将发生相同的仿射变换, 因此, 此时网络预测的热点图和偏移图, 与输入原图时预测的热点图和偏移图之间, 可构成一种额外的结构约束, 使网络更好地学习图像结构, 并基于此构建结构不变性损失, 最后模型将结合两部分损失得到标志点坐标的预测结果.
用于预测热点图和偏移图的深度神经网络结构如图2所示. 对于输入的医学影像, 神经网络提取不同分辨率的特征图, 同时为不同分辨率的特征图计算各个像素的重要程度, 以生成不同分辨率的注意力系数图, 实现加强对重要特征的关注, 削弱冗余特征的影响. 通过将U-Net网络每一层级的普通的上采样部分改为多分支空洞卷积结构, 使得网络可以同时感知多尺度的上下文特征, 进而能够更好地确定标志点位置. 网络将经注意力加权后的低层特征与对应层级的高层特征进行拼接后得到新特征, 将该特征输入解码器经过上采样再得到下一层级的高层特征, 通过上述方式, 网络不仅可以加强对重要特征的关注, 还可以同时考虑不同尺度的上下文信息, 提升了网络的特征提取能力.
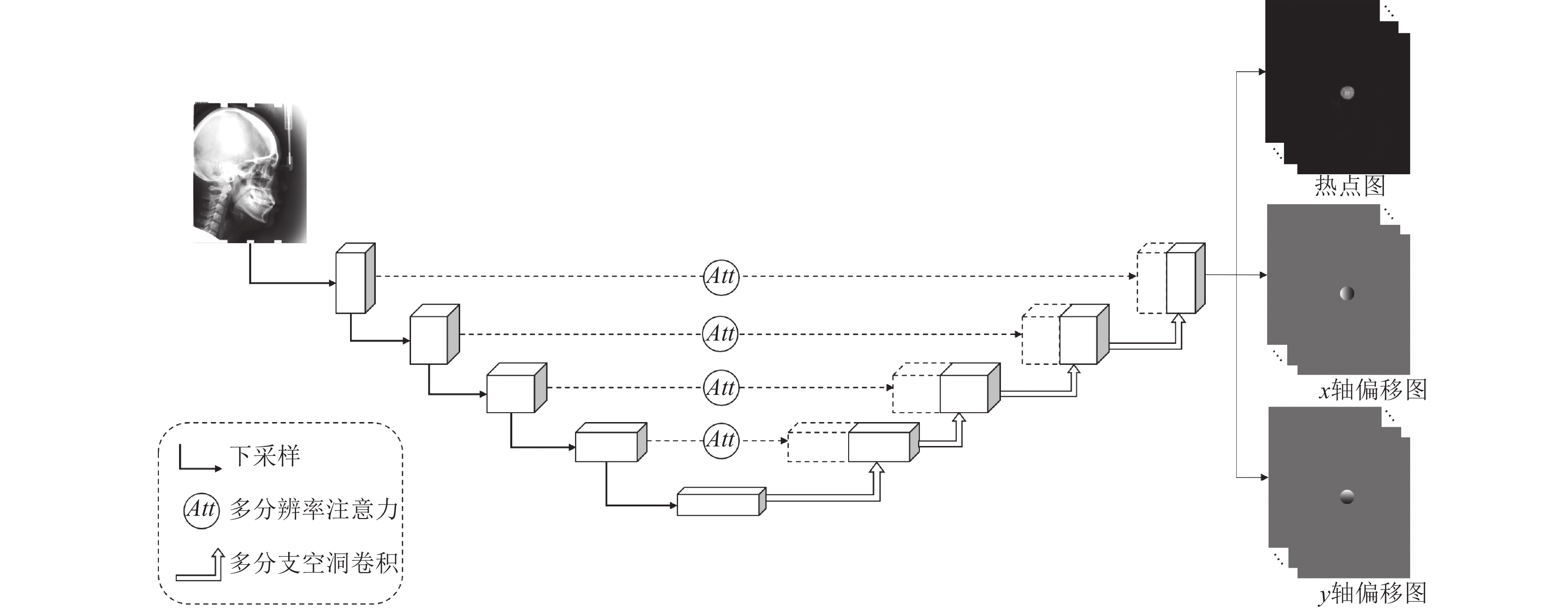
|
图 2 模型结构图 |
2.1 多任务U-Net网络
U-Net网络采用编码器-解码器结构, 编码器部分通过下采样操作得到多个分辨率的低级特征图, 解码器部分通过上采样操作将低级特征图逐层上升为与编码器相应层级相同的分辨率, 直至恢复为原图分辨率. 网络在每一层级采用跳跃连接(skip connection)来结合同层级的高级特征图和低级特征图, 并使用拼接后的特征图作为该层级的上采样层输入. 这种方式结合了低层特征所包含的信息, 避免直接使用最后一层特征进行损失计算, 通过利用多尺度特征有效提升网络精度.
本文使用多任务U-Net网络[4]同时预测热点图和偏移图, 对于图片X中坐标为
| Yhi(x,y)=exp[−12σ2((x−xi)2+(y−yi)2)] | (1) |
其x轴方向的相对偏移
| Yoxi=x−xiσ,Yoyi=y−yiσ | (2) |
本文中使用二值交叉熵损失函数
| Llabel=αLh(Yh,fh(X))+I(Yhi>λ)∑o∈{ox,oy}Lo(Yo,fo(X)) | (3) |
网络输出的热点图、x轴偏移图和y轴偏移图均为19个通道, 每个通道代表对一个标志点的预测信息. 在检测阶段, 根据网络预测的坐标偏移图, 对所有热点图值高于
受启发于等价标志点变换[21], 本文提出基于仿射变换的拓扑结构约束方法, 根据标志点之间拓扑结构的整体性质构建额外的约束, 使网络可以学习到更鲁棒的标志点特征.
给定图像
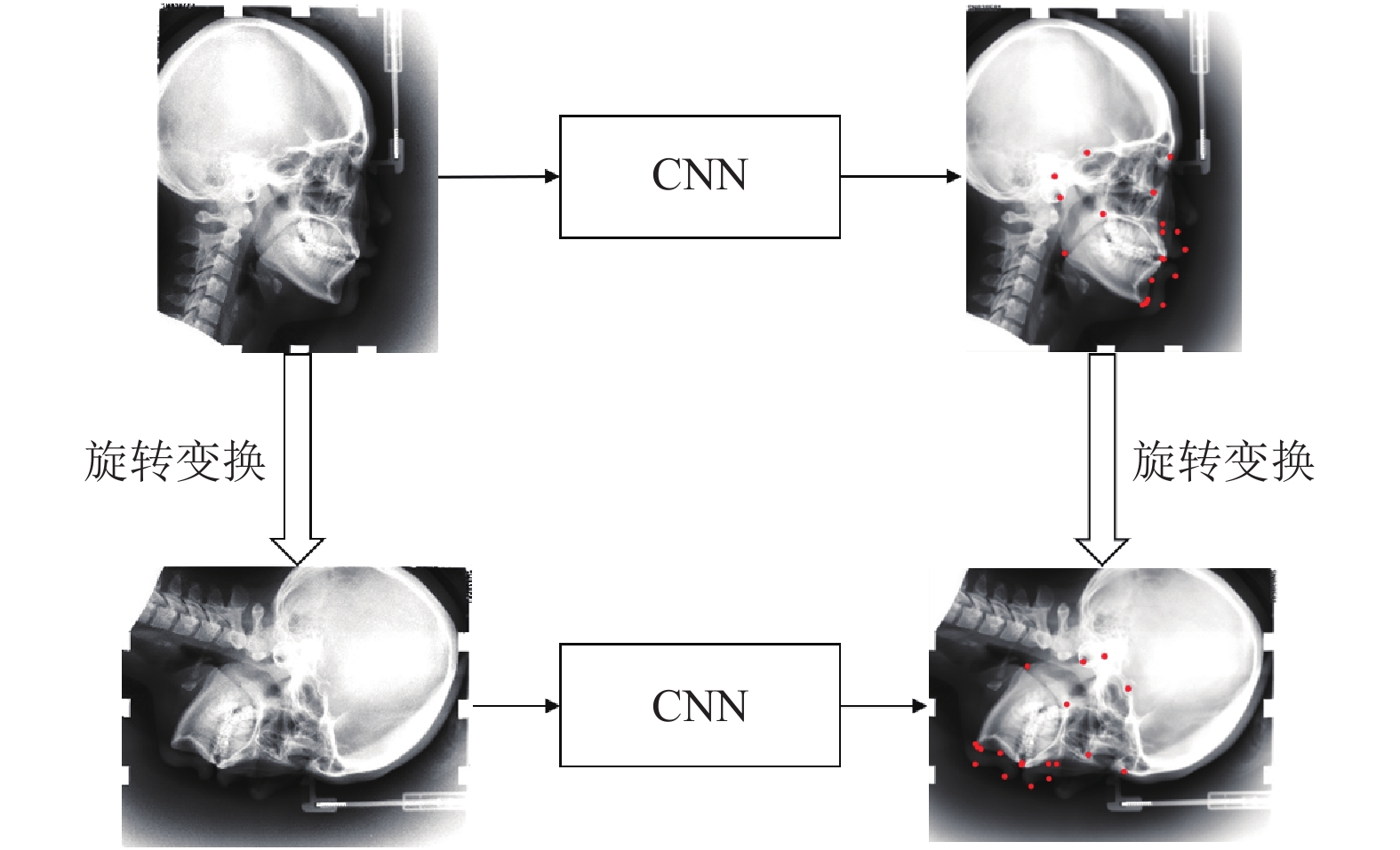
|
图 3 拓扑结构约束(以旋转为例) |
基于此, 利用图像中标志点所形成的拓扑结构经过仿射变换后自相似的性质, 通过分别对比图像变换前后网络输出的热点图以及偏移图之间的相似程度, 可以构成一种额外的约束, 使网络能够捕获标志点之间所蕴含的结构信息, 进而提取鲁棒性更强的特征, 本文中使用MSE损失函数来惩罚图像变换前后网络的预测差异来构建结构不变性损失, 如式(4)所示:
| Laffine=‖ | (4) |
该部分也需同时考虑热点图和偏移图的信息, 因此该部分的损失函数
| \begin{split} {L_{\rm structure}}=&\alpha {L_{\rm affine}}({f^h}, \theta ) \\ & +\mathbb{I}(Y_i^h > \lambda )\sum\limits_{o \in \{ {o_x}, {o_y}\} } {{L_{\rm affine}}({f^o}, \theta )} \end{split} | (5) |
此时, 模型总的损失函数如式(6)所示:
| L = {L_{\rm label}} + {L_{\rm structure}} | (6) |
U-Net网络可以获取不同分辨率的特征图, 进而得到不同比例下的标志点信息, 然而原始的U-Net网络只是简单地把低层特征和高层特征进行拼接, 无法实现加强对图像中重要区域的关注. 本文通过引入注意力门控(attention gate, AG)模块[5], 使编码器获取的低层特征在经过注意力门控调整之后, 再与解码器得到的高层特征进行拼接, 进而加强网络对显著特征的关注, 并抑制不相关的冗余特征的影响.
注意力门控模块搭载于U-Net网络的跳跃连接部分, 其结构如图4所示.
| Att = {{\textit{Sigmoid}}}\left( {{\psi _{1 \times 1}}\left( {{{ReLU}}\left( {{W_{{g_{1 \times 1}}}}\left( g \right) + {W_{{x_{1 \times 1}}}}\left( x \right)} \right)} \right)} \right) | (7) |
将
通过在不同层级的跳跃连接加入注意力模块, 可以获得不同分辨率下的注意力系数图. 在训练过程中, 注意力系数图将不断进行更新, 从而使网络可以逐渐修正重要特征的位置信息, 最终实现对不同分辨率特征图中重要区域的关注, 提升模型的特征提取能力.

|
图 4 注意力门控模块 |
2.4 多分支空洞卷积结构
在对医学影像进行解剖标志点定位时, 通常需要考虑器官位置及骨骼结构等额外信息, 故而需要增大网络的感受野以使模型能够捕获更多的上下文特征. 网络的感受野大小由卷积核的大小所决定, 而更大的卷积核在带来更大感受野的同时也引入了更多的参数. 空洞卷积[6]通过在卷积核中插入权值为0的空洞实现在不增加模型参数量的情况下增大网络感受野, 卷积核各值之间的间距称为扩张率, 对于扩张率为
| {\left( {k + \left( {k - 1} \right)\left( {r - 1} \right)} \right)^2} | (8) |
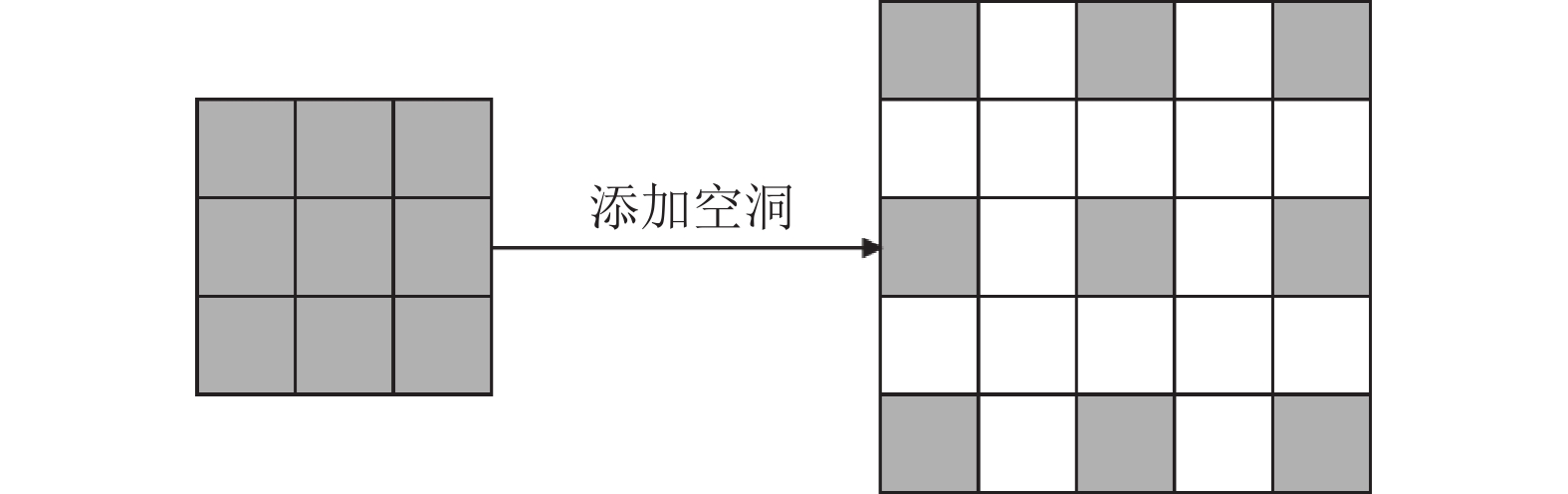
|
图 5 空洞卷积示例 |
本模块旨在利用多尺度感受野以获得更丰富的特征信息, 达到提升标志点定位精度的目的. 受启发于语义分割任务中常用的ASPP结构[6], 本文提出一种适用于医学影像标志点定位的多分支空洞卷积结构, 并将其应用于模型的解码器部分. 每个分支使用不同扩张率的空洞卷积层, 不同的分支提取不同尺度的上下文信息, 使解码器在融合低层特征与高层特征的过程中, 可以在更大程度上捕获像素特征之间的相关性, 进而更好地恢复图像的空间位置等信息.
定义多分支空洞卷积结构的参数为

|
图 6 多分支空洞卷积 |
将不同扩张率的空洞卷积提取的不同尺度的特征图分别经过批归一化和ReLU激活层后进行通道拼接, 再使用1×1卷积将拼接后的新特征降低通道数, 输入接下来的上采样层. 通过这种方式, 不仅解决了特征融合的过程中缺乏对上下文信息的利用这一问题, 并且在不增加参数量和计算量的基础上使网络可以更好地关注多尺度特征, 进而提升网络预测标志点的准确性.
3 实验及分析本文基于公开数据集进行实验, 首先探究损失函数中权重系数
X光头影测量分析可用于正颌外科的辅助诊断, 以弄清畸形的特征, 并进一步进行治疗设计及疗效预测. 本文使用IEEE ISBI 2015 Challenge提供的头影测量标志点定位公开数据集[22]进行实验, 该数据集收集了400位病人的400幅头影测量X光图片, 并分为150张训练集和250张测试集. 数据集中每幅图片的像素大小为1935×2400, 每个像素大小为0.1×0.1 mm2, 包含19个头颅解剖标志点的标记信息, 每个标志点由两名医生进行手动标记和检查. 数据集中的原始头影测量X光片及其上标注的19个标志点如图7所示, 19个标志点的详细信息如表1所示.
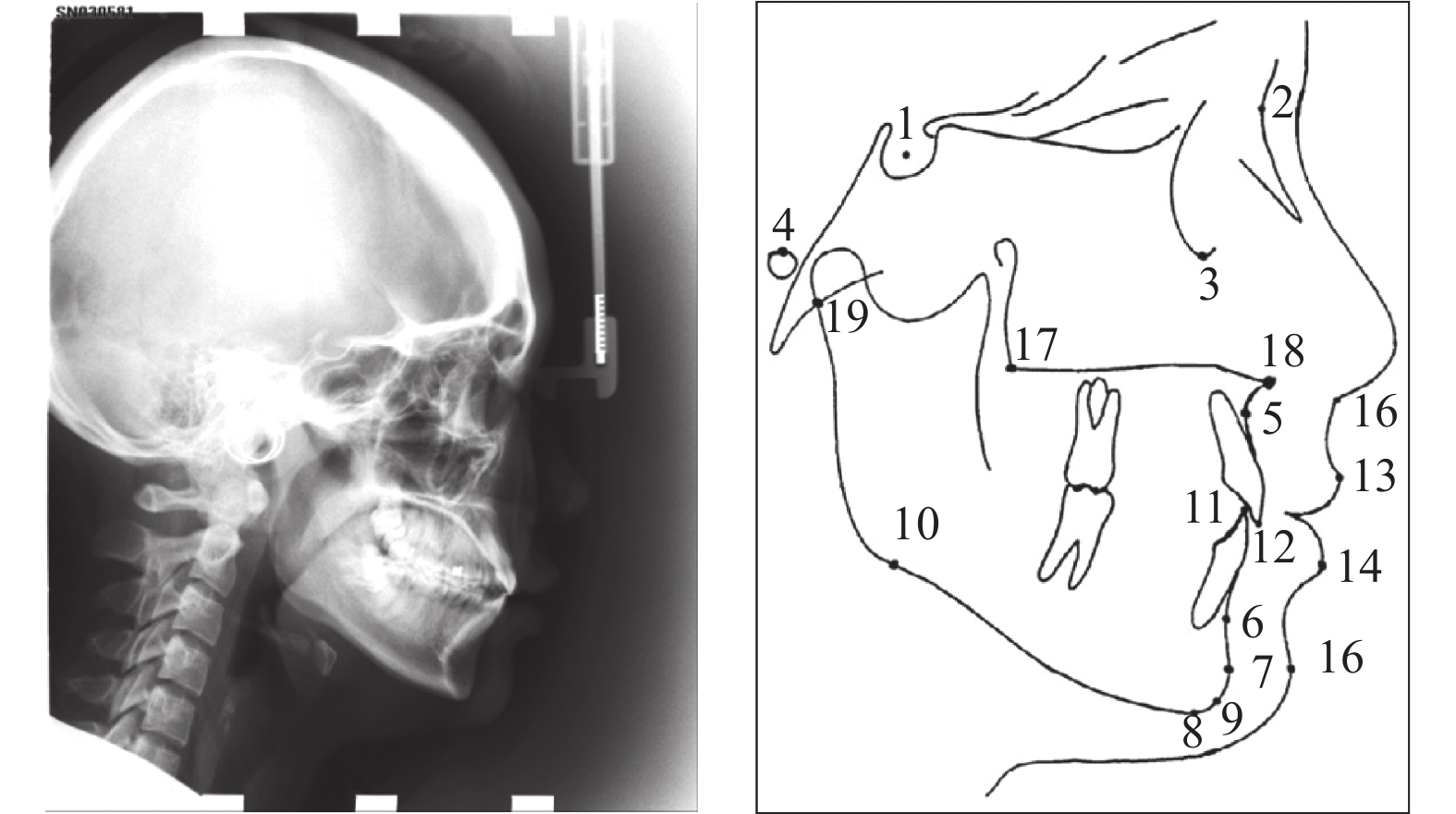
|
图 7 头影测量X光片及19个标志点 |
| 表 1 19个解剖标志点详细信息 |
3.2 实验设置
本文实验中网络的编码器部分使用ImageNet[23]上预训练的VGG-19网络[24]. 由于本文使用的数据集中图片分辨率较高, 因此将图片缩放至800×640大小, 并归一化至[−1, 1]区间. 模型在4个NVIDIA GeForce RTX 2080 Ti GPU上进行训练, 批大小设置为4, 初始学习率设置为0.001, 每150次迭代乘以0.1减小学习率, 权值衰减系数设置为0.000 1, 共迭代300次, 阈值
| 表 2 仿射变换参数 |

|
图 8 仿射变换示例 |
3.3 评价指标
本文中采用IEEE ISBI 2015 Challenge提供的平均径向误差和成功检测率作为评价指标, 以评估算法效果.
(1)平均径向误差(mean radial error,MRE): 用于衡量算法所获取的标志点位置与真实标志点位置之间的欧氏距离, 计算方式如式(9)所示, 其中
| MRE = \frac{{\displaystyle\sum\nolimits_{i = 1}^n {\displaystyle\sum\nolimits_{l = 1}^{19} {R_i^l} } }}{n}, R_i^l = \sqrt {{{(\Delta x_i^l)}^2} + {{(\Delta y_i^l)}^2}} | (9) |
(2)成功检测率(successful detection rate, SDR): 用于衡量算法检测标志点的精确度, 计算方式如式(10)所示, 其含义为如果预测的标志点和真实标志点之间欧氏距离不大于z mm, 则此标志点的检测将被视为精确检测, 否则被视为错误检测. 该指标越高表示算法的成功检测率越高. 为方便同主流方法进行对比, 本实验中使用与其他主流方法中相同的误差范围z, 即2 mm、2.5 mm、3.0 mm、4.0 mm.
| {P}_{{\textit{z}}}\text=\frac{\text{number\;of\;accurate\;detection}}{\text{number\;of\;detection}}\times 100\text{%} | (10) |
本部分实验用于探究损失监督学习与自监督学习的损失函数中权重系数
| 表 3 损失函数权重系数实验结果 |
实验结果表明,
本部分实验用于探究多分支空洞卷积模块的不同扩张率组合对实验结果的影响, 其中空洞卷积的卷积核大小设置为3, 即参数
从实验结果可以看出, 较大的扩张率对于模型性能有一定的提升作用, 其原因在于较大的扩张率可以更好地扩大模型的感受野, 使模型可以更多地感知上下文特征, 避免了普通卷积核仅使用局部特征带来的局限性. 当参数
在头影测量标志点定位的公开数据集上, 将本文所提出的算法与当前主流算法进行对比, 实验结果如表5所示, 表中加粗数字表示最优结果.
| 表 4 多分支空洞卷积参数实验结果 |
| 表 5 不同算法对比实验结果 |
从实验结果可以看出, 本文提出的模型效果在各个指标上均超过了当前主流算法, 这得益于模型在加入拓扑结构约束后, 能够更好地学习到医学影像中蕴含的解剖结构信息, 且多分辨率注意力机制和多分支空洞卷积的使用也使得模型可以更有效地提取图像特征, 从而提升标志点定位的准确程度.
3.7 消融实验本部分实验用于探究模型中各个模块对结果的作用, 以探究拓扑结构约束、多分辨率注意力机制和多分支空洞卷积对标志点自动定位效果的影响, 实验结果如表6所示, 表中加粗数字表示最优结果.
| 表 6 消融实验结果 |
从实验结果可以看出, 加入拓扑结构约束后, 模型效果优于未使用拓扑结构约束的效果, 这表明拓扑结构约束对于模型性能有提升作用, 且SDR指标的提高表明拓扑结构约束也提升了模型的鲁棒性; 加入多分辨率注意力机制后, 模型在各个指标上的表现均有提高, 这表明该机制有效增强了对图像中重要特征的关注, 并提升了模型预测的准确率; 引入多分支空洞卷积之后, 模型的性能进一步提高, 说明不同尺度的上下文信息有助于模型预测标志点位置. 消融实验结果验证了拓扑结构约束、多分辨率注意力机制和多分支空洞卷积对提升模型性能的有效性.
3.8 结果展示19个解剖标志点各自的MRE如图9所示, 从图中可以看出, 所有点的MRE均在2.5 mm以内, 17个点的MRE达到2.0 mm以内, 且1、7、8、9、12、15号标志点的MRE可以达到1.0 mm以内, 由此可见本文算法具有较高的定位精度, 而如4、16号标志点的MRE较高, 这可能是由于不同人的个体差异导致这两个标志点在不同人的影像上位置有一定偏差, 对这些标志点进行有针对性的优化也是未来的改进方向之一.
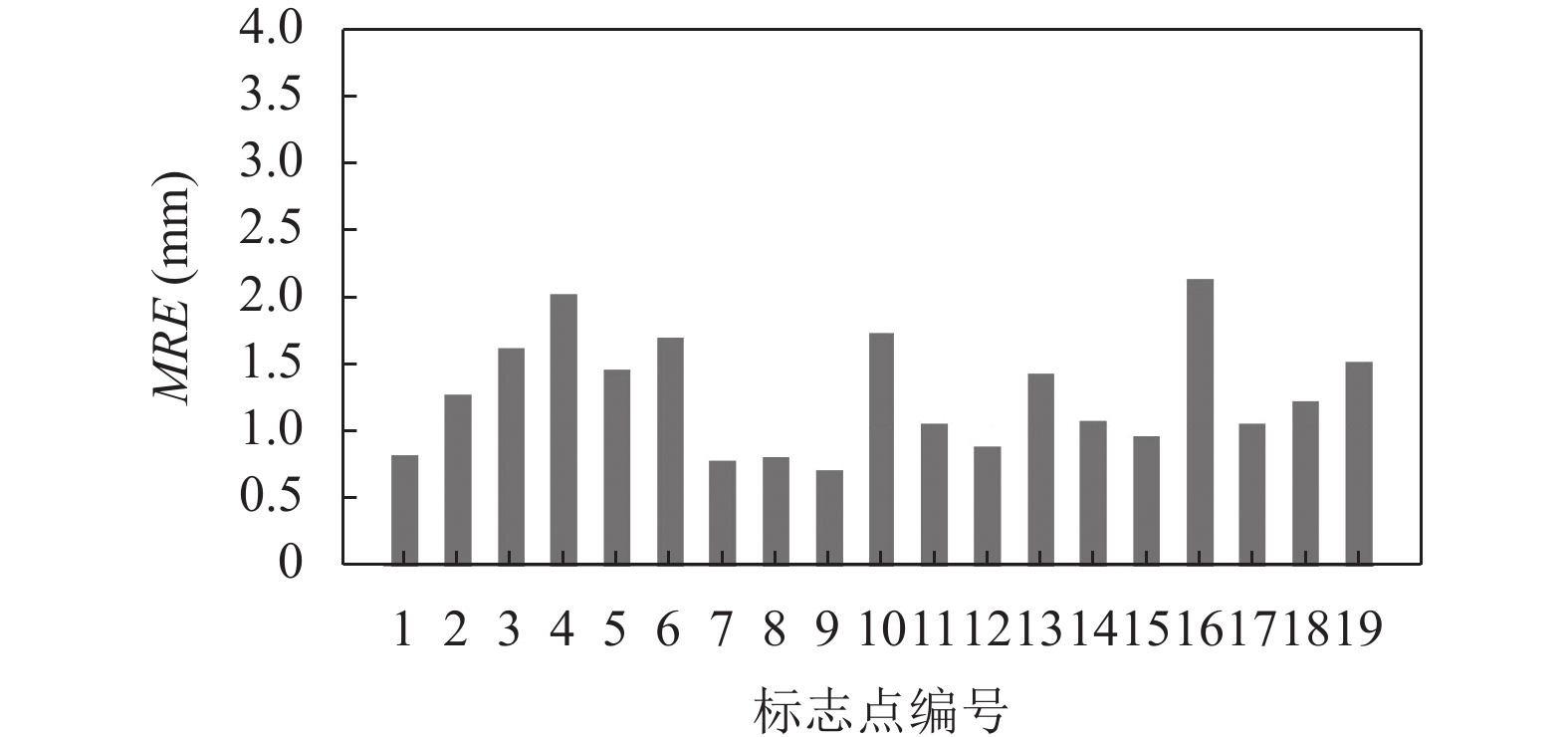
|
图 9 19个解剖标志点的MRE |
图10为部分测试集图片的解剖标志点定位效果展示, 第1排为原始图像, 第2排为真实标志点与预测标志点之间的误差对比图像, 其中蓝色为真实标志点, 绿色为预测标志点.
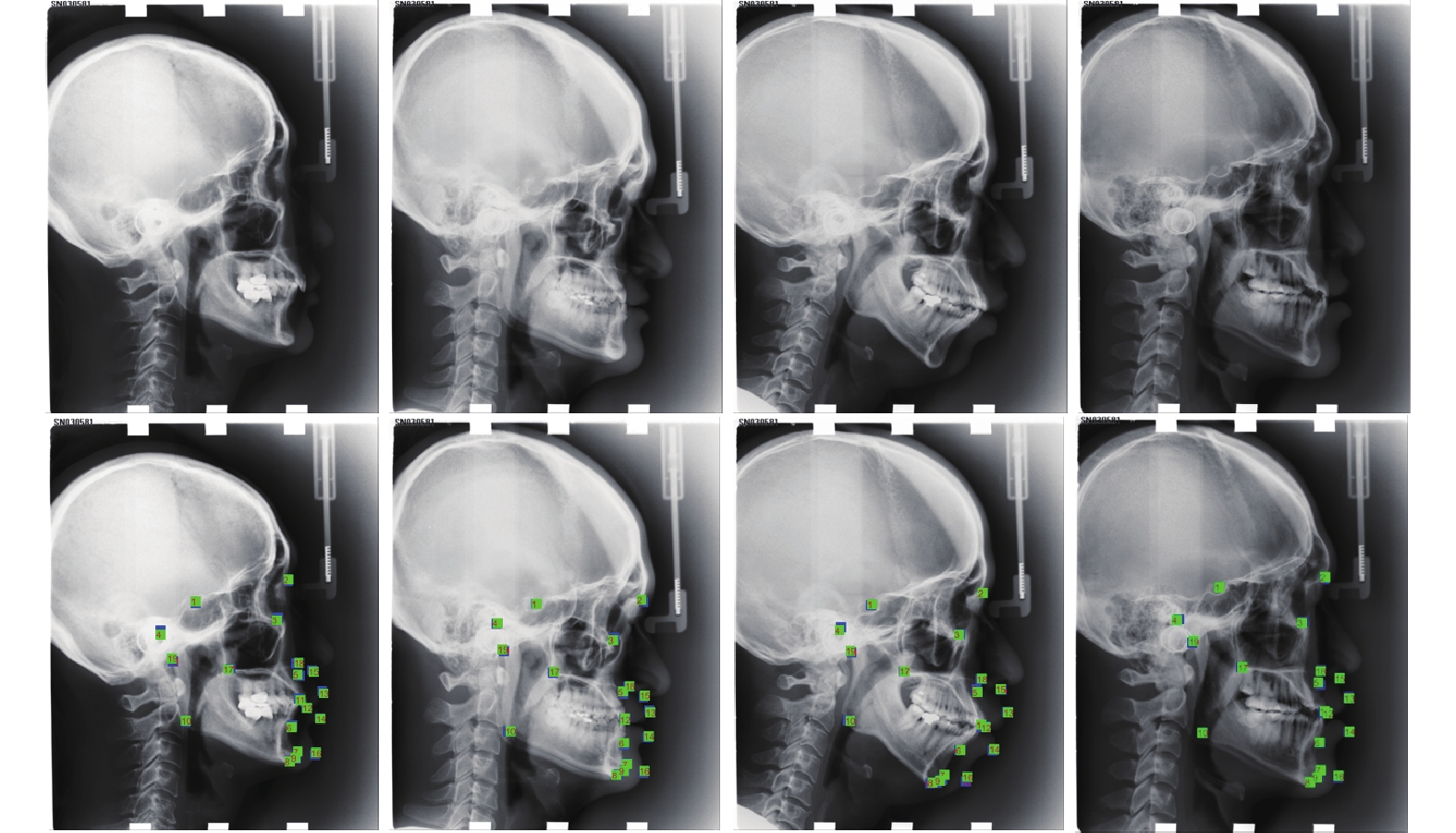
|
图 10 解剖标志点定位效果展示(真实标志点: 蓝; 预测标志点: 绿) |
由图10可见, 虽然不同影像中颅骨的形态有所不同, 但本文算法依然可以在较低的误差范围内定位标志点位置, 由此可见本文算法在医学影像解剖标志点定位任务上的优越性能.
4 总结医学影像标志点定位的准确性对于临床诊疗具有重要意义. 本文针对医学影像标志点定位任务当前所面临的问题, 提出了一种基于拓扑结构约束和特征增强的标志点定位算法, 根据标志点所形成的拓扑结构具有结构不变性的特点, 为网络添加额外约束, 使网络提取更鲁棒的特征, 以实现网络对解剖结构信息的利用; 多分辨率注意力机制的引入使模型更加关注重要的图像区域, 网络自动学习注意力系数图并为不同的空间特征分配不同的权重, 以提升模型的特征提取能力; 多分支空洞卷积结构在不增加模型参数量和计算量的情况下增大网络的感受野, 实现对不同尺度上下文信息的融合利用, 进一步提升了模型性能. 基于公开数据集的实验表明, 本文提出的算法获得了较高的识别精度, 为目前公开发表的论文中性能较优的水平, 具有定位效果好、鲁棒性强的特点. 未来在获得更多类型的医学影像数据后, 将在更广泛的数据集上验证本文算法的有效性.
| [1] |
上海交通大学人工智能研究院, 上海市卫生和健康发展研究中心, 上海交通大学医学院, 等. 中国人工智能医疗白皮书. https://www.080910t.com/wp-content/uploads/2020/03/2019-中国人工智能医疗白皮书.pdf. (2019-01-09).
|
| [2] |
Leonardi R, Giordano D, Maiorana F, et al. Automatic cephalometric analysis: A systematic review. The Angle Orthodontist, 2008, 78(1): 145-151. DOI:10.2319/120506-491.1 |
| [3] |
张蕊, 李锦涛. 基于深度学习的场景分割算法研究综述. 计算机研究与发展, 2020, 57(4): 859-875. DOI:10.7544/issn1000-1239.2020.20190513 |
| [4] |
Yao QS, He ZC, Han H, et al. Miss the point: Targeted adversarial attack on multiple landmark detection. Proceedings of the 23rd International Conference on Medical Image Computing and Computer Assisted Intervention. Lima: Springer, 2020. 692–702.
|
| [5] |
Oktay O, Schlemper J, Folgoc LL, et al. Attention U-Net: Learning where to look for the pancreas. arXiv: 1804.03999, 2018.
|
| [6] |
Chen LC, Papandreou G, Kokkinos I, et al. DeepLab: Semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFS. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4): 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [7] |
Gertych A, Zhang AF, Sayre J, et al. Bone age assessment of children using a digital hand atlas. Computerized Medical Imaging and Graphics, 2007, 31(4–5): 322-331. DOI:10.1016/j.compmedimag.2007.02.012 |
| [8] |
Yang D, Zhang ST, Yan ZN, et al. Automated anatomical landmark detection on distal femur surface using convolutional neural network. Proceedings of 2015 IEEE 12th International Symposium on Biomedical Imaging (ISBI). Brooklyn: IEEE, 2015. 17–21.
|
| [9] |
Bier B, Unberath M, Zaech JN, et al. X-ray-transform invariant anatomical landmark detection for pelvic trauma surgery. Proceedings of the 21st International Conference on Medical Image Computing and Computer Assisted Intervention. Granada: Springer, 2018. 55–63.
|
| [10] |
Chen RN, Ma YX, Chen NL, et al. Cephalometric landmark detection by attentive feature pyramid fusion and regression-voting. Proceedings of the 22nd International Conference on Medical Image Computing and Computer Assisted Intervention. Shenzhen: Springer, 2019. 873–881.
|
| [11] |
Chen RN, Ma YX, Liu LJ, et al. Semi-supervised anatomical landmark detection via shape-regulated self-training. Neurocomputing, 2022, 471: 335-345. DOI:10.1016/j.neucom.2021.10.109 |
| [12] |
Rueda S, Alcaniz M. An approach for the automatic cephalometric landmark detection using mathematical morphology and active appearance models. Proceedings of the 9th International Conference on Medical Image Computing and Computer-assisted Intervention. Copenhagen: Springer, 2006. 159–166.
|
| [13] |
Kafieh R, Mehri A, Sadri S. Automatic landmark detection in cephalometry using a modified active shape model with sub image matching. Proceedings of 2007 International Conference on Machine Vision. Isalambad: IEEE, 2007. 73–78.
|
| [14] |
Lindner C, Bromiley PA, Ionita MC, et al. Robust and accurate shape model matching using random forest regression-voting. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015, 37(9): 1862-1874. DOI:10.1109/TPAMI.2014.2382106 |
| [15] |
Thuong LT, Hieu PC. An approach for efficient detection of cephalometric landmarks. Procedia Computer Science, 2014, 37: 293-300. DOI:10.1016/j.procs.2014.08.044 |
| [16] |
Jetwani DP, Kumar S, Sardana HK. Cephalometric landmark identification using fuzzy wavelet edge detector. Proceedings of 2011 IEEE International Symposium on Medical Measurements and Applications. Bari: IEEE, 2011. 349–353.
|
| [17] |
Zhang J, Liu MX, Shen DG. Detecting anatomical landmarks from limited medical imaging data using two-stage task-oriented deep neural networks. IEEE Transactions on Image Processing, 2017, 26(10): 4753-4764. DOI:10.1109/TIP.2017.2721106 |
| [18] |
Oh K, Oh IS, Le VNT, et al. Deep anatomical context feature learning for cephalometric landmark detection. IEEE Journal of Biomedical and Health Informatics, 2021, 25(3): 806-817. DOI:10.1109/JBHI.2020.3002582 |
| [19] |
Payer C, Štern D, Bischof H, et al. Integrating spatial configuration into heatmap regression based CNNs for landmark localization. Medical Image Analysis, 2019, 54: 207-219. DOI:10.1016/j.media.2019.03.007 |
| [20] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [21] |
Honari S, Molchanov P, Tyree S, et al. Improving landmark localization with semi-supervised learning. Proceedings of 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 1546–1555.
|
| [22] |
Wang CW, Huang CT, Lee JH, et al. A benchmark for comparison of dental radiography analysis algorithms. Medical Image Analysis, 2016, 31: 63-76. DOI:10.1016/j.media.2016.02.004 |
| [23] |
Deng J, Dong W, Socher R, et al. Imagenet: A large-scale hierarchical image database. Proceedings of 2009 IEEE Conference on Computer Vision and Pattern Recognition. Miami: IEEE, 2009. 248–255.
|
| [24] |
Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition. arXiv: 1409.1556, 2014.
|
| [25] |
Urschler M, Ebner T, Štern D. Integrating geometric configuration and appearance information into a unified framework for anatomical landmark localization. Medical Image Analysis, 2018, 43: 23-36. DOI:10.1016/j.media.2017.09.003 |
 2022, Vol. 31
2022, Vol. 31



