脑胶质瘤是最常见的原发性颅内肿瘤, 占恶性脑肿瘤的81%, 虽然相对罕见, 但却造成了显著的死亡率[1]. 为了进行有效的治疗, 获取清晰和准确的医学影像是前提. 由于MR图像可以提供大脑精细的细节结构, 且不同模态(T1、T2、T1CE、FLAIR)的MR图像可以从不同的角度反映脑肿瘤引起的组织变化, 因此常用于脑肿瘤的检测和脑部疾病的诊断[2, 3]. 然而, 在医学上获取多模态数据存在着费时、价格昂贵和设备缺乏等问题, 使得多模态数据存在缺失[4, 5]. 因此, 如何通过其他简单的方法获得多模态数据成为了近来研究的热点.
近年来, 随着生成对抗网络(generative adversarial networks, GAN)[6]的提出, 引起了学者们对图像生成的广泛研究, 尤其是在图像缺乏的医学领域[7]. 基于GAN, 研究人员提出了多种图像合成方法. Radford等人提出了深度卷积生成对抗网络(deep convolutional generative adversarial networks, DCGAN)[8], 它将卷积神经网络应用于GAN中的生成器和判别器, 使得GAN可以更好地生成图像数据. Frid-Adar等人[9]基于DCGAN生成肝脏CT不同类别的病变斑块, Bermudez等人[10]基于DCGAN在少量数据集上生成了人眼无法辨别真假的脑MR图像. 由于DCGAN无法控制输出图像的类别, Mirza等人提出了条件生成对抗网络(conditional generative adversarial networks, CGAN)[11], 通过在生成器中加入了条件输入, 从而有效控制输出不同类别的数据. 在CGAN中, pix2pix[12]将输入的噪声向量和条件统一用一张图像来代替, 从而为配对的图像转换领域提供了一个通用的框架. 例如, Dar 等人[13]采用pix2pix来实现T1和T2脑MR图像的跨模态转换, Yu等人[14]通过整合边缘信息来改善pix2pix的MR图像生成结果. 为了提高图像生成质量, Pix2PixHD[15]在构造判别器时考虑了多尺度因素, 为每一个尺度的特征图分别训练一个判别器. 鉴于pix2pix无法解决无配对的图像转换问题, Zhu等人提出了CycleGAN[16], 它无需配对的图像便可以实现跨模态转换. 例如, Wolterink等人[17]使用CycleGAN将2D MR图像转换为CT图像; Chartsias等人[18]将CycleGAN用于未配对的图像到图像转换, 将心脏CT切片和分割图像生成心脏MR图像及其相应的分割掩模; Jiang等人[19]提出了一种肿瘤感知损失函数用于CycleGAN, 实现CT图像生成MR图像.
但是, 上述的方法只解决了诸如数据稀缺和单一模态的图像生成问题, 而MR医学图像通常包含4种模态, 如果面临另外几种(不只一种)模态缺失的情况, 利用传统方法, 则需要通过多个网络来分别生成, 而训练多个网络必然带来计算资源浪费和可扩展性差的问题. 另一方面, GAN的训练本身又具有脆弱和不稳定性[20, 21].
另一方面, 虽然卷积神经网络在诸多领域中取得了巨大成功, 但其性能的提升往往源于模型深度与宽度的增加, 这将导致计算量的提升. 近年来, 动态卷积[22]由于其能够在不增加网络深度或宽度的情况下提高模型的表达能力, 因此一经推出, 便得到了广泛研究[23-26]. 动态卷积可以自适应的为每个输入学习自己的卷积核参数, 如Yang等人[23]、Chen等人[24]和Zhang等人[25]提出通过注意力机制[27]来自适应结合提前初始化好的卷积权重、参数与注意力网络同时学习; Ma等人[26]提出在动态卷积CondConv[23]与注意力机制[27]的基础上直接生成卷积权重. 近年来, 动态卷积已广泛应用于分类[23–26]、分割[28–30]、检测[23, 24,26,31]和语言翻译[32]等领域, 但其在图像生成方面应用还很少.
基于上述问题, 我们将动态卷积和GAN结合用于多模态图像的生成, 提出了一种动态生成对抗网络(dynamic GAN, DyGAN). 新网络通过在卷积核上加入条件, 来控制生成不同模态的图像所对应的卷积核参数, 实现由一种模态同时生成多种模态的MR图像. 同时, 为了提高图像生成的质量, 本文又提出了多尺度判别器, 通过在一个网络中同时分别进行多种不同深度的下采样, 实现了综合判别多个尺度特征图的真假. 采用BRATS19数据集进行图像生成实验, 实验结果证明了本文方法的有效性.
2 相关工作 2.1 pix2pixpix2pix在有配对的图像转换领域, 可以一对一的将一种模态转换成另一种模态. 它由一个生成器G和一个判别器D组成, 其训练过程如图1所示. 在训练图像x生成y的过程中, x输入生成器G, 生成器G则最大可能的生成和它所对应真实图像y相似的图像G(x), 判别器D不仅负责判别生成的图像G(x)是否和y相似, 而且还要判别它是否和x配对. 这一判别过程是通过分别将真实图像x和生成图像G(x)、真实图像x和真实图像y输入判别器D实现的, 只有输入 {x, y}的组合, 判别器才会判断为真.
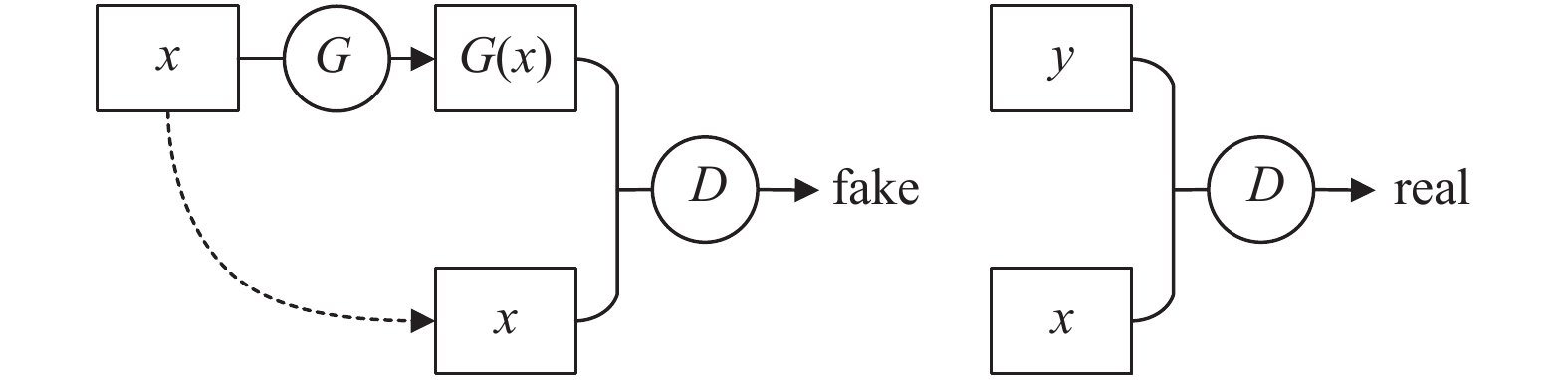
|
图 1 pix2pix训练过程 |
但是, pix2pix只能一对一的将一种模态转换成另外一种模态, 如果面临多种模态缺失的问题, 想要同时生成多种模态的MR图像, 则需要训练多个pix2pix网络才能实现. 如图2(a)所示, 如果将T2模态转化成其他3种模态(FLAIR, T1, T1CE), 则需要分别训练3个pix2pix网络.
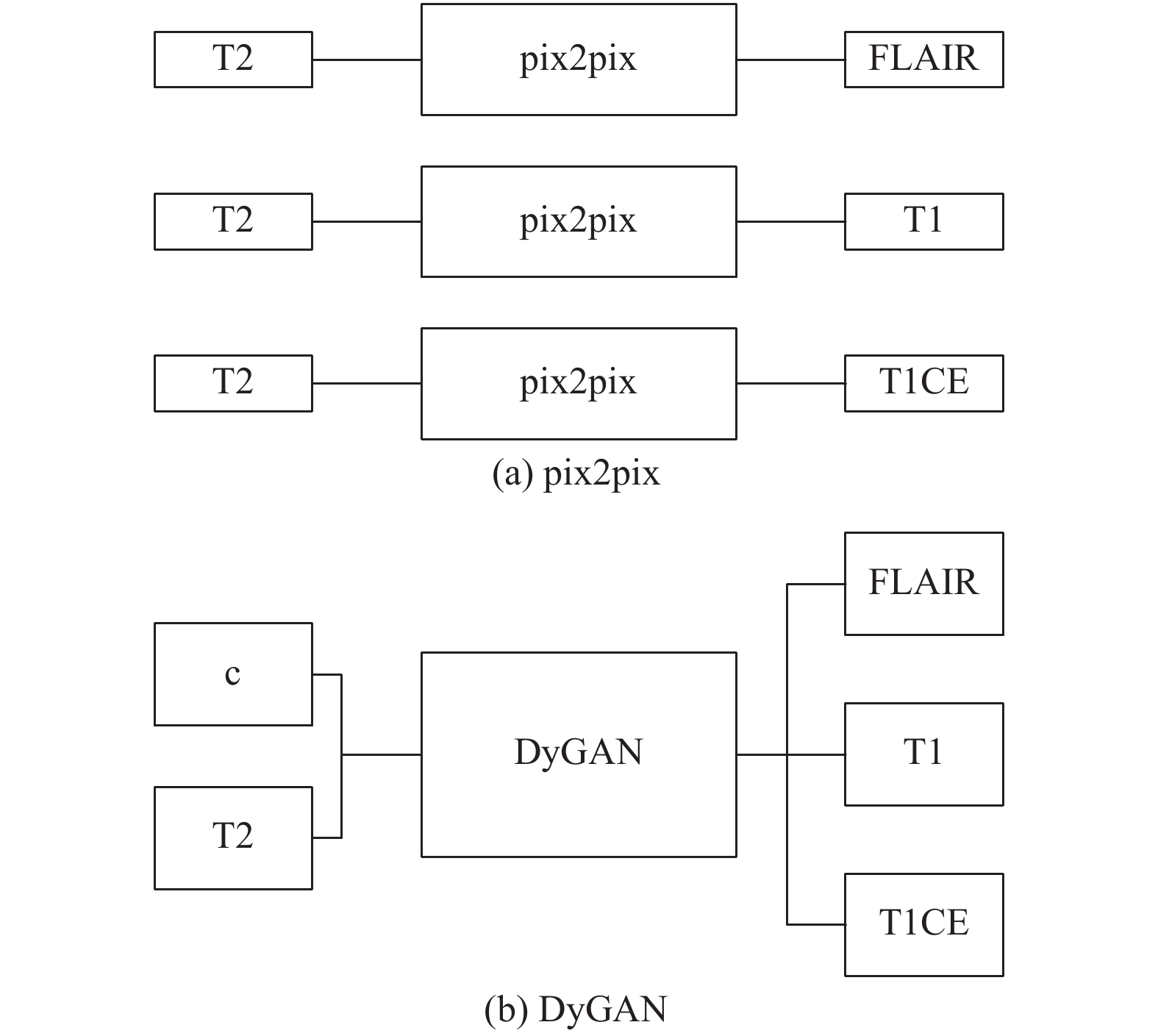
|
图 2 DyGAN与pix2pix多模态转化区别 |
2.2 CondConv
现有的动态卷积大致可以分为两类. 第一, 通过注意力机制来自适应的结合提前初始化好的卷积参数来完成动态变更, 如Yang等人[23]和Chen等人[24]提出的方法. 第二, 直接生成卷积参数, 如Ma等人[26]提出的方法. 若在生成对抗网络中采用第2种方法, 会导致参数生成网络最后一层的神经元个数过多. 因此, 本文采用第1种动态卷积的方法.
在第1种动态卷积的方法中, 最具代表性的是Yang等人[23]提出的CondConv, 其结构如图3所示. 前一层输出的特征图(prev layer output)经过全局平均池化(global average pooling, GAP)后, 再经过注意力层(attention), 最终生成w1, w2, w3, ···, wn分别对应的权重α1, α2, α3, ···, αn. 其中, w1, w2, w3, ···, wn为提前初始化好的卷积参数, 它们分别与对应的权重相乘然后再相加, 结合成一组新的参数W. 最后, 前一层的输出再与生成的W参数做卷积运算. 整个过程中w1, w2, w3, ···, wn与注意力层同时学习和训练, 因而对于每一个不同的输入样例都会生成自己特定的权重.
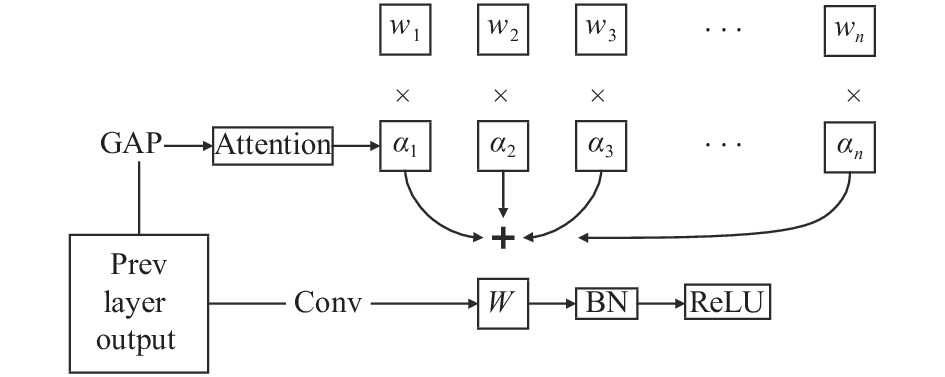
|
图 3 CondConv结构 |
3 动态生成对抗网络(DyGAN) 3.1 网络结构
在配对的图像转换领域, pix2pix虽然可以一对一的将一种模态的图像转换成另外一种模态, 但它却无法同时生成多种模态的图像. 针对该问题, 本文在pix2pix的基础上, 提出了动态生成对抗网络DyGAN, 通过结合动态卷积, 并加入任务标签这一条件, 将不同的模态转换定义为不同的任务, 来控制生成器输出不同模态的图像. 如图2(b)所示, 如果将T2模态同时转化成其他3种模态, 只需在DyGAN的输入中加入“c”这一条件, 便可同时生成3种所需的模态. DyGAN具体训练过程如图4所示, 图中以T2模态生成其他模态为例.
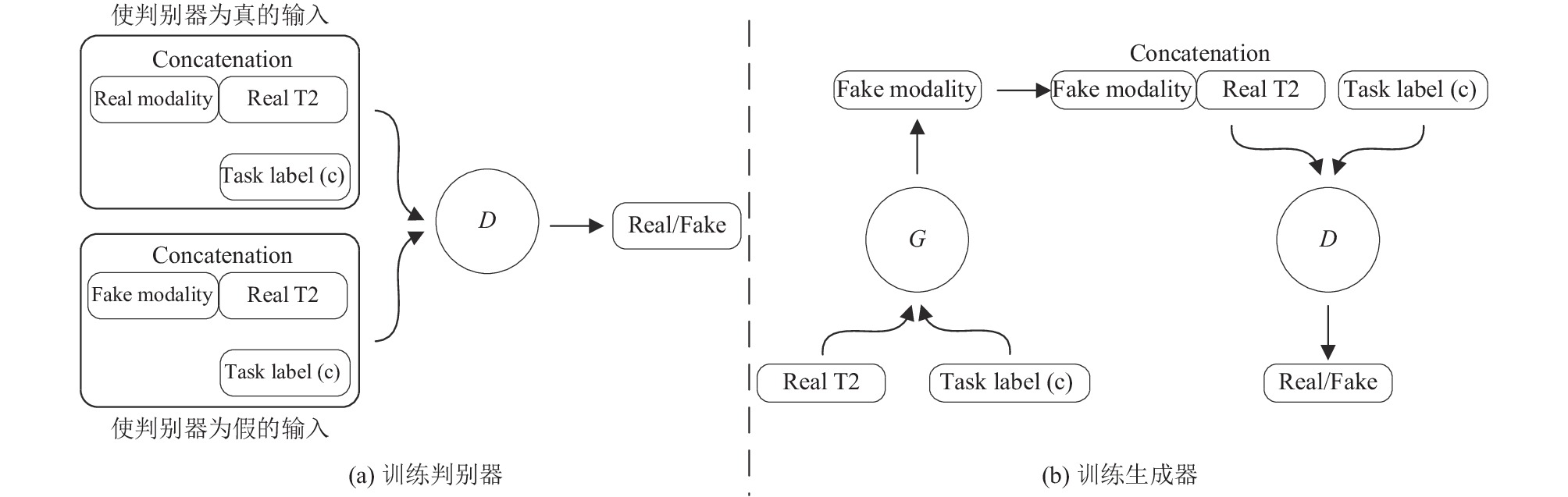
|
图 4 DyGAN训练过程 |
在训练判别器中, 上方矩形框中为使判别器判断为真的输入, 下方矩形框中为使判别器判断为假的输入. 与pix2pix类似, 其他模态真实的图像(real modality)与真实的T2 (real T2)相结合, 同时输入判别器才为真. 相反, 生成器生成其他模态的图像(fake modality)与真实的T2 (real T2)相结合, 输入判别器为假. 与pix2pix不同的是判别器D还需要在任务标签(task label)的控制下, 判断不同任务下图像的真假. 在训练生成器中, 在当前任务(task label)下, 将真实的T2 (real T2)输入生成器G, 生成器G使生成的图像(fake modality)越来越接近真实, 从而使判别器判断为真.
3.2 生成器网络框架 3.2.1 多模态标签在多任务多模态医学图像生成中, 比如T2同时生成其他3种模态T1、T1CE和FlAIR, 是同一张图像同时生成3种不同模态的医学图像. 为此, 我们通过对不同模态的图像编码, 并将该编码作为输入的条件. MR医学图像通常有4种模态, 由其中一种模态转化成另外3种模态可以有12种任务组合, 我们分别对这12种任务分别进行one-hot编码, 如表1所示. 与StarGAN[33]和MGAN[4]在GAN中加入条件不同, 它们都是在输入层直接控制在不同条件下的图像生成, 所有输入共享一套卷积核参数. 在本文方法中, 不同条件控制生成的是不同的卷积核, 不同模态的图像均有自己的卷积核参数.
| 表 1 多模态任务编码 |
3.2.2 DyConv结构
本文提出的多模态动态卷积结构如图5所示. 其中, 图5(a) 为动态卷积CondConv结构, 图5(b)为任务标签模块, 完成将不同的模态转换定义为不同的任务. 将编码好的任务标签c分别与全局平均池化后的张量进行拼接, 再经过注意力层, 可以控制生成不同任务所对应的动态卷积参数.

|
图 5 DyConv结构 |
3.2.3 编码解码网络
本文生成器采用U-Net网络结构[34], 该网络分为4次下采样和4次上采样两部分. 为了生成不同模态的图像, 本文考虑采用DyConv来替代U-Net中的卷积. 首先, 进行了3种不同情况的测试: 采用DyConv替代下采样卷积操作、采用DyConv替代上采样卷积操作、采用DyConv同时替代上下采样卷积操作3种情况, 结果表明后两种情况的效果最好, 而第2种情况的参数更少(可参见第4.4节实验部分). 其次, 因为输出的图像仅有一种模态, 因此我们认为下采样过程仅采用卷积操作即可, 在上采样过程采用动态卷积, 即可实现生成不同模态的任务. 为此, 在本文的结构中, 我们采用DyConv替代上采样过程中的卷积操作. 其结构如图6所示.
3.3 判别器网络框架在原生成对抗网络中, 判别器仅输出一个值(真或假)来对整幅图像进行评价, 显然缺乏对生成图像细节的评价. 为了解决这一问题, pix2pix提出了PatchGAN[12], 通过把整幅图像平均分成多个块, 首先针对每个块作评价, 然后取所有块评价的均值作为最终评价结果, 从而在一定程度上考虑了生成图像的细节. Pix2pixHD[15]在pix2pix的基础上, 设计了3个具有相同结构的判别器, 在3个不同尺度上分别判断生成图像的质量, 但显然存在复杂度高的问题.
为此, 在Pix2pixHD的基础上, 本文提出在一个判别器中完成对3个不同大小尺度的特征图进行判别, 其结构如图7所示. 首先, 生成的图像和真实的图像在通道上进行拼接, 一起输入判别器, 并进行3次步幅为2的下采样操作. 其次, 按照图7虚线框中所示的操作实现多尺度处理. 在虚线框所示的结构中, 第1层直接经过步幅为1的卷积操作. 第2层经过一次步幅为2的下采样后再经过一次步幅为1的卷积操作, 然后与第1层进行拼接. 最后一层经过两次步幅为2的下采样操作, 再经过一次步幅为1的卷积, 然后与前两层进行拼接. 第三, 经过步幅为1的卷积输出一个单通道的判别结果.
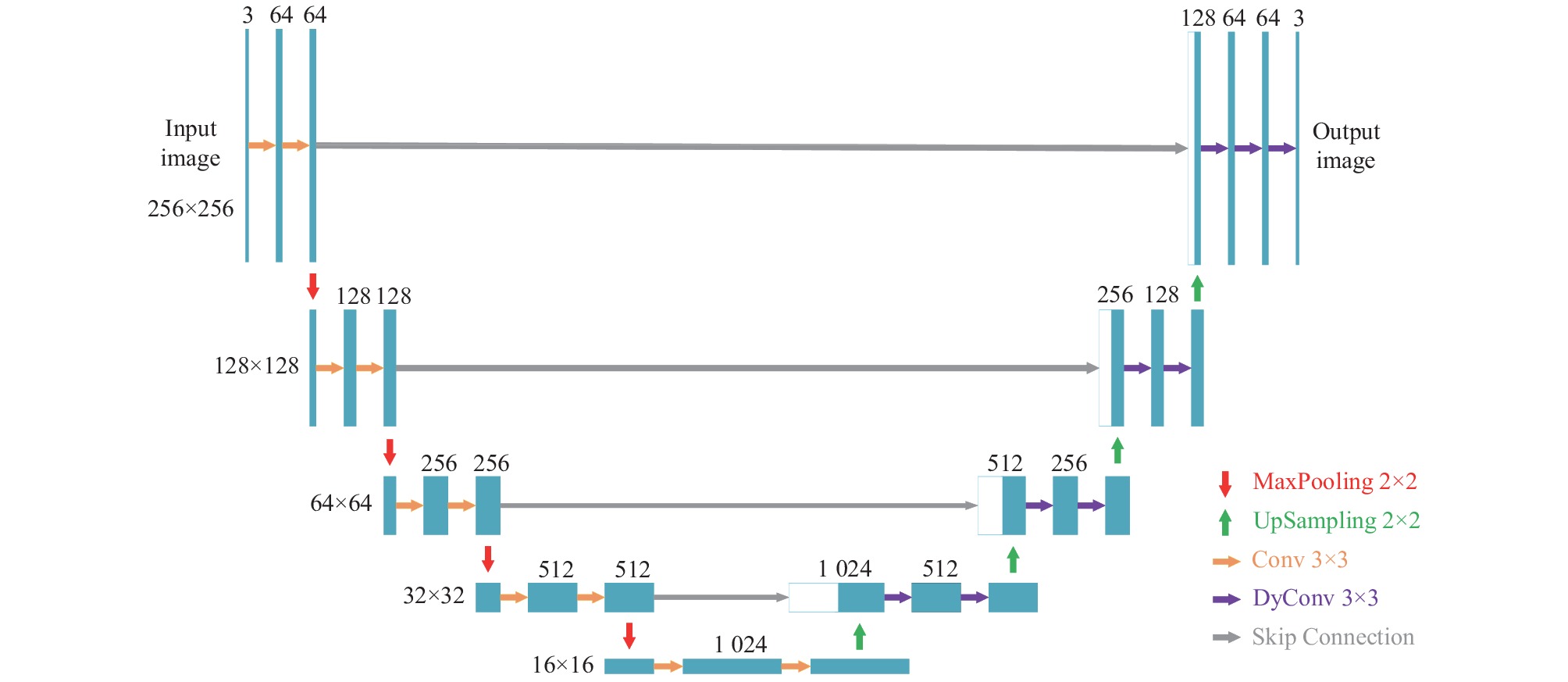
|
图 6 编码解码网络 |
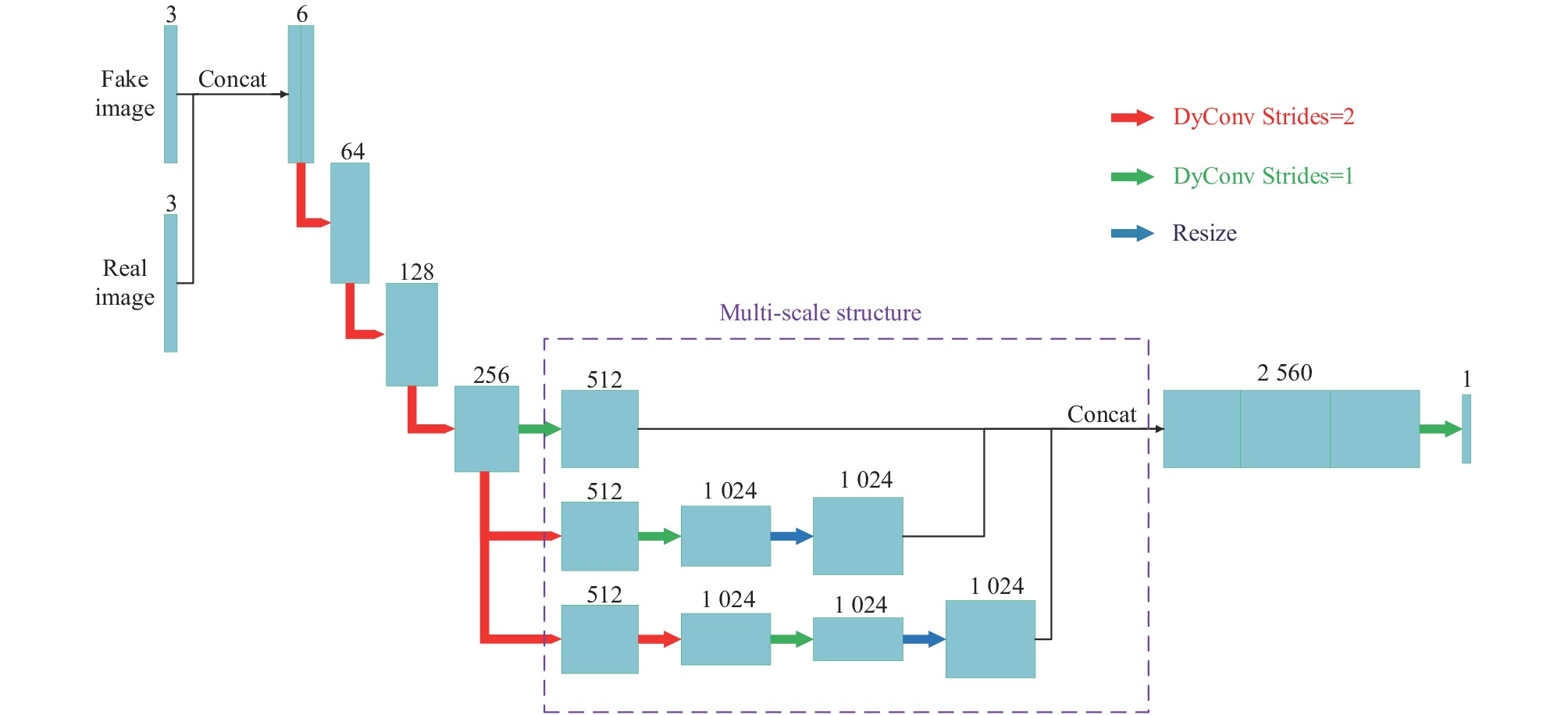
|
图 7 多尺度判别器网络结构 |
同时, 结合上面的生成器结构, 本文又测试了仅在生成器采用DyConv以及在生成器与判别器中均使用DyConv两种情况. 结果发现生成器和判别器均采用DyConv效果更好, 实验结果见表2. 因此, 我们在判别器中也采用DyConv替代了卷积操作, 文中将该方法简称为MS_DyGAN.
3.4 目标函数本文应用LSGAN[35]中提出的目标函数, 其判别器和生成器的目标函数定义分别如下:
| $ \mathop {\min }\limits_D {V_{\rm LSGAN}}\left( D \right) = \frac{1}{2}{E_x}\left[ {{{\left( {D\left( x \right) - 1} \right)}^2}} \right] + \frac{1}{2}{E_{\textit{z}}}\left[ {{{\left( {D\left( {G\left( {\textit{z}} \right)} \right)} \right)}^2}} \right] $ | (1) |
| $ \mathop {\min }\limits_G {V_{\rm LSGAN}}\left( G \right) = \frac{1}{2}{E_{\textit{z}}}\left[ {{{\left( {D\left( {G\left( {\textit{z}} \right)} \right) - 1} \right)}^2}} \right] $ | (2) |
| 表 2 多模态图像生成结果 |
由于本文基于pix2pix的方法, 训练网络需要配对的图像x和y, 且增加了任务标签“c”这一输入, 为此我们定义了新的目标函数:
| $ \begin{split} & \mathop {\min }\limits_D {V_{\rm GAN}}\left( D \right) = \frac{1}{2}{E_{x, y, c}}\left[ {{{\left( {D\left( {\left( {x, y} \right), c} \right) - 1} \right)}^2}} \right] \\ & +\frac{1}{2}{E_{x, c}}\left[ {{{\left( {D\left( {\left( {x, G\left( {x, c} \right)} \right), c} \right) - 1} \right)}^2}} \right] \\ \end{split} $ | (3) |
| $ \mathop {\min }\limits_G {V_{\rm GAN}}\left( G \right) = \frac{1}{2}{E_{x, c}}\left[ {{{\left( {D\left( {\left( {x, G\left( {x, c} \right)} \right), c} \right) - 1} \right)}^2}} \right] $ | (4) |
同时, 为了增加生成图像的准确性, 我们进一步加入了L1损失.
| $ {L_{L1}}\left( G \right) = {E_{x, y, c}}\left[ {{{\left\| {y - G\left( {x, c} \right)} \right\|}_1}} \right] $ | (5) |
最终, 新的目标函数定义为:
| $ {G^*} = \mathop {\min }\limits_G {V_{\rm GAN}}\left( G \right) + \mathop {\min }\limits_D {V_{\rm GAN}}\left( D \right) + \lambda {L_{L1}}\left( G \right) $ | (6) |
其中, λ为参数.
4 实验结果 4.1 数据集和实验设置本文实验采用BRATS19数据集. 该数据集包含259个HGG病例和76个LGG病例. 其中每个病例均有配对的T1、T2、T1CE和FLAIR四种模态的MR图像, 图像大小为240×240×155. 实验选择220个HGG病例和60个LGG病例作为训练集, 剩余的HGG和LGG病例作为测试集. 对于每个240×240×155大小的图像, 我们从脑占比最大且较清晰(75–85之间)的图像中切取出5张作为本实验的数据集. 由于T2磁共振图像是临床中广泛使用的模态, 因此本文的实验使用T2模态来合成其他3种模态的MR的图像.
在数据处理方面, 实验将切取的每张240×240大小的图像大小调整为256×256, 然后将原始数据线性缩放至[−1, 1]之间. 在参数初始化方面, 本文采用He等人[36]提出的初始化方法, 动态卷积的卷积核结合组数设置为3. 在网络训练方面, epochs设置为100, batch size为1, 优化器选择Adam, 学习率采用指数衰减策略, 初始学习率为0.000 2, 衰减率为0.98.
4.2 评价指标为评价生成图像的质量, 本文采用峰值信噪比(PSNR)和结构相似性(SSIM)两个指标. 其中, PSNR定义为:
| $ {\textit{PSNR}} = 10{\log _{10}}\left( {\frac{{{\textit{MAX}}_I^2}}{{{\textit{MSE}}}}} \right) $ | (7) |
| $ {\textit{MSE}} = \frac{1}{{mn}}\sum\limits_{i = 0}^{m - 1} {\sum\limits_{j = 0}^{n - 1} {{{\left[ {I\left( {i, j} \right) - K\left( {i,j} \right)} \right]}^2}} } $ | (8) |
其中, I和K表示大小为m×n的两幅图像.
SSIM定义如下:
| $ {\textit{SSIM}}_{\left( {x, y} \right)} = \frac{{\left( {2{\mu _x}{\mu _y} + {c_1}} \right)\left( {2{\sigma _{xy}} + {c_2}} \right)}}{{\left( {\mu _x^2 + \mu _y^2 + {c_1}} \right)\left( {\sigma _x^2 + \sigma _y^2 + {c_2}} \right)}} $ | (9) |
其中, μx为x的均值, μy为y的均值, σx2为x的方差, σy2为y的方差, σxy为x和y的协方差,
对于本文定义的目标函数, 我们首先通过实验来确定系数λ的取值, 以T2生成其他3种模态的图像为例, 实验结果如表3所示. 可以看出, 当λ取100时, 生成图像的多项指标显示为最优, 因此本文在提出的方法中λ均设置为100.
| 表 3 λ不同取值对应结果 |
4.4 单一模态MR图像生成
为了测试动态卷积的图像生成性能, 针对pix2pix的生成器, 我们比较了在生成器中下采样用动态卷积、上采样用动态卷积和上下采样均使用动态卷积、以及生成器上采样采用动态卷积和判别器也采用动态卷积4种情况, 实验结果如表4所示. 其中, only down表示下采样使用动态卷积, only up表示上采样使用动态卷积, down+up表示下采样和上采样均采用动态卷积, only up+D表示生成器中上采样用动态卷积、且判别器也使用动态卷积.
实验结果表明只上采样用动态卷积和上下采样均使用动态卷积效果相差不大, 但均优于基准模型pix2pix和下采样用动态卷积. 同时, 本文测试了在判别器中也使用动态卷积, 结果证明在生成器和判别器中均使用动态卷积的效果最好. 整个实验过程本文使用T2模态生成T1模态, 生成图比较如图8所示, 其中红色虚线框中为肿瘤区域.
| 表 4 单一模态图像生成结果 |
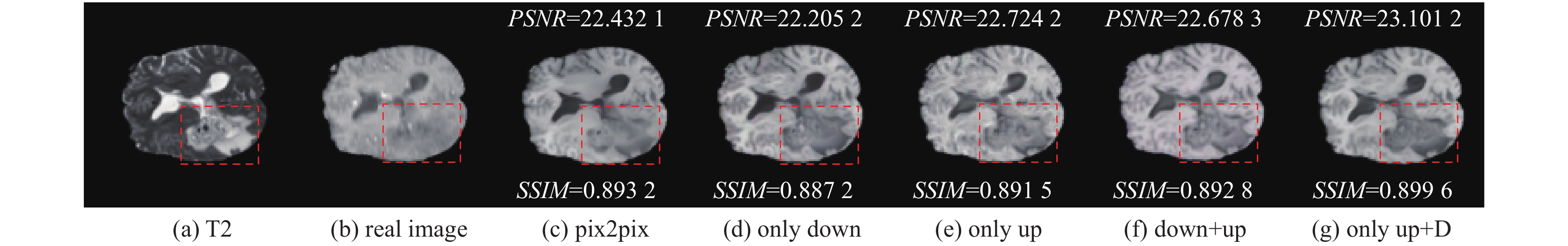
|
图 8 单一模态生成结果图示例 |
4.5 多模态MR图像生成
为了进一步验证本文方法的效果, 我们以T2模态生成其他3种模态图像为例, 将本文方法与pix2pix、MGAN进行了对比, 验证结果如表2所示. 3*pix2pix表示采用pix2pix模型生成其他3种模态数据需分别训练3个模型.
实验结果表明, 将DyConv与pix2pix相结合(DyGAN), 各项指标均优于MGAN. 不仅如此, 除了Flair模态的SSIM稍低于pix2pix, 其他指标也均高于pix2pix, 而且相比pix2pix本文是同时生成其他3种模态的图像. 除此之外, 本文提出的多尺度判别器的结构, 多项指标中显示它在DyGAN的基础上进一步提升了图像生成的质量. 图9给出了多模态图像的生成实例对比情况, 其中红色虚线框中为肿瘤区域.

|
图 9 多模态生成结果图示例 |
5 结论
针对多模态MR图像的生成方法难以通过一种模态同时生成其他所有模态的问题, 论文一方面将动态卷积应用于生成对抗网络, 提出了动态生成对抗网络模型; 另一方面, 通过任务标签的形式对不同模态的图像进行编码, 并将编码结果与动态生成对抗网络结合, 从而有效实现了由一种模态同时生成多种MR模态的数据. 在此基础上, 我们进一步提出多尺度的判别器网络, 提高了判别器的判别性能. 实验结果表明, 本文方法有效解决了难以同时生成多种模态数据的问题, 并同时提高了生成图像的质量.
| [1] |
Ostrom QT, Bauchet L, Davis FG, et al. The epidemiology of glioma in adults: A “state of the science” review. Neuro-Oncology, 2014, 16(7): 896-913. DOI:10.1093/neuonc/nou087 |
| [2] |
Yu BT, Zhou LP, Wang L, et al. 3D cGAN based cross-modality MR image synthesis for brain tumor segmentation. 2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018). Washington: IEEE, 2018. 626–630.
|
| [3] |
Cai L, Wang ZY, Gao HY, et al. Deep adversarial learning for multi-modality missing data completion. Proceedings of the 24th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining. London: ACM, 2018. 1158–1166.
|
| [4] |
Xin BY, Hu YF, Zheng YF, et al. Multi-modality generative adversarial networks with tumor consistency loss for brain MR image synthesis. 2020 IEEE 17th International Symposium on Biomedical Imaging (ISBI). Iowa City: IEEE, 2020. 1803–1807.
|
| [5] |
Li RJ, Zhang WL, Suk HI, et al. Deep learning based imaging data completion for improved brain disease diagnosis. Proceedings of the 17th International Conference on Medical Image Computing and Computer-Assisted Intervention. Boston: Springer, 2014. 305–312.
|
| [6] |
Goodfellow IJ, Pouget-Abadie J, Mirza M, et al. Generative adversarial nets. Proceedings of the 27th International Conference on Neural Information Processing Systems. Montreal: MIT Press, 2014. 2672–2680.
|
| [7] |
Kazeminia S, Baur C, Kuijper A, et al. GANs for medical image analysis. Artificial Intelligence in Medicine, 2020, 109: 101938. DOI:10.1016/j.artmed.2020.101938 |
| [8] |
Radford A, Metz L, Chintala S. Unsupervised representation learning with deep convolutional generative adversarial networks. 4th International Conference on Learning Representations. San Juan: ICLR, 2016. 1–16.
|
| [9] |
Frid-Adar M, Klang E, Amitai M, et al. Synthetic data augmentation using GAN for improved liver lesion classification. 2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018). Washington: IEEE, 2018. 289–293.
|
| [10] |
Bermudez C, Plassard AJ, Davis LT, et al. Learning implicit brain MRI manifolds with deep learning. Proceedings of SPIE 10574 Medical Imaging 2018: Image Processing. Houston: SPIE, 2018. 1–12.
|
| [11] |
Mirza M, Osindero S. Conditional generative adversarial nets. arXiv: 1411.1784, 2014.
|
| [12] |
Isola P, Zhu JY, Zhou TH, et al. Image-to-image translation with conditional adversarial networks. Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017. 5967–5976.
|
| [13] |
Dar SUH, Yurt M, Karacan L, et al. Image synthesis in multi-contrast MRI with conditional generative adversarial networks. IEEE Transactions on Medical Imaging, 2019, 38(10): 2375-2388. DOI:10.1109/TMI.2019.2901750 |
| [14] |
Yu BT, Zhou LP, Wang L, et al. Ea-GANs: Edge-aware generative adversarial networks for cross-modality MR image synthesis. IEEE Transactions on Medical Imaging, 2019, 38(7): 1750-1762. DOI:10.1109/TMI.2019.2895894 |
| [15] |
Wang TC, Liu MY, Zhu JY, et al. High-resolution image synthesis and semantic manipulation with conditional GANs. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 8798–8807.
|
| [16] |
Zhu JY, Park T, Isola P, et al. Unpaired image-to-image translation using cycle-consistent adversarial networks. Proceedings of the 2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017. 2242–2251.
|
| [17] |
Wolterink JM, Dinkla AM, Savenije MHF, et al. Deep MR to CT synthesis using unpaired data. Proceedings of the 2nd International Workshop on Simulation and Synthesis in Medical Imaging. Québec City: Springer, 2017. 14–23.
|
| [18] |
Chartsias A, Joyce T, Dharmakumar R, et al. Adversarial image synthesis for unpaired multi-modal cardiac data. Proceedings of the 2nd International Workshop on Simulation and Synthesis in Medical Imaging. Québec City: Springer, 2017. 3–13.
|
| [19] |
Jiang J, Hu YC, Tyagi N, et al. Tumor-aware, adversarial domain adaptation from CT to MRI for lung cancer segmentation. Proceedings of the 21st International Conference on Medical Image Computing and Computer-Assisted Intervention. Granada: Springer, 2018. 777–785.
|
| [20] |
Arjovsky M, Bottou L. Towards principled methods for training generative adversarial networks. Proceedings of the 5th International Conference on Learning Representations. Toulon: ICLR, 2017. 1–17.
|
| [21] |
Arjovsky M, Chintala S, Bottou L. Wasserstein GAN. arXiv: 1701.07875, 2017.
|
| [22] |
de Brabandere B, Jia X, Tuytelaars T, et al. Dynamic filter networks. Proceedings of the 30th International Conference on Neural Information Processing Systems. Barcelona: Curran Associates Inc. , 2016. 667–675.
|
| [23] |
Yang B, Bender G, Le QV, et al. CondConv: Conditionally parameterized convolutions for efficient inference. Advances in Neural Information Processing Systems 32: Annual Conference on Neural Information Processing Systems 2019. Vancouver: NeurIPS, 2019. 1305–1316.
|
| [24] |
Chen YP, Dai XY, Liu MC, et al. Dynamic convolution: Attention over convolution kernels. Proceedings of the 2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Seattle: IEEE, 2020. 11027–11036.
|
| [25] |
Zhang YK, Zhang J, Wang Q, et al. DyNet: Dynamic convolution for accelerating convolutional neural networks. arXiv: 2004.10694, 2020.
|
| [26] |
Ma NN, Zhang XY, Huang JW, et al. WeightNet: Revisiting the design space of weight networks. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 776–792.
|
| [27] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [28] |
Tian Z, Shen CH, Chen H. Conditional convolutions for instance segmentation. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 282–298.
|
| [29] |
He JJ, Deng ZY, Qiao Y. Dynamic multi-scale filters for semantic segmentation. Proceedings of the 2019 IEEE/CVF International Conference on Computer Vision. Seoul: IEEE, 2019. 3561–3571.
|
| [30] |
Wang H, Deng C, Ma F, et al. Context modulated dynamic networks for actor and action video segmentation with language queries. Proceedings of the AAAI Conference on Artificial Intelligence, 2020, 34(7): 12152-12159. DOI:10.1609/aaai.v34i07.6895 |
| [31] |
Pang YW, Zhang LH, Zhao XQ, et al. Hierarchical dynamic filtering network for RGB-D salient object detection. Proceedings of the 16th European Conference on Computer Vision. Glasgow: Springer, 2020. 235–252.
|
| [32] |
Wu F, Fan A, Baevski A, et al. Pay less attention with lightweight and dynamic convolutions. Proceedings of the 7th International Conference on Learning Representations. New Orleans: ICLR, 2019. 1–14.
|
| [33] |
Choi Y, Choi M, Kim M, et al. StarGAN: Unified generative adversarial networks for multi-domain image-to-image translation. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 8789–8797.
|
| [34] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [35] |
Mao XD, Li Q, Xie HR, et al. Least squares generative adversarial networks. Proceedings of the 2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017. 2813–2821.
|
| [36] |
He KM, Zhang XY, Ren SQ, et al. Delving deep into rectifiers: Surpassing human-level performance on imagenet classification. Proceedings of the 2015 IEEE International Conference on Computer Vision. Santiago: IEEE, 2015. 1026–1034.
|
 2022, Vol. 31
2022, Vol. 31



