2. 中国科学院 沈阳计算技术研究所, 沈阳 110168;
3. 山东大学 大数据技术与认知智能实验室, 济南 250100
2. Shenyang Institute of Computing Technology, Chinese Academy of Sciences, Shenyang 110168, China;
3. Laboratory of Big Data and Artificial Intelligence Technology, Shandong University, Jinan 250100, China
电子病历中蕴含着大量的医学实体和概念, 记录了大量的与患者健康状况相关的信息, 是一个丰富的医学知识宝库. 基于电子病例的关系抽取作为医学信息抽取领域的子任务, 旨在从非结构化的电子病历文本中抽取两个医学实体之间的关系, 是构建医学垂直领域知识图谱的关键步骤. 知识图谱的基本组成是形如<头实体, 关系类型, 尾实体>的三元组. 从电子病历中识别医学实体之间的语义关系, 构建医学领域知识图谱对下游医学任务具有重要意义.
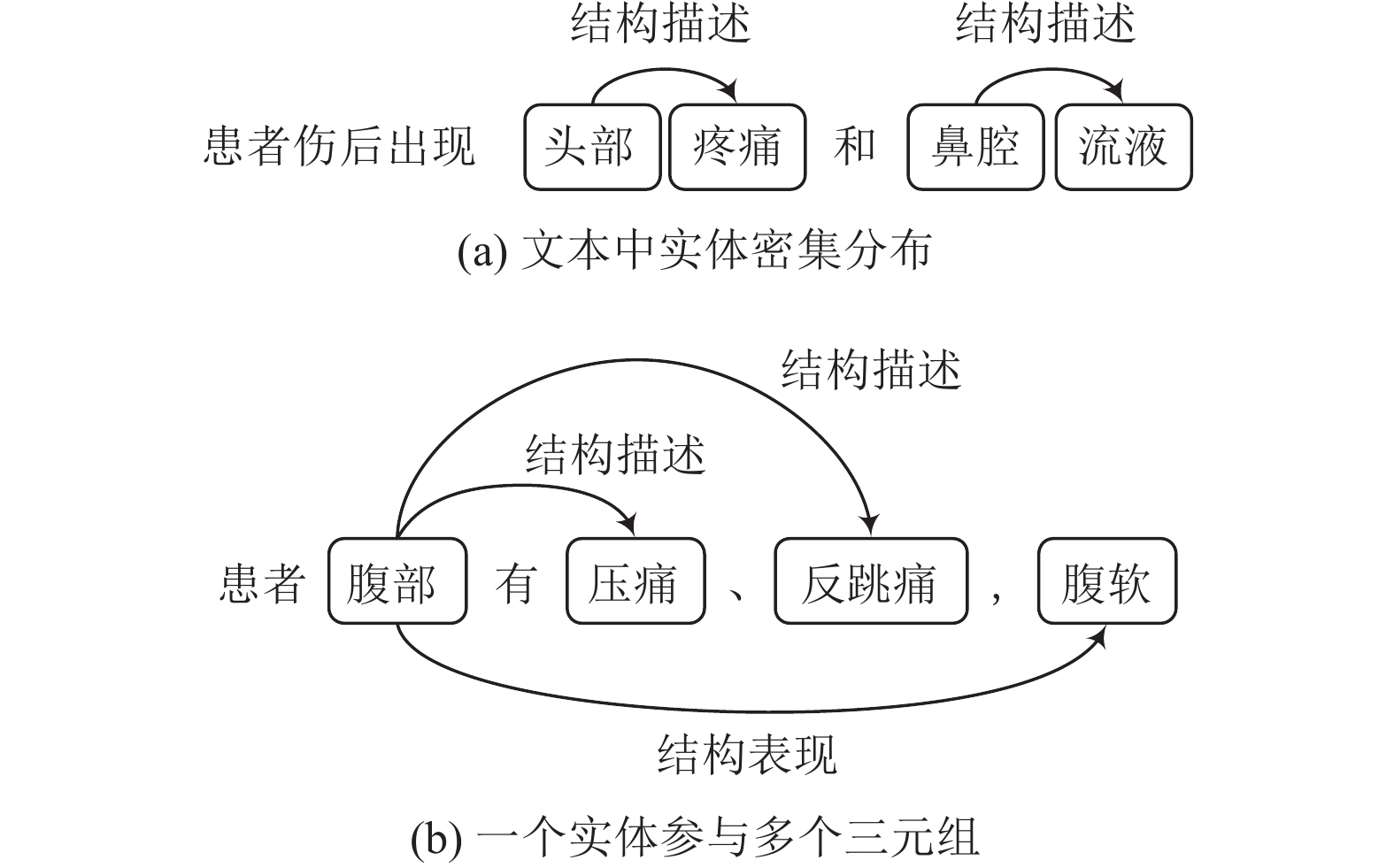
目前对电子病历中的关系抽取的研究主要集中在英文的电子病历, 主要是使用关系分类方法分析给定的两个实体的上下文, 从而判断两个实体间的关系所属的类别[1-3]. 然而, 中文电子病历中的医学实体的分布具有高度密集的特点. 例如在图1(a)所给出的例子中, “患者伤后出现头部疼痛和鼻腔流液”这个句子记录了患者伤后出现的症状表现, 包含[头部], [疼痛], [鼻腔], [流液]等医学实体, 实体间产生两组关系, 构成了两对医学关系三元组, 分别为<头部, 结构描述, 疼痛>和<鼻腔, 结构描述, 流液>, 其中“结构描述”是预定义的关系类型. 从上述的例子可以看出, [头部]和[疼痛]是一组有关系的实体对, 它们所在的上下文中分布着[鼻腔]和[流液]等医学实体, 这些实体概念的内容对分析[头部]和[疼痛]之间的语义关系并没有起到帮助作用, 当这些无关实体的数量变多时甚至可能会给关系分类模型引入噪声, 阻碍模型做出正确的分类决策.
|
图 1 中文电子病历关系三元组实体分布示意图 |
中文电子病历文本中医学实体密集分布的特点还会产生多个三元组共享一个实体的情况. 如图1(b)所示, 实体[压痛]和实体[反跳痛]都与实体[腹部]具有语义关系, 关系类型为“结构描述”, 实体[腹软]与实体[腹部]也具有语义关系, 关系类型为“结构表现”. 当一个实体参与了多个关系三元组时, 传统的关系分类器容易发生混淆. 综上所述, 中文电子病历文本的实体高密度分布的问题, 给现有的关系抽取模型带来了一定的挑战.
针对使用关系分类方法处理中文电子病历文本中的高密度实体分布问题所面临的挑战, 本文提出了一种基于条件提示与序列标注的关系三元组识别方法. 相较于传统的将实体对以及实体对所在的文本输入模型以求解实体对语义关系的关系分类方法, 本文提出的方法将关系抽取任务转换成一个基于条件提示信息的序列标注任务. 该方法的核心是给定先验条件作为提示, 建模条件提示信息与中文电子病历文本语义的依赖关系, 并从文本中寻找多个能够与条件提示信息相匹配的片段进行标注, 其中条件提示信息定义为关系三元组的头实体和关系类型词, 被抽取的多个片段是关系三元组中的尾实体. 比如在对图1(b)中的例子进行关系三元组抽取的过程中, 当条件提示信息为“腹部”和“结构表现”组成的术语时, 基于该术语作为先验条件, 可抽取出片段[腹软]. 当条件提示信息由“腹部”和“结构描述”组成时, 模型抽取内容则变成[压痛]和[反跳痛]两个片段. 即假如一个给定的头实体参与了多个关系三元组, 并且这些关系三元组的关系类型都是相同的, 那么模型的目标是从文本中识别出与给定的头实体和关系类型相关联的全部尾实体片段, 组成若干个关系三元组. 由于条件提示信息的存在, 电子病历文本中与条件提示信息无关联的医学实体将被过滤, 与条件提示信息相关联的医学实体才能被识别.
本文的主要贡献如下:
(1)组织构建了一批中文电子病历数据集, 在医学领域专家的指导下定义中文电子病历中的实体以及实体对之间的语义关系, 并请医学专业人员对实体和关系数据进行了人工标注.
(2)针对中文电子病历中实体密集分布的数据特征, 设计了一种基于条件提示与序列标注的关系三元组识别方法, 通过建模条件提示信息与文本序列字符特征的关联, 从文本中识别出与条件提示信息相关的尾实体片段, 从而实现关系三元组的识别. 在中文电子病历数据集上进行的实验证明了该方法的有效性.
2 相关工作目前在医学领域的关系抽取方法主要有基于规则匹配的方法, 基于特征工程的方法和基于深度学习的方法.
早期的关系抽取任务主要依靠人工分析文本特征构建模板, 用模板在新的文本中匹配符合既定规则的关系三元组, 这要求制定规则的人员拥有丰富的医学领域知识, 因此构建人工规则的代价十分昂贵. 文献[4]利用句子上下文的语法结构信息构建了10多个模板, 从医学文献中识别蛋白质实体以及实体之间的关系. 文献[5]设计了一种简化句法的方法, 将结构复杂的上下文句子分解为简单句, 通过规则从简单句中识别出具有相互作用的药物对. 对于不同的语料而言, 由于背景内容和语言结构的差异, 基于规则模板的方法难以在不同的语料之间迁移, 会导致关系抽取的召回率较低, 因此基于模板规则匹配的方法的泛化能力较差.
医学信息抽取任务的发展使得以特征工程为和核心的机器学习方法广泛应用于医学信息抽取. 2010i2b2/VA评测任务[6]的提出吸引了众多研究者关注电子病历中的关系抽取任务. 文献[7]手工提取词语特征、短语特征、句法特征等多种特征用于抽取化学-疾病数据集中的关系三元组. 文献[8]在支持向量机(support vector machine, SVM)的基础上融合句子结构信息, 在提取电子病历实体关系时考虑了句子结构的相似性. 相比于基于模板规则匹配的方法而言, 基于特征工程的方法具有很好的泛化能力, 能移植到不同的语料, 然而特征的选择会极大地影响模型最终的抽取性能, 并且提取语法、句法等特征往往需要用到外部工具, 外部工具自身的误差可能会传递到关系抽取模型中.
近年来, 深度学习方法在医学关系抽取任务中得到了广泛的应用, 其中比较经典的是基于卷积神经网络(convolutional neural network, CNN)和循环神经网络(recurrent neural network, RNN)的模型[9, 10]. 文献[11]将词嵌入向量和位置嵌入向量输入CNN模型进行药物相互作用的关系抽取. 文献[12]利用CNN获取句子的局部特征, 并结合最大池化抽取电子健康档案中的实体关系事实. 文献[13]利用残余卷积块降低了电子病历实体关系抽取中数据噪声带来的影响. 文献[14]提出了一种两阶段的方法提取医学文本中的实体和关系, 其中关系抽取模块利用CNN提取单词、实体类型以及位置嵌入的特征. 文献[15]利用层次RNN模型引入最短依赖识别药物关系. 为了弥补RNN处理长距离文本特征能力不足的缺陷, 其变种长短期记忆网络(long short-term memory, LSTM)得到了应用[16]. 文献[17]通过结合双向长短期记忆网络(bidirectional long short-term memory, Bi-LSTM)和多跳自注意力机制获取文本的多重向量表示, 提升捕捉医学实体之间复杂语义信息的效果. 文献[18]在深度学习框架中整合最短路径依赖和句子序列表示, 提升了关系抽取的性能. 基于深度学习的方法将文本转换为向量表示, 不需要手工提取复杂的特征.
对于开放领域的实体关系抽取任务, 研究者们提出了许多新范式. 文献[19]将实体关系抽取任务转换成阅读理解任务, 依据实体和关系生成不同的问题模板, 通过从上下文中抽取出能够回答该问题的片段的方法识别文本中的实体和关系. 文献[20]对重叠关系三元组问题进行了研究, 并提出了一种将文本中的主语实体映射成宾语实体的级联标注框架.
3 关系三元组识别模型为了应对中文电子病历文本中实体高密度分布带来的问题, 本文提出了一个基于条件提示与序列标注的关系三元组识别方法, 旨在通过捕捉由头实体和关系类型词组合成的提示信息与中文电子病历文本片段的关联, 并从文本中抽取出与条件提示信息相关联的片段, 被抽取出的片段作为尾实体与条件提示信息中的头实体和关系类型构成一个有效的关系三元组. 本文提出的模型如图2所示, 主要包括以下几个部分: 关系类型词编码、电子病历文本编码、条件信息交互融合以及解码输出. 模型的输入是电子病历文本、三元组的关系类型词以及三元组的头实体的掩码序列. 模型的输出是基于BIESO标注规范[21]标注的序列, 其中, B表示尾实体片段的开头, I表示尾实体片段的中间部分, E表示尾实体片段的结尾, 若尾实体片段为单字则标记为S, 其余的无关字符将被标注为O. 对于BIES标签, 标签的后面通过“-”连接尾实体所属的类别, 如“B-描述”“I-描述”“E-描述”, 表示该尾实体为一个“描述”类型的实体. 序列标注的结果经过处理后得到若干条形如<头实体, 关系类型, 尾实体>的关系三元组.
3.1 模型输入编码层如图2所示, 本文的模型设计了两个输入网络, 分别用于编码关系类型词和电子病历文本, 编码的token为单个中文字符. 为了使模型能够捕获电子病历文本中的每一个字相对于头实体的位置特征, 头实体的前后位置加入了特殊字符@作为位置标记. 在文本表示的过程中, 使用一个字向量矩阵, 将关系类型词和电子病历文本序列中的每个字映射成高密度的字嵌入向量.

|
图 2 基于条件提示与序列标注的关系三元组识别模型图 |
为了获得电子病历文本序列中每个字的特征, 将上一步获得的文本序列字嵌入向量表示
| $ \overrightarrow {{H_x}} = \overrightarrow {LSTM} ({E_x}) = [\overrightarrow {{x_1}} , \overrightarrow {{x_2}} , \cdots, \overrightarrow {{x_m}} ] $ | (1) |
| $ \overleftarrow {{H_x}} = \overleftarrow {LSTM} ({E_x}) = [\overleftarrow {{x_1}} , \overleftarrow {{x_2}} , \cdots, \overleftarrow {{x_m}} ] $ | (2) |
| $ {x_i} = [\overrightarrow {{x_i}} \oplus \overleftarrow {{x_i}} ] $ | (3) |
为了获得关系类型词的特征表示, 将字嵌入映射阶段获得的关系类型词字嵌入向量表示
| $ {H_r} = LSTM({E_r}) = [h_1^r, h_2^r, \cdots, h_n^r] $ | (4) |
其中, hrj表示第j个字经过LSTM编码后输出的隐藏层特征向量. 取
为了应对电子病历关系三元组识别中存在的实体密集分布以及一个实体参与了多个关系三元组的情况, 在本文提出的模型中设计了一个条件信息交互融合层, 用于建模由头实体+关系类型组成的条件提示信息与尾实体之间的依赖, 使得模型的输出层对电子病历文本序列进行标注的时候能够考虑先验条件, 依据不同的先验条件标注不同的尾实体片段. 条件信息交互融合层主要包含两个步骤: 1)创建条件提示信息; 2)条件提示信息与电子病历文本交互.
创建条件提示信息需要将头实体的特征表示和关系类型词的特征表示进行融合. 本文利用一个头实体掩码序列, 从电子病历文本序列的特征表示中获取头实体的特征表示. 假设在电子病历文本序列中, 头实体文本片段的位置跨度定义为
| $ {M_{\rm sub}}[i] = \left\{ \begin{gathered} 1, i \in {P_{\rm sub}} \\ 0, i \notin {P_{\rm sub}} \\ \end{gathered} \right. $ | (5) |
头实体的特征表示
| $ {h'_{\rm sub}} = \dfrac{1}{{end - start + 1}}\sum\limits_{i = start}^{end} {{H_{{x}}} \circ {M_{\rm sub}}} $ | (6) |
| $ {h_{\rm sub}} = {\text{tanh}}({W_{\rm sub}}{h'_{\rm sub}} + {b_{\rm sub}}) $ | (7) |
其中, start和end是头实体在文本中的起始位置和结束位置, tanh是激活函数,
| $ {h_{\rm ref}} = {h_{\rm sub}} + {h_{\rm rel}} $ | (8) |
为了从电子病历文本中识别出与条件提示信息相关联的尾实体片段, 让条件提示信息与电子病历文本产生交互是非常必要的, 这个过程能够让模型区分电子病历文本序列中每一个中文字符token相对于条件提示信息的关联程度, 从而使得模型能够依据不同的条件提示信息识别不同的尾实体片段. 本文将条件提示信息的特征表示与电子病历文本序列的每一个字符的特征表示进行拼接, 并利用Bi-LSTM网络进行编码, 获得更高级的融合条件提示信息的文本特征表示
| $ {v_t} = [{x_t};{h_{\rm ref}}] $ | (9) |
| $ \overrightarrow {{H_u}} = \overrightarrow {LSTM} ([{v_1}, {v_2}, \cdots, {v_m}]) = [\overrightarrow {{u_1}} , \overrightarrow {{u_2}} , \cdots, \overrightarrow {{u_m}} ] $ | (10) |
| $ \overleftarrow {{H_u}} = \overleftarrow {LSTM} ([{v_1}, {v_2}, \cdots, {v_m}]) = [\overleftarrow {{u_1}} , \overleftarrow {{u_2}} , \cdots, \overleftarrow {{u_m}} ] $ | (11) |
| $ {u_t} = [\overrightarrow {{u_t}} \oplus \overleftarrow {{u_t}} ] $ | (12) |
在解码过程中充分考虑标签之间的关联性有利于准确的识别实体的边界, 得到最优的标签序列. 本文利用条件随机场(conditional random fields, CRF) 作为解码输出层[22], CRF解码过程涉及一个转移矩阵V和一个状态序列Z. 转移矩阵V用于学习标签之间的依赖关系, Vij表示第i个标签转移到第j个标签的得分. 状态序列
| $ {{\textit{z}}_t} = {W_u}{u_t} $ | (13) |
其中,
| $ S(Z, Y) = \sum\limits_{t = 1}^m {{V_{{y_{t - 1}}, {y_t}}}} + \sum\limits_{t = 1}^m {{Z_{t, {y_t}}}} $ | (14) |
从输入序列Z解码得到每一个可能的预测序列Y的条件概率计算如式(15):
| $ P(Y|Z) = \frac{{{\text{exp}}(S(Z, Y))}}{{\displaystyle\sum\nolimits_{Y' \in {Y_Z}} {{\text{exp}}(S(Z, Y'))} }} $ | (15) |
其中,
本文从某三甲医院获取了一批门诊病历数据, 数据的形式为中文电子病历, 主要内容包括主诉、现病史、既往史、体格检查、辅助检查、初步诊断等. 选择了其中的2 000篇进行关系三元组识别任务的研究. 在医学领域专家的指导下, 定义了实体和关系的标注规范, 并组织一批医学专业人员对中文电子病历中的实体和关系进行人工标注. 对于中文电子病历中的实体, 本文确定了11种实体类型, 并设计了一套实体类型优先级规则, 实体对中具有较高优先级的实体将作为头实体, 较低优先级的实体将作为尾实体. 依据优先级由高到低, 这11种实体类型分别是疾病、结构、观察、表现、检查、描述、方位、限定、治疗、药物、用法. 对于中文电子病历中的实体对的关系, 本文将头实体类型和尾实体类型进行拼接, 定义为该实体对的关系. 在本文的研究中, 共确定了20种粗粒度的关系类型, 部分关系三元组schemas如表1所示.
| 表 1 部分关系三元组schemas |
本文设计了一个数据预处理算法, 依据中文电子病历文本以及对应的实体关系标注文件生成形如[关系类型, 文本片段, 头实体掩码序列, 标注序列]的数据样本, 具体方法如算法1.
算法1. 中文电子病历数据预处理算法
1) 利用滑动窗口方法, 将电子病历文本切分成若干个文本片段;
2) 对于每一个文本片段, 查找该片段中的实体对; 对于每一个实体对, 利用优先级规则区分头尾实体, 生成头实体掩码序列;
3) 检查该实体对能否形成有效的关系, 若有关系, 则创建标注序列, 其标注的内容是尾实体片段, 并生成数据样本;
4) 若该实体对无关系, 则将头实体的实体类型词和尾实体的实体类型词进行拼接, 检查拼接后的术语是否存在于预定义的关系集合中, 若存在, 则创建一个全为O的标注序列, 并生成数据样本, 若不存在, 则不生成数据样本;
5) 标注序列合并, 若生成的数据样本中, 某两条数据头实体、关系类型、文本片段均相同, 则将其标注序列中非O的部分合并.
4.2 实验设置本文实验使用的训练数据、验证数据和测试数据按照8:1:1的比例进行划分. 字向量利用随机初始化方法生成50维的向量, Bi-LSTM编码器的隐藏层的维度设置为128, 批处理的大小设置为64, 训练过程中的参数优化算法为Adam, 学习率设置为0.001, dropout设置为0.5以防止过拟合.
在实验中使用精确率(Precision), 召回率(Recall), F1值作为模型的评价指标.
| $ {{Precision}} = \frac{{TP}}{{TP + FP}} $ | (16) |
| $ {{Recall}} = \frac{{TP}}{{TP + FN}} $ | (17) |
| $ {{F}}1 = \frac{{2 \cdot {{Precision}} \cdot {{Recall}}}}{{{{Precision}} + {{Recall}}}} $ | (18) |
在实验过程中发现实体对没有关系的情况比较多, 为了实现样本均衡, 随机选择一部分无关系样本用于实验.
4.3 实验结果分析为了验证本文提出的模型的性能, 本文进行了对比实验. 在之前的研究中关系抽取任务通常被视为关系分类任务, 因此通常将CNN和RNN作为基准模型. 将本文模型分别与RNN模型、结合最大池化的CNN模型CNN-Max进行比较. 在构建对比模型的实验数据时, 对每一条文本片段插入位置标记<e1>、</e1>、<e2>、</e2>指示两个实体在文本片段中的起始位置和结束位置. 对比模型的输出层使用一个全连接层将编码特征映射成具体的关系类别. 实验结果如表2所示.
| 表 2 模型实验结果对比 |
从实验结果中可以发现, 本文提出的模型在精确率、召回率和F1值上分别达到0.979 6、0.976 5和0.977 7, 表现优于基准模型. 对比基础的RNN模型, 本文模型在精确率、召回率和F1值上分别提升了18.53%、16.64%和18.7%. 对比CNN-Max模型, 本文模型在精确率、召回率和F1值上分别提升了1.12%、1.13%和1.17%. 实验结果验证本文模型能有效的应用于识别中文电子病历中的医学关系三元组.
5 结论与展望本文设计了一种基于条件提示与序列标注的中文电子病历关系三元组识别方法, 将关系抽取任务建模成从电子病历文本中识别与条件提示信息相关的三元组尾实体片段的序列标注任务, 其中条件提示信息为头实体和关系类型组成的先验知识. 本文的模型聚焦于构建条件提示信息与文本序列的关联, 过滤掉文本序列中与条件提示信息无关的实体概念. 在中文电子病历上的实验结果表明, 本文模型的精确率达到97.96%, 召回率达到97.65%, F1值达到97.77%, 表现优于基准模型, 实现了对中文电子病历中的医学关系三元组的识别.
在未来的研究工作中, 计划将当前工作延伸至更具挑战性的场景, 如医学文献中的实体关系抽取. 为获得更丰富的文本序列表示, 可考虑加入预训练语言模型. 对于模型识别无关实体的情况, 可以考虑引入句法依赖, 限制识别实体片段的结果空间.
| [1] |
He B, Guan Y, Dai R. Convolutional gated recurrent units for medical relation classification. Proceedings of 2018 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). Madrid: IEEE, 2018. 646–650.
|
| [2] |
He B, Guan Y, Dai R. Classifying medical relations in clinical text via convolutional neural networks. Artificial Intelligence in Medicine, 2019, 93: 43-49. DOI:10.1016/j.artmed.2018.05.001 |
| [3] |
Raj D, Sahu S, Anand A. Learning local and global contexts using a convolutional recurrent network model for relation classification in biomedical text. Proceedings of the 21st Conference on Computational Natural Language Learning (CoNLL 2017). Vancouver: Association for Computational Linguistics, 2017. 311–321.
|
| [4] |
Blaschke C, Valencia A. The frame-based module of the SUISEKI information extraction system. IEEE Intelligent Systems, 2002, 17(2): 14-20. DOI:10.1109/MIS.2002.999215 |
| [5] |
Segura-Bedmar I, Martínez P, de Pablo-Sánchez C. A linguistic rule-based approach to extract drug-drug interactions from pharmacological documents. BMC Bioinformatics, 2011, 12(S2): S1. DOI:10.1186/1471-2105-12-S2-S1 |
| [6] |
Uzuner Ö, South BR, Shen SY, et al. 2010 i2b2/VA challenge on concepts, assertions, and relations in clinical text. Journal of the American Medical Informatics Association, 2011, 18(5): 552-556. DOI:10.1136/amiajnl-2011-000203 |
| [7] |
Alam F, Corazza A, Lavelli A, et al. A knowledge-poor approach to chemical-disease relation extraction. Database, 2016, 2016: baw071. DOI:10.1093/database/baw071 |
| [8] |
Zhai PJ, Huang X, Zhang BB, et al. Relation extraction based on fusion dependency parsing from Chinese EMRs. Scientific Programming, 2020, 2020: 8658040. DOI:10.1155/2020/8658040 |
| [9] |
Zeng DJ, Liu K, Lai SW, et al. Relation classification via convolutional deep neural network. Proceedings of COLING 2014, the 25th International Conference on Computational Linguistics: Technical Papers. Dublin: ACL, 2014. 2335–2344.
|
| [10] |
Zhang DX, Wang D. Relation classification via recurrent neural network. arXiv: 1508.01006, 2015.
|
| [11] |
Liu SY, Tang BZ, Chen QC, et al. Drug-drug interaction extraction via convolutional neural networks. Computational and Mathematical Methods in Medicine, 2016, 2016: 6918381. DOI:10.1155/2016/6918381 |
| [12] |
Tian B, Zhang Y, Liu KX, et al. Deep learning based information extraction framework on chinese electronic health records. Proceedings of the 30th International Conference on Software Engineering and Knowledge Engineering. Redwood City: Research Inc. and Knowledge Systems Institute Graduate School, 2018. 86–91.
|
| [13] |
Zhang ZC, Zhou T, Zhang Y, et al. Attention-based deep residual learning network for entity relation extraction in Chinese EMRs. BMC Medical Informatics and Decision Making, 2019, 19(2): 55. DOI:10.1186/s12911-019-0769-0 |
| [14] |
Suárez-Paniagua V, Zavala RMR, Segura-Bedmar I, et al. A two-stage deep learning approach for extracting entities and relationships from medical texts. Journal of Biomedical Informatics, 2019, 99: 103285. DOI:10.1016/j.jbi.2019.103285 |
| [15] |
Zhang YJ, Zheng W, Lin HF, et al. Drug-drug interaction extraction via hierarchical RNNs on sequence and shortest dependency paths. Bioinformatics, 2018, 34(5): 828-835. DOI:10.1093/bioinformatics/btx659 |
| [16] |
Zhang S, Zheng DQ, Hu XC, et al. Bidirectional long short-term memory networks for relation classification. Proceedings of the 29th Pacific Asia Conference on Language, Information and Computation. Shanghai: ACL, 2015. 73–78.
|
| [17] |
Zhang TX, Lin HF, Tadesse MM, et al. Chinese medical relation extraction based on multi-hop self-attention mechanism. International Journal of Machine Learning and Cybernetics, 2021, 12(2): 355-363. DOI:10.1007/s13042-020-01176-6 |
| [18] |
Li ZH, Yang ZH, Shen C, et al. Integrating shortest dependency path and sentence sequence into a deep learning framework for relation extraction in clinical text. BMC Medical Informatics and Decision Making, 2019, 19(1): 22. DOI:10.1186/s12911-019-0736-9 |
| [19] |
Li XY, Yin F, Sun ZJ, et al. Entity-relation extraction as multi-turn question answering. Proceedings of the 57th Annual Meeting of the Association for Computational Linguistics. Florence: Association for Computational Linguistics, 2019. 1340–1350.
|
| [20] |
Wei ZP, Su JL, Wang Y, et al. A novel cascade binary tagging framework for relational triple extraction. Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Online: Association for Computational Linguistics, 2020. 1476–1488
|
| [21] |
Yang J, Liang SL, Zhang Y. Design challenges and misconceptions in neural sequence labeling. Proceedings of the 27th International Conference on Computational Linguistics. Santa Fe: Association for Computational Linguistics, 2018. 3879–3889.
|
| [22] |
Lafferty JD, McCallum A, Pereira FCN. Conditional random fields: Probabilistic models for segmenting and labeling sequence data. Proceedings of 18th International Conference on Machine Learning. Williamstown: ACM, 2001. 282–289.
|
 2022, Vol. 31
2022, Vol. 31


