睡眠是人类各类生理活动进行过程中不可或缺的重要生理活动, 睡眠时间大约占了人的一生中33%的时间. 良好的睡眠是人们健康生活的重要保障, 保证人们进行正常的学习、生活和工作等生产生活方式, 维持人类基本的生理机能活动. 然而在现实生活中, 越来越多的人遭受到睡眠问题, 这极大干扰了人的正常生活. 而睡眠分期作为睡眠医学研究过程中的一个重要步骤, 可以有效监测、检查和评估睡眠质量, 为各类睡眠疾病诊断过程中提供了重要依据, 是诊断睡眠问题的关键前提. 旧时传统的人工判别睡眠分期主要是通过医学专家的视觉分析来完成, 专家需要花费很多时间去判定, 且仅靠专家肉眼观察分析, 效率非常低, 容易造成错误判断[1]. 利用深度学习和机器学习等技术现代信号处理技术对睡眠生理信号实现自动分期, 分期结果更加高效、客观, 这是当代研究自动化睡眠分期的主要方向趋势, 也为未来睡眠问题分析和研究提供了很多机遇和挑战. 机器学习主要是基于输入的自定义特征、训练出一个合适的分类算法模型, 根据睡眠各个阶段凸出的明显特征来分类实现自动睡眠分期. 其存在一定的局限性. 一是需要专家依靠先验知识手动设计特征. 二是设计的特征不能完全与睡眠阶段贴切相符, 目前还没有找到一个最优的睡眠特征与分类器的组合, 说明距离睡眠分期准确率的提高始终有“天花板”存在. 而深度学习能够从输入的脑电数据中自动进行特征表示, 可以通过睡眠脑电数据测试集中找到最优最合适的睡眠特征, 再结合分类器输出最终的睡眠分期结果. 但是深度学习算法得到的抽象特征往往不能被人们理解和解释, 网络层数越多, 得到的特征越复杂, 同时深度学习对于调参、网络层数设定、最优算法选择等方面需要花费很多时间精力, 对开发人员也有一定的专业知识储备要求. 然而当今社会中深度学习依然是解决很多优化分类问题的绝佳方法, 具有良好的泛化性能.
基于上述考虑, 本文选取CNN卷积神经网络与BiLSTM相结合, 利用卷积神经网络中提取的高维特征向量按时间作为BiLSTM的输入, 训练出一个可以捕捉脑电数据时间依赖性的神经网络模型, 不但可以利用卷积神经网络提取局部特征的优势, 而且能利用双向长短时记忆网络兼顾长时间序列全局特征的优势, 同时进行结构建模获取更高的分类准确率.
1.2 研究现状(1) 关于睡眠分期的研究
在睡眠分期之前, 首先要确定模型分析结果所要遵守的分期标准. 目前典型的睡眠分期标准有两个, 一个是《人类睡眠阶段标准化术语, 技术及划分系统手册》, 由Rechtschaffen和Kales提出并制定, 故简称R&K标准[2]. 另一个是由美国睡眠医学会针对R&K标准提出的修正版本, 简称AASM标准. R&K标准, 将整个睡眠过程划分为3个具有一定明显不同特征的分期阶段, 第1个是清醒期(W), 第2个是快速眼动期(REM), 最后一个是非快速眼动期(NREM). 非快速眼动期可以继续划分为4个时期, 即睡眠1期、睡眠2期、睡眠3期和睡眠4期, 一般标记为S1期、S2期、S3期和S4期, 其中, S1期和S2期为浅睡期(light sleep, LS), S2期和S3期为深睡期或慢波睡眠期(slow wave sleep, SWS). AASM标准, 与R&K标准进行比较, 其主要变化的是, 将非快速眼动期分为非快速眼动一期、非快速眼动二期和非快速眼动三期, 一般标记为N1期、N2期和N3期, 其中, N1期和N2期为浅睡期, N3期为深睡期(慢波睡眠期).
(2) 基于脑电信号(electroencephalogram, EEG)的睡眠分期现状
现阶段, 对于获取到的睡眠脑电信号, 如何从脑电信号中提取到合适的高级特征, 并根据特征将睡眠状态进行分类, 是脑电信号能否在睡眠分期中得到应用的关键性一步. 近年来, 许多研究提出了基于脑电信号的睡眠分期算法, 其中, Phan等人在2016年使用CNN神经网络提取原始的脑电信号中的特征, 但是并不是以神经网络端到端的学习形式进行特征的提取[3]. 随后, 有学者对脑电信号的特征提取进行了神经网络端到端的自动学习, 但睡眠分期的效果并不是很好[4,5], 考虑到脑电信号具有时序依赖性和连续性, 研究人员曾使用循环神经网络(recurrent neural network, RNN)进行睡眠分期, 实验结果表明RNN网络能够有效地提高睡眠分期的准确率. 然而, 该模型对于一些特殊的睡眠吋期的分期准确率并不是太高[6]. Supratak等人基于原始单通道 EEG 提出了结合卷积神经网络和双向长短期记忆网络的深度学习模型DeepSleepNet, 用于自动睡眠阶段评分, 总体准确率达到 82.00%[7]; Mousavi等人基于单通道 EEG 提出了由深度卷积神经网络组成的SleepEEGNet, 总体准确率达到 84.26%[8]; Phan等人提出分层递归神经网络 SeqSleepNet, 将自动睡眠分期作为序列到序列的分类问题来解决, 总体准确率达到87.10%[9]. 近年来, 随着CNN技术的探索发展, 其被广泛应用于语音识别处理、图像处理、机器视觉、自然语言等领域. 2011年Cecotti等人将CNN应用到基于脑电信号的P300 研究实验中[10], 而且也有将CNN与LSTM进行结合应用于时间序列的数据, 例如将 CNN-LSTM 进行电影类型的分类. 融合CNN与BiLSTM的模型算法还用于心律失常心拍分类中, 其识别准确率相对单独的CNN使用与单独的BiLSTM使用分别提高了13.97%和7.14%[11]. 利用CNN与BiLSTM网络特征结合进行文本情感分析, 也能有效地提高分类的准确率[12]. 在进行短文本相似度计算中, CNN与BiLSTM相结合的网络结构模型优于其他方法, 其准确率达到84.58%[13]. 针对上述研究, 可以发现CNN与BiLSTM相结合的混合神经网络在分类问题研究中具有明显的优点. 而睡眠数据作为一种具有时序信息的信号, 显然地, 实现睡眠自动分期如果要达到更佳的效果, 我们需要考虑到脑电信号前后数据信息之间的时序依赖性和联系性, 而CNN-BiLSTM神经网络可以更好地探究睡眠数据前后序列时间信息的关联和依赖, 所以本文中采用CNN-BiLSTM神经网络模型对睡眠脑电数据进行分期研究.
2 基于脑电信号的睡眠分期 2.1 预处理本文模型使用的睡眠数据是MIT-BIH生理信息库中Sleep-EDF的多通道脑电信号的真实数据, 数据记录了正常人的睡眠状态数据. 对脑电信号数据的预处理方法步骤如下.
(1) 首先提取公开数据集中两个EEG信号(Fpz-Cz通道和Pz-Oz通道)与医学专家人工判别的睡眠分期标签, 将其解析存储为pkl文件.
(2) 把连续30 s的睡眠数据划分为一个睡眠片段, 按照专家的判定将每个睡眠片段划分为WAKE、NERM和REM期, 根据对应的睡眠分期标签“W, 1, 2, 3, 4, R”转化为“0, 1, 1, 1, 1, 2”, 即分别对应着3分类睡眠阶段.
(3) 每次加载5个连续30 s的睡眠数据和判别标签, 对数据使用Mel带通滤波器处理, 可以得出相应的频谱Mel Spectrogram. 滤波后, 将其中每5段连续30 s的脑电信号数据归为一个组别, 构造成为特征矩阵.
2.2 CNN特征学习卷积神经网络(CNN)的核心思想是: 局部感知野、权值共享和下采样pooling层, 这3种核心思想共同作用, 减少了网络使用的内存量, 简单化网络参数, 提高了精度准确率和网络运算速度, 缓解了过拟合的网络问题[14]. 从功能结构划分来看, 卷积神经网络可以分为6个部分: 信息输入层、卷积运算层、归一化层、ReLU激活层、池化层和全连接层. 本文模型用到的CNN深度神经网络结构如图1所示.

|
图 1 CNN网络结构图 |
卷积运算层主要进行两个关键步骤操作: 数据局部关联和窗口滑动. 卷积运算层公式如下:
| $ {H_i} = {f_{{\rm{cov}}}}({H_{i - 1}} \otimes {W_i} + {b_i}) $ | (1) |
式中,
| $ {f_{{\rm{cov}}}}(x) = \max (0,x) $ | (2) |
这里使用max最大值函数对输入值和0进行比较取最大值. 激活函数可以加入非线性因素, 解决线性模型不能解决的问题, 同时提高模型鲁棒性和充分组合特征, 将特征图投射到新的一个特征空间, 有利于训练睡眠数据. 激活层后面紧接着的是池化层, 本文网络模型采用最大化池化方法, 池化层主要作用有特征降维、特征不变性和避免过拟合. 全连接层一般出现在池化层的后面, 用于分类, 对提取的特征进行整合处理[15]. 最后输出层采用Softmax函数作为激活函数, 输出EEG信号睡眠分期三分类结果.
2.3 LSTM特征学习LSTM长短期记忆神经网络本质上属于一种特殊的RNN神经网络. 与传统普通的RNN进行比较, LSTM能够学习长序列信息前后的依赖和关联关系, 记录间隔或延迟较长的历史信息和重要事件. 从结构来看, LSTM的结构中添加了一个细胞神经状态单元, 由门控制去选择性地让信息通过, 添加或者删除信息. LSTM神经网络具有3大特殊功能门, 即遗忘门、输入门和输出门[16]. LSTM详细结构架构图如图2所示.
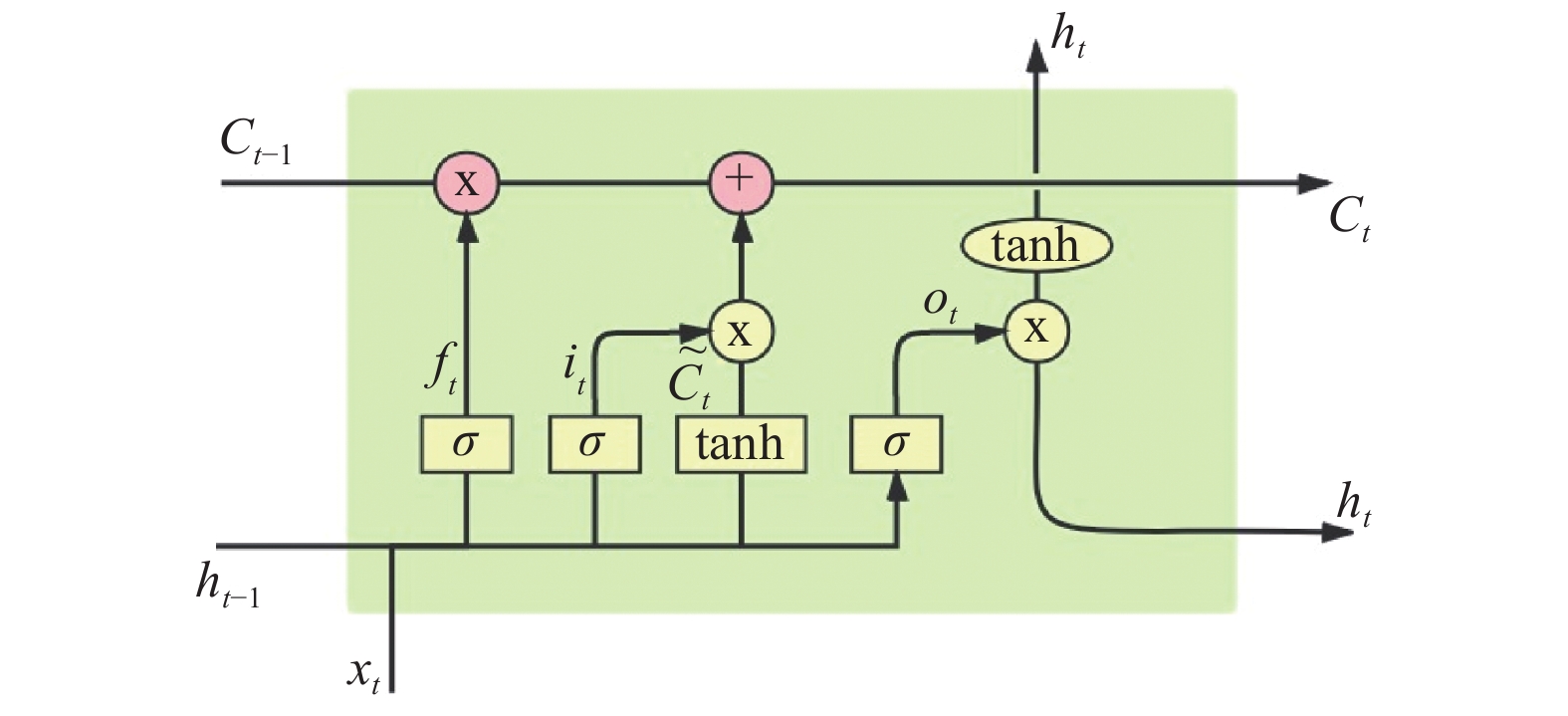
|
图 2 LSTM模型结构图 |
相关计算公式如下:
| $ {f_t} = \sigma ({W_f}[{h_{t - 1}}, {x_t}] + {b_f}) $ | (3) |
| $ {i_t} = \sigma ({W_i}[{h_{t - 1}}, {x_t}] + {b_i}) $ | (4) |
| $ {\tilde C_t} = \tanh ({W_c}[{h_{t - 1}}, {x_t}] + {b_c}) $ | (5) |
| $ {C_t} = {f_t} \times {C_{t - 1}} + {i_t} \times \tilde C $ | (6) |
| $ {o_t} = \sigma ({W_o}[{h_{t - 1}}, {x_t}] + {b_o}) $ | (7) |
| $ {h_t} = {o_t} \times \tanh ({C_t}) $ | (8) |
式中,
考虑到脑电EEG数据是非平稳、非线性数据, 传统的方法进行睡眠自动分期遇到了诸多困难. 目前来说, CNN适合处理网络结构数据如计算机视觉的图片二维数据. LSTM深度学习神经网络改善了RNN梯度消失和爆炸的问题, 与之相比更适合处理长时序序列前后类型如脑电信号的数据, 尤其在预估和处理长序列数据中间隔或延迟相对时间比较长的事件方面有极大的优势. 但是就目前来看, CNN和LSTM相结合形成的神经网络模型应用于一维生物信号(如EEG、EOG、EMG等)的分类相对较少.
本文深度学习神经网络模型主要是由卷积神经网络和双向长短时记忆神经网络两部分组成. CNN是深度学习神经网络的一个重要部分, 它的权值共享网络结构, 使神经网络模型的复杂度下降, 且图像数据输入到CNN网络中, 从而减少复杂的数据提取特征过程和重新建构数据过程, 上一层的局部数据区域通过窗口滑动和权值共享的卷积核运算操作可以得到下一层的特征, 这个特性使得CNN比其他神经网络模型对图像特征的提取、研究、表示与分析更加准确和高效. 在本文中, CNN 用来提取脑电数据的特征, 基于EEG信号的时频分析可以得出三维特征矩阵, 之后用来构建自动睡眠分期网络模型. 由于人的睡眠数据中包含着大量时间序列数据信息, 而且其在时域上也包含一定的各个不同的睡眠期的隐含特征信息, 本文考虑到每个睡眠周期前后是有关联的和有联系的, 所以采用的模型为卷积神经网络与双向长短时记忆神经网络(BiLSTM)的结合. 前向LSTM和后向LSTM的结合构成了双向长短时记忆网络 BiLSTM, BiLSTM从两个不同方向处理长序列的睡眠脑电数据信息, 可以更好地挖掘探索双向时序信息和捕捉前后时序数据信息关联和依赖性.
该网络可以自动提取脑电信号EEG的长时间序列信息. 该神经网络最后一层是Softmax分类器输出层, 用于输出各类睡眠分期结果的概率. 图3是本文的CNN-BiLSTM网络模型架构概括图.
首先将原始信号经过预处理, 计算每段30 s的时间序列EEG数据的Mel频谱图, 再将两个脑电通道的数据分别按照5组连续的30 s睡眠片段作为5组连续的时间关联数据输入到模型中. 输入层大小为(64, 47). 模型使用两个二维卷积层(Conv2d), 第一个卷积层的卷积核个数为8, 卷积核大小为(3, 3), 第二个卷积层的卷积核个数为16, 卷积核大小为(3, 3); 在两个卷积层之后进行批标准化(batch normalization); 通过ReLU激活函数; 再通过二维最大池化层(MaxPooling2d), 大小均为(4, 4); 为了避免训练过程中的过拟合问题[17], dropout层(ratio=0.2)需要连接到每个池化层后面; 再连接Flatten层, 将多维数据转化为一维数据, 实现数据从卷积层过渡到全连接层; 经过标准的Dense一维全连接层, 输出维度为30, 进行归一化和ReLU激活函数, 实现从卷积层到长短时记忆神经网络的过渡; 每5组连续的时间关联数据的睡眠信号经过卷积神经网络提取特征后, 得到(5, 60)输出维度; 模型使用一个双向的长短时记忆时间网络, 其中LSTM输出维度unit=15, 经过前后向LSTM的结合得到的数据维度为(5, 30); 最后将BiLSTM输出的特征经过Softmax层进行三分类任务, 将睡眠状态分为WAKE, NREM, REM三个时期.
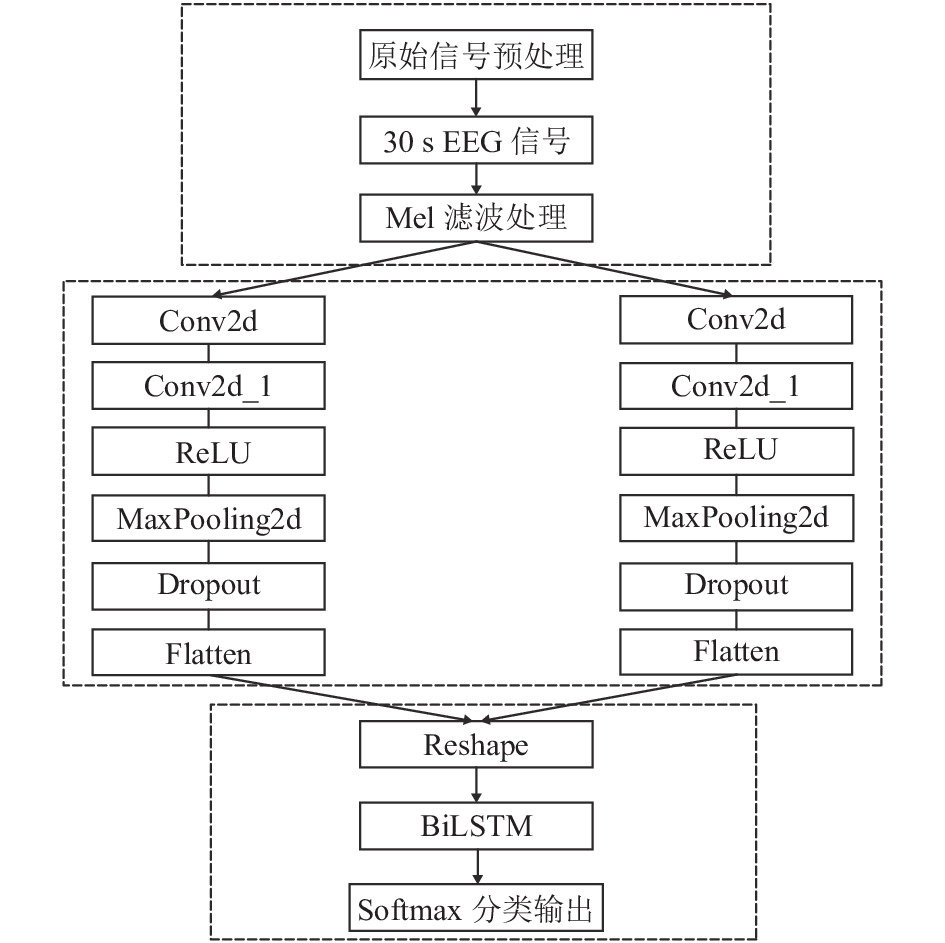
|
图 3 本文模型结构图 |
我们使用多分类的对数损失函数(categorica_crossentropy), 该损失函数与Softmax分类器相对应; 通过Adam优化损失函数, 能够动态地调整每个参数的学习率. 为了避免过拟合现象, 模型在训练过程中使用了早停机制, 若连续迭代3次损失率不下降, 则停止训练.
3 实验结果 3.1 实验数据及说明实验数据为MIT-BIH生理信息库Sleep-EDF多参数睡眠数据集, 包含197个整夜的多导睡眠图(polysomno graphic, PSG)睡眠记录, 其中包含EEG, EOG等生理数据, 每条数据均有明确的人工睡眠分期标签, 该数据集由睡眠盒式磁带对年龄在25–101岁的健康白人记录了大约20小时的PSG和利用射频发射的记录仪对22名白人男性和女性服用药物后在医院记录了大约9个小时的PSG组成.
根据 R&K 标准, 根据睡眠信息数据各阶段的不同特征, 我们可以将睡眠阶段分为3期, 即人体睡眠清醒期、快速眼动期和非快速眼动期. 本实验采用两个通道(EEG Fpz-Cz和EEG Pz-Oz)的数据组合, 记录频率为100 Hz, 划分连续30 s的睡眠脑电数据作为睡眠样本单元, 最后显示一个总的参考性的睡眠状态.
在本文实验中, 我们选择数据集中80%的数据作为数据训练集来训练网络模型, 10%的数据作为验证集验证模型的能力从而进行模型迭代训练, 10%的数据作为测试集评估模最终模型的泛化能力.
3.2 评价方法这里采用测试集的睡眠分期结果的准确率P来评价各阶段睡眠分期分类结果.
| $ P = {N_P}/N $ | (9) |
其中, 各睡眠分期(WAKE, NREM, REM)都用
采取子类平均准确
| $ AVG = \sum\limits_{i = 1}^3 {{P_i}/3} $ | (10) |
式中,
本文采用Sleep-EDF数据集的CNN-BiLSTM神经网络模型实现自动睡眠分期, 使用23个实验对象的脑电数据作为测试集对训练完成的模型进行精度检测, 最终预测结果的混淆矩阵如表1所示.
| 表 1 分期混淆矩阵表 |
混淆矩阵能够很好地检测分类的准确性, 其每一列代表预测的睡眠阶段数, 每一行代表的是实际的睡眠阶段数. 本文睡眠分期为三分类任务, 即得到的混淆矩阵为3×3矩阵, 其中, 对角线上的数值代表正确的分类数, 非对角线上的数据代表错误的分类数. 从表1可得, 测试集中实际为WAKE期睡眠状态的数据共有37244个, 模型将其中676个数据分类为NREM期, 105个数据分类为REM期; 测试集中实际为NREM期睡眠状态的数据共有15643个, 模型将其中391个数据分类为WAKE期, 817个数据分类为REM期; 测试集中实际为REM期睡眠状态的数据共有4228个, 模型将其中85个数据分类为WAKE期, 902个数据分类为NREM期.
从表中的混淆矩阵可以计算得出, 模型在WAKE期的准确率P可以达到97.9%, 在NREM期P可以达到92.3%, 都具有较高的准确率, 在REM期的准确率P为76.7%, 目前还有待提升. 图4给出了每个睡眠阶段时期的分期准确率数据显示.
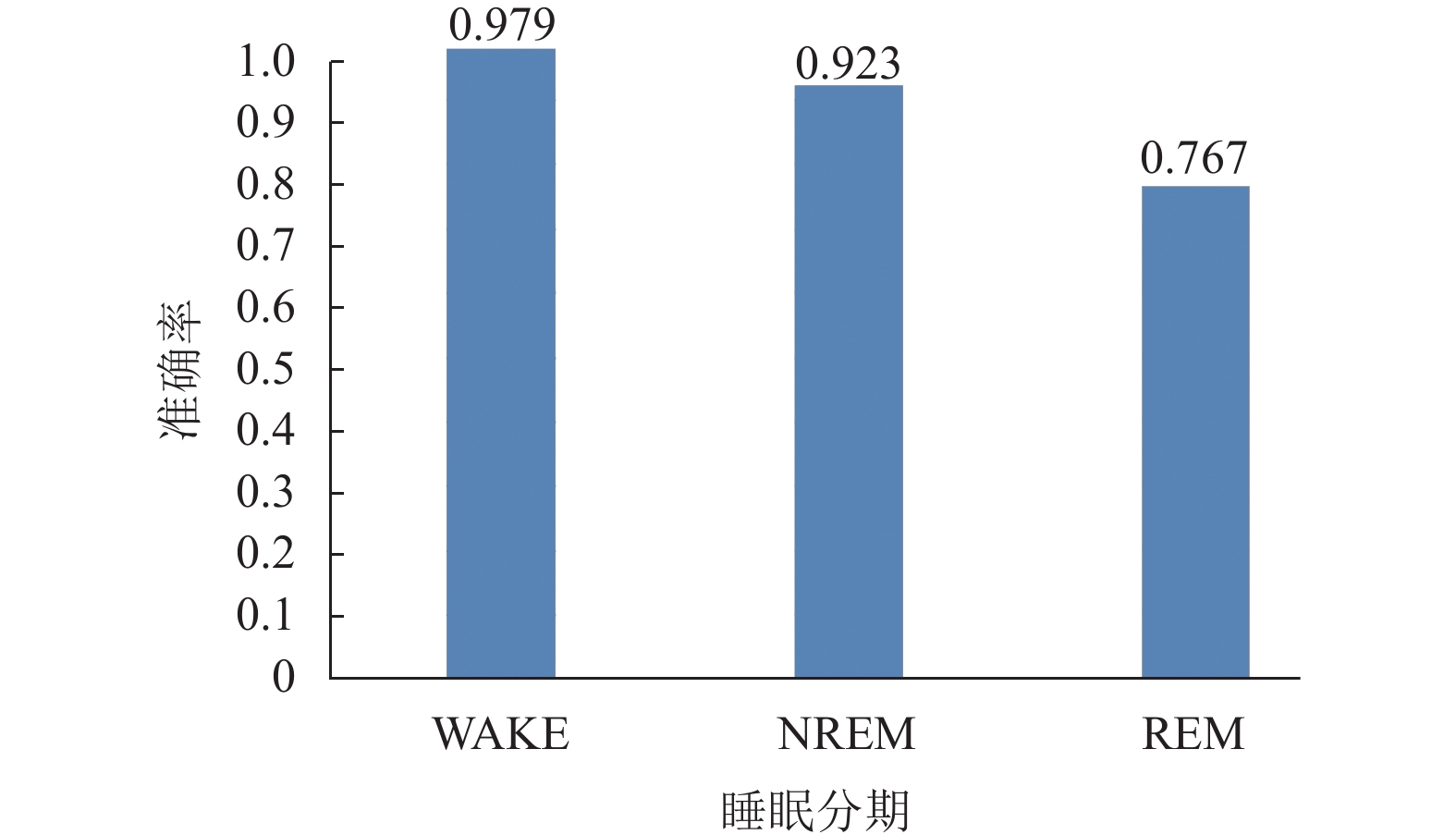
|
图 4 分期准确率 |
根据式(10)可以求得模型的平均准确率ACC= 89.0%. 从图4中可以看出, 3个睡眠时期都取得了较高的分期准确率. 进一步结合表1的分期结果混淆矩阵分析可得, 本文提出的睡眠分期算法能够较精确地识别出WAKE期和NREM期, 准确率分别达到97.9%和92.3%. 除此之外, REM期的准确率也达到了76.7%以上. 在对脑电信号进行自动睡眠分期的过程中, 本文所提出的分期算法的总体准确率可以达到89.0%, 这表明本文提出的算法模型在自动睡眠分期应用中具有良好的分期效果.
目前己经有很多的学者致力于对睡眠脑电EEG的自动分期研究, 为了能够更加全面地分析本文模型算法的分期结果, 本文将近年来有代表性的基于Sleep-EDF公共数据集的睡眠分期模型与本文的模型进行了对比分析, 如表2所示.
表2中, Tsinalis等人[18,19]使用机器学习算法Complex Morlet Wavelets+L-BFGS对Fpz-Cz和Pz-Oz两个通道进行五分类睡眠分期, 得到78.9%的准确率, 随后, 又使用 CNN 神经网络模型进行单通道睡眠信号五分类分期, 并从公开数据集选取20位实验对象的数据进行分析, 最终得到74.8%的准确率, 这与本文选取23位实验对象进行准确率评估较为接近; 除此之外, Hassan等人[20]使用Spectral features+K-W test+AdaBoost.M2的模型对Pz-Oz单个脑电通道进行四分类睡眠分期, 得到82.8%的准确率; Andreotti等人[21]使用ResNet模型对Fpz-Cz+EOG的脑电信号和眼电信号进行五分类睡眠分期, 得到86.1%的准确率; Phan等人[3]使用Image representation + MTCNN模型对Fpz-Cz+EOG的脑电信号和眼电信号进行五分类睡眠分期, 得到82.3%的准确率; Chen等人[22]使用Wavelet+SVM模型对单个Fpz-Cz脑电通道进行四分类睡眠分期, 得到86.8%的准确率. 对比表2中的结果, 单纯从算法的准确率上比较, 本文提出的算法在同一个数据集上的准确率达到一个较高的水平.
| 表 2 基于Sleep-EDF公共数据集的其它文献比较 |
可以看出, 本文提出的算法在睡眠分期的准确度上有了一定的突破. 与其它文献中的分期方法相比, 本文模型具有以下不同: ① 本文创新性地采用Mel滤波器进行睡眠脑电信号的预处理, 对每一个睡眠周期计算Mel频谱图, 通过Mel频谱对信号进行时频转换具有不错的效果; ② 本文采用了融合CNN和BiLSTM的神经网络模型, 充分挖掘了双向时间结构的信息, 实现更精确的特征提取; ③ 本文实现的是三分类算法, 在一定程度上也提高了模型的准确率.
为了更清楚地展示本文所提出的模型对于睡眠时期自动分类的结果, 本文将公开数据集的其中一名实验者数据, 也就是文件名为SC4001E0-PSG.edf的专家给定标签以及本文模型预测的结果进行分别绘制了睡眠时相图, 其中横轴为时间, 即持续了23小时的睡眠数据, 纵轴为3个睡眠期分类.
图5中给出了专家对这个持续23小时的实验睡眠数据进行了人工判定, 实验被试在不同的时间段分别处于不同的睡眠阶段, 根据人工判断结果, 在12:35–20:35时间段与03:35之后的时间内, 该名被试者一直处于WAKE期, 在20:35–03:35时间段内, 被试者进入睡眠状态, 其睡眠状态随着时间的改变在WAKE、NREM、REM三种睡眠状态下不断进行变化. 而图6中则是使用本文提出的模型对该睡眠数据进行分期的结果. 从模型进行分期的结果中可以看到, 模型对这个持续23小时的睡眠数据都进行了分期, 且在12:35–20:35时间段和03:35之后的时间内, 模型得到的分期结果也为WAKE期, 与图5基本接近; 在20:35–03:35时间段内, 模型得出的分期结果也在WAKE、NREM、REM三种睡眠状态下进行变化, 但与图5还存在一些差异.

|
图 5 专家分期结果时相图 |
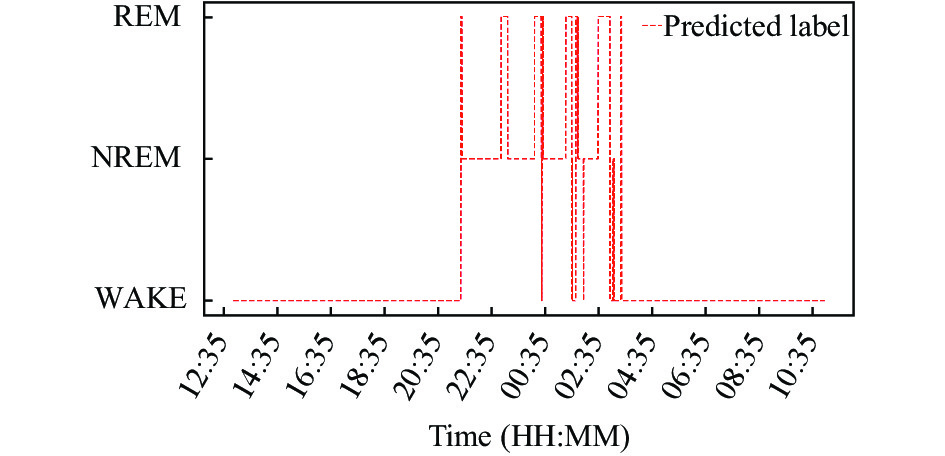
|
图 6 预测结果时相图 |
通过对比图5和图6, 可以看出本文自动睡眠分期模型与专家人工判别结果具有较大的重合性, 进一步验证了本文模型的准确性较高. 但在不同睡眠时期的过渡阶段, 模型判定的结果和专家分期结果有些不同, 考虑到处于过渡时期的睡眠可能具有不同时期的特征, 所以这一误差具有一定的合理性, 同时也为本文进一步研究发展指明了方向.
4 结语经过实验证明, 本文提出的基于算法对睡眠信号具有较好的分期性能. 算法选取Sleep-EDF数据集中双通道脑电数据Fpz-Cz和Pz-Oz进行分析, 使用Mel频谱对信号睡眠信号进行预处理, 结合卷积神经网络和双向长短时记忆神经网络进行时频域特征的提取, 目前在睡眠分期领域取得了比较好的性能. 实验结果表明, 本文模型在公开的睡眠数据集上取得了89.0%的整体准确率, 其中WAKE期的准确率达到97.9%, NREM期的准确率达到92.3%, REM期的准确率达到76.7%, 证明该模型具有良好的睡眠分期能力. 再通过将本文的工作与其他算法进行比较, 进一步验证了本文算法的有效性.
同时, 在对比其他不需要手动提取特征的模型时, 本文需要用到的生理信号更少, 只需要两个通道的脑电信号. 除此之外, 本文模型的网络结构更加简单, 训练速度也更快. 这对于不稳定、有起伏变化、非线性的脑电信号数据分析处理提供了新的研究思路想法和参考价值.
然而, 本文提出的算法还存在一定的缺点, 在不同睡眠时期的过渡阶段, 算法的判定结果与专家分期结果还存在一定的差异, 这表明了该算法的分期准确率还有一定上升的空间, 为本文进一步研究与发展指明了方向.
| [1] |
林少倩. 基于脑电信号的睡眠自动分期研究[硕士学位论文]. 杭州: 浙江工业大学, 2020, 2. |
| [2] |
Hobson JA. A manual of standardized terminology, techniques and scoring system for sleep stages of human subjects. Electroencephalography & Clinical Neurophysiology, 1968, 26(6): 644. |
| [3] |
Phan H, Andreotti F, Cooray N, et al. Joint classification and prediction CNN framework for automatic sleep stage classification. IEEE Transactions on Biomedical Engineering, 2019, 66(5): 1285-1296. DOI:10.1109/TBME.2018.2872652 |
| [4] |
Liang SF, Kuo CE, Hu YH, et al. Automatic stage scoring of single-channel sleep EEG by using multiscale entropy and autoregressive models. IEEE Transactions on Instrumentation and Measurement, 2012, 61(6): 1649-1657. DOI:10.1109/TIM.2012.2187242 |
| [5] |
Zhang JM, Wu Y. A new method for automatic sleep stage classification. IEEE Transactions on Biomedical Circuits and Systems, 2017, 11(5): 1097-1110. DOI:10.1109/TBCAS.2017.2719631 |
| [6] |
徐富献. 基于深度学习的单通道睡眠分期研究[硕士学位论文]. 厦门: 厦门大学, 2019, 16-23. |
| [7] |
Supratak A, Dong H, Wu C, et al. DeepSleepNet: A model for automatic sleep stage scoring based on raw single-channel EEG. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2017, 25(11): 1998-2008. DOI:10.1109/TNSRE.2017.2721116 |
| [8] |
Mousavi S, Afghah F, Acharya UR. SleepEEGNet: Automated sleep stage scoring with sequence to sequence deep learning approach. PLoS One, 2019, 14(5): e0216456. DOI:10.1371/journal.pone.0216456 |
| [9] |
Phan H, Andreotti F, Cooray N, et al. SeqSleepNet: End-to-end hierarchical recurrent neural network for sequence-to-sequence automatic sleep staging. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2019, 27(3): 400-410. DOI:10.1109/TNSRE.2019.2896659 |
| [10] |
Cecotti H, Graser A. Convolutional neural networks for P300 detection with application to brain-computer interfaces. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2011, 33(3): 433-445. DOI:10.1109/TPAMI.2010.125 |
| [11] |
杨浩, 黄茂林, 蔡志鹏, 等. 融合CNN和BiLSTM的心律失常心拍分类模型. 中国生物医学工程学报, 2020, 39(6): 719-726. DOI:10.3969/j.issn.0258-8021.2020.06.009 |
| [12] |
李洋, 董红斌. 基于CNN和BiLSTM网络特征融合的文本情感分析. 计算机应用, 2018, 38(11): 3075-3080. DOI:10.11772/j.issn.1001-9081.2018041289 |
| [13] |
郭浩, 许伟, 卢凯, 等. 基于CNN和BiLSTM的短文本相似度计算方法. 信息技术与网络安全, 2019, 38(6): 61-64, 68. |
| [14] |
罗森林, 郝靖伟, 潘丽敏. 基于CNN-BiLSTM的自动睡眠分期方法. 北京理工大学学报, 2020, 40(7): 746-752. |
| [15] |
Xiao YW, Wu J, Lin ZL, et al. A deep learning-based multi-model ensemble method for cancer prediction. Computer Methods and Programs in Biomedicine, 2018, 153: 1-9. DOI:10.1016/j.cmpb.2017.09.005 |
| [16] |
Gers FA, Schmidhuber J, Cummins F. Learning to forget: Continual prediction with LSTM. Neural Computation, 2000, 12(10): 2451-2471. DOI:10.1162/089976600300015015 |
| [17] |
程俊华, 曾国辉, 鲁敦科, 等. 基于Dropout的改进卷积神经网络模型平均方法. 计算机应用, 2019, 39(6): 1601-1606. DOI:10.11772/j.issn.1001-9081.2018122501 |
| [18] |
Tsinalis O, Matthews P M, Guo YK, et al. Automatic sleep stage scoring with single-channel EEG using convolutional neural networks. arXiv: 1610.01683, 2016.
|
| [19] |
Tsinalis O, Matthews PM, Guo YK. Automatic sleep stage scoring using time-frequency analysis and stacked sparse autoencoders. Annals of Biomedical Engineering, 2016, 44(5): 1587-1597. DOI:10.1007/s10439-015-1444-y |
| [20] |
Hassan AR, Bashar SK, Bhuiyan MIH. Automatic classification of sleep stages from single-channel electroencephalogram. Proceedings of 2015 Annual IEEE India Conference (INDICON). New Delhi: IEEE, 2015. 1–6.
|
| [21] |
Andreotti F, Phan H, Cooray N, et al. Multichannel sleep stage classification and transfer learning using convolutional neural networks. Proceedings of the 40th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Honolulu: IEEE, 2018. 171–174.
|
| [22] |
Chen T, Huang HY, Pan JH, et al. An EEG-based brain-computer interface for automatic sleep stage classification. Proceedings of the 13th IEEE Conference on Industrial Electronics and Applications (ICIEA). Wuhan: IEEE, 2018. 1988–1991.
|
 2022, Vol. 31
2022, Vol. 31



