肋骨骨折是骨伤科中十分常见的疾病, 其发生率大概在40%~80%[1], 并且有逐年上升的趋势. 而肋骨骨折同时导致肺炎、气胸等危及生命的并发症[2]. 当观察肋骨区域时, CT图像其优势在于可更充分、细致的观察到骨折区域, 能满足于临床肋骨骨折诊断要求[3, 4], 所以CT图像已成为临床上诊断骨折病症常用的方法[5]. 但目前针对CT图像的肋骨骨折诊断仍然是一项复杂的任务, 由于整个诊断过程依赖临床医生的诊断经验, 而单个病人的病理图像多达几百张, 甚至上千张, 且受不同设备、不同环境等因素的影响, 导致医生分析、诊断肋骨骨折的工作特别繁琐且艰难. 同时, 过大工作量易使医生产生疲劳现象, 使诊断肋骨骨折病症时产生误诊现象. 因此, 实现快速准确的肋骨骨折计算机辅助诊断技术对于临床治疗具有十分重要的意义.
随着近年来计算机技术的兴起, 利用图像处理方法辅助标注肋骨已成为众多学者不断研究的问题. 由于肋骨分布较广且形状较小, 许多针对肋骨CT图像的分割方法被提出, 如郑惠敏等人[6]提出一种肋骨投影N-维平面方法, 通过把肋骨投影到二维平面从而消除肋骨遮挡产生的影响, 另外应用增强滤波算子强化肋骨边缘分割出肋骨图像, 赵晓飞等人[7]提出应用区域增长方法进行肋骨追踪进而实现肋骨分割的算法. 其中有部分研究致力于帮助医生实现肋骨图像重建, 如王淑丽等人[8]应用多层螺旋CT扫描方法进行并行薄层及曲面重建, 算法可完整显示肋骨骨折位置与形态, 而杨东等人[9]提出了基于CT数据的肋骨骨折自动诊断系统, 通过改进的经典中心性算法以及曲面重建算法(CPR)对单根肋骨进行可视化, 帮助临床医生提高肋骨骨折的诊断效率, 但该系统只对部分骨折类型敏感. Urbaneja等人[10]使用前像图像与后向图像两种图像, 确定了识别肋骨骨折形状和位置的具体参数, 但该方法需要花费大量精力进行手工参数制定.
近年来, 卷积神经网络由于在图像识别的高性能被广泛应用, 针对医学图像方面应用于宫颈细胞核图像分割[11]、心血管造影图像分割[12]领域等. 国内外研究者将卷积神经网络应用于肋骨骨折识别研究, 如Hu等人[13]提出了切片分组聚合网络(slice grouping and aggregation network, SGANet), 首先通过三维(3D)卷积网络对不同切片进行聚合, 然后使用二维卷积网络提取各张切片特征进行分类. Chen等人[14]提出了空间相干性检测算法(spatial coherence based rib fracture detection, SCRFD), 首先从CT切片中将肋骨区域提取出来, 之后利用结合空间信息的卷积神经网络对该区域进行识别, 但该方法具有较差的鲁棒性. 而Zhou等人[15]提出了一种基于卷积神经网络的肋骨骨折辅助识别系统, 可自动定位并分类多种骨折类型, 该系统具有良好的检测性能, 同时将辅助诊断与人工诊断结果对比表明该系统可有效帮助医生提高诊断效率. Jin等人[16]提出了一种卷积神经网络模型FracNets, 进行人工标注后对三维肋骨图像进行检测, 实现了92.9%的检测灵敏度. 以上都是应用深度学习技术对肋骨骨折进行识别的研究, 但仍然存在部分不足: (1)识别算法需要的计算代价仍然巨大, 无法保证大量CT图像在临床诊断的实时检测速度. (2)整体网络未考虑到肋骨区域的多尺度信息, 导致分类精度不高.
为全面提取肋骨CT图像整体信息, 同时为达到医学设备对检测速度的要求, 本文提出了一种基于RF-Net的肋骨骨折识别算法.
本文的主要贡献有以下几点:
(1)使用生成对抗网络对肋骨CT图像进行数据增强作为额外训练集, 丰富图像的多样性, 解决医学图像匮乏而模型难以训练的难题.
(2)使用多分支模块RF-block构建了卷积神经网络RF-Net, 该模块可提取肋骨的多尺度特征信息, 着重关注肋骨区域的形状特征, 提高检测性能.
(3)提出压缩策略充分优化模型结构, 在减少参数量的同时提升分类性能. 同时在肋骨数据集上验证了所提出的方法领先于多种主流分类网络, 另外进行了充分的消融实验, 证明了各模块的有效性.
1 相关工作 1.1 MobileNetMobileNet由于其高效检测精度和检测速度符合移动设参数要求受到广泛关注. 其主体结构为深度可分离卷积(depthwise separable convolution, Dw conv), 深度可分离卷积将原有的标准卷积因式分解为深度卷积和点卷积, 其中深度卷积对输入特征图中不同通道分别使用一个卷积核得到多维特征图, 点卷积使用1×1卷积将多个特征图按顺序拼接, 而点卷积对深度卷积作用后的特征图进行线性组合起到升维或者降维的效果, 操作过如图1所示.
假设输入特征图为M×M, 输出通道数为C, 卷积核大小为N×N, 个数为K, 深度卷积计算量P1, 点卷积计算量P2分别为:
| $ {P_1} = M \times M \times N \times N \times C $ | (1) |
| $ {P_2} = M \times M \times N \times N \times C + M \times M \times C \times K $ | (2) |

|
图 1 深度可分离卷积 |
故深度可分离卷积参数量与标准卷积参数量比值Ratio为:
| $ Ratio{\text{ }} = \frac{{{P_1} + {P_2}}}{{M \times M \times N \times N \times C \times K}} = \frac{1}{M} + \frac{1}{{{N^2}}} $ | (3) |
当卷积核大小N×N为3×3时, 由式(3)可知深度可分离卷积相比于标准卷积可减少8至9倍的计算量, 故使用深度可分离卷积的MobileNet具有高效性.
1.2 传统数据增强方法医学数据具有一定的隐私性与复杂性, 因此数据量一般较少. 但深度学习模型的性能非常依赖于训练数据的数量, 更多的训练数据往往伴随着更好的深度学习模型, 相比较于直接收集数据, 在原有数据集上应用数据增强技术由于低成本的特性被广泛采用. 在CT图像中传统数据增强方式就是对图像进行随机仿射变换处理, 包含翻转、缩放、镜像、平移等方法, 而为获得更多的数据, 在实际应用中会同时将上述多种方法组合使用, 但需要注意的是, 传统数据增强方法虽然可以有效扩大数据集数量, 但无法改变图像本身的内容, 易使模型产生过拟合效应, 降低模型性能.
2 本文方法本文提出了一种基于RF-Net的肋骨骨折识别算法, 图2展示了该方法的流程图, 核心思想如下: 首先应用生成对抗网络对原始样本进行数据增广, 产生许多高质量的生成样本构建全新的训练集. 然后使用RF-block替换深度可分类卷积单元, RF-block可学习到多尺度特征, 进行特征融合后传递给下层网络, 加强骨折区域上下文信息之间的联系. 同时应用压缩策略对冗余模块进行优化. 本节将详细介绍基于生成对抗网络的数据增强方法实现过程以及RF-Net的整体结构.

|
图 2 论文框架图 |
2.1 基于生成对抗网络的数据增强方法
生成对抗网络是近年来用于生成随机数据的一种框架, 其由生成器(generative model, G)、辨别器(discriminative model, D)两种模型共同组成. 前者试图生成接近真实的数据, 而后者试图分辨真实图像与虚假图像, 在最理想的情况下, 整个网络的目的为生成器能够生成“以假乱真”的图像, 即模型的目标函数V(D, G)为:
| $ \begin{split} {\mathop {\min}\nolimits_{{G}}}{\mathop {\max}\nolimits_{{D}}}V(D, G) = &{{{E}}_{x\sim{{{p}}_{{\text{data }}}}(x)}}[\log D(x)] \\ &+ {{{E}}_{{\textit{z}}\sim{{{p}}_{\textit{z}}}({\textit{z}})}}[\log (1 - D(G({\textit{z}})))] \end{split} $ | (4) |
其中, z表示输入噪声变量, x表示真实样本数据, D(x)表示辨别器网络识别x是真实样本的概率, 而G(x)表示生成器产生的一个样本, pz表示从噪声中生成的概率分布, pdata表示从原始数据中学习到的概率分布, 函数的最优值即为pz=pdata, 此条件下辨别器无法正确分辨生成样本与真实样本, 同时生成器已经可以生成高质量的“完美”样本.
生成对抗网络产生了各种各样的变体模型, Mirza等人[17]提出了深度卷积生成对抗网络(deep convolutional generative adversarial nerwork, DCGAN), 由于其优秀的性能被广泛应用. 本文基于DCGAN构造了4层卷积网络的生成器与辨别器, 图3展示了该网络的整体结构. 生成器的输入为1×100的符合正态分布的向量, 经过反卷积层(deconvolution, DeConv)输出分辨率为32×32的图像.辨别器的输入为生成器产生的图像, 经过卷积层(convolution, Conv)输出辨别概率.
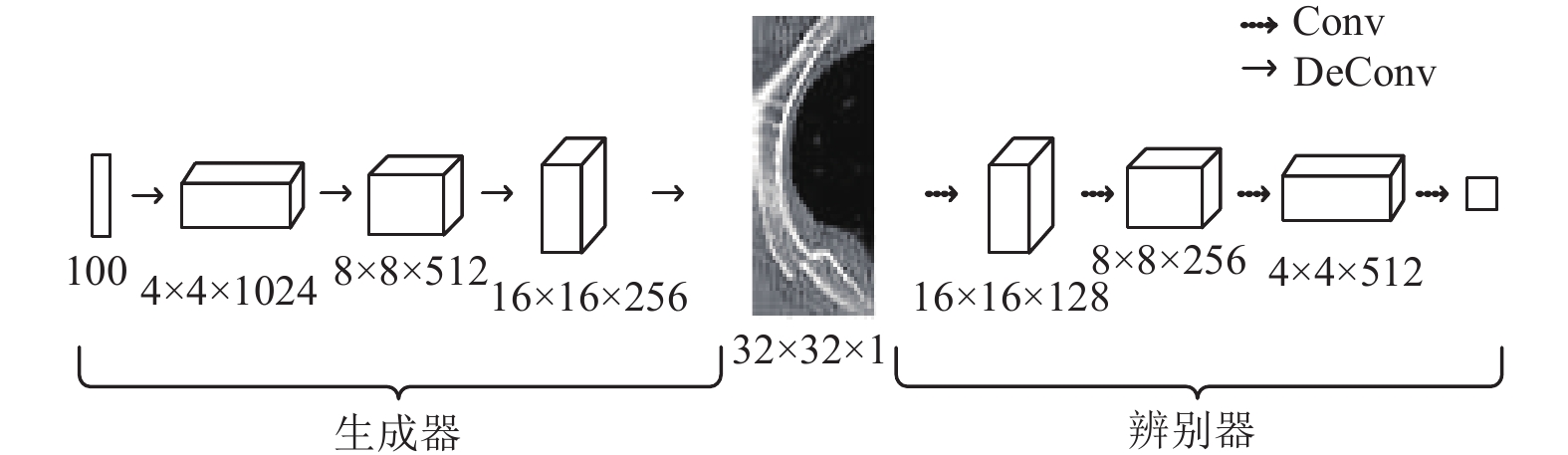
|
图 3 DCGAN网络 |
为验证基于生成对抗网络的数据增强方法实际效果, 本文将在实验部分说明具体细节, 同时通过消融实验证明此方法下各模型分类性能优于传统数据增强方法.
2.2 RF-Net由于肋骨骨折病症的巨大差异性, 且存在不同生理特征的干扰, 导致部分骨折情况复杂. 故需要结合多尺度信息对肋骨进行针对性学习. 本文使用MobileNet[18]作为基础架构, 结合RF-block与压缩策略提出了RF-Net, 提取出多尺度特征的同时对网络结构进行充分优化, 更高效的处理图像信息.
2.2.1 RF-block应用深度可分离卷积可有效加深网络以及提升推理效率. 但该单元仍存在部分不足, 由于单分支结构的局限性, 难以提取到强大的多尺度特征, 导致模型出现部分精度损失.
在此基础上, 本文提出了RF-block模块, 该模块由深度可分离卷积和1×1卷积组成, 分别作用于输入得到不同特征图, 使用Contcat操作进行特征融合, 此时不仅能获得肋骨的空间特征, 又可获得非线性的原始特征, 实现多尺度特征提取. 另外RF-block在前后通道图上进行跳跃连接(skip-connection), 有效避免梯度消失问题, 实现跨通道的信息整合.
RF-block的结构图如图4所示, 使用两个通道数为C/2的卷积层代替通道数为C的深度可分离卷积, 整体结构共分为池化单元与非池化单元, 非池化单元输入输出特征图尺寸相同, 而池化单元使用最大池化层进行下采样, 输出特征图尺寸减半, 有利于下层卷积更有效地处理特征. 相比较而言, 在参数量相同的条件下, RF-block不仅保留了深度可分离卷积的高效性, 同时使网络学习到更深层次的多尺度特征.

|
图 4 RF-block结构图 |
2.2.2 压缩策略
由于肋骨问题需在短时间内得到检测结果, 而单个病例具有大量CT图像, 故算法计算参数量应处于一定规模之下, 而MobileNet模型结构仍存在额外计算成本. 针对以上问题, 本文提出了模型压缩策略, 首先通过快速下采样操作[19]最大限度保留原有信息, 之后对高维结构进行优化.
作为卷积神经网络中的常用操作, 下采样层通过缩放操作成倍减小特征图的尺寸, 可大幅度降低模型计算量. 而上层输入特征图的尺寸可影响下层卷积操作所需要的计算规模, 简单分析可得, 越早进行下采样, 下层网络的计算规模越小, 故本文使用快速下采样方法对RF-Net整体结构进行优化, 方法示意图如图5所示.

|
图 5 快速下采样示意图 |
从图5中可看到, 原下采样网络交替经过3个非池化层与3个池化层, 每1个池化层前的非池化层易破坏原始肋骨特征信息, 而快速下采样方法连续经过3个池化单元, 直接使输入特征图尺寸缩小8倍. 与原网络相比较, 应用快速下采样策略后的网络能够保留更多全局信息. 由于快速下采样策略使原有特征图质量不可避免地受到影响, 导致网络高维卷积层存在多个空卷积核. 故可删除多个高维模块以弥补快速下采样方法的不足. 在本文问题下, 压缩策略可有效减少参数量的同时保留良好的特征提取能力, 实现细节如表1结构所示.
| 表 1 RF-Net结构参数 |
2.2.3 整体模块设计
表1列出输入尺寸为224×224的图像时RF-Net的具体网络结构参数. 其中s表示步长, C表示模块输出通道数. 网络骨干部分共分为5个阶段, 阶段[1, 2, 3, 4, 5]分别对应于输出通道数为[64, 128, 256, 512, 1024]的RF-block, 每个阶段的末尾均为RF-block的池化单元, 池化单元通过最大池化层降低特征图的尺寸而传递信息到下一阶段. 相比于MobileNet, RF-Net仅保留两个通道数为512的RF-block与一个通道数为1024的RF-block, 以解决高维信息的丢失问题. 具体来说RF-Net由9层模块构成, 包含1个带池化的卷积层, 4个带池化的RF-block以及4个未带池化的RF-block, 使用全局平均池化层(global average pooling, GAP)代替全连接层. GAP层对每一个特征图求均值, 后经过Sigmoid分类器得到分类结果. 实验证明RF-Net网络在降低计算成本的同时可有效提升肋骨识别精度.
3 实验 3.1 准备工作 3.1.1 肋骨数据集为验证本文算法的分类性能, 本文构建取自湖南省湘雅医院的临床肋骨数据集, 采集来自20个病例的500张CT医学图像. 图像分辨率为512×512, 切片厚度为1~5 mm. 每张样本都取自于肋骨骨折的病人且已进行去信息化处理. 首先标记出一块不超过32×32像素的矩形区域, 进行切片后形成单张图像作为实验样本, 标注过程如图6所示, 左侧图6(a)为标记出的非骨折图像, 右侧图6(b)为标记处的骨折图像. 图像共分为骨折与非骨折两种类别, 每标注一张骨折图像的同时标记一张对应的非骨折图像以保证训练样本数量的均衡. 经过标记操作数据集最后包含1468张图像, 骨折与非骨折类型数量相同均为734张.

|
图 6 数据集标注过程 |
3.1.2 数据预处理
将肋骨数据集按7:2:1比例划分为1027张训练集与294张测试集及146张验证集. 同时为使数据匹配网络模型输入, 将图像像素调整为224×224并转换为RGB 3通道格式.
使用基于生成对抗网络的数据增强方法扩增数据集, 其具体操作为: 使用单个类别图像分别训练DCGAN网络, DCGAN中生成器与辨别器相互对抗, 当网络已经学习到原图像的概率分布后, 生成器接收100维的随机向量而生成像素为32×32的图像, 对两种类别分别进行上述操作, 为保证样本数量的平衡, 共生成2054张骨折图像与2054张非骨折图像. 部分生成图像与原始图像分别如图7(a)和图7(b)所示, 上下图像具有相似的肋骨特征, 但不是简单的仿射变换, 丰富了数据集的多样性, 故认为生成图像适合用于扩充训练集.

|
图 7 肋骨CT图像 |
3.1.3 评价指标
为了客观且全面地评价网络的分类性能, 同时方便与其他算法进行比较, 本文采用准确率(accuracy, ACC)、灵敏度(sensitivity, SE)、特异度(specificity, SP)、ROC (receiver operating characteristic curve)曲线作为评价指标进行定量分析与比较, 将骨折视为阳性, 非骨折视为阴性, 各指标具体计算式为:
| $ ACC = \frac{{TP + TN}}{{TP + TN + FP + FN}} $ | (5) |
| $ SE = \frac{{TP}}{{TP + FN}} $ | (6) |
| $ SP = \frac{{TN}}{{TN + FP}} $ | (7) |
| $ TPR = \frac{{TP}}{{TP + FN}} $ | (8) |
| $ FPR = \frac{{FP}}{{TN + FP}} $ | (9) |
式中, FP表示假阳性的个数, TP表示真阳性的个数, FN表示假阴性的个数, TN表示真阴性的个数.其中ACC描述模型整体性能, SE描述模型检测骨折的性能, SP描述模型检测非骨折的性能, ROC曲线横轴为FPR (false positive rate), 表示在所有非此类别下被预测为正确的概率, 纵坐标为真阳率TPR (true positive rate), 表示在该类别下被预测出来的概率, 根据此数值做ROC曲线, AUC (area under curve)就是该曲线下面的面积, 曲线越靠近左上角, 表示AUC值越接近于1, 则算法的分类性能越好. 各项数据均取多次实验结果的平均值.
3.1.4 实验环境本论文实验基于Keras框架进行网络搭建及训练, 硬件平台为Intel(R) Core(TM) i7-8750H CPU @ 2.20 GHz, GPU为NVIDIA Geforce GTX 1060 (8 GB显存). 分类实验参数设置如下: 训练迭代次数epoch为100次, 批处理大小batch_size为64, 使用随机梯度下降法(stochastic gradient descent, SGD)进行优化, 初始学习率learning_rate为0.01. 动量参数为0.9, 学习率衰减值为0.0001. 生成对抗网络的实验参数设置如下: 训练迭代次数epoch为14000次, 批处理大小batch_size大小为32, 使用自适应学习率优化器Adam[20]进行优化, 初始学习率learning_rate设为0.0002, 衰减因子设为0.99.
3.2 结果与分析 3.2.1 实验结果与分析将RF-Net模型与现有的经典网络EfficientNet[21]、MobileNet、VGG16[22]、Inception-v3[23]、DenseNet[24]、ResNet-50[25]、ResNet-152[25]、Xception[26]在肋骨数据集上进行对比实验, 在训练完全收敛的情况下, 所有模型在测试集上对比结果如表2所示.
从表中可看出使用基于生成对抗网络的数据增强方法, 所有模型的准确率均高于90%, 表明该方法在肋骨问题上有一定优势, RF-Net在肋骨测试集上准确率、AUC、敏感度、特异性分别为0.9726, 0.9966, 0.9778, 0.9674. 4种指标均高于其他模型, 证明本文方法可更精确的检测出各骨折图像. 具体来说, 相较于MobileNet, 本文方法的准确率, AUC值分别提升1.09%, 1.05%, 参数量降低约65.6%. 相对目前最受欢迎的分类网络EfficientNet, 本文方法的准确率、AUC值增幅分别达到1.20%, 1.05%, 并且模型参数量降低约79.2%, 从而表明本文算法能够在低参数量的前提下兼顾更优的模型分类性能.
| 表 2 与多种分类模型在肋骨数据集上的性能对比 |
3.3 消融实验与分析 3.3.1 ISIC 2016
为验证本文模型的分类性能, 本文在二分类数据集ISIC 2016[27]进行消融实验, ISIC 2016是由ISIC协会发布的黑色素瘤检测皮肤数据集. 共包含900张训练集图像和379张测试集图像, 图像分辨率为224×224, 数据集的任务为检测图像中的黑色毒瘤为恶性或者良性. 实验预处理过程将对训练集图像进行标准化、翻转、平移等数据增强操作, 对测试集仅进行标准化处理.
表3为本文模型与ResNet-50、Inception-v3、DenseNet-201、MobileNet四种现有网络模型及其他论文中模型的对比结果. RF-Net模型在ISIC测试集上准确率与AUC值分别为0.8578与0.8208, 相对于MobileNet模型分别提高2.7%, 1.3%, 与CUMED算法[28]相比较, RF-Net准确率, AUC值分别提高0.3%, 2.0%. 另一方面, SDL算法[29]分类性能虽略高于本文算法, 但该增幅需要消耗巨大的计算资源. 该实验验证了本文模型在其他数据集上同样具有较优的特征识别能力, 能够快速学习与适应不同图像, 即模型具有较好的鲁棒性.
| 表 3 与已有方法在ISIC数据集上的性能对比 |
3.3.2 数据增强方法对比
为进一步评估基于生成对抗网络的数据增强方法在改善模型上的能力, 使用两种数据增强方法在和多个分类模型搭配在肋骨数据集上进行对比实验, 将传统方法实验训练集由原图像与传统方法生成的图像构成, 而本文方法实验训练集由原图像与生成对抗网络生成的图像构成, 使用相同测试集进行分类实验. 不同模型分别在两种方法的测试结果如图8所示.
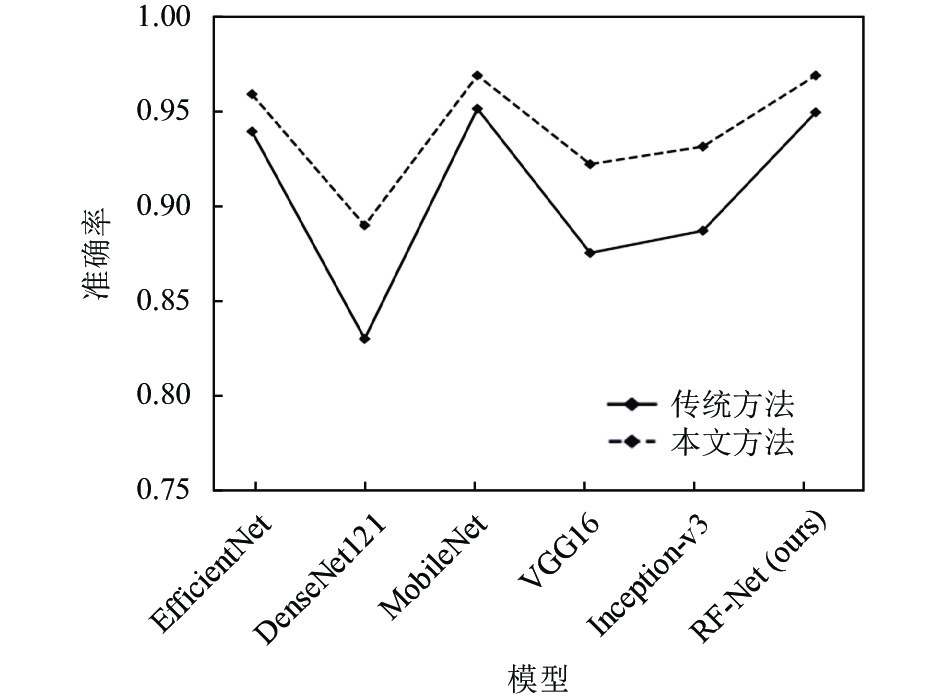
|
图 8 不同数据增强方法对比 |
图8中直线表示传统方法结果, 虚线表示本文方法结果, 从图中可看到本文数据增强方法处理后的模型分类性能显著优于传统数据增强方法, 准确率增幅均高于2.1%, 如RF-Net模型准确率增幅约为2.4%, 且该方法对部分网络提升更加显著, 如DenseNet-121模型分类准确率提升约达14.2%. 传统数据增强方法的分类性能较差, 原因是模型对部分训练数据产生过拟合效应, 而基于生成对抗网络的数据增强方法可有效解决该问题, 即生成对抗网络可自学习图像特征, 能够产生质量更好、特异性更强的图像, 加速模型训练从而有效提升分类性能.
3.3.3 模块有效性为验证本文RF-block、压缩策略的有效性, 在肋骨数据集上进行对比实验, 通过不同实验组合以验证各个模块对于模型性能的实际影响, 共设计MobileNet, MobileNet+ RF-block, MobileNet+压缩策略及RF-Net四种方案. 实验结果如表4所示.
实验结果表明, 应用RF-block后的模型准确率与AUC值分别提高0.38%, 0.57%, 表明RF-block能够结合多尺度特征来有效提升模型特征提取能力, 而应用压缩策略后的模型参数量减少2.20 M, 表明压缩策略可在保持性能的基础上有效减少模型参数量. 该对比实验验证了各个模块的可行性. 另外证明了不同模块能够结合各自优势, 有效改善模型对肋骨骨折图像的识别能力.
| 表 4 不同模块分类性能比对 |
4 结论
针对肋骨CT图像数据量大, 识别难度大等问题, 本文提出了一种基于RF-Net的肋骨骨折识别算法, 以实现计算机辅助诊断肋骨骨折病症. 本文方法首先使用生成对抗网络对肋骨图像进行数据增强以缓和学习过程中的过拟合现象, 然后应用RF-Net进行分类. 在肋骨数据集上的对比实验表明了本文方法优于多个主流深度学习模型. 此外, 消融实验进一步验证了本文提出的基于生成对抗网络的数据增强方法的高效性及RF-block模块和压缩策略的有效性.
本文的肋骨识别集中在类别较少的数据集上, 在后续的研究工作中, 将考虑对更多类型的肋骨骨折病症做进一步研究.
| [1] |
Cho SH, Sung YM, Kim MS. Missed rib fractures on evaluation of initial chest CT for trauma patients: Pattern analysis and diagnostic value of coronal multiplanar reconstruction images with multidetector row CT. The British Journal of Radiology, 2012, 85(1018): e845-e850. DOI:10.1259/bjr/28575455 |
| [2] |
Liman ST, Kuzucu A, Tastepe AI, et al. Chest injury due to blunt trauma. European Journal of Cardio-Thoracic Surgery, 2003, 23(3): 374-378. DOI:10.1016/s1010-7940(02)00813-8 |
| [3] |
葛虓俊, 吴昊, 滑炎卿, 等. 低剂量CT扫描诊断肋骨骨折的临床应用. 中华放射学杂志, 2011, 45(5): 492-495. DOI:10.3760/cma.j.issn.1005-1201.2011.05.016 |
| [4] |
Traub M, Stevenson M, McEvoy S, et al. The use of chest computed tomography versus chest X-ray in patients with major blunt trauma. Injury, 2007, 38(1): 43-47. DOI:10.1016/j.injury.2006.07.006 |
| [5] |
牛玉军, 何翠菊, 闫晓虹, 等. 肋骨骨折各种影像学诊断价值及对比分析. 中国现代医学杂志, 2006, 16(14): 2161-2165. DOI:10.3969/j.issn.1005-8982.2006.14.025 |
| [6] |
郑惠敏. 面向肋骨骨折诊断的图像处理方法研究[硕士学位论文]. 北京: 中国科学院大学, 2016.
|
| [7] |
赵晓飞. 一种新的可视化肋骨骨折诊断方法研究[硕士学位论文]. 杭州: 浙江大学, 2016.
|
| [8] |
王淑丽, 王林森, 孙鼎元, 等. 多层螺旋CT在肋骨微细骨折诊断中的应用价值. 中华放射学杂志, 2005, 39(12): 1289-1292. DOI:10.3760/j.issn:1005-1201.2005.12.013 |
| [9] |
杨东. 基于CT图像的肋骨骨折自动诊断系统[硕士学位论文]. 北京: 中国科学院大学, 2011.
|
| [10] |
Urbaneja A, De Verbizier J, Formery AS, et al. Automatic rib cage unfolding with CT cylindrical projection reformat in polytraumatized patients for rib fracture detection and characterization: Feasibility and clinical application. European Journal of Radiology, 2019, 110: 121-127. DOI:10.1016/j.ejrad.2018.11.011 |
| [11] |
张权, 陆小浩, 朱士虎, 等. 基于改进U-Net的宫颈细胞核图像分割. 计算机系统应用, 2021, 30(4): 39-45. DOI:10.15888/j.cnki.csa.007862 |
| [12] |
亢伉. 基于局部边缘特征水平集演化的主动脉CT图像分割. 计算机系统应用, 2019, 28(7): 180-183. DOI:10.15888/j.cnki.csa.006999 |
| [13] |
Hu YT, He XC, Zhang R, et al. Slice grouping and aggregation network for auxiliary diagnosis of rib fractures. Biomedical Signal Processing and Control, 2021, 67: 102547. DOI:10.1016/j.bspc.2021.102547 |
| [14] |
Chen M, Du P, Zhao JY. SCRFD: Spatial coherence based rib fracture detection. Proceedings of the 2018 5th International Conference on Biomedical and Bioinformatics Engineering. Okinawa: ACM. 2018. 105–109.
|
| [15] |
Zhou QQ, Wang JS, Tang W, et al. Automatic detection and classification of rib fractures on thoracic CT using convolutional neural network: Accuracy and feasibility. Korean Journal of Radiology, 2020, 21(7): 869-879. DOI:10.3348/kjr.2019.0651 |
| [16] |
Jin L, Yang JC, Kuang KM, et al. Deep-learning-assisted detection and segmentation of rib fractures from CT scans: Development and validation of FracNet. EBioMedicine, 2020, 62: 103106. DOI:10.1016/j.ebiom.2020.103106 |
| [17] |
Mirza M, Osindero S. Conditional generative adversarial nets. arXiv: 1411.1784, 2014.
|
| [18] |
Howard AG, Zhu ML, Chen B, et al. MobileNets: Efficient convolutional neural networks for mobile vision applications. arXiv: 1704.04861v1, 2017.
|
| [19] |
Qin Z, Zhang ZN, Chen XT, et al. FD-MobileNet: Improved MobileNet with a fast downsampling strategy. Proceedings of the 2018 25th IEEE International Conference on Image Processing (ICIP). Athens: IEEE, 2018. 1363–1367.
|
| [20] |
Kingma DP, Ba J. Adam: A method for stochastic optimization. Proceedings of the 3rd International Conference on Learning Representations. San Diego. arXiv: 1412.6980, 2014.
|
| [21] |
Tan MX, Le QV. Efficientnet: Rethinking model scaling for convolutional neural networks. Proceedings of the 36th International Conference on Machine Learning. Long Beach: PMLR, 2019. 6105–6114.
|
| [22] |
Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition. Proceedings of the 3rd International Conference on Learning Representations. San Diego, 2014.
|
| [23] |
Szegedy C, Vanhoucke V, Ioffe S, et al. Rethinking the inception architecture for computer vision. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas: IEEE, 2016. 2818–2826.
|
| [24] |
Huang G, Liu Z, van der Maaten L, et al. Densely connected convolutional networks. Proceedings of 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Honolulu: IEEE, 2017. 2261–2269.
|
| [25] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas: IEEE, 2016. 770–778.
|
| [26] |
Chollet F. Xception: Deep learning with depthwise separable convolutions. Proceedings of 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Honolulu: IEEE, 2017. 1800–1807.
|
| [27] |
Gutman D, Codella NCF, Celebi E, et al. Skin lesion analysis toward melanoma detection: A challenge at the international symposium on biomedical imaging (ISBI) 2016, hosted by the International Skin Imaging Collaboration (ISIC). arXiv: 1605.01397, 2016.
|
| [28] |
Yu LQ, Chen H, Dou Q, et al. Automated melanoma recognition in dermoscopy images via very deep residual networks. IEEE Transactions on Medical Imaging, 2017, 36(4): 994-1004. DOI:10.1109/TMI.2016.2642839 |
| [29] |
Zhang JP, Xie YT, Wu Q, et al. Medical image classification using synergic deep learning. Medical Image Analysis, 2019, 54: 10-19. DOI:10.1016/j.media.2019.02.010 |
 2022, Vol. 31
2022, Vol. 31


