新型冠状病毒(新冠肺炎)自2020年初被发现以来, 已在全球范围内迅速蔓延, 严重威胁着人类健康, 尽早发现并加以隔离治疗是遏制新冠肺炎蔓延的有效途径. 目前, 利用深度学习技术进行肺部CT图像的分析可以尽早检测出是否感染的情况[1-3], 但训练检测模型需要大量带标注的图像, 数据量过少容易出现过拟合的情况, 导致模型检测精度降低. 利用传统的医学成像设备进行采集标注不仅耗时、昂贵而且会导致患者暴露辐射以及涉及患者隐私等问题, 使得短时间内难以获取到大量的CT图像用于训练. 针对医学图像的缺乏问题, 很多研究人员致力于寻求有效的方法来实现医学图像的扩增和重建工作.
目前, 常用的图像扩增方式大都是基于深度学习的方法, 尤其是利用生成对抗网络(generative adversarial networks, GAN)[4]来扩增图像. 现有的CT图像生成研究多是集中于不同模态的转换之中, 例如从配对的MR图像向对应CT图像的转换[5, 6]. 较为经典有Nie等人[7]提出的基于上下文感知的生成对抗网络的CT图像合成方法; Zhang等人[8]提出的基于CNN并结合多种损失函数的GAN模型, 实现了MR图像和CT图像的相互转换; Emami等人[9]和Gholamiankhah等人[10]分别提出的使用residual和CNN修改的GAN网络实现从MR图像到目标CT图像的转换. 除了从配对MR图像生成CT图像外, 还有Ying等人[11]提出X2CTGAN模型, 利用双平面X射线图像来重建CT图像, 将肺部图像从基于2D的X射线图像映射为3D的CT图像.
由于不同模态图像的配对结果影响CT图像的生成质量, 而且获取配对准确的CT图像和MR图像也是非常困难且成本昂贵的. 为了解决该问题, 研究人员提出了DualGAN[12]和CycleGAN[13]架构, 从而为实现无需配对的不同模态图像转换带来了可能. 基于此, 吴香奕等人[14]提出使用CycleGAN实现未配对的盆腔部位MR图像和CT图像的相互转换.
Wolterink等人[15]提出使用CycleGAN模型实现从未配对的脑部MR图像生成CT图像. 以及Chartsias等人[16]提出使用CycleGAN实现心脏CT图像与MR图像之间的转换. 然而不同模态之间的转换方法虽然可以实现CT图像的生成, 却受限于需要同时具备两个不同模态图像. 在新冠疫情影响下, 训练检测模型需要具有感染区域多样性的CT图像来参与训练, 以应对未知CT图像感染区域的预测. 但目前并没有可以用来通过模态转换生成受感染的CT图像的其它模态数据, 而且通过此类方法也会存在丢失部分特征信息的问题, 因此难以实现批量扩增受感染的CT图像.
针对新冠CT图像稀缺现状, 本文以公开的COVID-19 CT数据集为基础, 提出一种新颖可编辑的三阶段CT图像生成方法(three-stage amplification method), 简称为TSA方法, 来扩增CT图像. 首先, 利用无监督的生成对抗网络扩增感染分割标签; 其次, 基于泊松编辑方法实现对病灶区域的融合; 最后, 利用有监督的条件生成对抗网络CGAN[17]将编辑标签生成对应的CT图像. 该方法一方面不需要其它模态配对图像即可实现CT图像的扩增; 另一方面, 通过扩增分割标签和编辑病灶区域, 可以实现图像数据的多样性; 同时, 基于标签生成对应的CT图像还可以减少人工标注的工作. 基于Kaggle公开的COVID-19数据集进行图像生成实验和分割实验, 结果证明了本文方法的有效性.
文章的其余部分组织如下: 在第2节中, 介绍了生成对抗网络的相关概念. 在第3节中, 介绍了本文提出的方法. 在第4节中, 介绍了实验数据集和生成分割实验结果并进行了分析. 在第5节中, 对本文研究工作进行了总结.
2 相关工作 2.1 生成对抗网络生成对抗网络GAN是一种无监督的深度学习方法, 自2014年被Goodfellow等人[4]提出以来, 在图像生成领域取得了突破性的进展. GAN的主要思想是一种二人零和博弈的策略, 包含了两个模型之间的竞争: 生成器G和鉴别器D, 其中生成器G的任务是学习真实样本的数据分布, 将输入的噪声数据z映射到真实的数据分布, 生成尽可能真实的数据G(z)来欺骗鉴别器, 达到让鉴别器分辨不出真假的目的. 而鉴别器D和普通的二分类网络的分类功能类似, 负责判断输入图像真假, 将生成器生成的图像与真实的图像尽可能的区分出来. 其结构如图1所示.

|
图 1 生成对抗网络GAN模型 |
通过最小化生成图像与真实图像之间的误差, 然后反馈到生成器和鉴别器更新优化各自的网络参数, 使得生成器可以生成鉴别器无法鉴别出来的更加真实的数据, 而鉴别器也通过不断更新优化网络参数达到最高的鉴别效果. 二者之间相互对抗不断优化, 提高生成器生成图像的质量以及鉴别器鉴别真假的能力, 经过对抗训练后达到一个动态平衡的状态, 认为此时生成器生成的图像质量最好. 其目标函数如式(1)所示.
| $ \begin{split} \mathop {\min }\limits_G \mathop {\max }\limits_D V(D, G) =& {E_{x{{\sim}}{P_{\rm data}}(x)}}[\log D(x)] \\ & + {E_{{\textit{z}}{{\sim}}{P_{\textit{z}}}({\textit{z}})}}[\log (1 - D(G({\textit{z}})))] \end{split} $ | (1) |
其中,
条件生成对抗网络CGAN是由Mirza等人[17]提出的添加条件的生成对抗网络模型, 此网络是在原始GAN模型的基础上引入约束条件, 用以指导数据生成过程. 这些条件变量可以基于多种信息, 例如类别、标签、轮廓等用于生成图像的部分数据指导, 使得生成图像满足给定的约束条件. CGAN可以看做是把无监督的GAN变成有监督的模型的一种改进, 这个简单直接的改进被证明非常有效, 并在后续的相关研究中得到了非常广泛的应用. 其优化目标如式(2)所示:
| $\begin{split} L_{\rm C G A N}(D, G) = E_{x, y}[\log D(x, y)] + E_{y, {\textit{z}}}[\log (1 - D(y, G({\textit{z}}, y)))] \end{split} $ | (2) |
其中, z表示输入, x表示真实数据, y表示引入的约束条件.
3 方法 3.1 方法架构本文提出的TSA方法分为扩增分割标签、泊松编辑融合图像、生成CT图像3个步骤. 整体框架结构如图2所示.
(1)标签扩增: 使用VAE-GAN[18]来扩增感染区域的分割标签, 增加数据多样性, 并以扩增标签作为分割标签, 可以减少标注过程.
(2)编辑标签: 利用泊松编辑的方法实现不同区域、数量分割标签的插入, 用于扩增分割标签的多样性以及生成CT图像的多样性.
(3) CT图像生成: 使用基于CGAN的图像生成模型, 然后引入ERUNet[19]作为生成器结构, 提高生成图像的质量.
3.2 标签扩增实现肺部CT图像精确分割, 需要足量带标签的数据, 为扩增带标注的CT图像的多样性, 本文提出通过扩增分割标签的多样性来实现生成感染区域多样性的CT图像.
由于标签区域相对较小, 不需要太复杂的网络来生成. 本部分采用VAE-GAN[18]扩增用于肺部CT图像感染区域分割的标签. VAE-GAN是变分自编码器(variational auto-encoder, VAE)和GAN的结合, 其中VAE网络包含编码器和解码器, 编码器对输入的图像

|
图 2 TSA方法架构 |

|
图 3 生成感染区域标签图像 |
首先, 对整张标签图像进行预处理, 由于感染区域比较分散, 在整张标签图像中所占比重较小, 通过阈值处理的方法提取感染区域的部分为独立的标签, 并将尺寸统一为64×64作为数据集; 然后, 使用GAN模型进行训练, 在保证生成图像质量最佳的情况下进行数据的扩增.
为减少实验误差, 对扩增和的感染区域分割标签进行去噪处理, 将其处理为跟原数据标签阈值相同的标签图像. 同时, 扩增的融合标签可以作为配对的分割标签, 这样可以减少对图像标注的过程.
3.3 编辑标签泊松编辑是Pérez等人[20]提出的一种可以用于图像处理的技术方法, 在图像拼接、图像融合[21]等方面得到了广泛应用. 本文主要利用泊松编辑来实现两幅图像的融合生成新的标签图像. 其方法主要思想是根据指定的边界条件利用构造泊松方程求解像素的最优值, 在保留源图像梯度信息的同时, 根据源图像的梯度信息以及目标图像的边界信息, 利用插值的方法重新构建出融合区域内的图像像素, 实现梯度域上的连续, 从而达到边界处的无缝融合, 将源图像与目标图像融合.
其数学基础在于狄利克雷边界条件下的泊松方程. 进行融合的图像在通道上进行计算, 并通过引导插值法来解泊松方程. 泊松编辑方法提出一个引导矢量场, 通过解泊松方程来进行准确的拉普拉斯算子插值. 如图4所示.

|
图 4 引导差值 |
定义
| $ \underset{f}{\mathrm{min}}{{\displaystyle \underset{\Omega }{\iint }\left|\nabla f\right|^{2}}}dxdy\text{, }\quad f{\left|{}_{\partial \Omega }=f^{\prime}\right|}_{\partial \Omega } $ | (3) |
其中,
| $ \underset{f}{\mathrm{min}}{{\displaystyle \underset{\Omega }{\iint }\left|\nabla f-v\right|^{2}}}dxdy\text{, }\quad f{\left|{}_{\partial \Omega }=f^{\prime}\right|}_{\partial \Omega } $ | (4) |
其中,
| $ \nabla f={div}(v),\Big.\quad f\Big|_{\partial \Omega}=\Big.f^{\prime}\Big|_{\partial \Omega} $ | (5) |
其中,
由于泊松编辑可以实现源图和任意目标区域的融合, 本部分进行融合的图像为感染区域的分割标签和肺部的分割标签. 在该过程中, 本文将GAN扩增的感染区域标签和肺部的分割标签进行编辑, 自行选择要融合的区域以及数量, 实现可编辑的标签融合, 扩增生成标签多样性. 为保证最终生成图像的质量, 对融合后的图像进去噪处理, 并提取CT图像的轮廓线作为约束条件, 添加到融合后的图像优化生成标签, 用于后续CT图像的生成. 如图5所示, 提取肺部CT图像的边界轮廓线, 然后将提取的轮廓线和扩增的标签与肺部分割标签进行融合, 最终形成用于生成对应CT图像的生成标签图像. 此方法可以大量增加用于训练的数据量, 扩增数据的多样性.
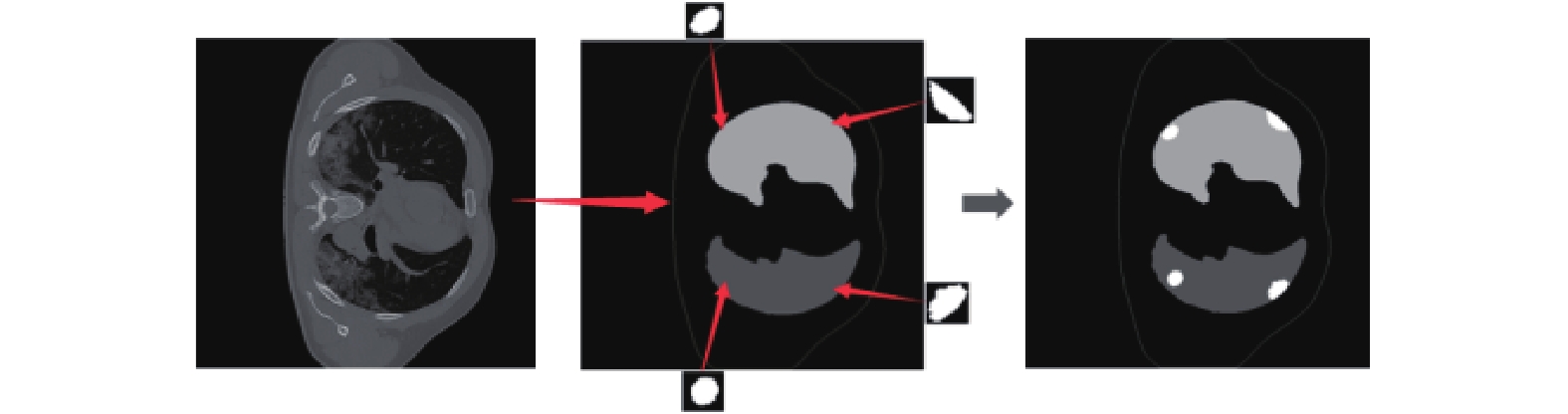
|
图 5 融合标签 |
3.4 CT图像生成
在实现标签图像到肺部CT图像的生成中, 我们使用基于条件生成对抗网络(CGAN)的Pix2Pix模型[22]来实现, 模型结构如图6所示. 不同于CGAN中以噪声z作为输入, Pix2Pix中使用条件y作为源图像输入到生成器G, 生成器G是基于编解码结构的网络, 负责学习输入图像到目标图像之间的映射, 生成既符合目标真实图像x的分布且满足约束条件的图像G(y). 鉴别器D通常为连续的卷积层构成的网络,D的输入为在通道上叠加后的G(y)和y以及x和y, 负责判断输入是否是真实的数据. 为了提高生成质量, 本文引入基于残差网络的ERUNet网络结构作为生成器, 其基本结构如图7所示. 其中, 图7(a)为残差结构[23], 图7(b)为ERUNet网络. 对比传统的UNet结构[24]和基于残差结构的ResUNet结构[25], ERUNet可以减少信息的损失, 保留更多的特征信息, 提高生成图像的质量.
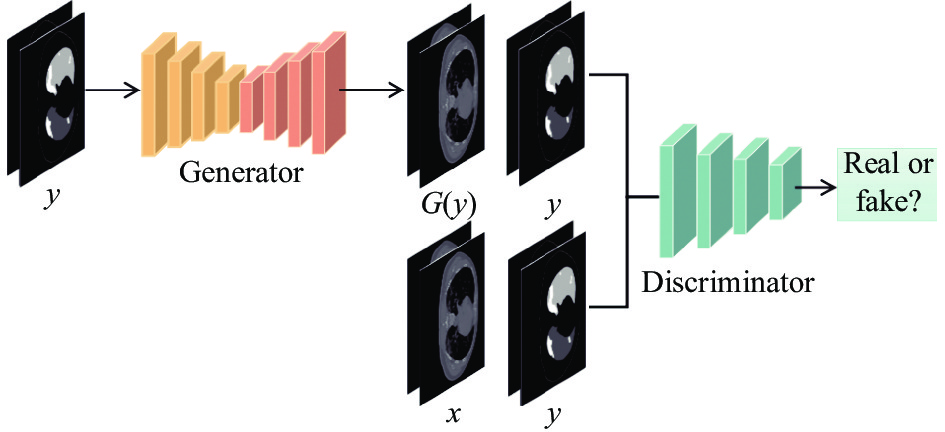
|
图 6 CT图像生成模型 |
实验中采用处理过的数据来训练, 让网络学习从生成标签到CT图像的映射, 实现将生成标签转换为真实的CT图像. 在生成网络模型达到最优的情况下, 将扩增的带有不同病灶区域的标签作为验证输入, 批量生成对应的感染区域多样性的CT图像.
该网络需要优化的目标函数包含两部分, 其一是CGAN的优化目标, 另一部分是L1距离, 如式(6)所示, 用于约束生成图像和真实图像之间的差异减少生成图像的模糊.
| $ {L_{\rm L1}}(G) = {E_{x, y}}\left[ {{{\left\| {x - G(y)} \right\|}_1}} \right] $ | (6) |
其中, y是输入标签, x是真实的数据, G(y)表示输入y后生成的数据. 因此该网络模型需要优化的总目标函数如下:
| $ {G^ * } = \arg \mathop {\min }\limits_G \mathop {\max }\limits_D {L_{\rm CGAN}}(G, D) + {L_{\rm L1}}(G) $ | (7) |

|
图 7 基于残差网络的ERUNet网络结构 |
4 实验和结果 4.1 数据集和预处理
实验中使用Kaggle公开的COVID-19分类分割挑战赛数据集作为研究的数据基础. 该数据集包含20例新冠肺炎患者的CT图和放射科专家对肺部感染区域标注的分割标签, 其中每例患者都有不同切片数量的图像, 如图8所示. 提取其中轮廓较为清楚包含所有带感染区域的切片以及未感染的正常切片作为生成实验的数据集, 共得到1 390张切片图像.
对提取的数据进行处理并划分数据集, 为方便计算将图片尺寸统一缩放至512×512. 实验中的数据集分为两部分: 第一部分为用于扩增感染区域的分割标签数据. 由于感染区域相对整张图像来说比重较小, 使用基于OpenCV的图像截取方法获取分割标签中感染区域的图像, 然后将尺寸统一为64×64, 用于感染区域分割标签的扩增.
图像生成模型训练的数据. 使用阈值处理方法去掉图像中无关的部分, 避免其它因素影响实验, 并提取CT图像的轮廓线添加至对应的分割标签上进行图像的配对保存(见图2). 接着从数据集中筛选出肺部区域较为明显, 感染区域相对容易区分的CT图像用于实验. 最后将CT图像与对应感染区域和肺部分割标签进行配对保存, 作为图像转换模型训练的数据集.
4.2 实验设置和评价指标实验在一台Windows 10系统, 显卡为RTX 3090, 显存为24 GB的服务器上进行, 使用Python 3.8, PyTorch 1.7搭建神经网络模型, 使用Matlab 2018a进行泊松融合; 采用Adam优化算法来优化网络参数, 学习率设置为0.001, 将数据按照8:2划分训练集和测试集, 输入和输出的图像尺寸均为512×512.

|
图 8 数据集样本示例 |
为验证本文提出的生成方法的性能, 采用峰值信噪比(PSNR)和结构相似性指数(SSIM)来对生成的图像进行评价. PSNR表示两张图像的均方误差MSE相对于(2n−1)2的信号最大值的平方, PSNR数值越大表示图像的失真越小, 其公式如式(8)所示:
| $ {\textit{PSNR}}=10 \log _{10}\left(\frac{\left(2^{n}-1\right)^{2}}{{\textit{MSE}}}\right) $ | (8) |
其中,
SSIM是一种衡量两幅图像相似度的指标, 其计算公式如式(9)所示:
| $ {{\textit{SSIM}}}=\frac{\left(2 \mu_{a} \mu_{b}+c_{1}\right)\left(\sigma_{a b}+c_{2}\right)}{\left(\mu_{a}^{2}+\mu_{b}^{2}+c_{1}\right)\left(\sigma_{a}^{2}+\sigma_{b}^{2}+c_{2}\right)} $ | (9) |
其中,
为了证明提出方法的有效性以及验证生成图像的质量, 我们对比使用UNet和ResUNet结构作为生成器与ERUNet结构生成的图像进行对比. 实验进行1 000轮的迭代训练, 经过400个epoch可以看到生成图像的质量已经比较清晰. 图9显示生成模型训练到相同轮次时输入同一张验证图片后3种不同的网络结构的生成效果. 根据直观对比可以明显看出, 本文提出的使用ERUNet生成器结构的网络生成图像质量相对更好, 进一步观察生成图像细节部分, 如图10中标注部分所示, UNet结构生成图像相对模糊且出现较多的噪声干扰, ResUNet结构生成图像背景和感染区域丢失较多的纹理信息, 相比之下ERUNet结构生成的图像具有更高级更加丰富的特征信息, 感染区域纹理更加明显, 因此更接近真实图像. 经过充分训练后保存最佳的生成模型用于后续的CT图像的扩增.
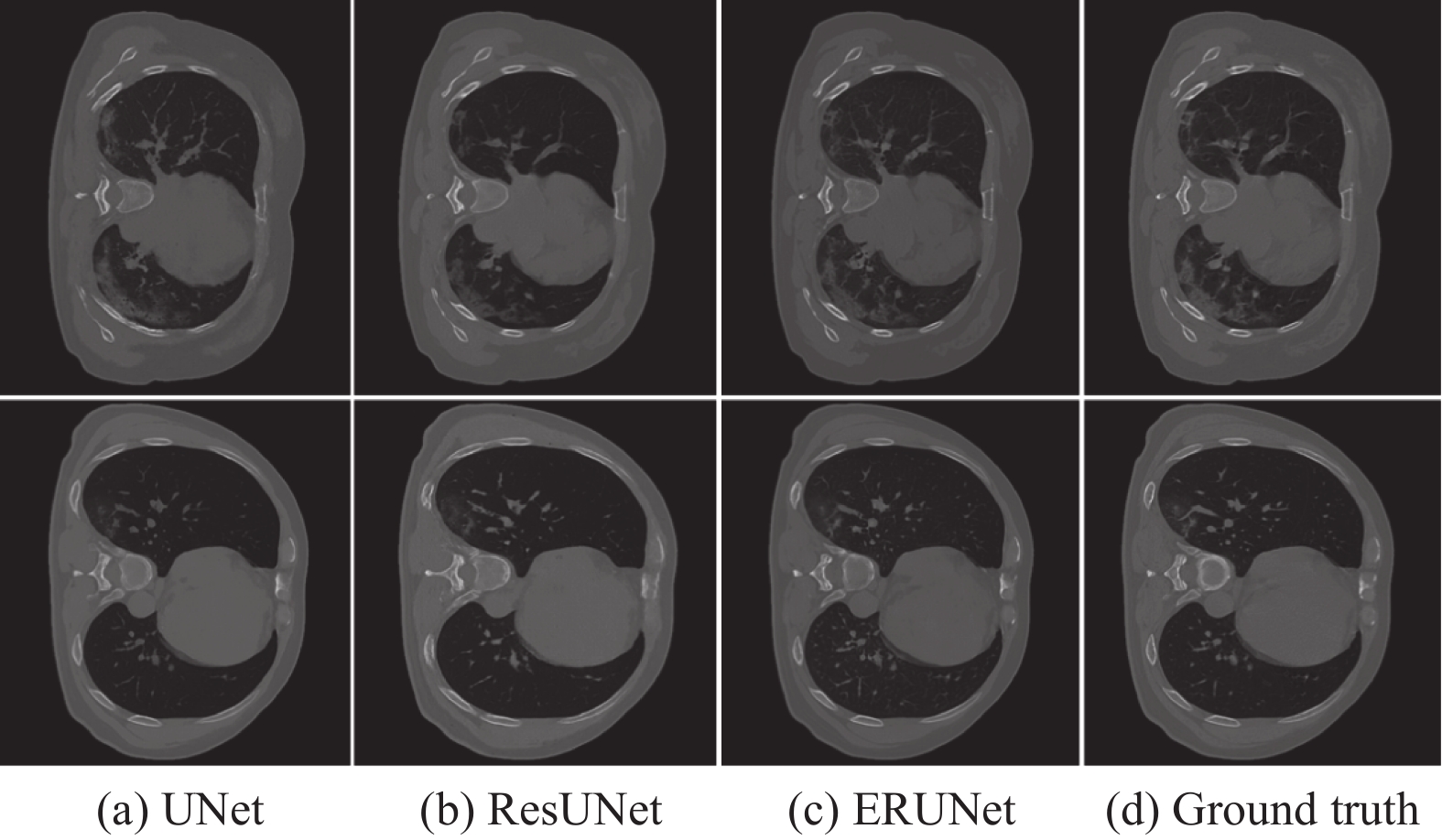
|
图 9 不同生成器结构生成图像对比 |

|
图 10 生成图像细节对比 |
本文对不同的生成器网络各进行3组交叉验证, 表1记录了生成时间以及生成图像的各项评价指标. 从表中可以看出, 本文提出的引入ERUNet网络作为生成器结构, 生成图像的评价指标PSNR和SSIM的值均为最高. 在生成时间上, 表中显示3个不同的网络作为生成器生成300张图像所需要的时间, 其中, UNet网络作为基础结构, 生成图像耗时较少, ERUNet作为改进的结构耗时高于UNet但优于ResUNet, 且生成图像质量最佳. 相比之下, 本文提出方法可以提高生成图像质量, 优化了生成图像的时间.
| 表 1 交叉验证生成图像指标 |
4.4 分割实验
为进一步验证所提方法在生成数据方面的有效性以及验证扩增数据对提高模型检测精度的影响, 本部分引入U2Net网络[26]作为分割模型进行CT图像的分割测试. U2Net网络是在UNet结构的基础上进行改进的结构, 每个模块均为独立的UNet结构, 可以进行多通道的提取特征以保留更多的图像特征信息. 通过对比添加不同数量实验中生成的CT图像进行分割实验来对比分割结果, 以验证本文提出的扩增方法的有效性. 如图11所示, 为本文所提方法扩增的CT图像示例图, 其中红圈标注的部分为生成的CT图像中具有不同位置和形状的感染区域.

|
图 11 扩增的CT图像 |
4.4.1 分割实验设置
将实验划分为两组进行, 第1组为不包含生成数据的原始CT图像的分割实验, 需要提取数据集中CT图像和配对感染区域分割标签并划分数据集, 其中训练集950张, 测试集300张; 第2组为训练集中添加生成图像的对比实验, 其中训练集图像为原始的950张和添加不同数量的生成图像, 测试集是300张图像. 在相同的实验条件下, 两组实验进行100个Epoch迭代训练后测试分割结果. 为了验证分割结果的精确度, 使用Dice来对分割的结果进行评价, 其定义如下:
| $ { Dice }=\frac{2 T P}{2 T P+F P+F N} $ | (10) |
其中, TP表示预测和标签都是正样本的数量,FP表示预测是正样本、但标签是负样本的数量; FN表示预测是负样本但标签是正样本的数量.
4.4.2 分割结果分析经过100个Epoch的训练后使用测试集进行验证, 实验结果如表2所示. 可以看出, 在未添加扩增数据的情况下, 测试集分割的平均Dice值可以达到0.728 7, 对比添加不同数量GAN扩增的训练数据的实验结果, 可以看出扩增训练数据后的分割结果得到显著的提升, 在添加300张扩增图像训练时分割的平均Dice值达到了最高的0.759 1. 在添加GAN扩增的400张和500张CT图像训练时, 提升的分割精度低于添加300张时的情况, 这可能是因为在添加300张生成数据时网络学习到有利于验证集分割的数据分割映射, 例如边缘或浅薄的感染区域, 而添加400和500张生成数据时, 由于数据不平衡问题, 使得网络更倾向于学习例如中心位置或特征明显的感染区域分割映射, 从而降低了其它边缘或浅薄复杂区域数据的分割能力, 这也表明数据的不平衡是分割工作中需要注意的问题.
| 表 2 对比添加生成数据分割结果 |
综上, 本文的实验结果进一步证明了所提生成方法有效性, 利用GAN来生成数据, 可以增加数据的多样性以及进行扩充数据用于训练, 随着不同扩增数据的增加, 对于病灶区域的分割准确度均高于基准未添加扩增数据的分割精度. 图12为测试集中3张CT图像分别在原训练集和添加不同数量生成数据的训练集训练后的模型分割结果对比, 可以看出在添加300张生成数据后分割模型获得了较好的分割性能. 相对于未添加生成数据的分割结果, 本文方法对于提高模型面对未知病灶区域分割的泛化能力和提高模型的分割精度显示出了很大的优势.

|
图 12 分割结果对比 |
5 结论
针对医学图像获取困难和不均衡的现状, 本文提出一种通过三阶段操作实现扩增CT图像的TSA方法, 避免了现有的不同模态图像之间转换方法的局限性, 有效的实现了可编辑的CT图像扩增. 经过对比实验显示, 所提出的生成方法不仅有效, 而且在扩增医学图像多样性方面显示出了较好的效果. 此外, 为进一步验证方法的有效性, 提出利用生成数据进行分割实验测试, 结果表明利用GAN来扩增数据用于训练对于提高模型的检测精度具有重要意义. 本文研究中存在的不足之处是使用的生成器网络生成图像的时间相对高于基准的UNet网络, 因此接下来要研究的方向也是在提高图像质量的前提下, 优化生成器网络尽可能的减少生成图像的时间.
| [1] |
Voulodimos A, Protopapadakis E, Katsamenis I, et al. Deep learning models for COVID-19 infected area segmentation in CT images. Proceedings of the the 14th Pervasive Technologies Related to Assistive Environments Conference. Greece: ACM, 2021. 404–411.
|
| [2] |
郭保苏, 庄集超, 吴凤和, 等. 基于CT图像卷积神经网络处理的新冠肺炎检测. 计量学报, 2021, 42(4): 537-544. DOI:10.3969/j.issn.1000-1158.2021.04.21 |
| [3] |
Saood A, Hatem I. COVID-19 lung CT image segmentation using deep learning methods: U-Net versus SegNet. BMC Medical Imaging, 2021, 21(1): 10-19. DOI:10.1186/s12880-020-00536-6 |
| [4] |
Goodfellow IJ, Pouget-Abadie J, Mirza M, et al. Generative adversarial networkss. Proceedings of the 27th International Conference onAdvances in Neural Information Processing Systems. Montreal: MIT Press, 2014, 3. 2672–2680.
|
| [5] |
Boni K N DB, Klein J, Vanquin L, et al. MR to CT synthesis with multicenter data in the pelvic area using a conditional generative adversarial network. Physics in Medicine & Biology, 2020, 65(7): 075002. |
| [6] |
王蕾, 李媛茜. 基于生成对抗网络的CT图像生成. 成都信息工程大学学报, 2021, 36(3): 286-292. |
| [7] |
Nie D, Trullo R, Lian J, et al. Medical image synthesis with context-aware generative adversarial networks. Proceedings of the 20th International Conference on Medical Image Computing and Computer-assisted Intervention. Quebec City: Springer, Cham, 2017. 417–425.
|
| [8] |
Zhang ZZ, Yang L, Zheng YF. Translating and segmenting multimodal medical volumes with cycle-and shape-consis tency generative adversarial network. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 9242–9251.
|
| [9] |
Emami H, Dong M, Nejad-Davarani SP, et al. Generating synthetic CTs from magnetic resonance images using generative adversarial networks. Medical Physics, 2018, 45(8): 3627-3636. DOI:10.1002/mp.13047 |
| [10] |
Gholamiankhah F, Mostafapour S, Arabi H. Deep learning-based synthetic CT generation from MR images: Comparison of generative adversarial and residual neural networks. arXiv:2103.01609, 2021.
|
| [11] |
Ying XD, Guo H, Ma K, et al. X2CT-GAN: Reconstructing CT from biplanar X-rays with generative adversarial networks. Proceedings of the 2019 IEEE/ CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019. 10611–10620.
|
| [12] |
Yi ZL, Zhang H, Tan P, et al. Dual GAN: Unsupervised dual learning for image-to-image translation. Proceedings of the 2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017. 2868–2876.
|
| [13] |
Zhu JY, Park T, Isola P, et al. Unpaired image-to-image translation using cycle-consistent adversarial networks. Proceedings of the 2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017. 2242–2251.
|
| [14] |
吴香奕, 曹锋, 曹瑞芬, 等. 基于循环一致性生成对抗网络的盆腔伪CT生成方法. 中国医学物理学杂志, 2021, 38(01): 21-29. DOI:10.3969/j.issn.1005-202X.2021.01.005 |
| [15] |
Wolterink JM, Dinkla AM, Savenije MHF, et al. Deep MR to CT synthesis using unpaired data. Proceedings of the Second International Workshop on Simulation and Synthesis in Medical Imaging. Québec City: Springer, Cham, 2017. 14–23.
|
| [16] |
Chartsias A, Joyce T, Dharmakumar R, et al. Adversarial image synthesis for unpaired multi-modal cardiac data. Proceedings of the Second International Workshop on Simulation and Synthesis in Medical Imaging. Québec City: Springer, Cham, 2017. 3–13.
|
| [17] |
Mirza M, Osindero S. Conditional generative adversarial nets. arXiv:1411.1784, 2014.
|
| [18] |
Larsen ABL, Sønderby SK, Larochelle H, et al. Auto-encoding beyond pixels using a learned similarity metric. Proceedings of the 33rd International Conference on International Conference on Machine Learning. New York: JPMLR, 2016. 1558–1566.
|
| [19] |
Kou CX, Li W, Yu ZK, et al. An enhanced residual U-Net for microa neurysms and exudates segmentation in fundus images. IEEE Access, 2020, 8: 185514-185525. DOI:10.1109/ACCESS.2020.3029117 |
| [20] |
Pérez P, Gangnet M, Blake A. Poisson image editing. ACM Transactions on Graphics, SIGGRAPH 2003 Papers. 2003, 22(3): 313–318.
|
| [21] |
Yu ZK, Yan RY, Yu YY, et al. Multiple lesions insertion: Boosting diabetic retinopathy screening through Poisson editing. Biomedical Optics Express, 2021, 12(5): 2773-2789. DOI:10.1364/BOE.420776 |
| [22] |
Isola P, Zhu JY, Zhou TH, et al. Image-to-image translation with conditional adversarial networks. Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017. 5967–5976.
|
| [23] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [24] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich: Springer, Cham, 2015. 234–241.
|
| [25] |
Zhang ZX, Liu QJ, Wang YH. Road extraction by deep residual U-Net. IEEE Geoscience and Remote Sensing Letters, 2018, 15(5): 749-753. DOI:10.1109/LGRS.2018.2802944 |
| [26] |
Qin XB, Zhang ZC, Huang CY, et al. U2-Net: Going deeper with nested U-structure for salient object detection
. Pattern Recognition, 2020, 106: 107404. DOI:10.1016/j.patcog.2020.107404 |
 2022, Vol. 31
2022, Vol. 31


