2. 中国科学院大学, 北京 100049;
3. 山东大学 大数据技术与认知智能实验室, 济南 250100;
4. 中康健康科技有限公司, 广州 510620
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Laboratory of Big Data and Artificial Intelligence Technolgy, Shandong University, Jinan 250100, China;
4. Sinohealth Technology Limited, Guangzhou 510620, China
近年来, 随着国家对医疗健康的重视, 人们对自身健康的关注度也越来越高, 很多企业和研究单位都开始深入智能医疗与健康领域, 其中包括腾讯、阿里、京东、百度以及各AI医疗企业等, 共同推动了智能诊疗、医疗问答、临床辅助决策等技术的发展. 在医学场景中, 已经可以直观地感受到医学文本数量明显的增加, 其中医学文本包括医学文献、临床检测报告、电子病历记录、医疗保险记录等, 这些医学文本中包含了大量的可以挖掘利用的信息, 而这些数据中大多是非结构化或者半结构化, 如何更好地对这些数据进行有效的分析和利用, 是当前的研究热点和难点.
术语标准化能够帮助数据更合理的分析和利用并且提升下游任务的应用效果. 本文主要研究中文医疗文本的术语标准化. 医学术语标准化是将非正式的医学术语如“经皮髂骨成形术”, 映射到正式的医学概念, 如概念“骨盆成形术”, 然后再对应到相应的医学编码上. 这项任务在医学领域非常重要, 在临床上, 关于一种疾病、药品、症状等都有各种不同的写法(包含非正式、非标准的形式还有误写等), 如果都能够归一到对应的术语上来, 它能够推动AI技术在医学应用系统上的落地, 如“CDSS (临床决策诊疗系统)”“DRGs (诊断相关分组管理系统)”等[1]. 并且这项技术在辅助诊疗、公共卫生检测、医疗检索等方面有巨大的作用.
在广大研究者的积极推动下, 关于术语标准化的研究经过了如下的几个阶段: 基于规则和字符词典匹配的方法[2, 3]、基于机器学习的方法[4]、基于深度学习的方法[5, 6]. 早期的基于规则的方法, 由于人工消耗较大且只能在特定的语料上达到满意的效果, 所以在处理比较复杂的数据时往往达不到预期. 后来随着机器学习、深度学习的发展, 人力构建规则的成本消耗得到很大的缓解, 相应术语标准化的准确率也获得了极大的提升. 由于深度学习方法的非线性建模能力更强、能够利用语义信息等优点, 所以在术语标准化的任务上的效果也能达到更好. 随着预训练模型BERT[7]的诞生, 因为它是通过未标注维基百科数据训练得到, 包含了丰富的先验知识和语义信息, 所以在术语标准化任务上利用预训练模型会比传统的深度神经网络如LSTM[8]有更优越的性能.
目前来说, 在使用BERT进行术语标准化任务时一般会采用的方法为直接排序和先召回再排序两种方式. 后一种方式能够相对减少排序时间的开销, 本次研究也是基于先召回再排序的思想.
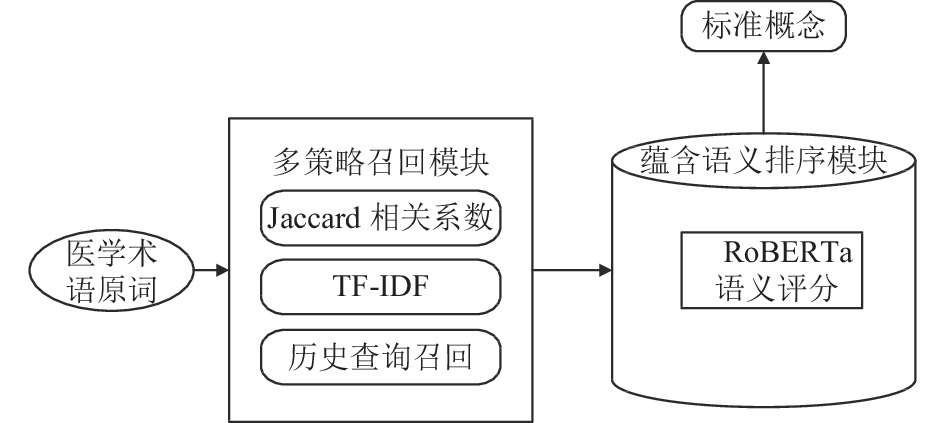
本文在此基础上使用多策略召回排序的思路, 如图1. 在第1阶段尽可能把正确的概念召回, 第2阶段使用蕴含语义评分模型将术语原词与候选概念进行语义相似度排序, 筛选出得分最高的概念. 同时, 之前的中文医疗领域术语标准化研究都是以ICD9或者ICD10 (international classification of diseases, ICD)为标准, 本文首次在SNOMED CT (the systematized nomenclature of human and veterinary medicine clinical terms)标准的数据上进行研究探索, 验证了本方法的有效性及使用SNOMED CT探索术语标准化的可能. 该实验结果证明, 本文提出的方法具有很强的实用性.

|
图 1 整体算法流程图 |
2 相关工作
本文主要的研究是在以中文为核心的医疗领域的术语标准化, 对于医学文本中的不规范的表达也就是术语原词, 经过标准化之后映射到正确的概念编码上来. 每一个标准的概念都有一个概念编码. 有表1不规范的表达, 最后经过术语标准化算法, 最终对应到正确的SNOMED CT编码.
医学术语标准化任务的目标是将医学文本中抽取的非标准的医学表达映射到正确的医学概念编码上, 以便于直接或给下游医学任务利用. 迄今为止, 已经积累了很多关于医疗领域的术语标准化研究工作.
早期的医学术语标准化工作主要是采用规则和机器学习的方法. 文献[9] 引入了一种新的基于编辑距离的方法来进行疾病名称标准化, 在SemEval 2014疾病名称标准化比赛[10]中排名第二. 文献[11] 使用了5种规则的NLP技术提升生物医学文本中的疾病名称标准化水平, 分别提升了MetaMap[12]和Peregrine系统[13]的效果. 文献[14] 提出一种多层筛选系统, 通过定义10种不同优先级的规则来度量术语原词和实体库中概念的相似性. 文献[15]利用线性模型对候选术语与概念名称之间的相似性进行评分, 并且运用了从训练集学习候选实体和概念名称的相似性的策略. 文献[16]在文献[15]的基础上采用了基于低秩矩阵近似的降维技术, 减少了参数量, 同时提升了疾病名称标准化在NCBI疾病语料上的效果.
| 表 1 术语原词-标准概念对应表 |
近些年来, 随着深度学习的蓬勃发展, 来源于神经网络的深度学习技术在术语标准化任务上的表现不断突破, 这种不依赖于规则和人工特征的方案逐渐成为主流. 基于深度学习的术语标准化任务相关研究工作有: 文献[17]使用了不同语料训练的Word2Vec[18]语义向量表示, 并且利用卷积神经网络(CNN)和循环神经网络(RNN)提取特征, 大大超越了以TF-IDF、BM25、向量相似度为基线的术语标准化水平. 文献[19]在文献[18]的基础上增加了医疗健康相关的文本训练获得医疗专业的词嵌入(word embedding), 从而更好地表示医学概念的语义特征, 在数据集上获得新的SOAT (state of the art). 文献[20]提出了一种疾病名称和术式名称结合的多任务医学术语标准化框架, 利用多视角CNN提取特征, 并且对两个任务引入权重共享层, 利用疾病名称和术式名称之间的相关性更好地进行术语标准化. 文献[21]针对术语标准化任务构建了端到端的模型结构, 运用结合attention[22]的双向LSTM和GRU结构[23]提取候选实体特征, 与UMLS系统中的标准词概念特征拼接, 运用Softmax函数进行评分, 证明了比单纯使用CNN结构进行术语标准化有更好的效果. 文献[24]考虑到标注医疗数据需要丰富的专业知识和时间开销, 提出了一种利用共病网络embedding的疾病名称标准化的无监督方法, 接近了经典有监督学习的准确性.
但是以上方法都存在一个问题: 初始的词嵌入并不能表示一词多义, 词向量的特征包含不够丰富, 随着预训练模型的提出, 在术语标准化任务上有了如下的研究.
文献[25]采用基于字符级ELMo向量[26]与传统的Word2Vec词向量拼接共同表示最终的词向量的, 以获得包含更丰富信息的词向量. 并利用BiLSTM提取候选实体的特征, 最终结果超越了以BIGRU-attention进行术语标准化的SOAT. 文献[27]将标准化任务视为一个分类问题, 在3个不同的数据集上进行实验, 对医学概念标准化任务的模型进行细粒度的评估. 通过BERT、ELMO、RNNs模型进行语义表示, 分别对比在术语标准化上的效果, 得出BERT在医学概念标准化上有更好的效果、神经网络的结构会影响术医学概念标准化的准确性等结论. 文献[28]比较BERT/Bio-BERT/ClinicalBERT在生物医学实体标准化任务上的准确性, 结论得出对预训练模型进行微调可以显著提升生物医学实体标准化水平. 文献[29]提出了一种生成和排序的框架解决医学术语标准化问题. 第一阶段使用Lucene工具生成候选对象, 之后使用BERT进行候选实体打分.
基于规则的方法需要根据不同的场景设定不同的规则, 费时费力同时可移植性不强. 基于机器学习的方法虽然在一定程度上缓解了人工消耗, 但由于缺乏语义信息的局限性且不能考虑上下文信息, 它不能在更为复杂的医学术语标准化任务上表现得很好. 深度学习在文本建模上具有强大的表征能力, 不仅可以更好地表示词语和文本, 还可以学习到词语的上下文关系和重要词语的信息[30]. 随着预训练语言模型的诞生, 因为其在上下文中可以获得更为丰富的语义特征, 且使用基于预训练语言模型的方法在很多自然语言处理任务上都达到了最好的水平, 所以现在利用预训练模型模块实现医学术语标准化任务也成为了主流.
本文的研究也是基于预训练模型提高医学术语标准化任务准确率. 由于在医学术语标准化第一阶段的召回过程中单一的方法往往不能够覆盖大部分正确概念, 为此本文提出了多策略召回的方案, 极大提升了第1阶段正确概念的召回率. 结合第2阶段使用RoBERTa-WWM-ext[31]进行蕴含语义排序, 术语标准化最终的准确性得到有效提高.
3 模型介绍 3.1 问题定义在基于SNOMED CT标注的术语标准化数据集中, 设标准概念数量为
术语原词为
本文提出的模型, 总体分为两部分, 下面会对这两部分分别介绍.
第1部分是多策略召回阶段, 多策略召回分为3个小模块. 通过计算术语原词与术语库中所有概念的Jaccard相关系数, 取Jaccard相关系数最高的作为候选实体的一部分. 同时也在所有的概念经过分词之后训练一个TF-IDF模型, 这样就能获得所有的分词权重, 之后把术语原词作为一个query, 计算query与所有概念的相关性, 取相关性最高的概念作为候选实体. 同时结合历史召回方法对候选实体进行召回.
第2部分是蕴含语义排序模块, 使用了RoBERTa-wwm-ext模型, 计算术语原词与候选实体的语义相似性蕴含分数, 再进行蕴含分数排序, 选取得分最高概念, 图2展示了整体的模型架构图.
|
图 2 整体模型框架图 |
3.2.1 候选概念多策略召回模块
这个召回模块主要由3个小部分组成: 历史召回、TF-IDF相关性召回、Jaccard相关系数召回模块, 下面会介绍各小部分的召回原理, 图3展示了多策略召回的具体流程.

|
图 3 候选概念多策略召回 |
TF-IDF召回原理: TF-IDF是一种高效的计算特征权重的算法, 其可以用来解决短文本的相似度的问题. 本文将它用来作为术语原词和概念库中的概念相关性比较的算法, 通过此算法将术语原词匹配到部分最相关的概念.
首先要计算概念库中特征词的权重, 将概念库中的每个概念进行分词, 对于概念库中概念中的特征词其特征权重的计算公式如式(1):
| TF-IDF(Ci,wj)=TF(Ci,wj)×IDF(Ci,wj)=N(wj)n×logmM(wj)+1 | (1) |
其中,
接下来计算术语原词与概念库中每个概念的相关性得分, 对术语原词
| score(s,Ci)=score([v],[w])=∑TF-IDF(Ci,wj), wj∈[v],wj∈[w] | (2) |
Jaccard相关系数召回原理: Jaccard相关系数主要计算符号度量的个体之间的相似程度. 对于术语原词
| J(A,B)=|A∩B||A∪B|=|A∩B||A|+|B|−|A∩B| | (3) |
历史召回原理: 历史召回的算法也是采用的TF-IDF, 它是对训练数据中的术语原词进行召回而不是直接对概念库中的标准词进行召回, 其优势在于能更加充分的利用训练集中的信息. 它直接对训练集中所有的术语原词进行计算获得特征分词的权重, 之后计算待标准化术语与训练集中术语原词的相关性, 最后取相关性最高的top-k个术语原词对应的标准概念作为候选概念集. 图4展示了历史召回的算法原理.

|
图 4 历史召回算法流程图 |
训练集中数据是以“术语原词-概念”的形式出现, 所以找到最匹配的术语原词, 也就间接地找到了需要的概念, 这种方法能够在召回阶段更充分利用数据.
3.2.2 蕴含语义排序模块候选实体排序阶段, 要对第1阶段召回的所有概念进行打分排序, 这样才能选择与术语原词最对应的概念作为答案. 候选实体排序模块由RoBERTa-wwm-ext蕴含语义相似性评分模块构成.
RoBERTa-wwm-ext作为蕴含分数计算模型, 主要是通过训练一个二分类模型, 之后预测原词和术语库中的概念之间的蕴含分数(其中把预测为1的概率作为蕴含分数), 对术语库中的每个概念进行评分. RoBERTa-wwm-ext是在BERT的基础上进行的改进, 以下主要介绍二者的区别.
BERT采用预训练-微调的模式, 问世以来在解决实体识别、文本分类、自然语言推断等多个自然语言处理获得了SOTA, 给学术界提供了很多的参考.
BERT的全称是(bidirectional encoder representa-tion from Transformers), 本文中作为语义排序的基础模型. 在BERT之前预训练语言模型有ELMo (embedding from language model)和GPT (generative pre-training), 但是这两种方法都只采用了一个预训练目标, 而且没有充分的利用上下文信息. BERT采用Transformer的encoder作为基本组成, 能够充分结合上下文本信息进行有效的训练. 与早期提出的训练语言模型的目标“预测下一个词”不同的地方在于BERT在单词级别和句子级别设置了两个目标: 掩码语言模型(masked language model, MLM)与预测下一句(next sentence predict, NSP)模型. 其中MLM可以理解为完型填空做法的思路, 模型随机会mask每个句子中15%的词, 利用上下文信息来预测这些词. MLM具体做法是80%的词用[mask]替换原来的词, 10%的词随机取一个词替代mask的词, 10%词保持不变. 预测下一句训练过程的具体做法是选取一些句对与, 其中50%的数据是的下一句, 剩余50%是从语料库中随机选择, 通过对句对进行二分类训练来学习句子间的关系. 通过这两个目标训练出的BERT模型, 具有很强的字词级别的表征能力.
RoBERTa-wwm-ext与BERT模型的基本结构基本相同, 改进更多的是从训练集和训练策略角度来提升, 主要有以下几点: 首先相对于BERT的静态掩码机制采取了动态掩码机制, 在BERT 中训练数据时, 一条样本只进行一次随机 mask, 在训练时 mask 的位置都保持不变, 动态mask在每次训练前会动态生成一次mask, 这种方法提高了模型输入的随机性, 使模型可以学习更多的句式. 另外它使用了更大的batch size进行训练, 被实验验证有更好的效果. 同时采用字节对编码(BPE)进行文本数据处理, 使用了更多的数据同时进行训练, 且取消了NSP任务, 提升了效率. 且采用了WWM(全词掩码) 策略, 相较于BERT的单字掩码, 先进行分词, 如果有词中的部分字符被mask, 那么整个词都将会被mask, 这样做RoBERTa-wwm-ext能够更好地学习词级别的信息[31-33].
本文使用RoBERTa-wwm-ext模型将语义间相关性判别转化为一个二分类模型, 图5展示了RoBERTa-wwm-ext模型结构作为语义蕴含评分模块.

|
图 5 RoBERTa-wwm-ext语义蕴含评分模型结构 |
分别按照相应的策略构造<术语原词, 概念, 1>和<术语原词, 非正确概念, 0>的正负样本, 用此数据来训练蕴含语义评分模型. 将样本输入到模型中具体格式为“[CLS]手足抽搦[SEP]手足抽搐症[SEP]”, 经过RoBERTa-wwm-ext编码获得[CLS]的隐藏层状态表示, 接着输入到全连接层, 经过Softmax函数打分, 其中[CLS]和[SEP]分别表示用于分类的令牌、分隔术语原词和候选概念的令牌.
蕴含语义评分模型使用Softmax作为分类回归函数, 模型采用交叉熵损失函数进行优化, 使用类别为1的概率作为语义蕴含分数. 其中蕴含分数及最后预测对应概念的计算过程如式(4)–式(5):
| score(s,gi)=Softmax l=1(FFNN(RoBERTa-wwm-ext(E1,⋯,EN))) | (4) |
| y=argmax(score(s,G)) | (5) |
其中, FFNN表示前馈神经网络层,
本文需要比较的是术语原词和概念库当中概念的语义相似性, 虽然使用的是二分类的方法, 但是需要二分类模型评出概念相似程度的高低, 涉及到排序, 所以其精度要求更高, 难度上比传统二分类任务的0.5作为阈值更难. 所以对构造二分类模型的训练样本一定要经过特殊处理, 这样模型才能够更好地学习到语义的相关性.
方法1. 构造语义模型数据集方法
1)构建空的数据列表datas=[], 将其作为语义模型训练需要的数据;
2)设置困难负样本数量k, 随机负样本数量m, 正样本数量n;
3)训练集术语原词使用Jaccard相关系数召回概念库中得分最高的前k个负样本, 将k个样本处理为<org, neg, 0>并入datas;
4)训练集术语原词从概念库中随机抽取m个负样本, 将m个样本处理为<org, neg, 0>并入datas;
5)训练集术语原词重复n条作为正样本, 将n个样本处理为<org, neg, 1>并入datas;
6)对datas进行随机打乱;
4 实验过程与结果评估 4.1 实验数据SNOMED CT是目前国际上认可的且比较全面的医学术语集, 其内容包括了临床所需的基本信息. SNOMED CT 的概念表收录了大量具有唯一含义并经过逻辑定义的概念, 分类编入18个顶级概念轴(hierarchy)中, 分别包括临床发现、操作/介入、身体结构等[34].
本次实验的数据是以SNOMED CT为标准, 医疗相关人员进行标注, 获得“术语原词-概念”标注数据9 000余条, 此次标注中选取SNOMED术语库中在医学文本中常见的概念15 001个SNOMED CT概念作为概念标准, 这些数据均来自现实的医学场景. 按照比例6:2:2划分为训练集、开发集、测试集, 数据集中的真实数据如表2.
| 表 2 数据集中的数据形式 |
数据集中最长的术语原词为“库兴氏综合征(由于各种原因引起的肾上腺糖皮质激素慢性分泌过多, 表现为肥胖伴有高血压等一系列症状)”, 最短的术语原词为“腿”.
4.2 评价指标 4.2.1 总项评价指标对于分类任务, 总体的评价指标有如下几个标准: 准确率、精确率、召回率、F1值, 其计算方式如式(6)–式(9):
| accuracy=TSN | (6) |
| precision=TPTP+FP | (7) |
| recall=TPTP+FN | (8) |
| F1=2×precision×recallprecision+recall | (9) |
式(6)中,
本文是术语标准化任务作为候选概念召回和候选概念排序两阶段任务来实现的, 所以在这两个分项中也有不同的指标来衡量他们的效果. 候选概念召回阶段使用召回率(
| recall=n∑i=1(Gi∩Ti)N | (10) |
其中,
本文中使用了RoBERTa-wwm-ext作为蕴含语义评分模型, 使用了Adam作为优化器, 实验采用内存大小为11 GB, 一张2080ti GPU显卡. Dropout rate 设置为0.1, 学习率为2E–5, batch_size为64, 隐藏层维度为768, 最大的句子长度为64.
4.3.2 结果分析在第一阶段召回主要做实验对比单召回方式, 以及本文中多策略召回的效果, 表3展示了不同方法的候选概念召回率.
| 表 3 候选概念召回率 |
表3的结果来看, 本文提出的召回策略具有明显优势. 单一方法的召回都不能够完全覆盖正确的概念. 结合多策略召回, 其中每个召回方式都取前10最高得分能够达到0.983的召回率, 基本能够覆盖正确答案, 所以将它作为蕴含语义排序模型的输入部分. 在候选概念蕴含语义排序阶段, 为了获得更强的语义排序模型, 本文采用了不同的策略构建负样本. 在训练语料中引入了困难负样本, 再结合不同的训练样本比例, 得到当前效果最佳的蕴含语义模型训练方案. 不同构造样本的策略效果如表4.
| 表 4 样本构建对结果影响 |
表4中, P代表训练集中的正样本重复次数, Nrandom数代表随机负样本数量,Nhard表代表困难负样本数量. 为了维持训练集正负样本的比例, 所以始终将训练正负样本维持在1:1与1:2. 从蕴含语义排序的结果来看, 在一定范围内引入困难负样本会显著的提升蕴含语义排序模型的效果并且增加正、负样本的数量也可以提升模型的效果. 为了比较不同的语义模型蕴含排序效果的差别, 本文分别在同结构的网络模型进行了对比实验. 在召回策略相同、正负样本构造分别是(P, Nrandom, Nhard)=(20, 20, 20)的条件下, 各语义蕴含模型的效果如表5.
| 表 5 部分同类型模型蕴含语义排序效果对比 |
从结果来看, 在使用相同的召回策略且蕴含语义模型的正负样本构建策略均相同的情况下, 使用RoBERTa-wwm-ext作为蕴含语义排序模型展现了它有更强的蕴含语义表征能力, 并且效果比同类其他模型的效果更好. 这是由于其模型的训练方式和丰富的训练语料带来的优势, 实验结果也展现了本方法在以SNOMED CT为标准的医学术语标准化上的可行性及优越性.
5 结论与展望本文在解决医学术语标准化的问题上, 提出了一种结合RoBERTa与多策略召回的方法, 该模型使用RoBERTa-wwm-ext作为蕴含语义排序模型. 首次在医疗标准SNOMED CT标注的数据上进行实验验证, 证明了本方法的有效性, 为其他从事SNOMED CT标准进行的术语标准化工作者提供了参考. 本文将医学术语标准化分为两个阶段来执行, 第1阶段是多策略召回, 第2阶段是蕴含语义排序. 在多策略召回阶段, 由于医学术语的表达多样化与口语化的特点, 往往通过一种召回方法召回候选概念效果欠佳, 而本文提出的多策略的召回方法可以召回98.3%的正确候选概念. 在蕴含语义排序阶段, 为了构建强大语义模型, 本文引入了困难负样本进行训练, 并且构造不同数量的正负样本比例确定蕴含语义模型的训练方式, 最终蕴含语义排序模型的效果得到极大提升. 通过对比本文模型和其他同类型模型的基于SNOMED CT医学术语标准化效果, 本文提出的模型有更高的准确性, 准确率达87.8%.
由于医疗领域的术语一字之差可能完全表达的是两个不同的概念、术语原词与概念之间没有交集等情况. 在以后的工作中希望能够引入外部信息, 或者根据医疗数据特点引入特征词典来提高医学术语标准化的水平, 同时也希望对一个术语对应多个概念或者多个术语对应多个概念的方向去展开研究.
| [1] |
陈漠沙, 仇伟, 谭传奇. 基于BERT的手术名称标准化重排序算法. 中文信息学报, 2021, 35(3): 88-93. DOI:10.3969/j.issn.1003-0077.2021.03.008 |
| [2] |
Tsuruoka Y, McNaught J, Tsujii J, et al. Learning string similarity measures for gene/protein name dictionary look-up using logistic regression. Bioinformatics, 2007, 23(20): 2768-2774. DOI:10.1093/bioinformatics/btm393 |
| [3] |
Lee HC, Hsu YY, Kao HY. AuDis: An automatic CRF-enhanced disease normalization in biomedical text. Database, 2016, 2016: baw091. DOI:10.1093/database/baw091 |
| [4] |
Xu J, Lee HJ, Ji ZC, et al. UTH_CCB system for adverse drug reaction extraction from drug labels at TAC-ADR 2017. Proceedings of TAC 2017. Gaithersburg: TAC, 2017. 1–6.
|
| [5] |
Li HD, Chen QC, Tang BZ, et al. CNN-based ranking for biomedical entity normalization. BMC Bioinformatics, 2017, 18(11): 385.
|
| [6] |
Luo YF, Sun WY, Rumshisky A. A hybrid normalization method for medical concepts in clinical narrative using semantic matching. AMIA Joint Summits on Translational Science Proceedings, 2019, 2019: 732-740. |
| [7] |
Devlin J, Chang MW, Lee K, et al. BERT: Pre-training of deep bidirectional transformers for language understanding. Proceedings of 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. Minneapolis: Association for Computational Linguistics, 2018. 4171–4186.
|
| [8] |
Sundermeyer M, Schlüter R, Ney H. LSTM neural networks for language modeling. Proceedings of the 13th Annual Conference of the International Speech Communication Association. Portland: ISCA, 2012. 194–197.
|
| [9] |
Ghiasvand O, Kate RJ. UWM: Disorder mention extraction from clinical text using CRFs and normalization using learned edit distance patterns. Proceedings of the 8th International Workshop on Semantic Evaluation. Dublin: Association for Computational Linguistics, 2014. 828–832.
|
| [10] |
Pradhan S, Elhadad N, Chapman WW, et al. SemEval-2014 task 7: Analysis of clinical text. Proceedings of the 8th International Workshop on Semantic Evaluation. Dublin: Association for Computational Linguistics, 2014. 54–62.
|
| [11] |
Kang N, Singh B, Afzal Z, et al. Using rule-based natural language processing to improve disease normalization in biomedical text. Journal of the American Medical Informatics Association, 2013, 20(5): 876-881. DOI:10.1136/amiajnl-2012-001173 |
| [12] |
Aronson AR. Effective mapping of biomedical text to the UMLS metathesaurus: the MetaMap program. Proceedings of AMIA 2001, American Medical Informatics Association Annual Symposium. Washington: AMIA, 2001. 17–21.
|
| [13] |
Schuemie MJ, Jelier R, Kors JA. Peregrine: Lightweight gene name normalization by dictionary lookup. Proceedings of the BioCreative 2 Workshop. Madrid, 2007, 131-140. |
| [14] |
D’Souza J, Ng V. Sieve-based entity linking for the biomedi-cal domain. Proceedings of the 53rd Annual Meeting of the Association for Computational Linguistics and the 7th International Joint Conference on Natural Language Processing. Beijing: Association for Computational Linguistics, 2015. 297–302.
|
| [15] |
Leaman R, Doǧan RI, Lu ZY. DNorm: Disease name normalization with pairwise learning to rank. Bioinformatics, 2013, 29(22): 2909-2917. DOI:10.1093/bioinformatics/btt474 |
| [16] |
Leaman R, Lu ZY. Automated disease normalization with low rank approximations. Proceedings of BioNLP 2014. Baltimore: Association for Computational Linguistics, 2014. 24–28.
|
| [17] |
Limsopatham N, Collier N. Normalising medical concepts in social media texts by learning semantic representation. Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Berlin: Association for Computational Linguistics, 2016. 1014–1023.
|
| [18] |
Mikolov T, Chen K, Corrado G, et al. Efficient estimation of word representations in vector space. arXiv: 1301.3781, 2013.
|
| [19] |
Lee K, Hasan SA, Farri O, et al. Medical concept normalization for online user-generated texts. Proceedings of 2017 IEEE International Conference on Healthcare Informatics (ICHI). Park City: IEEE, 2017. 462–469.
|
| [20] |
Luo Y, Song GJ, Li PY, et al. Multi-task medical concept normalization using multi-view convolutional neural network. Proceedings of the 32nd AAAI Conference on Artificial Intelligence. New Orleans: AAAI, 2018. 5868–5875.
|
| [21] |
Tutubalina E, Miftahutdinov Z, Nikolenko S, et al. Medical concept normalization in social media posts with recurrent neural networks. Journal of Biomedical Informatics, 2018, 84: 93-102. DOI:10.1016/j.jbi.2018.06.006 |
| [22] |
Bahdanau D, Cho K, Bengio Y. Neural machine translation by jointly learning to align and translate. arXiv: 1409.0473, 2014.
|
| [23] |
Chung J, Gulcehre C, Cho KH, et al. Empirical evaluation of gated recurrent neural networks on sequence modeling. arXiv: 1412.3555, 2014.
|
| [24] |
Zhang YZ, Ma XJ, Song GJ. Chinese medical concept normalization by using text and comorbidity network embedding. Proceedings of 2018 IEEE International Conference on Data Mining (ICDM). Singapore: IEEE, 2018. 777–786.
|
| [25] |
Subramanyam KK, Sangeetha S. Deep contextualized medical concept normalization in social media text. Procedia Computer Science, 2020, 171: 1353-1362. DOI:10.1016/j.procs.2020.04.145 |
| [26] |
Peters ME, Neumann M, Iyyer M, et al. Deep contextualized word representations. Proceedings of 2018 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. New Orleans: Association for Computational Linguistics, 2018. 2227–2237.
|
| [27] |
Miftahutdinov Z, Tutubalina E. Deep neural models for medical concept normalization in user-generated texts. Proceedings of the 57th Annual Meeting of the Association for Computational Linguistics: Student Research Workshop. Florence: Association for Computational Linguistics, 2019. 393–399.
|
| [28] |
Ji ZC, Wei Q, Xu H. BERT-based ranking for biomedical entity normalization. AMIA Joint Summits on Translational Science Proceedings, 2020, 2020. 269–277.
|
| [29] |
Xu DF, Zhang ZY, Bethard S. A generate-and-rank framework with semantic type regularization for biomedical concept normalization. Proceedings of the 58th Annual Meeting of the Association for Computational Linguistics. Association for Computational Linguistics. Online: ACL. 2020. 8452–8464.
|
| [30] |
孙曰君, 刘智强, 杨志豪, 等. 基于BERT的临床术语标准化. 中文信息学报, 2021, 35(4): 75-82. DOI:10.3969/j.issn.1003-0077.2021.04.011 |
| [31] |
Cui YM, Che WX, Liu T, et al. Pre-training with whole word masking for Chinese BERT. IEEE/ACM Transactions on Audio, Speech, and Language Processing, 2021, 29: 3504-3514. DOI:10.1109/TASLP.2021.3124365 |
| [32] |
Liu Y, Ott M, Goyal N, et al. RoBERTa: A robustly optimized BERT pretraining approach. arXiv: 1907.11692, 2019.
|
| [33] |
王曙燕, 原柯. 基于RoBERTa-WWM的大学生论坛情感分析方法. 计算机工程, 2021, 1-9. (2021-10-18)[2022-01-13]. DOI:10.19678/j.issn.1000-3428.0062008 |
| [34] |
郭玉峰, 刘保延, 崔蒙, 等. SNOMED CT内容简介. 中国中医药信息杂志, 2006, 13(7): 100-102. DOI:10.3969/j.issn.1005-5304.2006.07.058 |
 2022, Vol. 31
2022, Vol. 31



