骨折是日常生活中常见的一种骨科疾病, 如果不及时采取治疗或治疗方式欠妥会影响患者术后康复, 困扰患者的健康生活. 对于普通骨折, 有经验的医生可以通过计算机断层扫描来定位骨骼、判断骨折类型与受损程度. 当骨折程度较为严重或执行大型手术时, 就需要对骨骼建模生成三维模型辅助医师开展术前规划, 因此对骨骼的精确分割意义重大.
骨骼医学图像的分割算法发展至今, 大多以传统图像处理方法为主. 由于人体组织对X射线的吸收程度不同, 显现在CT图像上的灰度值就不同. 骨骼做为高吸收区, 成像多为白影, 因此国内外提出了许多使用阈值分割的算法. 如李银波等[1]提出的一种自适应阈值方法对骨骼切片的分割, 结合区域生长和Chebyshev滤波逼近的方法, 对阈值进行改善. Kang等[2]提出一种迭代阈值法对骨骼进行分割, 首先对原始CT做形态学操作, 然后不断迭代阈值点, 优化分割结果. Bakthula等[3]提出一种结合图像增强与聚类的骨骼分割方法, 首先对待聚类的图像做优化, 提高聚类准确性. 以上算法均存在过分依赖于数据的好坏, 以及分割精度较低的问题.
随着卷积神经网络的发展, 其强大的特征提取能力使得深度学习技术成功在医学图像领域大放光彩. 与传统的分割算法相比, 神经网络算法拥有大量可学习神经元参数与非线性的表达, 可以很好地去除CT数据中的噪声, 提取骨骼特征. Shelhamer等[4]提出一种全卷积网络(FCN), 开启了语义分割的热潮. FCN使用卷积层代替全连接层, 并通过转置卷积将特征图的大小变换回输入图像的尺寸, 同时将浅层网络的特征信息与深层网络中的特征信息相加来提高分割精度, 从而实现对任意尺寸图像进行像素级别的分割. Ronneberger等[5]对 FCN模型进行改进, 提出了U-Net模型. U-Net模型采用对称的编码与解码格式, 将下采样与上采样的特征相互融合来弥补丢失的信息, 充分地利用了空间信息与语义信息, 使得分割结果更加精细, 其在医学图像分割领域中表现优异. He等[6]为解决梯度消失等问题, 提出了特征直接映射的残差结构, 为更深的网络层数提供了支持. Hu等[7]提出了Se-Net模型, 该模型专注于特征图的通道维度, 通过对特征图挤压与扩张获取特征与特征之间的关系, 可以让网络更加专注于信息量大的通道, 实现了通道注意力. Oktay等[8]将U-Net与注意力机制相结合提出Attention U-Net, 通过注意门模块将网络专注于目标物体区域, 实现精细分割.
神经网络与骨骼分割结合的算法也有很多. Vania等[9]通过使用数据处理和简单的卷积神经网络结合实现对脊柱骨骼的自动分割. Klein等[10]通过对U-Net改进实现对全身骨骼的分割. Noguchi等[11]通过使用新型的数据增强方式结合卷积神经网络实现对人体骨骼的分割. 以上工作都较传统算法有了大幅度提升, 但大多依赖于数据的增强来提升准确度, 对网络模型的改进不足, 骨骼的分割精度远远不够. 因此为了解决以上问题本文提出一种新的分割框架. 本文主要贡献包括: 1)针对下肢CT数据提出一种新的骨骼分割方法. 2)基于空洞卷积思想, 结合多尺度金字塔模块进一步增强对骨骼信息的提取. 3)基于空间与通道注意力机制, 改进高低维度特征融合不充分的问题, 提升了模型的分割精度.
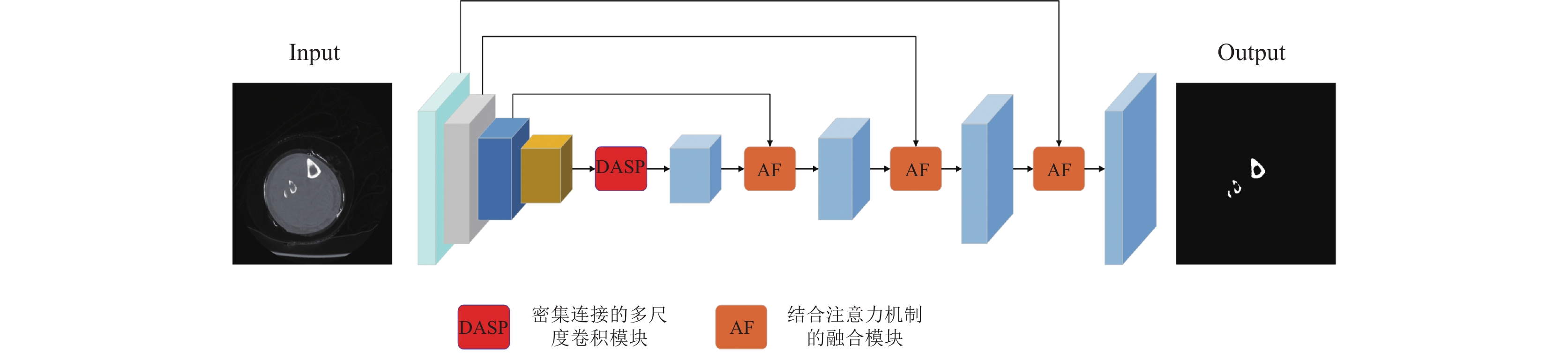
1 分割算法人体下肢骨骼CT数据的形状不同, 大小不一, 尤其在骨骼两端因为其骨密度较小, 导致出现于其他组织区分度不大的情况, 传统的神经网络难以对其精确分割. 本文提出的AFU-Net是将医学领域中表现优异的U-Net作为基础网络, 针对骨骼数据特点与U-Net自身缺点出发进行改进. AFU-Net的整体模型如图1所示.
|
图 1 AFU-Net网络框架图 |
网络编码阶段又称为下采样阶段使用卷积、批归一化、ReLU激活函数以及尺寸为2×2的最大池化构成的骨干模块. 每次下采样后通道数会增加一倍, 尺寸会缩小为原来的1/4. 因训练过程中对样本数据进行裁剪, 所以将U-Net的4次下采样缩减为3次. 图像分割的整体流程: 首先使用编码器提取骨骼图像语义特征; 其次通过密集连接的多尺度卷积模块获取更丰富的特征信息; 最后将解码器的输出与编码器的输出通过结合注意力机制的融合模块进行融合来弥补下采样丢失的信息, 多次上采样直到恢复为原图分辨率为止.
1.1 密集连接的多尺度卷积模块相对于U-Net, 本网络下采样次数较少, 导致卷积操作的感受野较小, 获取信息具有局限性, 对于深层的语义信息提取效果较差. 为了弥补感受野小的缺陷, 提出了使用密集连接的多尺度卷积模块(DASP)模块来增强对不同尺寸的信息提取能力, 有效地扩大了感受野的同时还避免了多次下采样带来的信息丢失. DASP模块思想来源于密集卷积模块[12]和ASPP模块[13], 并对其原有模块进行化简与融合, 提高对骨骼特征的提取能力. DASP模块如图2所示.

|
图 2 密集连接的多尺度卷积模块 |
空洞卷积操作是在卷积核中插入空洞, 使得在参数量不变的情况下, 网络可以获得更大的感受野. 图2中d表示空洞率即卷积核中插入空洞的个数, 当空洞率为1时空洞卷积与常规卷积操作相同. ASPP模块通过平行引入不同空洞率的空洞卷积来实现多尺度的信息提取, 有效地提升了分割精度. 但是ASPP也存在相应的问题: 空洞卷积因为空洞的存在, 采样点较为分散导致大量信息被抛弃; 当空洞率较大时, 小目标物体的分割就会受到影响. 因此本文将不同空洞率的路径的输入输出以通道维度密集连接起来, 有效地避免了信息丢失的同时带来了较大的感受野. 密集连接后的模型可以同时实现串联和并联的卷积操作, 使得网络可以自适应的获取骨骼信息. 最后使用普通卷积操作将合并后的特征图通道降维至与输入通道相同. DASP可以表示为:
| Xi=Convdi3×3([Xi−1,Xi−2,⋯,X1]) | (1) |
| Y=Conv3×3([X3,X2,X1,X]) | (2) |
其中, X代表输入, Xi代表中间操作的输出, Y代表最终输出, di代表空洞率, Conv代表空洞卷积操作, []代表通道维度连接. 可以看到每一层的输入都是前面所有中间操作的输出通道连接, 最后通过卷积操作降低维度作为输出. 空洞率的选取也决定着信息提取的好坏, 较差的空洞率组合会带来网格效应. 根据混合空洞卷积理论[14]和本实验对比结果, 我们选取大小为3, 5, 7的空洞率组合.
1.2 结合注意力机制的融合模块U-Net网络针对下采样信息丢失的问题采用跳层连接来解决, 将浅层特征与深层特征拼接融合后, 作为上采样的特征图. 其优点在于操作简单, 可以弥补大量丢失信息, 但其仍有不足: 一方面使得解码器无法很好地利用浅层空间信息, 另一方面引入的空间信息也会破坏原有的语义信息. 因此合理的利用不同维度的信息, 对提高分割精度有重要意义. 为了解决这个问题, 本文提出了结合注意力机制的融合模块(AF).
AF模块结合了常见的模块融合方法与相应的注意力机制, 采用不同的方式实现跳层连接, 因此AF模块可以看作由通道注意力分支与空间注意力分支成, 可以更加充分地将高低维度特征融合在一起. AF模块如图3所示, 融合模块的整体流程如下.
1) 将高维特征经过双线性插值放大尺寸, 实现与低维度特征具有相同分辨率.
2)将高维度特征H按通道维度随机平分生成H1, H2.
3) 将低维度特征经过卷积核为1×1的卷积层实现跨域操作生成L.
4) H1与L通过通道注意力分支与普通卷积操作后生成Fc.
5) H2
6) Fc与Fs融合后通过CBR模块进行通道降维, 作为模块输出.
通道注意力分支为经典的压缩激励模块[7], 该模块旨在获取特征通道之间的关系. 低维度特征即使通过1×1的卷积实现跨域操作后, 其中仍包含大量的无用信息, 若直接与高维度特征融合, 会存在破坏语义信息的可能性. 因此本文通过通道注意力机制来抑制无用的信息通道. 通道注意力流程为: 首先对特征图每一个通道做全局平均池化来获取当前通道的全局特征, 实现特征压缩; 其次使用两个大小为1×1的卷积操作和ReLU激活函数代替原文中的全连接层并学习通道与通道之间的关系; 最后将学习后的特征经过Sigmoid变换后作为通道的权重, 实现特征激励, 以此来突出重要的骨骼信息. 通道分支可用式(3)和式(4)表示.
| ωC=Conv1×1(Conv1×1(GAP([H1,L]))) | (3) |
| FC=Sigmoid(ωC)×[H1,L] | (4) |

|
图 3 结合注意力机制的融合模块 |
Conv表示卷积核为1×1的卷积操作; GAP表示为全局平均池化; []表示为通道维度的连接. 为了实现减少参数量和模型复杂度, 第1个卷积层的输出通道缩小r倍, 第2个卷积层的输出通道数放大r倍即恢复至原来大小. 实验结果表明, CT数据集分割任务的降维系数r取16时效果最佳.
空间注意力分支旨在获取特征图空间上的关系, 从自身学习通道共享的空间权重参数, 以此来突出骨骼特征[15]. 空间注意力分支通过将不同维度的特征图相加来实现语义信息与空间信息融合, 再通过注意力机制, 将网络的关注重心放在骨骼信息上. 整体流程为: 首先将高低维度特征元素相加; 对相加后的特征做通道上的最大池化与平均池化; 将获取到的两个特征图通道连接后经过一个卷积核大小为7×7的卷积层, 生成通道数为1的特征图; 最后将生成的特征图经过Sigmoid变化后作为高低维度融合后的空间权重, 以此来实现更好地实现特征融合. 空间分支可用式(5)和式(6)表示.
| ωS=Conv([(AvgPool(H2+L),MaxPool(H2+L)]) | (5) |
| FS=Sigmoid(ωS)×(H2+L) | (6) |
最后将空间注意力分支和通道注意力分支的结果做通道连接后, 采用与编码阶段的骨干模块相似的CBR (Conv2d, BN, ReLU)模块实现通道降维. AF模块的输出可以用式(7)表示.
| FOUT=CBR([FS,FC]) | (7) |
本文实验在自建下肢CT数据集与公开数据集Peréz-Carrasco上开展对比实验, 详细介绍了骨骼分割实验的数据处理、实验设置、评价指标与实验结果.
2.1 数据处理实验使用的数据均来自常州市图形图像与骨科植入物数字化技术重点实验室提供的人体下肢CT数据集(简称CT数据集). 对CT数据集沿Axial方向切片后生成共8 000张dcm格式的二维CT图像. 将数据集以8:2比例划分为训练集与验证集. 标签数据为专业医师采用Mimics手工标注得到, 转换后生成只含0、1值的PNG图片. CT数据集图像大小为512×512, 将原始CT图像通过Pydicm库设置为适合查看骨骼数据的窗宽窗位后导出, 图4表示原始CT图像与医师标注的金标准对比图.

|
图 4 CT数据与金标准 |
观察原始数据集可知, 骨骼数据在原始图中占比较小且相对集中, 导致图中大部分为无用的信息, 所以训练过程中对数据集进行有规划的随机裁剪, 可以节省训练时间和起到数据增强的作用. 规划的随机裁剪目的为将原图裁剪成大小为128×128、包含较多骨骼图像的小尺寸训练样本. 因此裁剪区域需要满足式(8)的约束.
| N裁剪N原图⩾1i×0.2+0.2 | (8) |
N表示为当前区域骨骼像素的总数; i表示当前随机裁剪的次数, i取值[1, 100]. 图片的随机剪裁次数越多, 对其剪裁区域的约束要求越低. 为了扩充数据集样本的多样性和提高模型的泛化性, 除了使用上述的裁剪方法, 本实验还使用了随机旋转、随机水平反转和光度失真3种数据增强的方法.
2.2 实验设置实验所使用硬件平台为16 GB的Tesla T4显卡, 使用的深度学习开源框架为PyTorch 1.8.0, Python版本为3.6, CUDA版本为11.0. 本文采用SGD优化器来对模型参数训练优化, 并使用Poly学习策略对学习率进行调整, 具体定义如式(9). 其中初始学习率lr设置为0.001, power为0.9, 最大训练次数total_epoch为100, 并设置batch_size为64.
| lr×(1−epochtotal_epoch)power | (9) |
针对骨骼数据集的特点, 损失函数的选取使用了以Dice系数损失函数为主, Focal损失函数[16]为辅的策略. CT数据集中骨骼数据相对其余组织占比较少, 使用交叉熵损失函数会将忽略骨骼数据带来的梯度, 从而导致较差的模型优化. Dice损失函数的优化不会根据类别像素占比来决定, 因此选择Dice损失函数作为主要的损失函数. 同时Dice损失函数也具有训练不稳定, 梯度变化剧烈等情况, 因此选择配合Focal损失函数使用. Focal损失函数最早使用于目标检测中正负样本比例严重失衡的场景, 有利于困难样本的挖掘和学习, 针对骨密度较低的难样本具有较好的优化作用. 因此损失函数可用式(10)表示, α表示辅助损失函数的权重, 经实验, 当α取0.5时效果最好.
| Lloss=LDice+αLFocal | (10) |
本文使用Dice系数(Dice)、交并比(IoU)系数、召回率(Recall)、精确率(Precision)来评判模型分割质量. 其计算方式如式(11)–式(14)所示:
| Dice=2TP/(2TP+FP+FN) | (11) |
| IoU=TP/(TP+FP+FN) | (12) |
| Recall=TP/(TP+FN) | (13) |
| Precision=TP/(TP+FP) | (14) |
其中, TP(true positives)、TN(true negatives)、FP(false positives)、FN(false negatives)依次表示为预测和标签均为骨骼像素点数量(真阳性)、预测和标签均为背景像素点数量(真阴性)、预测为骨骼和标签为背景的像素点数量(假阳性)、预测为背景和标签为骨骼的像素点数量(假阴性). 以上指标范围均为0–1之间, 越靠近1表示模型的预测能力越强.
2.4 实验结果汇总本实验选择了在语义分割领域中具有代表性的网络模型: U-Net[5]、Attention U-Net[8]和BiSeNet[17]作为对比网络模型. 在CT数据集上多次实验计算平均值作为最终实验结果, 实验结果如表1所示. 由表1可知在Dice系数上, AFU-Net相较于U-Net提高5个百分点. 相较于同样使用注意力机制提高精度的Attention U-Net提高了2个百分点. 在其余指标上AFU-Net均有不同程度的提高, 证明了AFU-Net模型能够更为准确地对下肢CT数据的骨骼进行精确分割.
| 表 1 CT数据集实验结果(%) |
图5展示了不同模型的分割结果, 从左至右依次为: 原始数据、医师标注的金标准、U-Net、Attention U-Net、BiSeNet和AFU-Net. 可以看到在简单样本中U-Net、Attention U-Net、BiSeNet和AFU-Net均有较好的表现, 但在低密度骨骼区域和有部分小目标碎骨的难样本中AFU-Net分割得更为准确.

|
图 5 CT数据集实验结果对比 |
为进一步证明AFU-Net网络具有较好的泛化性, 我们选取公开数据集Peréz-Carrasco等[18]来验证网络的有效性. 它是由20位患者的27套不同部位的骨骼CT数据集组成, 年龄分布在16–93岁. Peréz-Carrasco数据集对于深度学习算法而言, 训练样本数量较少, 但AFU-Net同样表现得较为良好.
为了确保实验结果准确, 本实验参考原文中的实验设置, 随机选取18套数据作为训练集, 9套数据作为验证集. 取原文中的算法实验结果来进行对比, 对比结果如表2所示.
| 表 2 Peréz-Carrasco数据集实验结果(%) |
本文算法在数据量较小的Peréz-Carrasco数据集上, 仍能取得较好的分割结果. 相较于文献[18] Dice系数提升了4%, IoU系数提高了5%. AFU-Net的分割耗时依赖于算力的好坏, 但较为普通的显卡推理速度也优于原始算法, T4显卡下推理一张图片耗时0.2 s, 相对于文献[18]有较大提升.
CT骨骼数据的精确分割有利于实现骨骼的三维重建, 进而开展后续骨折修复等术前规划. 因此本文将分割好的CT切片利用开源库VTK实现重建, 重建结果如图6所示. 图6(a)为U-Net网络重建结果图, 展示了整体结构与两处细节放大图. 由图可得: 因一整套CT数据集中包含大量简单样本, 所以在整体外观上两者相差不大. 对比细节可得: U-Net建模结果丢失了细小的碎骨, 以及相对于AFU-Net重建结果有许多空洞. AFU-Net分割度较高, 重建效果良好, 可以为后续术前规划工作提供精确的3D模型支持.

|
图 6 分割结果三维重建对比图 |
2.5 消融实验
为进一步证明AFU-Net所提出的模块在CT骨骼数据集上均能有效地提高分割精确度, 本文还做了消融实验. 我们选取U-Net作为基础网络, 依次添加本文新模块来实验分割效果, 实验设置超参数均保持一致, 结果如表3所示.
| 表 3 AFU-Net消融实验结果(%) |
根据表3我们可以得出:
(1)密集连接的空洞卷积模块有效地提高了骨骼的语义提取能力;
(2)基于注意力机制的融合模块能够更加有效地融合不同维度的信息;
(3) 密集连接的空洞卷积模块与基于注意力机制的融合模块结合后, 模型仍可以收敛与优化参数.
3 结论与展望针对现有骨骼图像分割算法存在的分割不精细、细小碎骨易忽略、低对比度骨骼图像难识别的问题, 本文提出一种改进的U-Net分割算法. 首先使用多尺度空洞卷积层来增大感受野, 提取更多的语义信息, 并将不同尺度的. 卷积输出密集连接改善空洞卷积带来的采样点分散问题. 其次使用空间注意力与通道注意力相结合的特征融合模块, 让网络更加专注骨骼信息的同时还解决了不同维度特征融合不充分的问题, 有效地提高了骨骼分割的精准度. 文中通过多个对比实验证明所提出的AFU-Net对于下肢CT数据集具有较好的分割效果. 但是目前算法仍有不足如将CT数据集切分后会丢失部分Axial轴信息和模型参数量较大. 因此后续工作将会考虑利用CT数据的3D信息提高算法精确度, 以及使用模型剪枝和知识蒸馏来压缩模型, 在保证算法性能的同时提高算法实时性.
| [1] |
李银波, 洪波, 高上凯, 等. 人体CT切片图像中骨骼的分割. 生物医学工程学杂志, 2004, 21(2): 169-173. DOI:10.3321/j.issn:1001-5515.2004.02.001 |
| [2] |
Kang HC, Shin YG, Lee J. Automatic segmentation of skin and bone in CT images using iterative thresholding and morphological image processing. IEIE Transactions on Smart Processing and Computing, 2014, 3(4): 191-194. DOI:10.5573/IEIESPC.2014.3.4.191 |
| [3] |
Bakthula R, Agarwal S. Segmentation of bone pixels from EROI image using clustering method for bone age assessment. Proceedings of the SPIE 9789, Medical Imaging 2016: PACS and Imaging Informatics: Next Generation and Innovations. San Diego: SPIE, 2016. 97890H.
|
| [4] |
Shelhamer E, Long J, Darrell T. Fully convolutional networks for semantic segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [5] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [6] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [7] |
Hu J, Shen L, Albanie S, et al. Squeeze-and-excitation networks. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(8): 2011-2023. DOI:10.1109/TPAMI.2019.2913372 |
| [8] |
Oktay O, Schlemper J, Le Folgoc L, et al. Attention U-Net: Learning where to look for the pancreas. arXiv: 1804.03999, 2018.
|
| [9] |
Vania M, Mureja D, Lee D. Automatic spine segmentation from CT images using convolutional neural network via redundant generation of class labels. Journal of Computational Design and Engineering, 2019, 6(2): 224-232. DOI:10.1016/j.jcde.2018.05.002 |
| [10] |
Klein A, Warszawski J, Hillengaß J, et al. Automatic bone segmentation in whole-body CT images. International Journal of Computer Assisted Radiology and Surgery, 2019, 14(1): 21-29. DOI:10.1007/s11548-018-1883-7 |
| [11] |
Noguchi S, Nishio M, Yakami M, et al. Bone segmentation on whole-body CT using convolutional neural network with novel data augmentation techniques. Computers in Biology and Medicine, 2020, 121: 103767. DOI:10.1016/j.compbiomed.2020.103767 |
| [12] |
Huang G, Liu Z, van der Maaten L, et al. Densely connected convolutional networks. 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Honolulu: IEEE, 2017. 2261–2269.
|
| [13] |
Chen LC, Papandreou G, Kokkinos I, et al. DeepLab: Semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4): 834-848. DOI:10.1109/TPAMI.2017.2699184 |
| [14] |
Wang PQ, Chen PF, Yuan Y, et al. Understanding convolution for semantic segmentation. 2018 IEEE Winter Conference on Applications of Computer Vision (WACV). Lake Tahoe: IEEE, 2018. 1451–1460.
|
| [15] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 3–19.
|
| [16] |
Lin TY, Goyal P, Girshick R, et al. Focal loss for dense object detection. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(2): 318-327. DOI:10.1109/TPAMI.2018.2858826 |
| [17] |
Yu CQ, Wang JB, Peng C, et al. BiSeNet: Bilateral segmentation network for real-time semantic segmentation. Proceedings of the 15th European Conference on Computer Vision. Munich: Springer, 2018. 334–349.
|
| [18] |
Pérez-Carrasco JA, Acha B, Suárez-Mejías C, et al. Joint segmentation of bones and muscles using an intensity and histogram-based energy minimization approach. Computer Methods and Programs in Biomedicine, 2018, 156: 85-95. DOI:10.1016/j.cmpb.2017.12.027 |
| [19] |
Bae E, Yuan J, Tai XC. Global minimization for continuous multiphase partitioning problems using a dual approach. International Journal of Computer Vision, 2011, 92(1): 112-129. DOI:10.1007/s11263-010-0406-y |
| [20] |
Zhang Y, Matuszewski BJ, Shark LK, et al. Medical image segmentation using new hybrid level-set. 2008 5th International Conference BioMedical Visualization: Information Visualization in Medical and Biomedical Informatics. London: IEEE, 2008. 71–76.
|
 2022, Vol. 31
2022, Vol. 31



