2. 武汉工程大学 智能机器人湖北省重点实验室, 武汉 430205;
3. 悉尼大学 生物医学工程学院, 悉尼 2006
2. Hubei Key Laboratory of Intelligent Robot, Wuhan Institute of Technology, Wuhan 430205, China;
3. School of Biomedical Engineering, University of Sydney, Sydney NSW 2006, Australia
大豆作为一种豆科植物, 是一种能够提供油脂和蛋白质的农作物, 在许多国家和地区, 都作为主要农作物. 例如, 大豆作为巴西出口的主要农作物产品, 在巴西经济中具有重要意义[1]. 由于大豆是美国最大的动物蛋白饲料来源和第二大植物油来源[2], 因此是美国极为重要的农作物. 由于在当前不利的环境条件下人口基数不断增长, 对大豆育种、生长、发育和产量加以研究以提供充足食物这一任务具有特别重要的意义.
大豆品种(cultivar)种类繁多, 农业专家也在继续开发具有高生产率和高利润率的新的大豆品种. 在低投入和有机耕作条件下, 品种选择和育种重新成为提高作物生长性能的主要工具[3]. 因此, 大豆品种的鉴定和识别在大豆品种的评估、筛选和生产过程中起着至关重要的作用[4].
许多研究[5-8]已经证明了叶片图像模式对于植物物种(species)识别的可用性, 常使用的叶片特征有形状, 纹理, 叶脉和颜色. 然而, 大豆叶片是否也可以作为大豆品种识别的重要线索, 还是一个有趣但具有挑战性的问题. 许多植物物种具有明显不同的叶片图像模式, 并且大多数叶片图像模式可以很容易地从人类视觉感知中识别(请参见物种叶片图像数据库Leaf100[9], ICL[10]和MEW2012[11]中的示例), 与之不同的是, 大豆叶片图像模式在不同的品种之间, 具有很高的相似性(见图1), 这甚至导致即使是领域内的专家也很难简单通过叶片图像模式区分它们.
最近, 人们进行了一些从叶片图像模式中探索大豆品种信息的可用性的尝试[12, 13], 但是方法依赖于手工提取的特征, 例如形状, 纹理和叶脉等. 由于手工提取的特征在捕获大豆品种之间的细微差异方面的性能有限, 在处理大豆品种识别, 这种非常细粒度的模式识别问题方面, 有着很大的局限性, 很难获得令人满意的分类准确率.
深度学习是一类机器学习技术, 它能够通过自动提取信息特征来克服手工方法提取特征的局限性. 深度学习中用于识别任务的典型方法是由不同神经层(例如卷积神经层, Softmax层)的堆栈组成, 并且随着网络训练的不断迭代, 每一层都可以逐步学习更具代表性的抽象特征. 最近的许多研究证明了深度学习技术可有效地实现叶片物种识别任务[14], 但是将深度学习应用于大豆品种识别, 这一类极具挑战性的细粒度的叶片图像识别问题还未见报道.

|
图 1 来自12个不同大豆品种的叶片样本 |
本文专注于使用深度学习技术来研究大豆的品种识别问题. 我们提出了用于大豆品种识别的目标转换注意力网络(Transformation Attention Network, TAN), 它结合了注意力机制和仿射矩阵变换, 分别用于放大叶片图像上的兴趣区域和纠正叶片姿势. 其中, 注意力机制可以实现图像中细粒度特征提取, 该特征包含能够区分不同的品种的有效信息, 而姿势矫正过程可以调整叶片图像姿势以减轻由旋转和平移引起的方差问题. 为研究该方法的有效性, 我们构建了由240个品种组成的大豆叶片图像数据库, 每个类别中有10个样本, 并使用构建的大豆叶片栽培品种数据集对提出的模型进行了评估, 以证明其在研究叶片栽培品种识别任务方面的性能优于最新的方法, 并确认了注意力机制和叶片姿势矫正过程是栽培品种识别的两个关键点.
2 相关工作在过去的几十年中, 已经产生了大量基于叶片图像模式的植物物种识别方法. 这些方法主要可以分为手工方法和深度学习方法两类. 前者从叶片图像中手动提取特征进行分类. 后者使用深度学习模型直接从原始图像数据中学习叶片特征.
当前许多手工提取的叶片特征已被用于区分植物种类, 如叶片形状与轮廓已被视为重要特征, 并为人类专家作为区分植物的主要线索. 近年来已经提出了叶片形状描述子并在植物物种分类任务中取得了较高的分类精度. 分层弦切法(HSC)[9]使用轮廓线段的所有点相对于其切割线段的字符串的空间分布信息来描绘轮廓特征, 通过改变轮廓片段的长度, 构建了多尺度的轮廓弦切描述子, 它具有较高的轮廓描述能力以及较低的时间复杂度, 适合需要竞争性和准确性的大型形状数据库检索. 为了描述叶片形状还设计了不同形式的曲率量度方法, 例如积分不变量[15], 多尺度距离度量[5], 三角形表示[16]等. 此外, 图像矩和傅立叶描述符也广泛用于叶片形状特征的表征[11, 17,18].
叶片纹理是区分植物种类的另一个重要线索. 流行的纹理描述子(例如Gabor滤波器[19, 20], 灰度共现矩阵[21], 局部二进制模式(LBP)[22])已被应用于基于叶片的植物物种识别. 而叶脉作为一种特殊的纹理结构模式可以用作补充线索, 以进一步提高物种分类的准确性. Charters等[23]设计了一个新的描述子, 称为Eagle, 它使用相邻区域之间的边缘模式来表征整个图像叶脉结构, 他们的实验结果表明, Eagle描述子可以与SURF局部描述子相结合并在瑞典叶图像数据集上获得6%的性能提升. Larese等[24]提出在分割的叶脉上计算大量形态特征的方法, 并验证了其在豆类种类识别与分类任务中的有效性.
近年来, 随着深度学习技术的发展, 一些研究人员已努力将深度学习技术应用于分析叶片图像模式. Zhang等[25]构建了一个7层卷积神经网络(CNN), 用于对32种植物的叶片进行分类. 为了提高模型的泛化能力, 他们在研究中使用了局部响应归一化方法加以改进, 研究证明了深度学习方法在叶子分类中的有效性. Grinblat等[7]专注于应用深度学习方法通过叶片的叶脉模式进行植物识别研究, 实验中特别考虑了3种豆科植物的分类: 白豆, 红豆和大豆. Lee等[26]研究了利用深度学习技术直接从叶片图像输入数据的原始表示中学习出有效的描述子的方法, 并从反卷积网络(DN)中获得了所选特征的直觉, 研究中他们利用传统的手工特征抽取算法进行了性能对比, 实验结果表明基于卷积神经网络学习功能的描述子可以提供更好的叶片特征表示, 且取得了更好的识别精确率. 为了提供有效的植物物种自动分类方法, Tan等[27]提出了一种利用叶脉形态计量学对植物物种分类的新颖的卷积神经网络模型, 称为D-leaf模型, 该模型将多个预训练的网络模型(如经过预训练的AlexNet和经过微调的AlexNet)结合在一起用于特征提取, 然后通过将特征转移到支持向量机、K-NN、卷积神经网络等分类器方法中进行比较, 验证了D-leaf模型进行分类的效率.
现有的大多数用于叶片图像分析的方法都只关注植物的种属识别. 同时, 最近, 也有一些研究人员探索叶片图像模式是否可以进一步用于大豆品种识别. Larese等[14, 24]提取、描述并分析了叶脉网络所包含的特征以进行3个不同的大豆品种分类, 并取得了可接受的分类结果. Wang等[13]首次尝试同时使用来自大豆植物不同部位的叶片图像特征描述子, 将整合后的联合叶片模式特征用于进行大豆品种分类, 在研究过程中, 通过多尺度滑弦测量来描绘每个叶片图像模式特征, 他们报告的实验结果证明了大豆叶片图像中蕴含的品种信息的可用性.
值得注意的是, 尽管当前已有研究大豆品种识别方面的工作, 但是由于细粒度图像模式识别的挑战性, 他们对大豆品种的识别准确度明显低于对物种的识别准确度. 在本文中, 我们尝试使用深度学习来解决基于叶片图像模式的大豆品种识别问题. 据我们所知, 这是一个使用深度学习技术进行大豆品种分类的首次尝试, 本研究工作, 也将会进一步推动从物种识别到品种识别的叶片识别研究.
3 目标转换注意力网络我们的目标是设计一个用于准确识别叶片品种的深度神经网络, 该网络不仅可以规范叶片的姿势, 而且可以提取图像细粒度的特征, 从而能够鲁棒而精确的描述不同大豆品种之间的细微差异. 图2展示了我们提出的用于大豆品种识别的目标转换注意力网络的基本框架, 它由卷积神经网络主干和我们提出的目标转换注意力网络模块组成. 下面介绍该网络模型的细节.
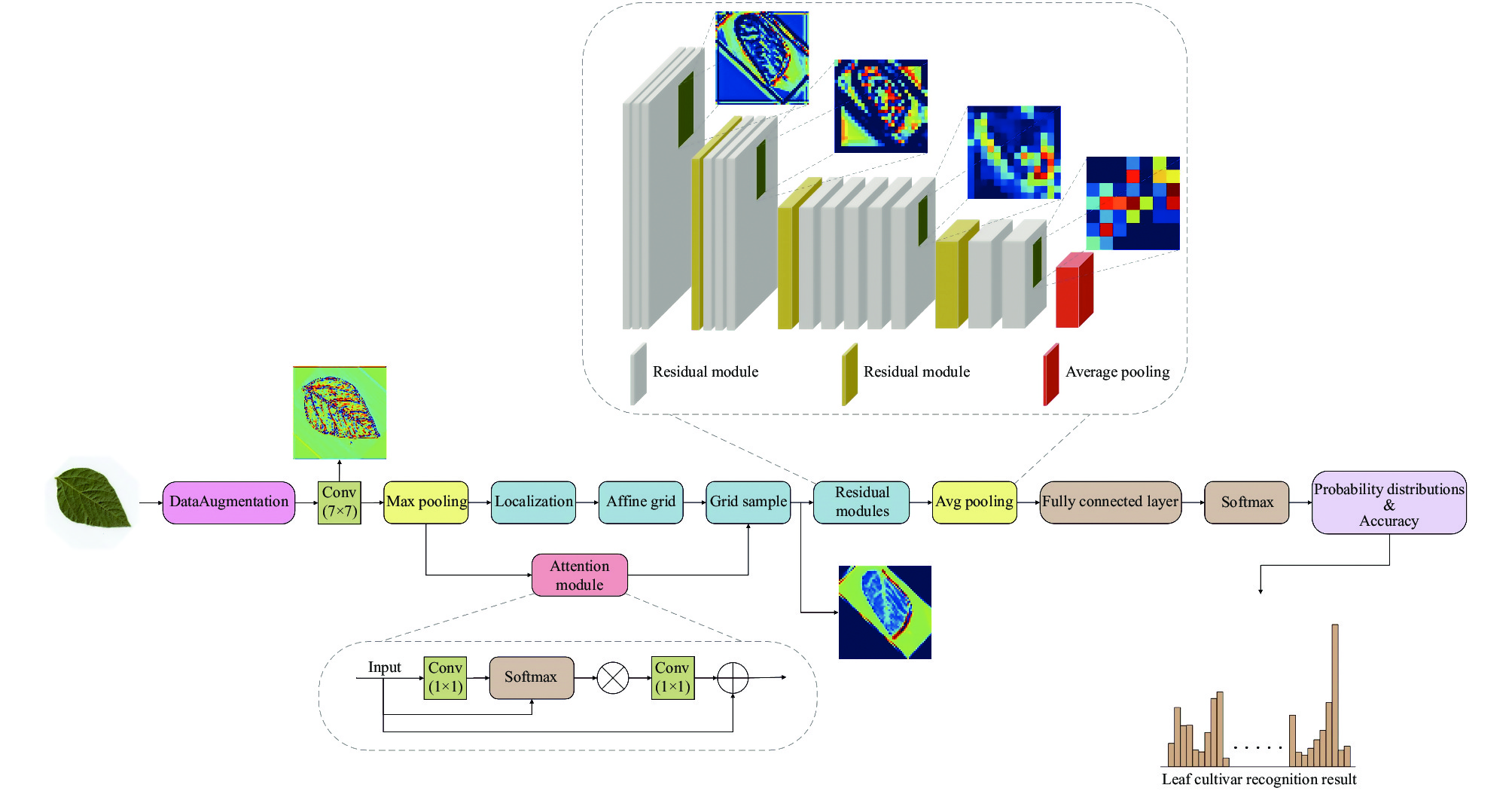
|
图 2 目标转换注意力网络的结构图 |
3.1 卷积神经网络框架
我们的目标是设计一种端到端的体系结构, 该体系结构可以从叶片的图像中鲁棒且准确的提取有用的叶片特征. 尽管当前图像分类研究中已经提出了许多新型网络, 但是由于ResNet[28]已经在叶片分类任务中被多次成功应用[12, 29], 并被证明了其有效性. 因此我们选择ResNet[28]作为目标转换注意力网络的基本框架. 考虑到用于农业的人工智能算法通常是在资源有限的环境中执行的, 因此我们选择使用具有可接受的性能水平和合理的资源要求的ResNet-34加以研究并改进.
本文中使用的ResNet-34包括4个运行阶段, 其中每个阶段都包含特定数量的残差模块[28]. 根据其中使用的shortcut类型, 残差模块被分为两个不同的类别. 在我们的论文中, 我们严格遵循ResNet-34的正式构建方法搭建神经网络.
3.2 目标转换注意力网络文献[8, 26]证明了添加附加的辅助信息可以进一步改善模型的性能, 已被广泛用于叶片图像物种识别任务. 例如, 边界框注释[8, 26]被广泛用于突出具有区分能力的叶脉和纹理信息. 手工裁剪的补丁块[30]被用于辅助叶脉的描述并消除形状特征带来的变异. 尽管这些方法在叶片种类识别方面取得了巨大成功, 但收集并标注这些辅助信息需要花费大量的精力, 并且非常耗时.
当将这种方法用于叶片图像品种分类任务时, 这一困难尤为突出. 例如, 如图3所示, 与不同物种的叶片图像不同, 由于这些大豆品种叶片属于同一个大豆物种, 大量品种的叶片图像在外观特征上非常相似, 甚至某些品种的叶片只在叶脉纹理结构上有细微的差别. 因此, 即使是农业植物学专家很难为叶片图像进行标记以提供附加信息. 此外, 如果图像被专家错误地标记, 甚至会造成深度神经网络的性能大大降低.
除了上述问题, 由于叶片在图像中的位置是多种多样的, 因此叶片图像的姿势也是重要的影响因素, 可能会对识别精度产生不利的影响. 此外, 叶片的形状可能由于生长环境或疾病而变形, 因此这种叶片缺少一些形状特征, 这类因素会对图像识别带来重要影响.
上述两个问题在品种识别任务中广泛存在, 并且不可避免地对最近提出的人工智能算法的结果产生负面影响. 为了应对这些问题, 我们提出了目标转换注意力网络, 它同时利用了注意力机制和仿射变换. 注意机制旨在通过自动定位感兴趣的区域来区分不同品种叶片图像的细微差别, 从而可以为品种识别提供更多细节信息. 以这种方式, 所提出的方法免除了使用手动标记和附加信息的麻烦. 仿射变换校正是从输入图像本身推断出适当的仿射变换矩阵, 然后将其通过缩放和旋转操作来校正输入叶片的姿势, 这样就可以将叶片放置在更容易识别的形态上. 需要注意的是, 这两个模块是互为补充的, 它们提供的功能对于叶片栽培品种数据集的识别至关重要.
目标转换注意力网络(见图2)核心模块由4个部分组成: 注意力模块(attention module), 定位网络(localization), 网格生成器(affine grid)和采样器(grid sample). 网络具体工作流程如下:
步骤1. 对输入图像经过预处理及数据扩充(data augmentation), 统一图像的大小与格式, 并按照一定的比例设计训练集和测试集后, 再将之输入卷积神经网络早期
步骤2. 研究中的注意力模块应用的是结合了空间注意力和通道注意力的混合注意力模型. 对于原始输入特征图, 该模块经过堆叠多个卷积核为1×1的卷积层生成大小为
步骤3. 为了解决输入叶片图像中目标姿态不同的问题, 以卷积神经网络早期运行过程中卷积层生成的原始特征图为输入进入定位网络, 这个定位网络是轻量级的卷积神经网络, 包括两个卷积核大小为
步骤4. 网格生成器调用Python的affine grid模块并输入定位网络提供的
| ${(x_i^t,y_i^t)^{\rm{T}}} = T(G) = \left[ {\begin{array}{*{20}{c}} {\begin{array}{*{20}{c}} {{\theta _{11}}}&{{\theta _{12}}}&{{\theta _{13}}} \end{array}} \\ {\begin{array}{*{20}{c}} {{\theta _{21}}}&{{\theta _{22}}}&{{\theta _{23}}} \end{array}} \end{array}} \right]{(x_i^s,y_i^s,1)^{\rm{T}}}$ | (1) |
这些更新的坐标将被传入采样器模块用于纠正叶子图像的姿势. 这是因为这些姿势校正的坐标传入采样器是用于对注意力增强的特征图进行采样, 以生成注意力特征增强的姿势校正的特征图.
步骤5. 使用Python的Grid Sample采样器模块的作用是通过网格生成器传入的采样坐标
步骤6. 最后, 将注意力机制增强的姿势校正特征图提供给卷积神经网络主干网络和全连接层(fully connected layer), 以进行进一步的特征提取和分类. 这些特征图相较于原始的输入拥有更多信息, 因此可以提高我们的网络在叶片品种识别任务上的性能. 此外, 使用文献[28]中所示的Softmax损失来训练该网络.
4 实验结果和讨论为了验证大豆叶片图像模式中品种信息的可用性, 并验证所提出的目标转换注意力网络对大豆品种识别的有效性, 我们建立了一个大豆品种栽培叶片图像数据库, 并将我们的方法与包括手工方法和深度学习方法在内的几种最先进的方法进行了比较, 以验证本文提出的方法的优越性.
4.1 数据集虽然目前有一些网上公开的叶片图像库, 如Leaf100[9], MEW2012[11], ICL[10], 可供实验研究, 但这些数据库都是作为物种识别研究的, 还没见到可供品种识别的叶片图像库. 我们收集不同大豆品种的叶片来建立大豆品种叶片数据库. 该数据库包含从240个大豆品种植株中收集的2400片叶子. 对于每个品种, 我们从不同植株的下部随机采集10片叶子. 所有叶片图像均使用具有600 dpi高分辨率和48位真彩色设置的EPSON V850 Pro扫描仪获得. 图3给出了每个品种的叶片示例, 图4则给出了前10个品种的所有叶片样本作为示例. 与网上公开的叶片图像数据库Leaf100[9], MEW2012[11], ICL[10]相比, 大豆品种栽培叶片数据库具有更高的类间相似性, 因为所有叶片属于同一物种, 这使其成为研究叶片品种识别的一个新的且具有挑战性的数据集.

|
图 3 取自大豆品种叶片图像库的240个品种的示例样本(每个品种一个样本) |
4.2 实验对比的方法
在我们的实验中使用了6种对比方法, 包括4种传统的手工方法: 局部二进制模式(LBP)[22], 分层弦切法(HSC)[9], 定向梯度直方图(HOG)[31]和多尺度滑弦匹配(MSCM)[13]. 此外还有两种深度学习方法, 分别是滤波器学习区分网络(DFL)[32]和导航-自指导-审查网络(NTS)[33], 这两种深度网络在对图像进行细粒度分类都有较为优异的结果, 它们被用于作为我们提出的模型的对比基准. 我们的方法和其他两个深度学习方法DFL和NTS的预训练模型分别是ResNet-34, VGG16和ResNet-50. 对于所有对比实验, 我们使用大豆品种叶片图像库中每个品种的前6个叶片图像用作训练样本(共有1440个样本), 其余960个叶片图像作为测试样本. 下面是参与对比的所有方法的介绍.

|
图 4 大豆品种叶片图像数据库中前10个品种的所有样本, 每行显示了同一大豆品种的所有图像 |
HOG: 梯度方向直方图[31]是一种梯度信息的统计方法. 由于梯度信息主要存在于图像目标的边缘区域, 因此可以通过研究梯度的分布或边缘的方向密度来适当地描述目标的外观和形状信息. 该方法已广泛用于植物叶片识别[34, 35].
HSC: 分层弦切线[9]旨在提取叶片的轮廓特征, 其中轮廓段的特征在于轮廓点相对于其弦的空间分布信息. 该方法是最新的形状描述方法, 可以提供快速而准确的叶片图像分类结果.
LBP: 局部二进制模式[36]被证明是最成功的局部外观描述符之一, 用于捕获小的纹理细节和外观. 由于叶片具有非常精细的纹理和叶脉特征, 而这些特征是叶片图像分类的最重要标志之一, 因此许多研究[22, 37-39]已将LBP技术应用于叶片图像识别任务.
MSCM: 多尺度滑弦匹配[13]是一种最近发表的方法, 致力于使用叶片图像模式来区分大豆品种. 它以从粗糙到精细的层次顺序表征叶片图像模式, 并测量描绘叶片图像的外部形状和内部外观的同步模式. 为了增强品种描述的判别力, 它结合了植物不同部位的叶片描述符, 使得来自不同位置的信息相互补充, 是一种有效的大豆品种鉴定方法.
DFL-NET: DFL-NET模型[32]是一种端到端深度学习方法, 该方法使用具有卷积滤波器监督和非随机层初始化的非对称多流结构. 旨在通过卷积过滤器来增强图像特征中级表示与学习, 以便在无须其他辅助信息情况下使用补丁程序获取不同级别信息和图像的全局外观, 并且网络可以提取特定于类别的判别性补丁程序.
NTS-NET: NTS-NET模型[33]由导航代理模块, 教师代理模块和审查程序代理模块组成. 通过计算图像中多个区域的特征向量来训练该导航器网络. 它可用于检查导航代理模块在教师代理模块的指导下检测到的信息量大的区域, 并反馈建议信息, 以便导航器网络可以查看和融合这些特征向量用于分类任务.
DFL-NET和NTS-NET在公共细粒度数据集CUB-200-2011上都获得了较高的分类精度[40]. 因此, 我们选择它们作为代表性的深度学习方法进行对比实验.
4.3 结果与分析表1给出了我们的方法和所有参与比较的方法在大豆叶片图像库上的实验结果. 可以看出, 我们的方法达到了令人鼓舞的71.90%的分类准确度, 证明了叶片图像模式的品种信息的可用性, 并验证了叶片图像模式对大豆品种识别的有效性.
| 表 1 本文提出的方法与其他手工特征和深度特征方法在大豆叶片图像数据库上的分类准确率 |
从表1可以看出, 我们的方法的分类准确率分别比4种手工特征方法HOG[31]、HSC[9]、LBP[36]、MSCM[13]高出50.10%, 48.98%, 47.50%和35.55%. 与深度学习方法相比, 我们的方法的分类准确率分别比NTS[33]和DFL[32]高14.20%和16.59%. 这些结果表明, 我们的方法在表征叶片图像模式方面效果更好, 并且更适合于大豆品种的识别.
实验结果还表明, 在大豆叶片品种识别问题上, 深度学习方法的分类精度比传统的手工方法高出18.96%, 该结果说明, 与手工特征方法相比, 深度学习方法在捕获不同品种的叶片图像模式非常细微的差异方面效果更好. 值得注意的是, 尽管DFL-NET[32]和NTS-Net[33]这两种深度学习模型在其他细粒度分类任务中均取得了良好的准确性, 但它们比本文提出的方法分别低了16.59和14.20个百分点. 其原因分析总结如下:
DFL[32]使用全局最大池为给定图像的每个补丁块生成响应权重. 然后, 将具有相应特征向量的响应权重用于描述给定图像, 以进行最终图像分类. 但是, 由于每个补丁块的特征图中会存在精细叶脉特征所携带的代表性信息丢失的情况, 因此DFL的网络设计不适用于叶片品种识别. 与DFL相比, 我们的网络使用了注意力机制, 该机制能够为每个像素分配权重得分, 这种策略使我们的网络能够专注于细粒度的叶脉和边缘像素, 从而改善模型的性能.
NTS-NET[33]的成功使用取决于深度网络自动检测到并设置大型边界框, 这些边界框是网络自动关注的感兴趣的矩形区域, NTS-NET在其中提取关键性的特征. 通常情况下, 这种边界框区域中的特征很容易识别, 特别是当测试图像中的对象由许多不同的部分组成时. 叶片叶脉信息是该数据集叶片十分独特的部分, 不能简单地用大的边界框来简单地描述叶片叶脉特征模式. 此外, 当叶子的姿势不正常时, 特别是当在数据集的收集过程中其略微旋转和平移时, 矩形边界框很容易标记错位置. 我们的网络通过使用叶姿势校正模块解决了这个问题, 因此我们的模型优于NTS-NET.
5 结论与展望本文针对大豆品种识别这一极具挑战性的叶片图像模式的细粒度识别问题, 提出一种称为目标转换注意力网络的深度学习模型. 为了有效地从叶片图像中提取细粒度的特征表示, 使用注意力机制来捕获叶片图像的细微的特征差异, 然后使用仿射变换来纠正叶片的姿势, 使得方法更加鲁棒. 我们构建了一个由240个大豆品种图像组成的叶片图像数据库, 每个品种有10个样本, 以验证叶片图像模式中品种信息的可用性, 并验证我们的方法对大豆品种识别的有效性. 实验结果表明, 我们的方法相比于最先进的手工方法和深度学习方法具有更高的分类精度, 并且还证实了叶片图像模式是大豆品种识别的重要线索.
| [1] |
Moreira A, Moraes LAC, Fageria NK. Variability on yield, nutritional status, soil fertility, and potassium-use efficiency by soybean cultivar in acidic soil. Communications in Soil Science and Plant Analysis, 2015, 46(19): 2490-2508. DOI:10.1080/00103624.2015.1085555 |
| [2] |
Liang WZ, Kirk KR, Greene JK. Estimation of soybean leaf area, edge, and defoliation using color image analysis. Computers and Electronics in Agriculture, 2018, 150: 41-51. DOI:10.1016/j.compag.2018.03.021 |
| [3] |
van Bueren ETL, Myers JR. Organic crop breeding: Integrating organic agricultural approaches and traditional and modern plant breeding methods. In van Bueren ETL, Myers JR eds. Organic Crop Breeding. Hoboken: John Wiley & Sons, Ltd., 2012. 3–13.
|
| [4] |
Cavassim JE, Filho JCB, Alliprandini LF, et al. AMMI analysis to determine relative maturity groups for the classification of soybean genotypes. Journal of Agronomy, 2013, 12(4): 168-178. DOI:10.3923/ja.2013.168.178 |
| [5] |
Wang B, Brown D, Gao YS, et al. MARCH: Multiscale-arch-height description for mobile retrieval of leaf images. Information Sciences, 2015, 302: 132-148. DOI:10.1016/j.ins.2014.07.028 |
| [6] |
Siravenha AC, Carvalho SR. Plant classification from leaf textures. Proceedings of 2016 International Conference on Digital Image Computing: Techniques and Applications. Gold Coast: IEEE, 2016. 1–8.
|
| [7] |
Grinblat GL, Uzal LC, Larese MG, et al. Deep learning for plant identification using vein morphological patterns. Computers and Electronics in Agriculture, 2016, 127: 418-424. DOI:10.1016/j.compag.2016.07.003 |
| [8] |
Zhu XL, Zhu M, Ren HG. Method of plant leaf recognition based on improved deep convolutional neural network. Cognitive Systems Research, 2018, 52: 223-233. DOI:10.1016/j.cogsys.2018.06.008 |
| [9] |
Wang B, Gao YS. Hierarchical string cuts: A translation, rotation, scale, and mirror invariant descriptor for fast shape retrieval. IEEE Transactions on Image Processing, 2014, 23(9): 4101-4111. DOI:10.1109/TIP.2014.2343457 |
| [10] |
Hu RX, Jia W, Ling HB, et al. Multiscale distance matrix for fast plant leaf recognition. IEEE Transactions on Image Processing, 2012, 21(11): 4667-4672. DOI:10.1109/TIP.2012.2207391 |
| [11] |
Novotný P, Suk T. Leaf recognition of woody species in Central Europe. Biosystems Engineering, 2013, 115(4): 444-452. DOI:10.1016/j.biosystemseng.2013.04.007 |
| [12] |
Chen X, Wang B. Invariant leaf image recognition with histogram of Gaussian convolution vectors. Computers and Electronics in Agriculture, 2020, 178: 105714. DOI:10.1016/j.compag.2020.105714 |
| [13] |
Wang B, Gao YS, Yuan XH, et al. From species to cultivar: Soybean cultivar recognition using joint leaf image patterns by multiscale sliding chord matching. Biosystems Eng-ineering, 2020, 194: 99-111. DOI:10.1016/j.biosystemseng.2020.03.019 |
| [14] |
Larese MG, Bayá AE, Craviotto RM, et al. Multiscale recognition of legume varieties based on leaf venation images. Expert Systems with Applications, 2014, 41(10): 4638-4647. DOI:10.1016/j.eswa.2014.01.029 |
| [15] |
Kumar N, Belhumeur PN, Biswas A, et al. Leafsnap: A computer vision system for automatic plant species identification. Proceedings of the 12th European Conference on Computer Vision. Berlin Heidelberg: Springer, 2012. 502–516.
|
| [16] |
Mouine S, Yahiaoui I, Verroust-Blondet A. A shape-based approach for leaf classification using multiscaletriangular representation. Proceedings of the 3rd ACM Conference on International Conference on Multimedia Retrieval. Dallas: ACM, 2013. 127–134.
|
| [17] |
Horaisová K, Kukal J. Leaf classification from binary image via artificial intelligence. Biosystems Engineering, 2016, 142: 83-100. DOI:10.1016/j.biosystemseng.2015.12.007 |
| [18] |
Kalyoncu C, Toygar Ö. Geometric leaf classification. Computer Vision and Image Understanding, 2015, 133: 102-109. DOI:10.1016/j.cviu.2014.11.001 |
| [19] |
VijayaLakshmi B, Mohan V. Kernel-based PSO and FRVM: An automatic plant leaf type detection using texture, shape, and color features. Computers and Electronics in Agriculture, 2016, 125: 99-112. DOI:10.1016/j.compag.2016.04.033 |
| [20] |
Cope JS, Remagnino P, Barman S, et al. Plant texture classification using Gabor co-occurrences. Proceedings of the 6th International Symposium on Visual Computing. Berlin Heidelberg: Springer, 2010. 669–677.
|
| [21] |
Tang Z, Su YC, Er MJ, et al. A local binary pattern based texture descriptors for classification of tea leaves. Neurocomputing, 2015, 168: 1011-1023. DOI:10.1016/j.neucom.2015.05.024 |
| [22] |
Naresh YG, Nagendraswamy HS. Classification of medicinal plants: An approach using modified LBP with symbolic representation. Neurocomputing, 2016, 173: 1789-1797. DOI:10.1016/j.neucom.2015.08.090 |
| [23] |
Charters J, Wang ZY, Chi ZR, et al. Eagle: A novel descriptor for identifying plant species using leaf lamina vascular features. Proceedings of 2014 IEEE International Conference on Multimedia and Expo Workshops. Chengdu: IEEE, 2014. 1–6.
|
| [24] |
Larese MG, Namías R, Craviotto RM, et al. Automatic classification of legumes using leaf vein image features. Pattern Recognition, 2014, 47(1): 158-168. DOI:10.1016/j.patcog.2013.06.012 |
| [25] |
Zhang CY, Zhou P, Li CH, et al. A convolutional neural network for leaves recognition using data augmentation. Proceedings of 2015 IEEE International Conference on Computer and Information Technology; Ubiquitous Computing and Communications; Dependable, Autonomic and Secure Computing; Pervasive Intelligence and Computing. Liverpool: IEEE, 2015. 2143–2150.
|
| [26] |
Lee SH, Chan CS, Mayo SJ, et al. How deep learning extracts and learns leaf features for plant classification. Pattern Recognition, 2017, 71: 1-13. DOI:10.1016/j.patcog.2017.05.015 |
| [27] |
Tan JW, Chang SW, Abdul-Kareem S, et al. Deep learning for plant species classification using leaf vein morphometric. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2020, 17(1): 82-90. |
| [28] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [29] |
Too EC, Li YJ, Njuki S, et al. A comparative study of fine-tuning deep learning models for plant disease identification. Computers and Electronics in Agriculture, 2019, 161: 272-279. DOI:10.1016/j.compag.2018.03.032 |
| [30] |
Lee SH, Chan CS, Wilkin P, et al. Deep-plant: Plant identification with convolutional neural networks. Proceedings of 2015 IEEE International Conference on Image Processing. Quebec City: IEEE, 2015. 452–456.
|
| [31] |
Dalal N, Triggs B. Histograms of oriented gradients for human detection. Proceedings of 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition. San Diego: IEEE, 2005. 886–893.
|
| [32] |
Wang YM, Morariu VI, Davis LS. Learning a discriminative filter bank within a CNN for fine-grained recognition. Proceedings of 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 4148–4157.
|
| [33] |
Yang Z, Luo TG, Wang D, et al. Learning to navigate for fine-grained classification. Proceedings of the 15th European Conference on Computer Vision. Cham: Springer, 2018. 438–454.
|
| [34] |
Xiao XY, Hu RX, Zhang SW, et al. HOG-based approach for leaf classification. Proceedings of the 6th International Conference on Intelligent Computing. Berlin Heidelberg: Springer, 2010. 149–155.
|
| [35] |
Olsen A, Han S, Calvert B, et al. In situ leaf classification using histograms of oriented gradients. Proceedings of 2015 International Conference on Digital Image Computing: Techniques and Applications. Adelaide: IEEE, 2015. 1–8.
|
| [36] |
Ojala T, Pietikainen M, Maenpaa T. Multiresolution gray-scale and rotation invariant texture classification with local binary patterns. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2002, 24(7): 971-987. DOI:10.1109/TPAMI.2002.1017623 |
| [37] |
Turkoglu M, Hanbay D. Leaf-based plant species recognition based on improved local binary pattern and extreme learning machine. Physica A: Statistical Mechanics and its Applications, 2019, 527: 121297. DOI:10.1016/j.physa.2019.121297 |
| [38] |
Turkoglu M, Hanbay D. Plant recognition system based on deep features and color-LBP method. Proceedings of the 2019 27th Signal Processing and Communications Applications Conference. Sivas: IEEE, 2019. 1–4.
|
| [39] |
Janahiraman TV, Yee LK, Der CS, et al. Leaf classification using local binary pattern and histogram of oriented gradients. Proceedings of the 2019 7th International Conference on Smart Computing & Communications. Sarawak: IEEE, 2019. 1–5.
|
| [40] |
Wah C, Branson S, Welinder P, et al. The Caltech-UCSD birds-200-2011 dataset. Pasadena: California Institute of Technology, 2011.
|
 2021, Vol. 30
2021, Vol. 30



