随着现代科技的飞速发展, 大量数据从各个领域中呈爆炸式不断产出. 同时因为这些数据中含有大量不相关、冗余信息, 在进行数据挖掘时, 预处理技术中的特征选择便变得极为重要和极具挑战性[1].
目前特征选择已经在文本分类[2]、模式识别[3-5]、癌症分类[6]和故障诊断[7]等领域内成功应用. 特征选择可定义为从数据集中去除不相关和冗余的特征, 从而增强后续学习算法性能的过程[8]. 特征选择方法可根据两个标准来分类: 搜索策略和评估标准[8]. 特征子集可以通过两种模型获取: 过滤式[9]模型和封装式[10]模型. 过滤式模型在搜索过程中仅考虑数据集本身, 而封装式模型需在搜索过程中结合学习算法. 因此过滤式模型花费时间成本较小, 但准确率不稳定. 反观封装式模型, 由于需要结合学习算法, 所以准确率相对更高, 同时也更耗时. 此外搜索最优子集的方式也是特征选择中的一个关键问题, 具体可分为遍历、随机搜索和启发式搜索. 其中遍历虽然一定可以获取最优子集但是不适用于大型数据集[11]. 随机搜索则因为没有数学理论引导搜索方向导致搜索性能不稳定[12]. 而启发式算法使用启发式信息指导搜索. 虽然不能保证找到最优解, 但可以再合理的时间内获取可接受的解[13]. 近年已经有许多研究对不同特征选择算法进行了对比, 如在文献[14]中, 使用了3种不同的分类器评估了8种标准的特征选择方法. 文献[10]使用了K最近邻算法作为分类器, 系统地研究了鲸鱼优化算法(Whale Optimization Algorithm, WOA)并将基于WOA的封装式特征选择算法和三种标准的启发式特征选择算法进行比较证明了WOA的强大性能. 文献[15]中将融合启发式算法和3种标准的封装式特征选择算法进行了对比以证明融合启发式算法的潜力.
综上, 本文基于过滤式模型和封装式模型各自的特点, 将这两种模型结合, 构建一种基于最大互信息系数和皮尔逊相关系数的、使用遗传算法进行超参数自动优化的两阶段特征选择融合算法(A feature selection fusion method based on Maximal Information Coefficient and Pearson correlation coefficient with parameters automatic optimized by Genetic Algorithms, MICP-GA), 通过结合使用最大互信息系数、皮尔逊相关系数的过滤式模型和以分类准确率为目标的封装式模型来克服过滤式模型准确率较低, 传统相关系数处理非线性关系能力较弱以及封装式模型的时间成本过高的问题. 第一阶段根据最大互信息系数获取各特征和标签之间的相关度评分, 该评分综合考虑线性和非线性相关度, 再设置相关度阈值以剔除不相关特征. 第二阶段, 通过皮尔逊相关系数获取特征子集特征之间的线性冗余度评分, 同样设置冗余度阈值来删除冗余特征. 最后, 将特征子集的分类准确率作为评价标准, 使用遗传算法自动优化前两步中的超参数, 达到综合减少特征数目和维持甚至提高特征子集分类精度的效果, 并自动获取最优特征子集.
1 基础理论 1.1 信息熵与最大互信息系数定义1. 信息熵[16]克服了对信息随机变量不确定性的度量, 设X为离散随机变量, 那么X的信息熵H(X)为:
| $H(X) = - \sum\limits_{i = 1}^m {p({x_i})} \log p({x_i})$ | (1) |
定义2. 条件熵表示为当随机变量Y单独发生时, 随机变量X发生的条件概率分布, 可通过式(2)表示:
| $H(X|Y) = \sum\limits_{{y} \in Y} {p({y})\sum\limits_{{x} \in X} {p({x}|{y})} } \log p({x}|{y})$ | (2) |
定义3. 互信息用于检测两随机变量中所含的信息量和互相关联程度. 互信息可通过式(1)和式(2)用熵表示:
| $I(X;Y) = H(X) - H(X|Y) = H(Y) - H(Y|X)$ | (3) |
最后依据式(2)和式(3)可得:
| $0 \le I(X;Y) \le \min \{ H(X),H(Y)\} $ | (4) |
Reshef等人[17]提出的最大互信息系数(Maximal Information Coefficient, MIC)无需对数据分布进行任何假设便可评估变量间的函数关系和统计关系. 该算法具有普适性和均匀性两大特点, 普适性指当数据规模足够大时, MIC算法能有效捕捉到大规模有意义的关系, 而并不会仅局限于某种函数关系; 均匀性则指对于不同类型函数关系, 当给予相同噪声时, MIC算法给出相同或相近的结果变化. 最大互信息系数Imax(X;Y)可通过互信息和熵计算得出:
| ${I_{\max }}(X;Y) = \frac{{I(X;Y)}}{{\min \{ H(X),H(Y)\} }}$ | (5) |
皮尔逊相关系数(Pearson Correlation Coefficient, PCCs)由Karl Person提出, 因其计算简单、运算速度快而被广泛用于度量在数据预测、故障诊断和参数估计等领域中两变量间的线性相关程度, 其取值范围是[−1,1]. 两变量间相关系数的绝对值越大, 表明两者的相关度越高, 当取值为0时表示两个变量不相关. 具体相关系数数值大小和相关判断结果的对应关系见表1.
通常皮尔逊相关系数用希腊字母ρ表示. 其具体定义为两个变量之间的协方差和标准差的商, 其中cov表示协方差, E为数学期望:
| $\begin{split} {\rho _{X,Y}} &= \frac{{{\rm{cov}} (X,Y)}}{{{\sigma _X}{\sigma _Y}}}= \frac{{E((X - {{{\mu }}_X})(Y - {{{\mu }}_X}))}}{{{\sigma _X}{\sigma _Y}}} \\ &=\frac{{E({{XY}}) - E(X)E(Y)}}{{\sqrt {E({X^{{2}}}) - {E^2}(X)} \sqrt {E({X^{{2}}}) - {E^2}(Y)} }}\end{split}$ | (6) |
| 表 1 相关性判断准则 |
1.3 遗传算法
遗传算法的灵感来自生物的遗传过程, 该算法的解被称为染色体或个体. 二进制遗传算法的每个染色体包含二进制值为0或1的基因, 这些基因决定了每个个体的属性. 一系列的染色体组成了一个种群. 每个染色体的性能通过适应度函数来评估, 适应度值较高的染色体被选作父代, 并通过交叉步骤结合产生新的后代. 再对新的种群进行变异处理来增加个体的随机性, 降低陷入局部最优的可能[18].
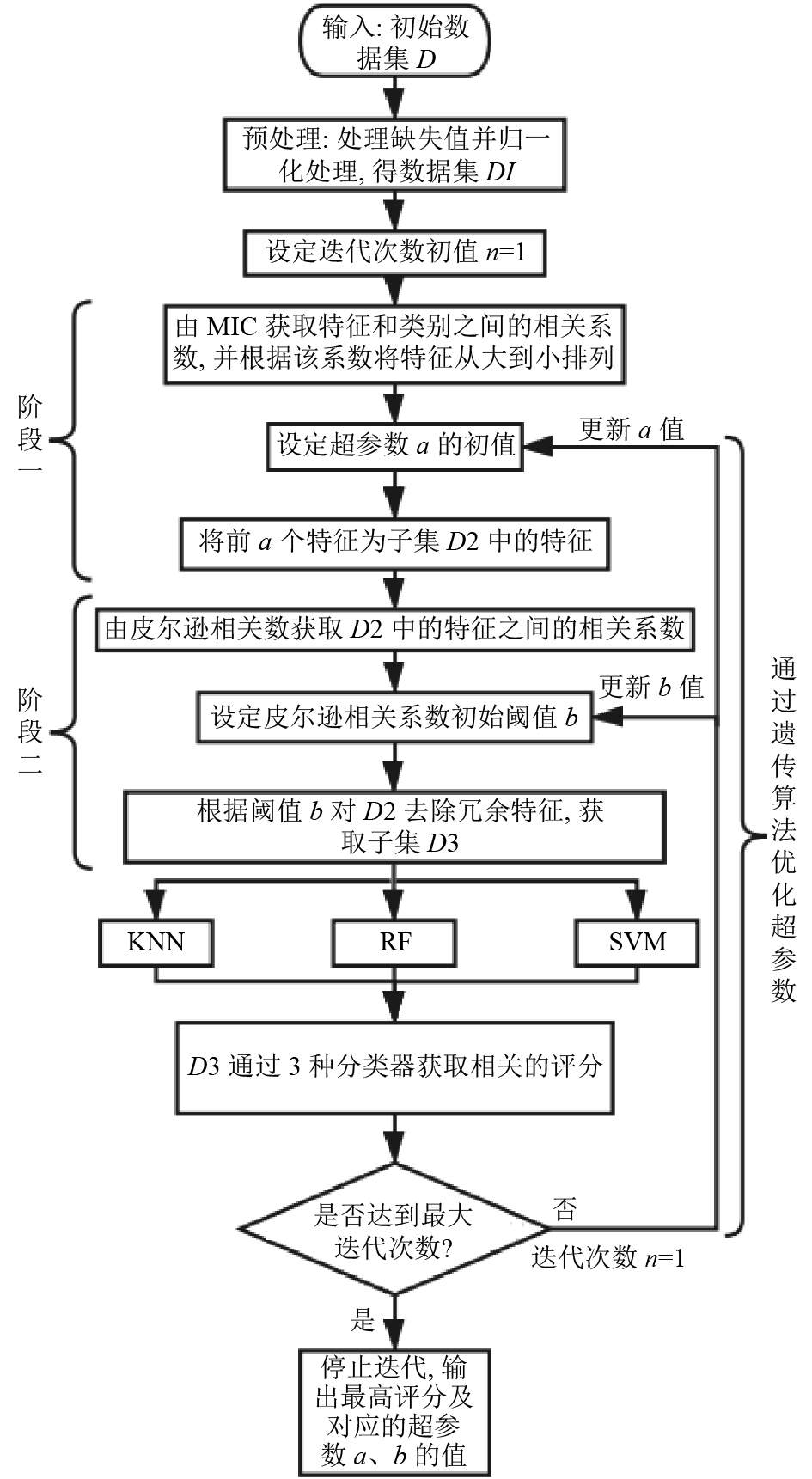
2 MICP-GA基础理论MICP-GA算法兼顾过滤式和封装式特征选择算法的优点, 同时利用遗传算法对前述步骤中的超参数进行自动优化来获取最优特征子集. 该算法的实现流程如图1所示.
2.1 运用最大互信息系数获取各特征和标签的相关度本阶段利用最大互信息系数进行初次特征选择. 第一阶段特征选择的具体步骤如算法1所示.
该方法使用最大互信息系数准确找出和类别线性相关、非线性相关的特征, 剔除不相关的特征, 但筛选出的特征之间仍可能存在线性冗余. 为解决该问题, 继续进行第二步特征选择.
2.2 基于皮尔逊相关系数的特征去冗余对第一步选取的特征子集D2
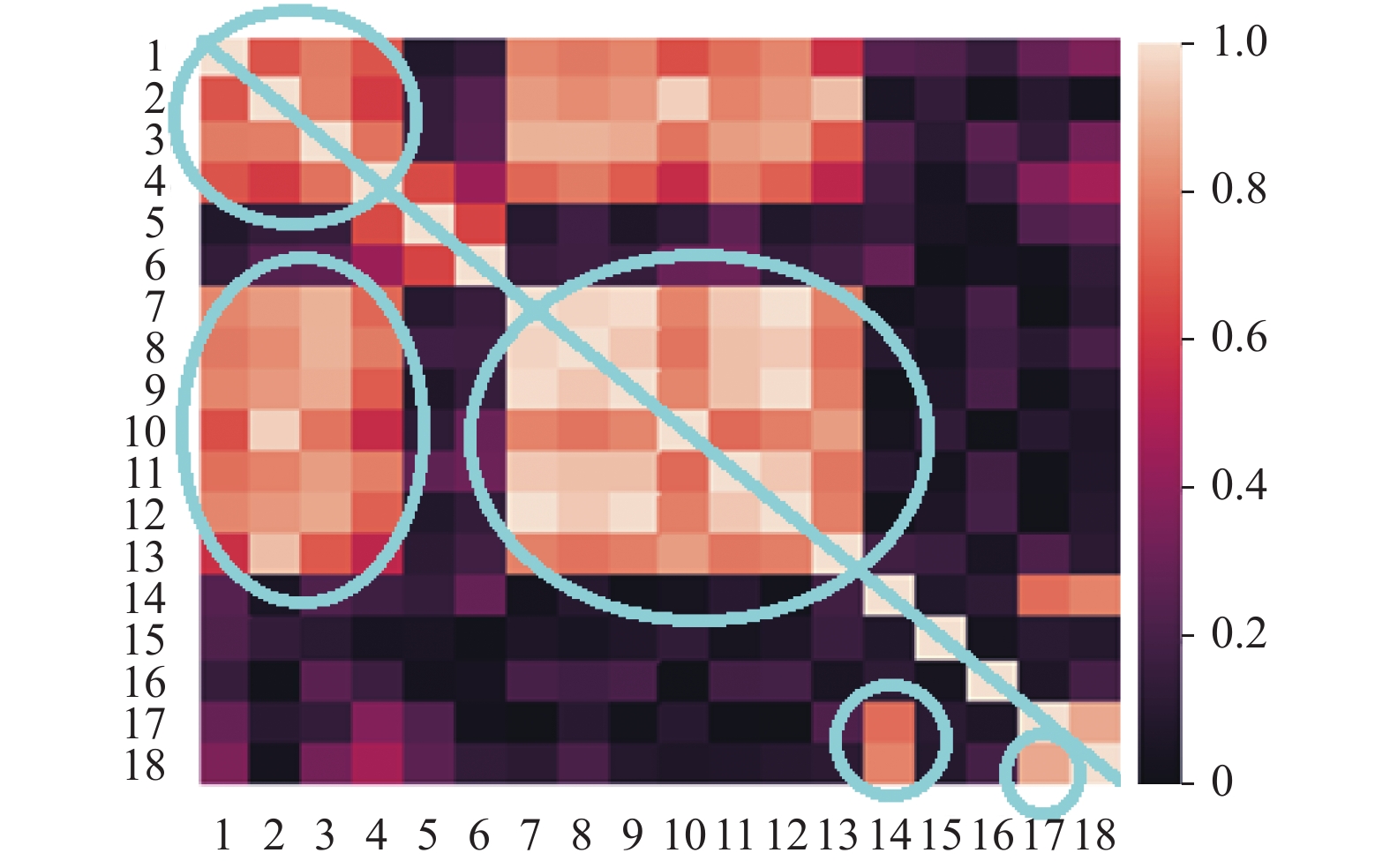
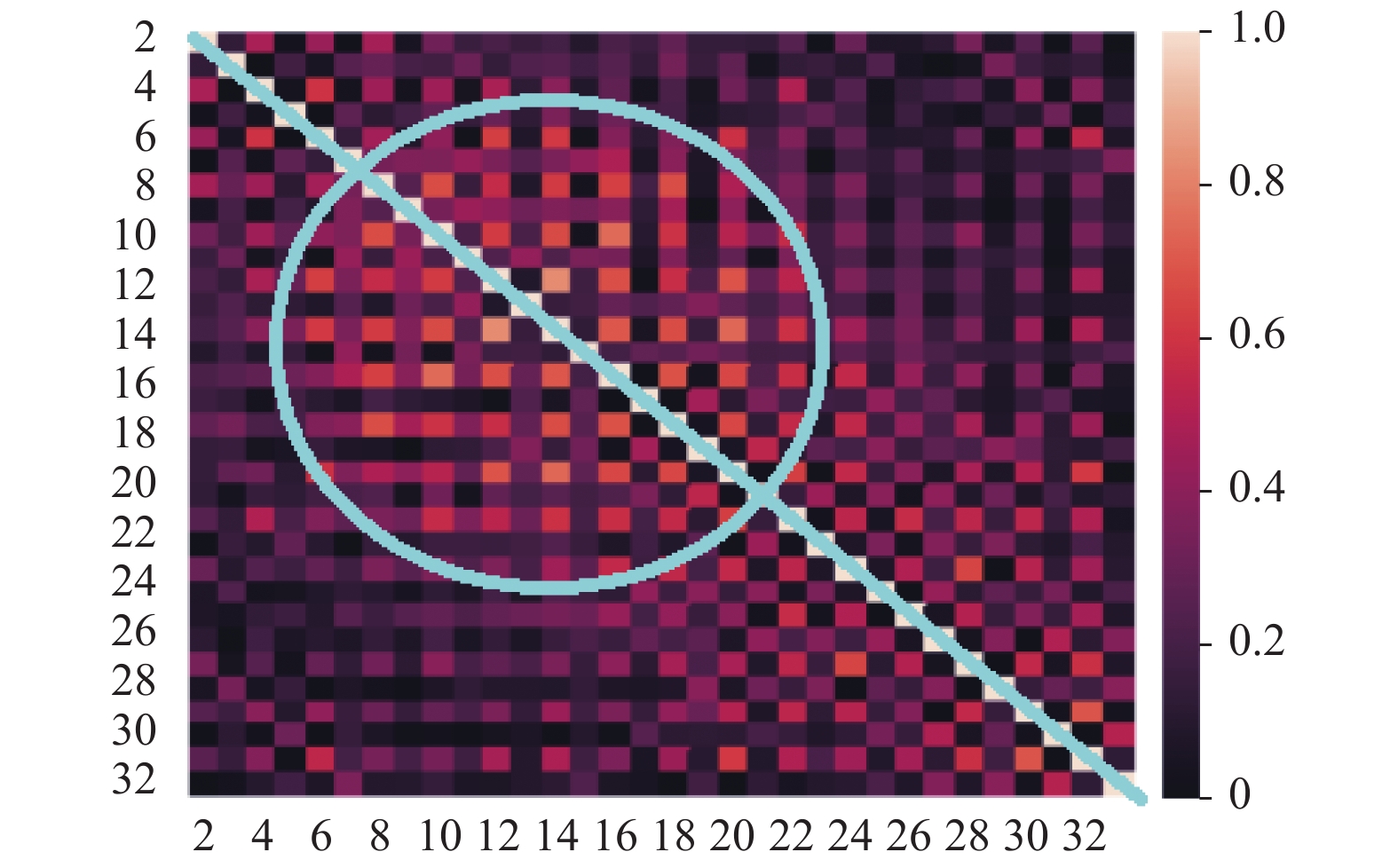
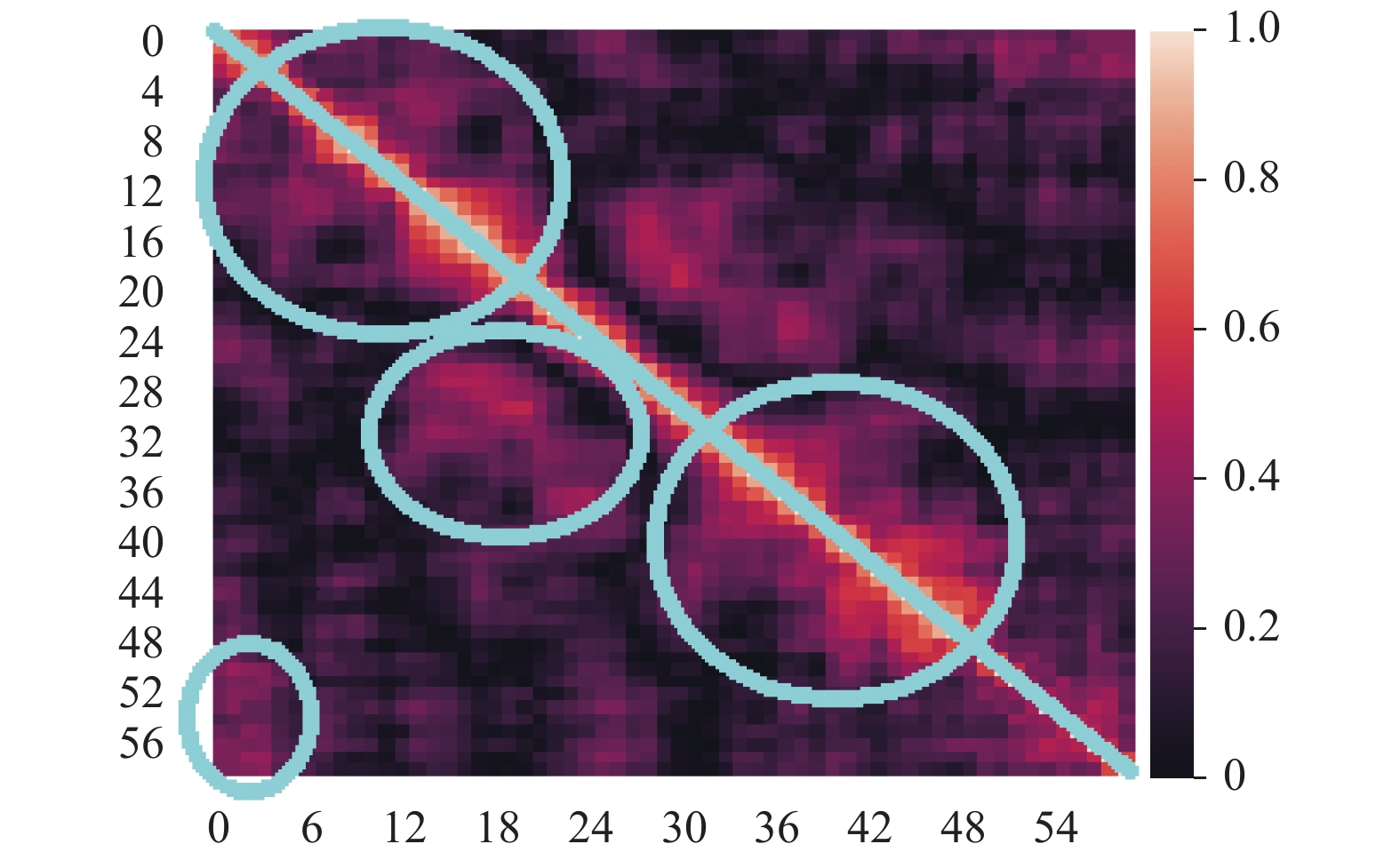
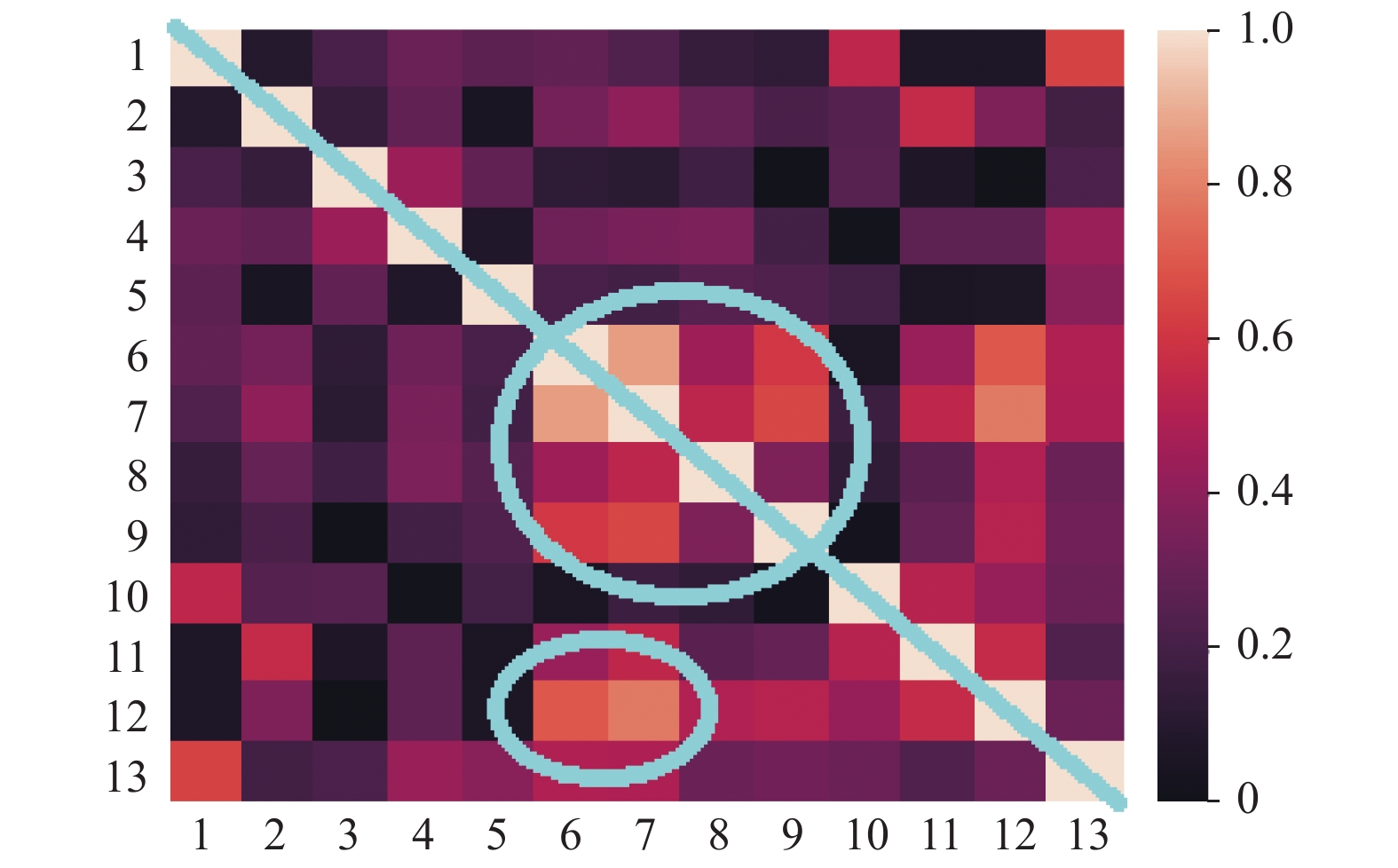
由前述可知, 第一阶段中的超参数a和第二阶段中的阈值b这两个参数值对分类结果有着重要的影响. 接下来, 以UCI标准数据集中的Vehicle数据集为例进行说明: 该数据集初始特征共计18维, 如在第一阶段特征选择时设定a为18, 则保留全部特征. 现对所选的Vehicle、Ionosphere、Wine和Sonar数据集中的特征的皮尔逊相关系数进行可视化处理, 如图2~图5所示. 热力图中, 两两特征之间的皮尔逊相关系数的取值范围是[0,1], 对应着图中不同的颜色. 此外, 由于热力图沿着正对角线(如图2~图5中从左上贯穿至右下的蓝线所示)对称, 所以仅观察热力图的左下部分即可. 由图可知, 其中若干区域(如图2~图5中蓝色圈注所示)中特征之间的皮尔逊相关系数较大, 说明该区域内的特征之间存在线性冗余. 可采用基于皮尔逊相关系数的特征选择算法有效解决该问题.
|
图 1 整体算法流程图 |
|
图 2 Vehicle数据的皮尔逊相关系数热点图 |
|
图 3 Ionosphere数据的皮尔逊相关系数热点图 |
|
图 4 Sonar数据的皮尔逊相关系数热点图 |
|
图 5 Wine数据的皮尔逊相关系数热点图 |
2.3 使用遗传算法实现超参数的自动优化
本文采用遗传算法自动优化3.1和3.2节中的超参数a和b. 遗传算法优化超参数的步骤描述如算法3所示.
此外染色体个数NIND设为5. 最大迭代次数MAXGEN设为20. 子代和父代个体不相同的概率GGAP设为0.9. 遗传算法选择方式SELECTSTYLE选用轮盘赌选择法rws. 基因变异的概率PM由源代码中的0.1修改为由式(7)获取, 令PM随着迭代次数t的增加而不断减小, 使得算法在搜索前期扩大搜索空间, 不容易陷入局部最优解, 而且在搜索后期也不会因为PM过大导致子集状态有较大突变.
| $PM = 0.9-\frac{{(t - 1) \times 0.9}}{{MAXGEN - 1}}$ | (7) |
综上所述, 本文提出的遗传算法可对前两步骤中的待优化超参数a、b自动进行优化, 并以适应度函数Fitness作为目标函数, 将Fitness取得最小值时对应的a、b作为最优参数.
3 实验分析
为检测上文提出的MICP-GA算法的可靠性, 本文选取了5个UCI标准数据集. 表2和表3分别列出了这些测试数据集的具体信息以及直接将各数据集初始数据用于不同分类器时的分类准确率.
| 表 2 测试数据集 |
在测试中, 使用Python3进行编程, 并且使用常规的十折交叉验证用于检验. 接下来使用3种特征选择方法对上述数据集进行测试和比较, 3种方法分别是MICP-GA、单独用MIC特征选择以及单独使用皮尔逊相关系数特征选择. 在测试过程中, 将3种特征选择方法与三种典型分类器, K最近邻分类器(K-Nearest Neighbors, KNN)、随机森林分类器(Random Forest, RF)和支持向量机(Support Vector Machine, SVM)结合后, 对上述数据集进行测试. 结果显示平均最高准确率如表4所示, 平均特征选取率如表5所示, 平均适应度函数如表6所示. 对比表3和表4的分类准确率, 可见本文提出的算法对原始数据的分类准确率有明显提升, 说明MICP-GA在搜索最优特征选择的组合时能够有效跳出局部最优, 确保搜索到的解是近似全局最优解, 同时说明MICP-GA充分考虑特征和类别之间的包括线性和非线性的关系, 解决了传统相关系数处理非线性关系表现不好的问题, 并根据MIC评分来判断删除每对冗余特征中的哪一个, 较好地结合了运用MIC和PCCs的特征选择从而使得分类效果比单独使用MIC或PCCs时更优. 同时, 结合表5中基于不同特征选择方法的分类器的平均特征选取率, 也表明了该算法在降低数据维度方面有较优的表现, 能够有效剔除和类别不相关的特征以及冗余特征, 为后续学习算法节省大量的运算成本, 提升了运行效率. 综上所述, 本文提出的算法充分考虑了特征和类别之间的线性相关性以及特征之间的冗余性, 能够有效对数据进行降维同时保证数据集的分类准确率保持不变甚至提升分类准确率.
| 表 3 基于不同分类器的初始数据集的平均分类准确率 |
| 表 4 基于不同特征选择方法的分类器的平均最高准确率 |
| 表 5 基于不同特征选择方法的分类器的平均特征选取率 |
| 表 6 基于不同特征选择方法的分类器的平均适应度值 |
4 结论与展望
特征选择算法的优劣对模型的预测准确率有着重要影响, Filter可以快速高效地去除冗余特征及不相关特征, 但是无法保证获取的特征子集准确率达标. Wrapper更倾向于获取准确率更高的特征子集, 所以其时间成本一般远高于前者. 本文针对以上两种方法的特点, 将其进行结合提出了MICP-GA方法, 所提算法兼顾了两者的优点并且在UCI标准数据集中也有较好的表现. 但是因为结合了遗传算法以及最大互信息系数, 导致时间复杂度比传统特征选择方法更高, 根据文献[10]可见传统的全局搜索算法在性能和运算复杂度上并没有较大差别, 所以没有将GA替换成其他全局搜索算法. 而如果使用单解的搜索算法如模拟退火(Simulated Annealing, SA)算法替换GA确实可以较快获取解, 但鉴于SA极其容易陷入局部最优, 因此不考虑用SA搜索最优特征子集. 所以如何加快搜索速度是后续进一步的研究课题.
| [1] |
Cai J, Luo JW, Wang SL, et al. Feature selection in machine learning: A new perspective. Neurocomputing, 2018, 300: 70-79. DOI:10.1016/j.neucom.2017.11.077 |
| [2] |
Yang JM, Liu YN, Liu Z, et al. A new feature selection algorithm based on binomial hypothesis testing for spam filtering. Knowledge-Based Systems, 2011, 24(6): 904-914. DOI:10.1016/j.knosys.2011.04.006 |
| [3] |
Wang YY, Peng WX, Qiu CH, et al. Fractional-order darwinian PSO-based feature selection for media-adventitia border detection in intravascular ultrasound images. Ultrasonics, 2019, 92: 1-7. DOI:10.1016/j.ultras.2018.06.012 |
| [4] |
Zhu XF, Li XL, Zhang SC, et al. Robust joint graph sparse coding for unsupervised spectral feature selection. IEEE Transactions on Neural Networks and Learning Systems, 2017, 28(6): 1263-1275. DOI:10.1109/TNNLS.2016.2521602 |
| [5] |
Chang XJ, Yang Y. Semisupervised feature analysis by mining correlations among multiple tasks. IEEE Transactions on Neural Networks and Learning Systems, 2017, 28(10): 2294-2305. DOI:10.1109/TNNLS.2016.2582746 |
| [6] |
Zhu PF, Xu Q, Hu QH, et al. Multi-label feature selection with missing labels. Pattern Recognition, 2018, 74: 488-502. DOI:10.1016/j.patcog.2017.09.036 |
| [7] |
Yan XA, Jia MP. Intelligent fault diagnosis of rotating machinery using improved multiscale dispersion entropy and mRMR feature selection. Knowledge-Based Systems, 2019, 163: 450-471. DOI:10.1016/j.knosys.2018.09.004 |
| [8] |
Liu H, Motoda H. Feature selection for knowledge discovery and data mining. Boston, MA: Springer, 1998, 7-8. |
| [9] |
Hancer E, Xue B, Zhang MJ. Differential evolution for filter feature selection based on information theory and feature ranking. Knowledge-Based Systems, 2018, 140: 103-119. DOI:10.1016/j.knosys.2017.10.028 |
| [10] |
Mafarja M, Mirjalili S. Whale optimization approaches for wrapper feature selection. Applied Soft Computing, 2018, 62: 441-453. DOI:10.1016/j.asoc.2017.11.006 |
| [11] |
Guyon I, Elisseeff A. An introduction to variable and feature selection. Journal of Machine Learning Research, 2003, 3(6): 1157-1182. |
| [12] |
Lai C, Reinders MJT, Wessels L. Random subspace method for multivariate feature selection. Pattern Recognition Letters, 2006, 27(10): 1067-1076. DOI:10.1016/j.patrec.2005.12.018 |
| [13] |
Boussaïd I, Lepagnot J, Siarry P. A survey on optimization metaheuristics. Information Sciences, 2013, 237: 82-117. DOI:10.1016/j.ins.2013.02.041 |
| [14] |
Abusamra H. A comparative study of feature selection and classification methods for gene expression data of glioma. Procedia Computer Science, 2013, 23: 5-14. DOI:10.1016/j.procs.2013.10.003 |
| [15] |
Mafarja MM, Mirjalili S. Hybrid whale optimization algorithm with simulated annealing for feature selection. Neurocomputing, 2017, 260: 302-312. DOI:10.1016/j.neucom.2017.04.053 |
| [16] |
Ma CW, Ma YG. Shannon information entropy in heavy-ion collisions. Progress in Particle and Nuclear Physics, 2018, 99: 120-158. DOI:10.1016/j.ppnp.2018.01.002 |
| [17] |
Reshef DN, Reshef YA, Finucane HK, et al. Detecting novel associations in large data sets. Science, 2011, 334(6062): 1518-1524. DOI:10.1126/science.1205438 |
| [18] |
Ghamisi P, Benediktsson JA. Feature selection based on hybridization of genetic algorithm and particle swarm optimization. IEEE Geoscience and Remote Sensing Letters, 2015, 12(2): 309-313. DOI:10.1109/LGRS.2014.2337320 |
 2020, Vol. 29
2020, Vol. 29



