在人工智能技术日趋成熟的今天, 人们将人工智能技术越来越多的应用在各个领域. AI+医疗是当下最火热的人工智能应用场景之一, 并且AI在乳腺癌, 糖尿病等预防和治疗方面, 创造了诸多突破和成就. 宫颈癌发病率高, 且病变周期长, 早期发现治疗效果好[1,2], 目前临床检测仍为人工筛选, 耗时, 昂贵且准确率低[3]. 前期筛查是对癌变进行预防和控制的关键途径[4,5]. 我国宫颈癌早期普查工作量十分繁重, 然而病理医生数量却严重不足. 因此, 采用AI技术进行宫颈细胞病理辅助诊断在癌前病变诊断中具有重要意义[6].
2009年, Pan SJ等[7]提出了一种元启发式算法进行宫颈细胞分类, 将遗传算法与KNN相结合, 从宫颈细胞图像中构建了20个特征, 使用遗传算法进行最优特征子集的选取, 使KNN算法进行分类, 并证明了有效性. 2014年, Chankong等[8]提出一种宫颈癌细胞自动分割和分类的方法, 利用FCM聚类技术将单细胞图像分割为细胞核, 细胞质, 并进行特征提取, 利用人工神经网络进行分类, 并与其他分类器结果进行比较, 证明了人工神经网络的分类效果与其他分类器相比, 精度较高. 2015年, Kaaviya等[9]提出了一种新的分类方法对宫颈细胞进行分类. 为了提高宫颈细胞分类结果, 采用集成方法, 集成了3个分类器的决策, 并使用五折交叉验证进行评估.
2010年, 暨南大学的范金坪[10]提出基于矢量量化的C-V模型进行彩色宫颈图像分割, 利用遗传算法进行特征选择, BP神经网络算法进行原始特征子集以及最优特征子集分类, 验证特征选择的有效性. 2018年, 四川大学的缪欣等[11]提出了基于神经网络集成模型的宫颈细胞分类算法, 集成神经网络相对于单个神经网络误识别率明显下降. 2018年, 胡卉等[12]提出了基于卷积神经网络对宫颈细胞进行分类. 验证了卷积神经网络用于宫颈细胞分类的可行性.
基于以往研究学者对宫颈细胞识别方法的研究, 可以总结出传统的算法都是先经过细胞分割, 其次从分割后的图像中人工提取细胞图像的特征, 然后设计算法进行特征降维等操作, 最后选取合适的分类器进行识别[13-15]. 此类方法通常要求在分割阶段有较高的分割准确率, 否则会对后续的特征提取产生影响, 其次选择特征提取需要人工来决定, 这就使得研究人员首先具备一定的病理知识, 尽管如此, 人工选取的特征也不一定具有代表性, 这就导致识别效果不好. 基于此, 本文因此采用深度学习算法中的DCNN来进行特征提取[16], 以及细胞识别分类的研究[17,18]. DCNN将卷积计算同BP神经网络相结合的神经网络, 具有特征自动特取以及分类识别的功能[19]. 卷积的引入使其能够感知图像局部细节[20,21], 提取数据的局部特征, 其权值共享的特性减少了网络参数运算量[22], 且无需考虑图像中特征出现的位置, 因此, 其在图像识别领域具有显著的优势[23]. 基于此, 本文将采用深度卷积神经网络模型进行宫颈细胞图像识别, 以解决特征提取不完善的问题, 从而提高宫颈细胞图像识别的准确率以及效率, 实现智能化识别.
1 研究方法针对宫颈细胞自动分类的研究过程, 主要分为图像集的预处理, 模型的选取, 模型的训练以及模型的测试. 在图像集的预处理阶段, 对图像进行灰度化, 去噪增强等操作[24], 接着对处理后的图强提取ROI区域, 并根据神经网络的输入要求统一尺寸.
在模型选取阶段, 本文采用深度卷积神经网络对宫颈图像进行分类, 卷积神经网络是一种具有不同功能网络层的深度学习算法, 其分为输入层, 特征提取层, 以及分类结果输出层[25].
输入层一般是带有分类标签的图像数据, 需要根据不同网络的要求来统一尺寸[26]. 在这一层一般是要对输入的图像数据进行预处理, 使之适应网络计算要求. 常用的预处理分为: 去均值, 归一化, PCA降维.
特征提取层包含卷积层, 激励层, 池化层. 卷积层采用类似滑动滤波器一样的滑动窗口对图像进行特征提取, 每个神经元的输入与前一层的局部接受域相连, 并提取该局部的特征, 且局部权重共享[27]. 权值共享减少了网络参数运算量, 降低了模型的复杂性, 且无需考虑图像中特征出现的位置. 激励层的主要作用是将卷积层的输出结果做一个非线性的映射, 一般采用ReLU函数作为激励层的激活函数, 它具有收敛速度快, 且梯度计算简单的特点. 池化层处于两个相连的卷积层中间, 用于压缩传输数据与参数, 减小分类过程中出现过拟合[28]. 池化的方法一般分为平均池化和最大池化, 本文采用最大池化的方法进行压缩传输图像. 池化层在卷积神经网络中的作用是用来识别经过位移, 缩放变换以及其他形式扭曲且不发生性质变化的图像[29].
分类结果输出层即特征映射层, 由图像特征与图像类别变迁的映射关系, 本文采用Sigmoid函数作为输出层的激活函数. Sigmoid函数的输出范围在(0,1)之间, 具有指数函数的平滑性, 在分类输出结果中, 越接近于1, 说明该类的可能性越大. Sigmoid函数使得特征映射具有位移不变性[30].
本文选取VGG16网络作为基础模型来进行改进, VGG卷积神经网络是2014年被提出的, 其在图像分类以及图像检测中表现很好, 并在2014年ILSVRC比赛中取得了很好的成绩, 其准确率达到了92.3%. 在VGG模型中, VGG-16表现良好, 且应用较多, 它是一个具有16层深度的模型, 模型结构如图1所示.
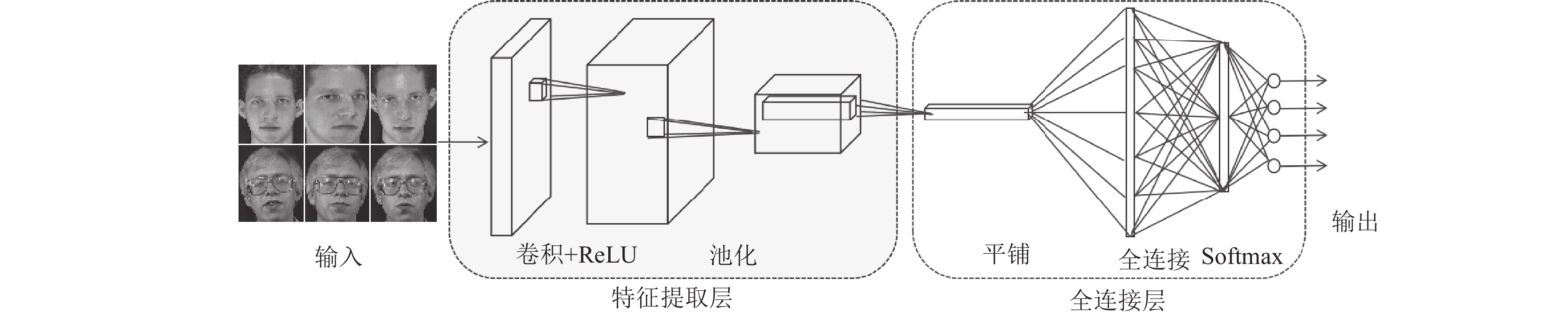
|
图 1 CNN模型图 |
在模型训练阶段, 由于宫颈细胞图像的数量较少, 为了提高分类准确率, 本文采用迁移学习的方法对网络进行预训练, 采用ImageNet数据集训练模型, 将得到的模型参数作为宫颈细胞分类模型的初始化参数, 由于ImageNet是一个具有1000类的数据集, 而宫颈细胞的分类只有7类, 所以将原始模型的特征映射层Softmax层进行修改, 并对全连接层参数进行调整, 以加快收敛速度, 提高准确率. 模型如图2所示.
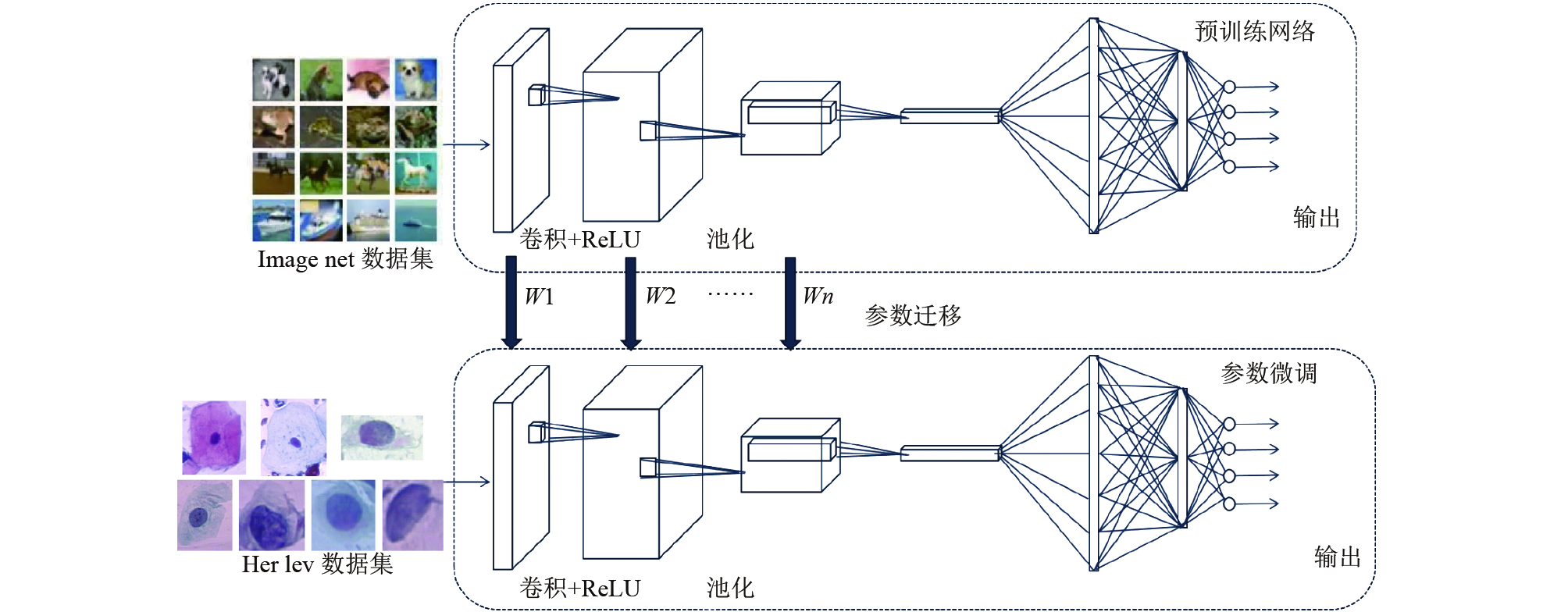
|
图 2 网络训练过程 |
在模型测试阶段, 对于训练好的模型, 将测试集输入模型, 得出分类准确率.
2 实验过程 2.1 数据集介绍本文采用的宫颈细胞数据集为公开的HerLev图像集, 该图像集是通过数码相机和显微镜在赫列夫大学医院制作的. 图像分辨率是0.201 μm/像素. Herlev的数据集共包含917幅图像, 每幅图像包含一个子宫颈细胞, 分为7类, 这7个类属于两大类别: 类1~3为正常, 类4~7为异常类, 每一类的数据情况如表1所示. 其中每个类别的确定由两名细胞技术人员和一名医生共同诊断, 这样以最大限度地提高诊断的准确性, 避免个人主观带来的误差.
图3为宫颈细胞数据集中部分细胞的例子, 图3(a)–图3(g)为正常细胞到异常细胞, 可以看出, 异常细胞相比较正常细胞细胞核明显增大, 且颜色变深, 核质比明显变大可见, 但是相邻种类的细胞变化较小, 比如图3(e)与图3(f)外观相似, 这对于CNN来说, 要想分辨两类, 是有相当大的难度.
| 表 1 HerLev数据集介绍 |
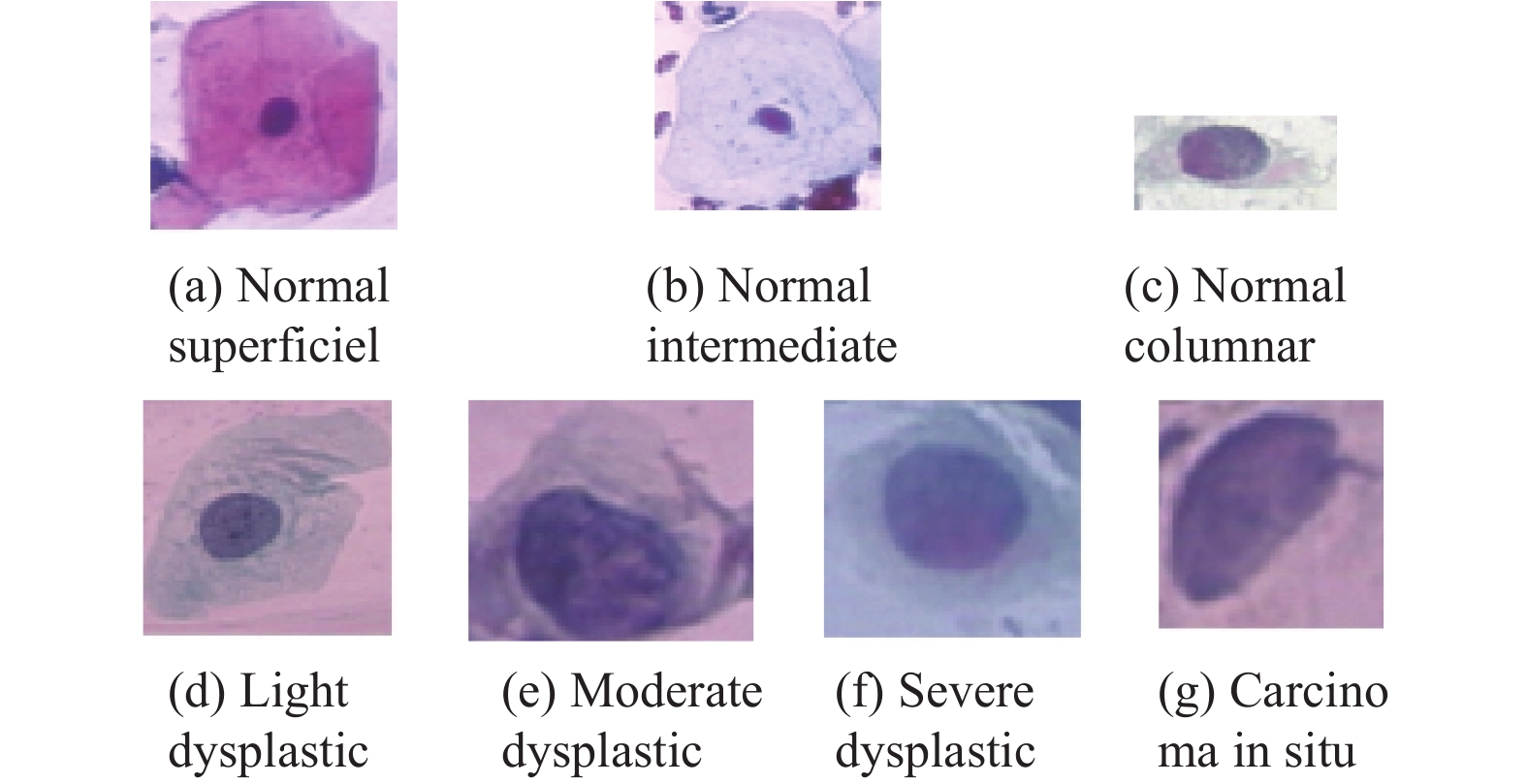
|
图 3 宫颈细胞图像 |
2.2 数据预处理
(1)提取ROI区域
由于网络需要统一大小的图片输入尺寸, 而HerLev数据中的尺寸大小不一, 且长宽比差均很大. 若依照传统方法改变图像尺寸, 会导致图像内部特征改变, 从而影响分类效果且分类模型不具有普适性, 因此本文采取提取ROI区域的方式进行适应网络尺寸. 宫颈细胞不同类别的差距主要来自于细胞核的差异, 医生在判别细胞异常与否也是通过对细胞核的分析来确定, 因此本文从原始图像中裁剪出固定大小的细胞核区域作为网络输入图像. 具体做法为以细胞核质心为中心, 裁剪出128×128大小的图像, 如图4所示.
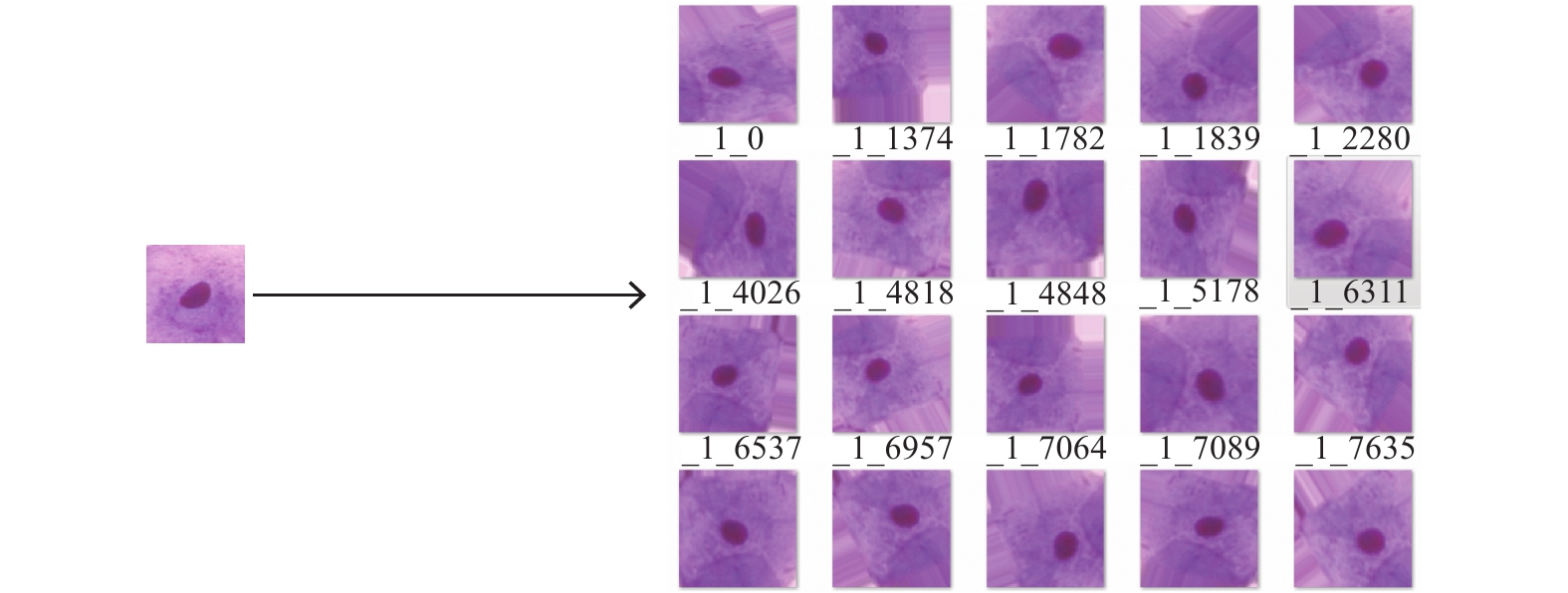
(2)图像集扩充
由于网络训练需要大量的数据集, 且每一类的样本量要均衡. 宫颈数据集共917张, 分为7类, 最少的一类有70张, 最多的一类有198张. 需要扩充数据集的量, 并且解决不同类别样本量不均衡的问题. 由于宫颈细胞具有旋转不变性, 本文通过平移旋转的方式对数据集进行扩充, 将正常细胞数据量扩充为之前的20倍, 异常细胞扩充为之前的10倍, 并按照网络的输入要求进行边缘填充. 扩充操作如图5所示.
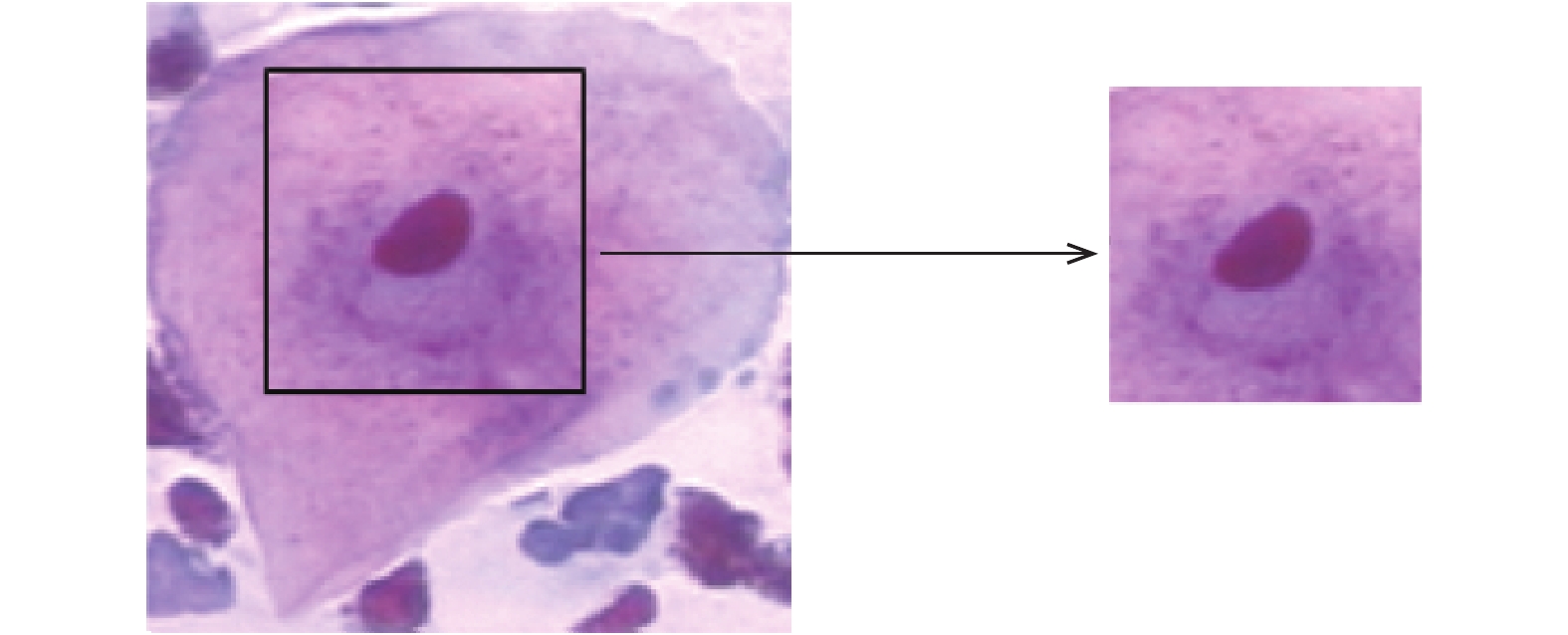
|
图 4 ROI区域选取 |
|
图 5 图像集扩充 |
2.3 网络设计
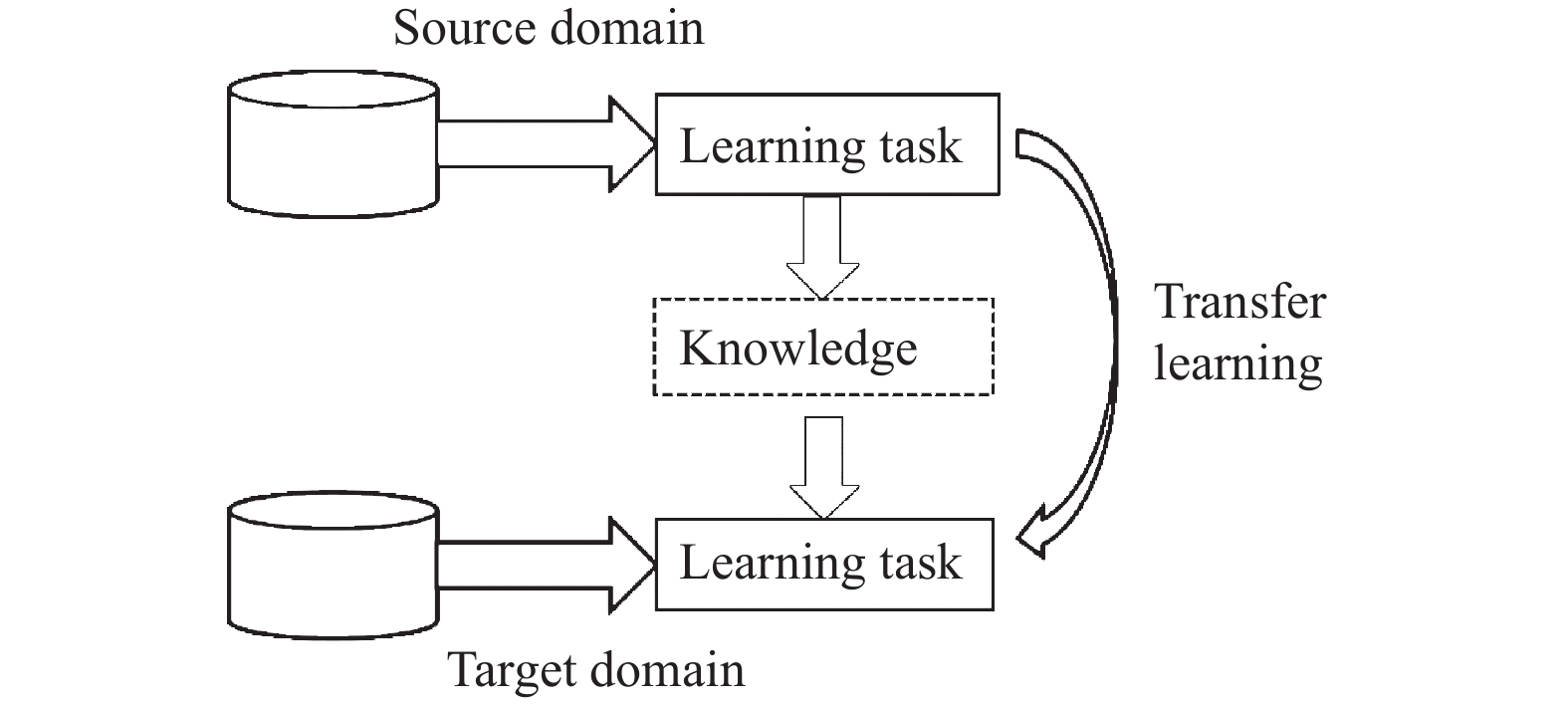
由于宫颈细胞数据量较小, 直接训练网络花费时间较长且效果不理想, 本文采用迁移学习的方法, 使用ImageNet进行网络预训练. 迁移学习指的是采用现有的或者已有的知识去解释或者学习另一相关领域的知识, 其目标是完成知识在相关领域之间的迁移. 迁移学习基本原理如图6所示.
|
图 6 迁移学习过程 |
对于卷积神经网络而言, 一般在如下情况下益采用迁移学习的方法. (1)新的数据集较小且与旧数据集差距大; (2)新数据集较大且与旧数据集相似. 本文情况符合第一种, 即宫颈细胞的数据集较小, 因此在网络的训练过程中, 可以先利用其他大型数据集对网络进行预训练, 以获得网络的初始化参数. 迁移学习使用预训练深度神经网络作为学习新任务的起点, 由之前的随机初始化变为预训练网络参数作为初始化, 只需要对网络进行较少的训练, 或者只用微调剩余网络层, 这样很大程度上降低了网络训练的时长. 因此, 迁移学习不仅增强了网络对小数据集的学习能力, 还可以加快网络的收敛速度.
由于本文使用的预训练网络模型为VGG16模型, 所以改进的网络参数大部分与VGG16网络参数一致, VGG16由13个卷积层, 5个池化层, 2个全连接层以及1个Softmax层. 本文设计的网络卷积层同VGG16网络卷积层相同, 卷积层参数如表2所示.
| 表 2 VGG16卷积层参数 |
由于越靠近最终的Softmax分类层, 网络的特征跟原始数据集越相关, 所以在迁移网络参数的时候, 只复用卷积层的参数, 并根据具体应用数据集进行更改全连接层以及Softmax层参数.
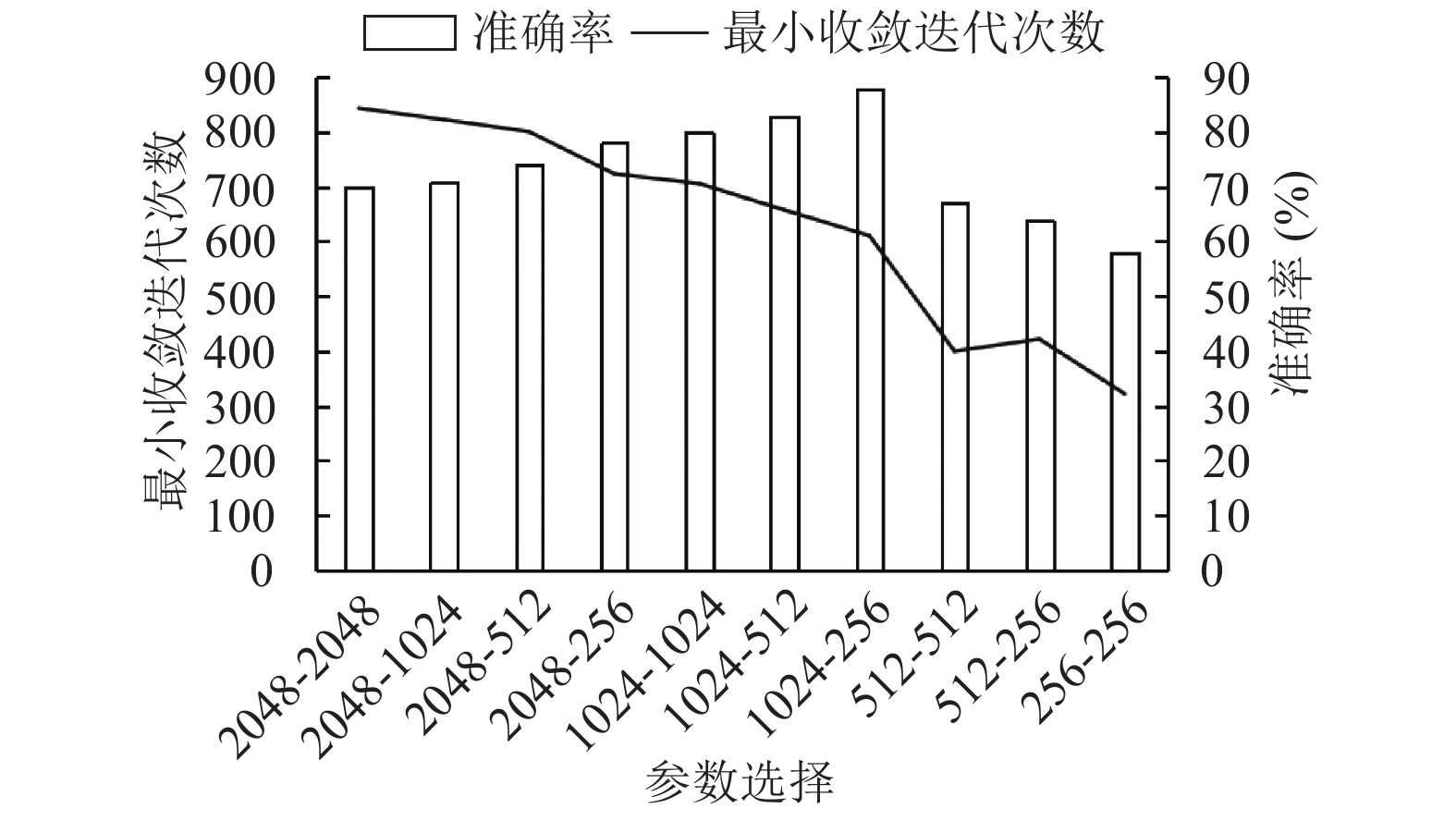
在VGG16网络中全连接层神经元个数分别为4096-4096-1000, 全连接层FC1的权重参数量为7×7×512×4096=102 760 448, FC2的权重参数量为4096×4096=16 777 216, Softmax层的权重参数量为4096×1000=4096 000. 参数量以及相应的计算量显然是非常大的, 这是因为ImageNet数据集中总共有14 197 122幅图像, 总共分为21 841个类别. 相比较宫颈细胞分类是一个非常大的数据集, 所以全连接层的神经元个数相对较多, 如果采用原始VGG16的全连接层神经元个数来实现宫颈细胞的分类, 参数量过大, 容易造成过拟合, 只对训练集产生较好的分类效果, 因此为了适应本文的应用, 需要对网络进行修改, 本文在相同学习率, 相同迭代次数的情况下, 改变全连接层神经元个数, 分别取2048, 1024, 512, 256作为全连接层参数值. 通过对比测试集准确率来选择最优的全连接层参数设置. 结果如图7所示.
|
图 7 全连接层参数选择 |
经过实验发现, 减少全连接层神经元个数不仅加快了网络的收敛速度, 并且在一定程度上提高了宫颈细胞的分类准确率. 本文通过实验对比, 发现全连接层参数为1024-256时, 分类准确率最高. 因此, 将VGG16的全连接层神经元个数改为1024-256.
Softmax层的参数取决于数据集的类别数, 原始VGG16网络是用来处理1000类的数据, 而本文主要用于宫颈细胞二分类和七分类, 故进行二分类时将Softmax层神经元个数改为2, 进行七分类时将Softmax层改为7.
由于只改变了全连接层的参数, 所以卷积层参数计算量是相等的, 所以更改后的VGG16网络参数计算量只跟全连接层相关, 如表3所示.
| 表 3 参数计算量对比 |
2.4 网络优化
为了提高模型的泛化能力, 加快收敛速度, 本文在已有的模型损失函数中加入正则化, 公式如下:
| $\theta {\rm{ = arg }}\mathop {{\rm{min}}}\limits_\theta \frac{1}{N}\sum\limits_{i = 1}^N {\left( {L\left( {\mathop {{y_i}}\limits^ \wedge ,y} \right) + \lambda R\left( \omega \right)} \right)} $ | (1) |
式中,
(1) L1正则化
L1正则化是指正则化项为模型系数w的L1范数, 如式(2)所示. 由于正则项在零点不可微, 因此权重因子趋近于零, 这就使一些对分类结果贡献较低的特征所对应的系数为0. 所以使用L1正则化可以对模型进行特征选择.
| $ {{{R}}_{{\rm{L}}1}}(\omega ) = {\left\| \omega \right\|_1} $ | (2) |
(2) L2正则化
L2正则化是指正则化项为模型系数w的L2范数, 如式(3)所示. L2正则化中模型系数为二次方, 因此与L1不同, L2使系数的取值趋于平滑. 由于引入L2正则化使的loss最小时, 模型参数也是最小的, 从而降低了模型的复杂度, 降低了模型出现过拟合的可能性.
| $ {{{R}}_{{\rm{L}}2}}(\omega ) = \left\| \omega \right\|_2^2 $ | (3) |
对于卷积神经网络对宫颈细胞分类的有效性进行评价是非常有必要的, 在分类任务中常用的评价指标有以下几项: 准确率(Accuracy, Acc), 精确率(Precision, P), 召回率(Recall, R)和F1-score. 以TP, FN, FP, TN分别表示分类过程中的值, 以二分类为例, 具体如表4所示.
| 表 4 混淆矩阵 |
准确率的定义对于给定的测试数据集, 分类器正确分类的样本数与总样本数N总之比, 即测试集中分类正确的宫颈细胞个数占总测试集的百分比.
| $ {Acc= \dfrac{{TP + TN}}{{{N_{\text{总}}}}}} $ | (4) |
精确率指的是正类分类正确的个数与分类器检索到的正类总数之比. 该值体现了分类器是否分类正确.
| $ P= \dfrac{{TP}}{{TP + FP}} $ | (5) |
召回率指的是正类分类正确的个数与实际正类别总数之比. 该值体现了分类器分类是否完全, 所以召回率也叫做查全率.
| $ R = \dfrac{{TP}}{{TP + FN}}{\rm{ }} $ | (6) |
F1-score指的是精确值和召回率的调和均值, 一般情况下, 要求精确率和召回率都要比较高, 但是实际情况中, 精确率高的时候, 召回率就低, F1值就是评价精确率和召回率的调和参数
| $ F1 - {{score}} = \dfrac{{2{{*P*R}}}}{{{{P + R}}}} $ | (7) |
在医学领域, 准确率是诊断疾病的重要评价指标, 但是相比较于准确率, 误识别率更是所要关注的, 即将异常细胞分类为正常细胞的概率.
| $ FPR= \frac{{{{FP}}}}{{{{FP + FN}}}} $ | (8) |
上述介绍了二分类过程中各个评价指标值的计算方法, 在多分类中采用混淆矩阵的表示方法来计算各个指标的值. 可以将多分类的计算看作是当前类和其他类, 从而转换成二分类进行计算. 多分类的准确率如下:
| ${p_i} = \frac{{T{P_i}}}{{T{P_i} + \displaystyle\sum\nolimits_{k = 1}^n {\left( {k \ne i} \right)} }}$ | (9) |
本文通过对HerLev数据集的预处理将图像集扩充至11 590张, 将扩充后的数据集以6:2:2的比列划分为训练集, 验证集, 测试集. 其中训练集为6954张, 验证集和测试集分别为2318. 采用VGG-16卷积神经网络卷积层对宫颈图像进行特征提取, 采用全连接层进行宫颈图像分类. 训练过程中, 设置mini-batch为32, 训练集共6954张, 故一个epoch的迭代次数iteration值至少为218次, epoch值设置为1000. 学习率Lr初始值为0.0001, 然后随着迭代次数的增加, 减小学习率. 学习率按照如下公式进行递减:
| ${{Lr}} = {0.95^{{\rm{epoch\_num}}}} \times {{l}}{{{r}}_0}$ | (10) |
式中,
对宫颈细胞图像分别进行七分类和二分类, 二分类结果如表5所示.
由表5可以看出: 二分类的准确率较高, 且正常细胞与异常细胞的准确率大致相同, 召回率异常细胞比正常细胞更高, 这说明网络对异常细胞的分类更为准确, 即有较少的异常案例被分类为正常细胞. 这也为神经网络能够进行细胞检测提供了依据, 但是在临床上, 对异常细胞的检测要更高, 异常细胞的召回率要尽量接近1, 所以卷积神经网络识别宫颈细胞目前只能作为辅助决策手段, 不能完全代替医生. 宫颈细胞的二分类混淆矩阵如图8所示.
| 表 5 宫颈细胞二分类结果 |

|
图 8 二分类混淆矩阵 |
由图8可以看出: 正常细胞中有38张误分类为异常细胞, 异常细胞中有25张图片被分类为正常细胞, 相对于测试集图片的数量, 分类错误的图像是很少的, 尤其是异常细胞的图像, 这也说明卷积神经网络在宫颈细胞分类中的优越性, 但是仍需医生进行二次筛选, 以达到误识别率最低, 所以需要对正常宫颈细胞以及异常宫颈细胞进行更细致的划分, 以达到最佳辅助决策的效果.
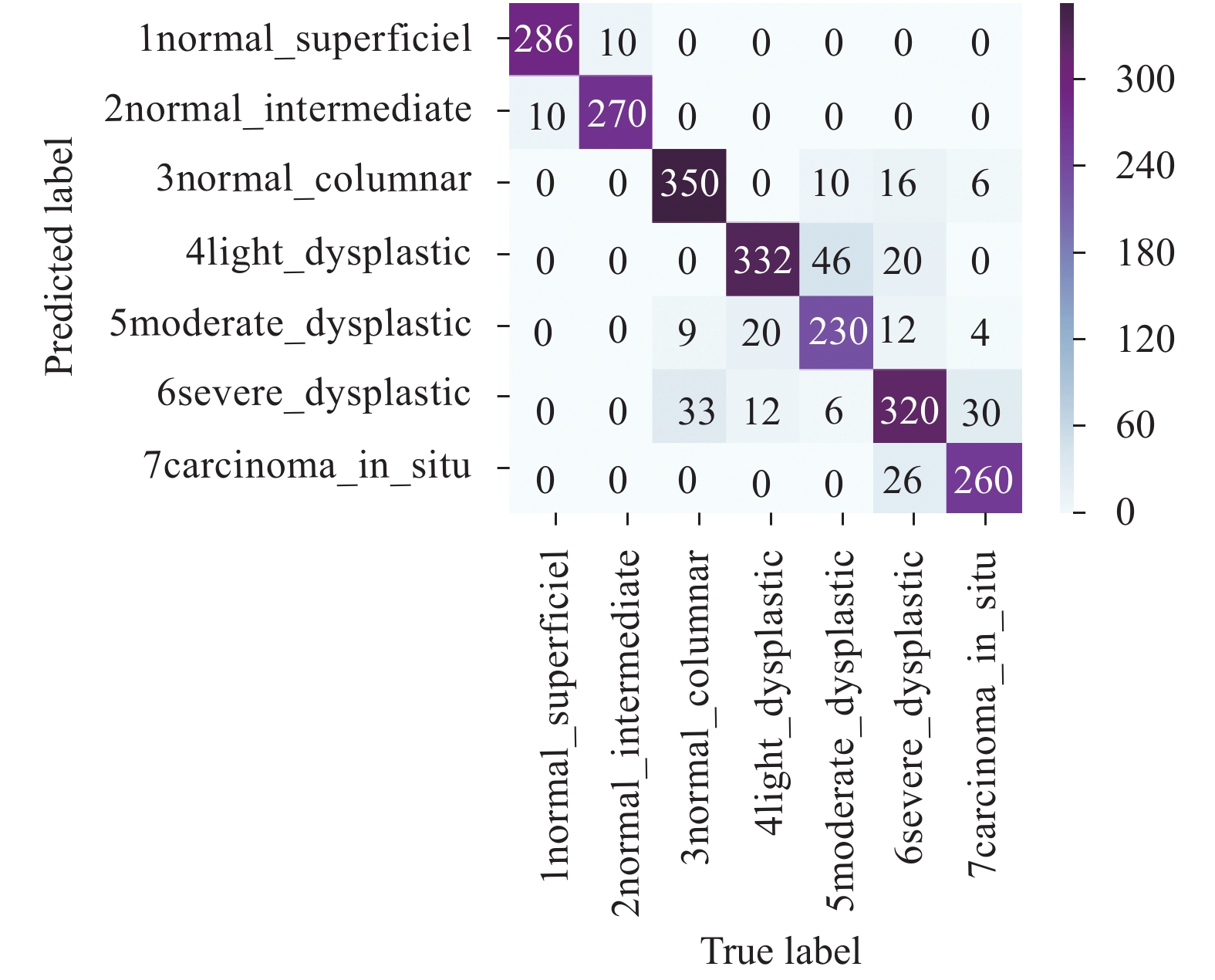
宫颈细胞八分类的混淆矩阵结果如图9所示, 通过对混淆矩阵计算得到七分类各个评价指标的结果, 如表6所示.
通过图9以及表6可以看出: 前两类的分类准确率相对其他类较高, 分别为0.966和0.964且未将这两类正常细胞错误分类为异常细胞, 两类细胞的召回率分别为0.966和0.964, 也是这7类细胞中最高的. 而第4, 5, 6类异常细胞的分类准确率较低, 分别为0.834,0.836和0.798, 但是, 错误分类多存在于相邻两类之间, 因此可以可以人工对第4类细胞进行筛选从而将异常细胞的误判率降到最低.
将本文卷积神经网络分类的方法, 同其他人工提取数据特征再通过设计分类器分类的算法相比较, 得出的结果如表7所示.
|
图 9 七分类混淆矩阵 |
| 表 6 宫颈细胞七分类结果 |
| 表 7 不同方法分类对比结果 |
从对比结果可以看出, 本文方法在细胞二分类的准确率相对较高, 高出目前最好的提升树分类器算法XGBoost, AdaBoost, Bagging等, 这些算法均为目前机器学习领域最为常用的分类算法, 在数据的分类表现优异. 这是因为采用数据特征对细胞进行分类, 其结果会受图像分割准确率的影响, 且分类特征为人工选取, 不具有代表性, 而本文通过卷积神经网络自动提取细胞图像的分类特征, 不受人工主观影响, 所以本文方法在二分类的准确率相对较高. 在七分类的准确率对比结果中可以看出: 本文方法的提升较大, 这是因为原始数据集中每个类别的数据量不均衡, 在数据分类中解决这类数据偏斜的问题, 往往需要对数据进行过采样, 欠采样或者调整预测概率的阈值, 这样模型变得复杂, 且不易得到稳定的分类模型. 本文在数据集预处理阶段, 基于细胞图像翻转不变性, 对图像进行平移翻转操作扩充了数据集, 解决了样本量不平衡的问题, 但是召回率仍相对较低, 这也是之后仍需改进的地方.
4 结论与展望本文通过卷积神经网络对宫颈细胞图像进行自动分类, 在预处理阶段通过图像裁剪, 图像集扩充解决了样本分布不平衡的问题, 并使用迁移学习初始化网络参数, 加快收敛, 最后对网络进行优化, 加入L1正则化进行特征的筛选, 简化网络, 并加入L2正则化来避免过拟合. 实验结果表明: 使用卷积神经网络对细胞图像进行分类可以得到较好的准确率, 分类准确率相比较人工提取特征分类器效果较好, 且分类结果不受分割图像准确率的影响, 模型分类效率高, 在一定程度上帮助医生进行医疗决策, 减少用人成本, 提高诊断准确率.
| [1] |
William W, Ware A, Basaza-Ejiri AH, et al. A review of image analysis and machine learning techniques for automated cervical cancer screening from pap-smear images. Computer Methods and Programs in Biomedicine, 2018, 164: 15-22. DOI:10.1016/j.cmpb.2018.05.034 |
| [2] |
Wang XW, Zheng B, Li SB, et al. Automated detection and analysis of fluorescent in situ hybridization spots depicted in digital microscopic images of pap-smear specimens. Journal of Biomedical Optics, 2009, 14(2): 021002. DOI:10.1117/1.3081545 |
| [3] |
Woolford L, Chen MZ, Dholakia K, et al. Towards automated cancer screening: Label-free classification of fixed cell samples using wavelength modulated Raman spectroscopy. Journal of Biophotonics, 2018, 11(4): e201700244. DOI:10.1002/jbio.201700244 |
| [4] |
Hyeon J, Choi HJ, Lee BD, et al. Diagnosing cervical cell images using pre-trained convolutional neural network as feature extractor. Proceedings of 2017 IEEE International Conference on Big Data and Smart Computing. Jeju, Republic of South Korea. 2017. 390–393.
|
| [5] |
Sharma B, Mangat KK. An improved nucleus segmentation for cervical cell images using FCM clustering and BPNN. Proceedings of 2016 International Conference on Advances in Computing, Communications and Informatics. Jaipur, India. 2016. 1924–1929.
|
| [6] |
Zhang L, Kong H, Liu SX, et al. Graph-based segmentation of abnormal nuclei in cervical cytology. Computerized Medical Imaging and Graphics, 2017, 56: 38-48. DOI:10.1016/j.compmedimag.2017.01.002 |
| [7] |
Pan SJ, Yang Q. A survey on transfer learning. IEEE Transactions on Knowledge and Data Engineering, 2010, 22(10): 1345-1359. DOI:10.1109/TKDE.2009.191 |
| [8] |
Chankong T, Theera-Umpon N, Auephanwiriyakul S. Automatic cervical cell segmentation and classification in pap smears. Computer Methods and Programs in Biomedicine, 2014, 113(2): 539-556. DOI:10.1016/j.cmpb.2013.12.012 |
| [9] |
Kaaviya S, Saranyadevi V, Nirmala M. PAP smear image analysis for cervical cancer detection. Proceedings of 2015 IEEE International Conference on Engineering and Technology. Coimbatore, India. 2015. 1–4.
|
| [10] |
范金坪. 宫颈细胞图像分割和识别方法研究[博士学位论文]. 广州: 暨南大学, 2010.
|
| [11] |
廖欣, 郑欣, 邹娟, 等. 基于深度卷积神经网络的宫颈细胞病理智能辅助诊断方法. 液晶与显示, 2018, 33(6): 528-537. |
| [12] |
胡卉, 蔡金清. 基于深度卷积神经网络的宫颈细胞涂片的病变细胞分类. 软件工程, 2018, 21(8): 19-22. |
| [13] |
赵越, 曾立波, 吴琼水. 卷积神经网络的宫颈细胞图像分类. 计算机辅助设计与图形学学报, 2018, 30(11): 2049-2054. |
| [14] |
夏为为, 夏哲雷. 基于卷积神经网络的宫颈癌细胞图像识别的改进算法. 中国计量大学学报, 2018, 29(4): 439-444. |
| [15] |
李文杰. 一种多分类器融合的单个宫颈细胞图像分割、特征提取和分类识别方法研究[硕士学位论文]. 桂林: 广西师范大学, 2016.
|
| [16] |
Sajeena TA, Jereesh AS. Automated cervical cancer detection through RGVF segmentation and SVM classification. Proceedings of 2015 International Conference on Computing and Network Communications. Trivandrum, India. 2015. 663–669.
|
| [17] |
Gençtav A, Aksoy S, Önder S. Unsupervised segmentation and classification of cervical cell images. Pattern Recognition, 2012, 45(12): 4151-4168. DOI:10.1016/j.patcog.2012.05.006 |
| [18] |
卢磊. 基于联合特征PCANet的宫颈细胞图像分类识别方法研究[硕士学位论文]. 桂林: 广西师范大学, 2017.
|
| [19] |
鲁武警. 基于Snake分割和SVM的宫颈细胞识别研究[硕士学位论文]. 济南: 山东大学, 2015.
|
| [20] |
郭磊. 基于卷积神经网络的宫颈细胞病变图像识别研究[硕士学位论文]. 桂林: 广西师范大学, 2017.
|
| [21] |
余宽. 基于机器学习的病理细胞辅助检测方法研究[硕士学位论文]. 成都: 电子科技大学, 2017.
|
| [22] |
Zhao LL, Li K, Wang M, et al. Automatic cytoplasm and nuclei segmentation for color cervical smear image using an efficient gap-search MRF. Computers in Biology and Medicine, 2016, 71: 46-56. DOI:10.1016/j.compbiomed.2016.01.025 |
| [23] |
Tareef A, Song Y, Cai WD, et al. Automatic segmentation of overlapping cervical smear cells based on local distinctive features and guided shape deformation. Neurocomputing, 2017, 221: 94-107. DOI:10.1016/j.neucom.2016.09.070 |
| [24] |
Bora K, Chowdhury M, Mahanta LB, et al. Automated classification of pap smear images to detect cervical dysplasia. Computer Methods and Programs in Biomedicine, 2017, 138: 31-47. DOI:10.1016/j.cmpb.2016.10.001 |
| [25] |
Rahmadwati, Naghdy G, Ros M, et al. Cervical cancer classification using Gabor filters. Proceedings of the 2011 IEEE First International Conference on Healthcare Informatics, Imaging and Systems Biology. San Jose, CA, USA. 2011. 48–52.
|
| [26] |
Teeyapan K, Theera-Umpon N, Auephanwiriyakul S. Application of support vector based methods for cervical cancer cell classification. Proceedings of 2016 IEEE International Conference on Control System, Computing and Engineering. George Town, Malaysia. 2016. 514–519.
|
| [27] |
Zhang L, Lu L, Nogues I, et al. DeepPap: Deep convolutional networks for cervical cell classification. IEEE Journal of Biomedical and Health Informatics, 2017, 21(6): 1633-1643. DOI:10.1109/JBHI.2017.2705583 |
| [28] |
Garcia-Gonzalez D, Garcia-Silvente M, Aguirre E. A Multiscale Algorithm for Nuclei Extraction in Pap Smear Images. Pergamon Press, 2016.
|
| [29] |
Devi MA, Ravi S, Vaishnavi J, et al. Classification of cervical cancer using artificial neural networks. Procedia Computer Science, 2016, 89: 465-472. DOI:10.1016/j.procs.2016.06.105 |
| [30] |
Phoulady HA, Zhou M, Goldgof DB, et al. Automatic quantification and classification of cervical cancer via Adaptive Nucleus Shape Modeling. Proceedings of 2016 IEEE International Conference on Image Processing. Phoenix, AZ, USA. 2016. 2658–2662.
|
 2020, Vol. 29
2020, Vol. 29



