伴随互联网和大数据技术的发展, 很多患者在感到身体不适时, 首先会到医疗问答网站上提问和查询疾病相关的问题, 同时许多医生也会到医疗问答网站去回答患者的疑问, 这类网站已经成为联系患者和医生之间的枢纽. 在线医疗问答社区的发展使得我们获取医学知识的渠道多样化, 有助于患者了解自己的健康状况, 同时也有助于健康医学知识的普及. 国内如39健康网, 寻医问药, 春雨医生等网站, 不仅提供基础的疾病知识和医学知识, 而且每天还积累了大量的问答数据, 这些医疗文本数据中包含大量有意义的信息, 如寻医问药从2004年开始一直到现在, 已经积累了大量真实的信息, 并且每天都在产生数万条的问答数据. 这些医疗文本数据将汇聚成非常客观的大数据, 数据中包含有大量的真实案例和医生的诊疗建议. 在这些数据中蕴含着比较丰富的医疗价值. 但这些数据是非结构化的状态, 无法进行更深的数据挖掘, 实现数据的充分利用. 为了更好的利用这类数据, 抽取和挖掘更有价值的医疗信息, 需要把非结构化的数据进行结构化, 而命名实体识别是结构化文本中的第一步, 而且该类文本的命名实体识别可以为医疗问答的研究和应用打好基础. 本文利用医疗问答网站积累的数据, 进行了命名实体识别的研究.
2 命名实体识别相关研究实体是文本的基本信息元素, 是构成文本的基础. 命名实体识别(Named Entity Recognition, NER)是自然语言处理的一项基本任务, 主要是从一段文本中找出实体, 并对实体出现的位置和类别进行标记. NER概念的提出是在MUC-6 (Message Understanding Conference)会议上[1], 最初的提出是作为信息提取的重要任务之一. 通用的命名实体识别任务, 主要是在一段文本中识别出人名, 地名, 专业机构, 时间和数字(货币, 百分数)等. 在特定的领域, 可以用来识别特殊领域的实体如医疗领域和金融领域等. 命名实体识别技术包括许多不同的方法: 基于词典和规则的方法; 基于统计学习的方法; 还有将二者混合的方法. 常见的统计学习的方法有支持向量机(SVM)、最大熵模型、贝叶斯分类等, 这些方法把NER任务看成分类问题. 此外, 还有隐马尔可夫模型(HMM)和条件随机场(CRF), 这类模型把NER任务当做序列标注问题处理. 随着深度学习的发展, 最近几年出现了大量的基于神经网络的模型, 并取得了较好的效果, 最具代表性的是BiLSTM-CRF模型[2], 该模型在各个公共数据集上均取得了不错的效果. 在RNN的输出层连接CRF层, 这种结构已经成为命名实体识别模型的常用结构. 目前对于医疗文本命名实体识别的研究主要集中在电子病历, 医学文献, 医学书籍等, 而互联网医疗问答社区文本的研究并不多, 国内最近几年也有研究者开始关注这方面的研究, 比如苏娅等[3]使用CRF在自建数据集上进行研究, 抽取的目标实体共5类, 分别包括疾病、症状、药品、治疗方法和检查, 通过采用逐一添加特征的方式训练模型, 模型精确率达到81.26%, 召回率60.18%. 张帆等人[4]设计深度神经网络应用到在线医疗文本实体识别上, 抽取的目标实体也是5类. 深度神经网络模型同CRF等方法相比减少了很多人工特征, 并且提高了精确率和召回率.
3 算法模型设计本文以BiLSTM-CRF作为基准模型, 设计了两种不同的命名实体识别模型, 并且在自构建的数据集上进行验证, 均取得了不错的效果.
3.1 BiLSTM-CRF模型双向循环神经网络(BiLSTM)由两个单向的循环神经网络构成, 两个网络中一个随时间正向, 另一个随时间逆向, 逆向网络的实现本质上把输入序列进行逆转, 然后输入到正向网络中. BiLSTM的优势是可以在当前节点获取正反两个方向的特征信息, 即能捕捉到未来信息的特征, 也能捕捉到过去信息的特征. 但是, 两个方向的循环神经网络并不会共享一个隐状态, 正向LSTM的隐状态传给正向的LSTM, 逆向LSTM的隐状态传给逆向的LSTM, 两个方向的循环神经网络之间没有连接, 两个输出会共同连接到输出节点合成最终输出. 方向不同的两个循环神经网络, 都可以展开成为普通的前馈网络, 使用反向传播算法(BPTT)进行训练. 双向循环神经网络被用在许多序列标注任务上.
条件随机场(CRF)模型是一种概率无向图模型, 可以解决序列标注任务, 命名实体识别可以看做是序列标注任务, 即给定观察序列X={x1, x2, …, xn}的条件下, 求Y的概率. 随机变量Y={y1, y2, …, yn}, Y是隐状态序列. 数学表达式为P(Y|X). 在命名实体识别上使用的CRF主要是CRF线性链, CRF建模的数学公式如式(1)和(2).
| $P(y|x) = \frac{1}{{Z(x)}}\exp (\sum\nolimits_{k = 1}^K {w_k} f_k(y, x))$ | (1) |
| $Z(x) = \sum\limits_y {\exp \sum\nolimits_{k = 1}^K {w_kf_k(y, x)} } $ | (2) |
上式中fk是特征函数, wk是特征函数的权重, Z(x)是归一化因子. 条件随机场可以看成是定义在序列上的对数线性模型, 能够使用极大似然估计方法求参数. 目前已经有了一些优化算法进行该问题的求解, 比如梯度下降法, 改进的迭代尺度法和拟牛顿法等. 模型在进行解码时可以利用维特比算法, 这是一种动态规划算法, 在给定观察序列的条件下, 求出最大的标记序列的概率. BiLSTM与CRF的结合, 本质上是把BiLSTM的输出作为CRF的输入, BiLSTM层输出的是每一个标签的预测分值, 这些分值会输入到CRF层. 其过程可描述为利用BiLSTM解决提取序列特征, 再使用CRF利用句子级别的标记信息进行训练, 单独使用BiLSTM也可以完成命名实体识别, 可以从BiLSTM的输出中挑选最大值对应的标签, 作为该单元的标签, 但是这不能保证每次预测的标签都是合法的, 比如对于{B, I, O}体系的标注, 标签序列是“I-Organization I-Person”和“B-Organization I-Person”, 很显然这是错误的. 如果在BiLSTM的输出层接入CRF层后, 相当于对最后的预测标签加入了约束, 保证输出的标签是合法的, 这些约束会在训练的过程学习到, 对于BiLSTM-CRF模型的学习方法同样可以使用极大似然估计方法.
可以把双向循环神经网络的输出看成打分矩阵, 称为P矩阵. 对于输入语句X=(x1, x2, x3, …, xn), P是一个n×k的矩阵, k是输出标注y的个数, Pi, j表示句子中第i个词被标记为第j个标签的概率. 句子的预测标注序列可以表示为: y=(y1, y2, y3, …, yn). 定义y矩阵的打分函数的计算式(3).
| $score(X, y) = \sum {\sum\nolimits_{i = 0}^n {A_{y_i, y_{i + 1}} + \sum\nolimits_{i = 1}^n {P_{i, y_i}} } } $ | (3) |
Ai, j是看成转移打分矩阵, 代表从标注i转移到标注j的得分. y0和yn分别代表句子开始和结束的标签, 标注矩阵A是一个k+2阶的方阵. 通过式(4)计算y在给定x下的条件概率p(y|x), 其中YX代表对于给定的句子X所有可能的标签序列, 损失函数可以定义为式(5), 并在训练的过程中极大化正确标签序列概率的对数值.
| $P(y|X) = \frac{{\exp (score(X, y))}}{{\sum\nolimits_{\tilde y \in YX} {\exp (X, \tilde y )} }}$ | (4) |
| $L = \log (P(y|X))$ | (5) |
在模型训练完成后可以通过式(6)进行模型预测, 其中y*是集合中使得得分函数score取最大值的序列.
| $y* = \mathop {\arg \max }\limits_{\tilde y \in YX} score(X, \tilde y )$ | (6) |
以上是BiLSTM-CRF的基本原理, 本文在设计的BiLSTM-CRF模型结构图如图1. 首先将输入语句经过一个embedding层, 之后连接到BiLSTM层, 在BiLSTM层后连接映射层, 并进行逻辑回归, 该层的输出会输入到下一层CRF层. 为了提高模型的泛化能力, 在embedding层和BiLSTM层之间加入了droupout层.
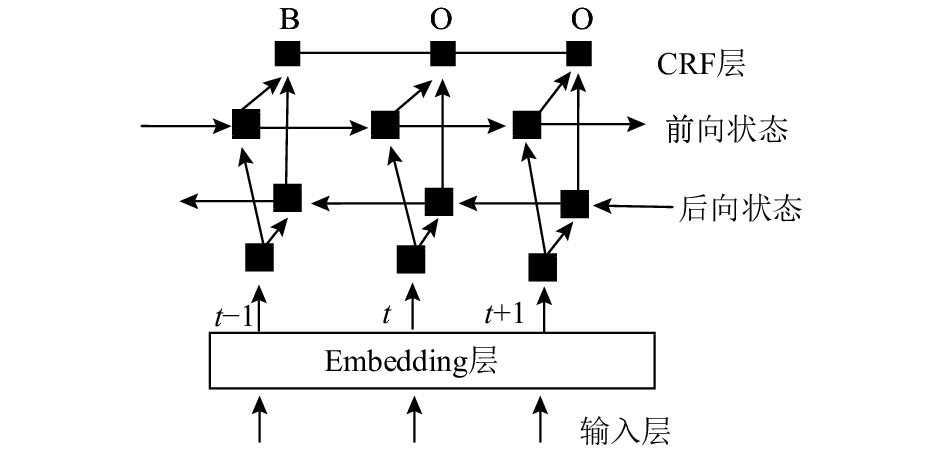
|
图 1 BiLSTM-CRF模型结构图 |
3.2 IndRNN-CRF模型
独立循环神经网络(Independently Recurrent Neural Network, IndRNN)[5]由Li S等人提出, 同传统的RNN不同的是IndRNN的神经元之间是独立的. 传统的RNN的隐藏层数学公式如式(7), 其中W是N×M的矩阵, U是N×N的矩阵, N是RNN中的神经元节点数.
| $h_t = \sigma (Wx_t + Uh_t - 1 + b)$ | (7) |
在传统的RNN中每个神经元都和上一时刻的全部神经元发生联系(U的行向量与ht–1向量的乘积, ht–1是t–1时刻的隐状态), 也就是神经元之间是不独立的. 而IndRNN结构神经元之间的连接仅发生在层与层之间, IndRNN的数学表达式可以在上面式(7)进行改造后得到如式(8). 其中U和ht–1是点积, 此时的U不是矩阵, 而是一个N维的向量, t时刻的每个神经元只和t–1时刻自身相联系, 与其他的神经元无关. 这也是独立循环神经网络名称的由来. 为了在神经元之间发生联系, 至少需要进行两层的堆叠.
| $h_t = \sigma (Wx_t + U \otimes h_{t - 1} + b)$ | (8) |
模型中的第n个神经元的隐藏状态hn,t, 可由式(9)计算得出, 其中wn和un分别是输入权重和t–1到t时刻的连接权重的第n行, 每个神经元只接受前一步它自己的隐藏状态和输入传来的信息. 这与传统的RNN是不同的, 这种结构提供了一种循环神经网络的新视角, 随着时间的推移(通过u), 独立的聚集空间模式(通过w), 不同神经元的相关性可以通过多层堆叠来实现, 下一层的神经元处理上一层所有神经元的输出. 模型同样采用梯度后向传播算法进行优化, IndRNN进一步缓解了随时间累积的梯度爆炸或消失的问题, 梯度可以在不同的时间步上有效的传播, 可以使得网络叠加更深.
| $h_{n, t} = \sigma (w_nx_t + u_nh_{n, t - 1} + b_n)$ | (9) |
在本文中, 将BiLSTM-CRF模型中的BiLSTM换成多层的IndRNN, 提出了一种新的模型Multi-IndRNN-CRF, 本文中IndRNN有4层, 之后拼接CRF, 模型示意图如图2, 在Embedding层后输出的数据, 会进行dropout, 每层IndRNN输出后都会进行BatchNormalization防止数据发生严重偏移, 同时防止梯度爆炸. IndRNN-CRF的损失函数和LSTM-CRF模型一样, 参数的学习方法依然是极大似然估计.

|
图 2 4-IndRNN-CRF结构图 |
3.3 IDCNN-BiLSTM-CRF模型
膨胀卷积(Dilated Convolution, 简称DCNN)是Yu F, Koltun V在2015年提出的[6], 经典卷积的filter, 作用在矩阵的一片连续区域上做滑动, 而膨胀卷积是在filter中增加了膨胀宽度, 在输入矩阵上做滑动时会跳过膨胀宽度中间的数据. filer矩阵大小不变, filter最终获取到了更广的输入矩阵的数据. DCNN的示意图如图3.
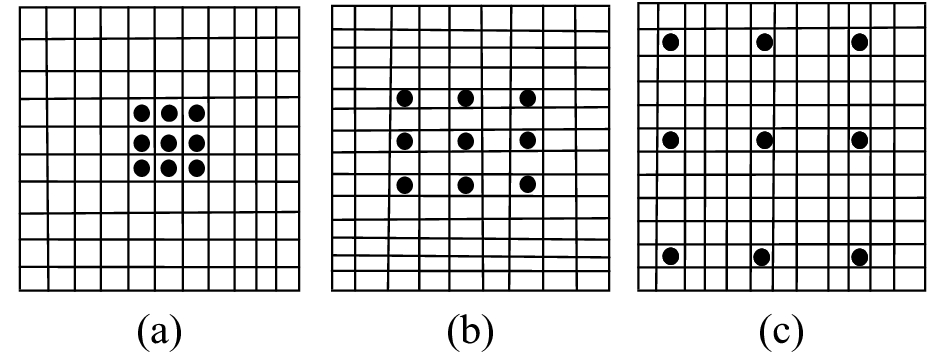
|
图 3 DCNN示意图 |
图3中的(a)图对应3×3的1-dilated convolution, 同经典的卷积操作一样; (b)图对应3×3的2-dilated convolution, 卷积核的大小仍然是3×3, 空洞大小是1, 可以理解成卷积核的大小是7×7, receptive filed是7×7; (c)图是4-dilated convolution操作, receptive filed是15×15的感受野.
Strubell E等人[7]提出了IDCNN(Iterated Dilated Convolution, IDCNN)模型, 用在实体识别任务上取得了不错的效果. 膨胀的宽度随着层数的增加呈现为指数增加, 但参数的数量是线性增加的, 这样接受域很快就覆盖到了全部的输入数据. 模型是4个大小相同的膨胀卷积块叠加在一起, 每个膨胀卷积块里的膨胀宽度分别为1, 1, 2的三层膨胀卷积. 把句子输入到IDCNN模型中, 经过卷积层, 提取特征, 其基本框架同BiLSTM-CER一样, 由IDCNN模型的输出经过映射层连接到CRF层.
尽管IDCNN模型可以使得接受域扩大, 但不会像双向循环神经网络, 可以从序列的整体提取正向和反向特征, 但循环神经网络不能很好的兼顾到局部特征, 本文提出模型IDCNN-BiLSTM-CRF既能兼顾全局特征(通过BiLSTM), 又能兼顾局部特征(通过IDCNN).
模型的基本结构描述如下: 首先输入语句经过embedding层, 输出字向量, 字向量并行输入到IDCNN模型和BiLSTM模型, 经过两个模型后, 将输出的向量进行拼接后形成向量特征, 然后经过映射层后输入到CRF层. IDCNN在提取局部特征的同时能够兼顾到部分全局特征, 但不会像BiLSTM能够很好的提取全局特征, 因此将IDCNN输出的向量特征作为对局部特征的弥补, 拼接在BiLSTM的向量特征上, 模型示意图如图4. 其中Dilated CNN block中有三个卷积层, 没有池化层, 第一层为1-dilated convolution, 第二层为1-dilated convolution, 第三层为2-dilated convolution. 将4个block(对应图中的DCNN-i)堆叠, 当有数据输入后. 先经过embedding层, 然后输入到DCNN-1, 从DCNN-1的输出有两个去向, 一是与其他DCNN的输出拼接后形成最终的特征向量, 另一个输出变成DCNN-2的输入, 依次类推. IDCNN模型的输出和BiLSTM模型的输出进行拼接后, 经过一个映射层, 再将值输入到CRF中, 模型图见图4. 模型的训练以及参数的学习方法同BiLSTM-CRF.
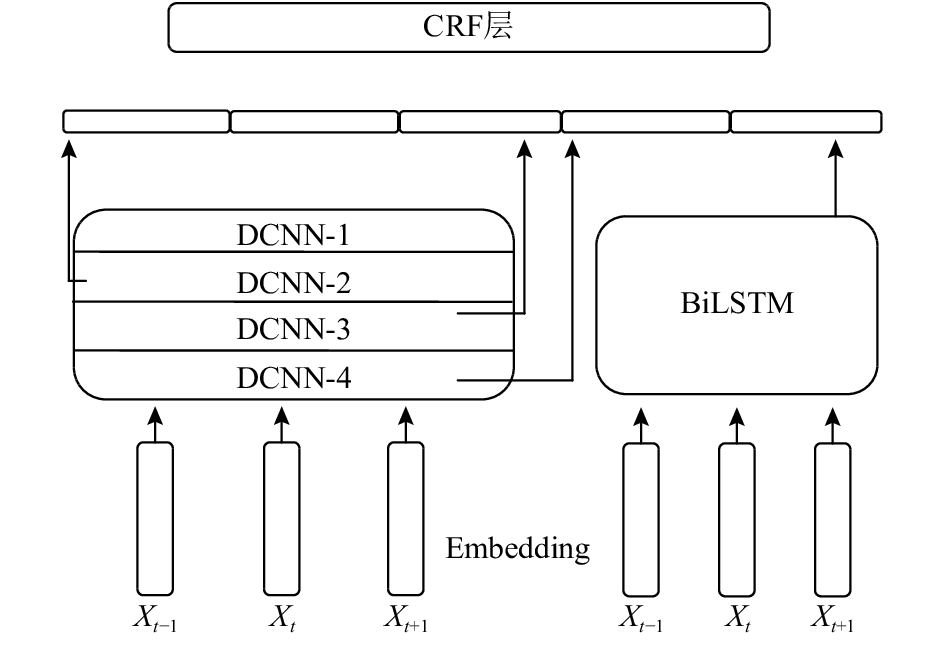
|
图 4 IDCNN-BiLSTM-CRF模型结构图 |
4 数据处理和标注
使用Scrapy框架编写爬虫, 从医疗问答网站爬取数据. 爬取的网站分别是“寻医问药”, “39健康网”, “快速问医生”等网站. 在各个网站的问答板块收集咨询者的问题, 总计收集数据约1200万条, 大约2.27 GB. 从收集的数据中挑选8027条数据作为训练集, 1972条作为测试集. 采用{B, I, O}标注体系, 对医疗文本进行人工标注, 具体格式为B-X, I-X和O. B代表实体开始, I代表实体中间或结束部分, O代表非实体. 标注的实体类别参考杨锦峰等人[8]在论文中提出的方案, 分为四类实体: 疾病、症状、检查和治疗. X代表命名实体的类别, 分别为DISEASE、SYMPTOM、TREATMENT、CHECK四个不同的标识, 代表疾病、症状、治疗和检查. 该任务的标记一共有9 (=4×2+1)类标签, 在标注时把药物归结到治疗实体. 表1给出了标注的示例.
| 表 1 命名实体类别 |
实际标注的格式如下: 对于语句“我有点发烧, 浑身无力, 是感冒了吗?”, 标注为{O, O, O, B-SYMPTOM, I-SYMPTOM, O, B-SYMPTOM, I-SYMPTOM, I-SYMPTOM, I-SYMPTOM, O, O, B-DISEASE, I-DISEASE, O, O, O}, 其中语句中包含的标点符号作为非实体, 标注为“O”.
5 实验结果和分析 5.1 实验条件本文实验是在Linux平台下使用Python 3.5语言在tensorflow框架进行开发, 硬件环境如下: Intel i7的cpu, 16 GB内存以及NVIDIA GTX-1070显卡.
使用预训练的字向量对embedding层进行初始化, 预训练字向量的过程如下: 首先将下载的1200万条问句, 按照字级别进行字向量的训练. 训练的模型使用的是开源工具Word2vec, 该工具是Toms Mikolov在2013年开发的工具包, Word2vec使用CBOW模型[9–11](连续词袋模型). 对于word2vec参数的设定如下: 字向量的维度设置为200, 窗口大小为5, 训练次数为20, 其余参数默认.
5.2 模型参数设置对于BiLSTM-CRF模型参数的设定: BiLSTM的隐层节点为300, 模型中的droupout层参数设置为0.5, 采用Adam优化算法, 学习率设置为0.001, batch size的大小为64, epoch的大小为100.
对于IndRNN-CRF模型参数的设定: IndRNN的隐层节点为300, 共有4层IndRNN, 模型中的droupout层参数设置为0.5, 采用Adam优化算法, 学习率设置为0.001, batch size的大小为64, epoch的大小为100.
对于IDCNN-BiLSTM-CRF模型参数的设定: BiLSTM的隐层节点为300, IDCNN的filter个数为100, 模型中的droupout层参数设置为0.5, 采用Adam优化算法, 学习率设置为0.001, batch size的大小为64, epoch的大小为80.
5.3 实验结果和分析实验结果的评价指标有3个, 分别为精确率, 召回率和F值. 计算公式如式(10), (11), (12).
| $Precision(P)=\frac {\text{系统正确识别的实体个数}}{\text{系统识别的实体个数}}$ | (10) |
| ${ {Re} } call(R)=\frac {\text{系统正确识别的实体个数}}{\text{文档中的实体个数}}$ | (11) |
| $F - measure = \frac{{2 \times P \times R}}{{P + R}}$ | (12) |
| 表 2 BiLSTM-CRF的实验结果 |
对比实验结果可以看出, IndRNN-CRF模型在精确率上比基准模型BiLSTM-CRF高, 召回率的值为0.6848, 相比于模型BiLSTM-CRF的召回率比较低. IDCNN-BiLSTM-CRF模型在精确率, 召回率和F1值上均超过了基准模型BiLSTM-CRF. 图5, 图6和图7分别是模型BiLSTM-CRF, IndRNN-CRF和IDCNN-BiLSTM-CRF的Loss曲线图, 纵坐标代表Loss值, 横坐标代表的是迭代次数. 从图中可以看出在经过了24 000次的迭代后模型BiLSTM-CRF的Loss值大于2.0, 模型IndRNN-CRF和IDCNN-BiLSTM-CRF的loss值小于2.0, 其中模型IndRNN-CRF的loss值最低.
| 表 3 IndRNN-CRF的实验结果 |
| 表 4 IDCNN-BILSTM-CRF的实验结果 |
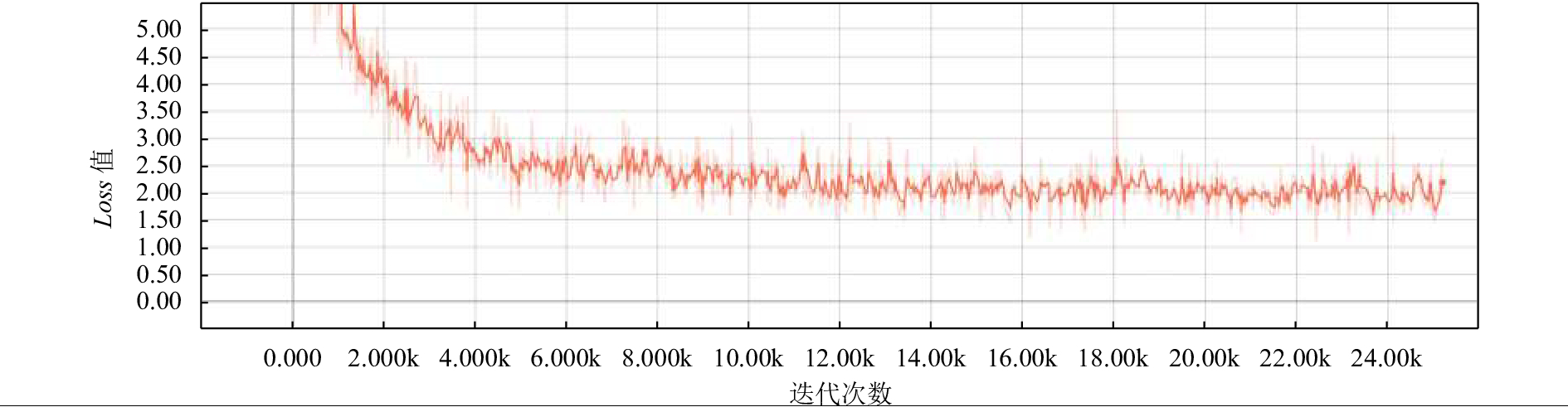
|
图 5 BiLSTM-CRF的loss-step曲线图 |
由于IDCNN-BiLSTM-CRF模型的总体性能最好, 可以用在互联网在线问诊医疗文本的实体识别上, 该模型也可用在医学文献, 电子病历等文本的命名实体识别上. 模型IndRNN可以用在对精确率要求较高, 但对召回率要求不高的任务中.
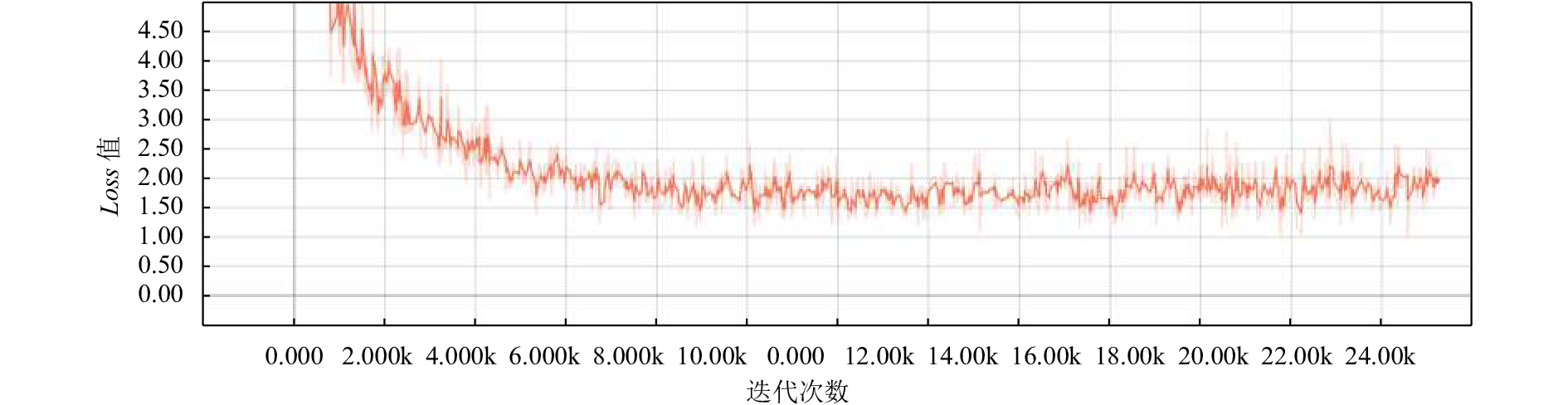
|
图 6 IndRNN-CRF的loss-step曲线图 |
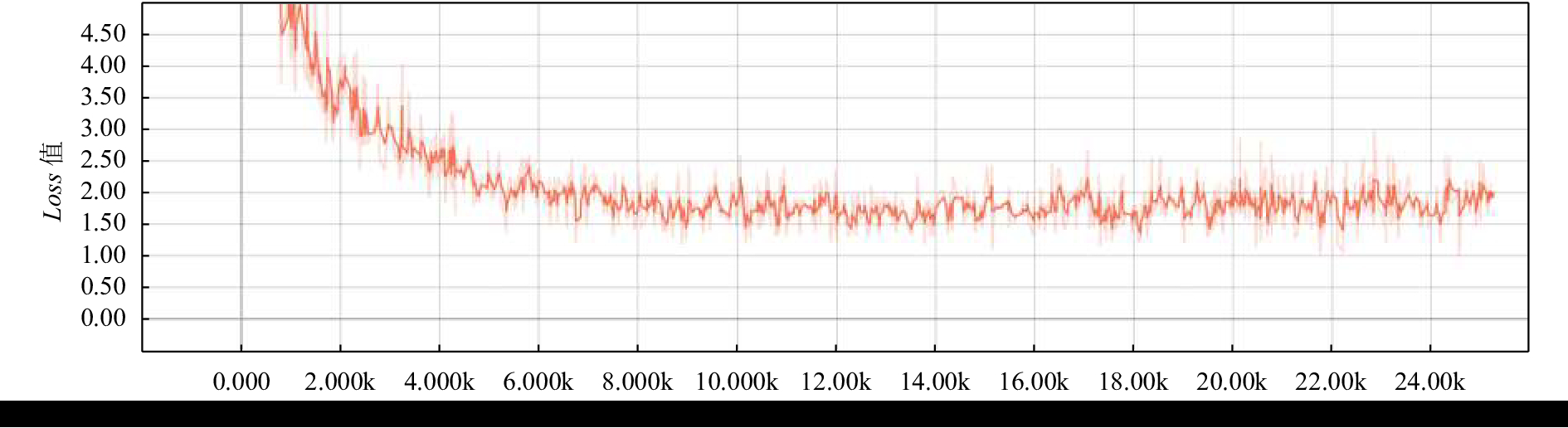
|
图 7 IDCNN-BILSTM-CRF的loss-step曲线 |
6 结论与展望
本文针对在线问诊医疗文本, 利用深度学习技术设计了两种不同的神经网络模型, 进行医疗文本命名实体识别的研究, 共识别4类医疗实体: 疾病, 症状, 治疗和检查. 对基于字级别的命名实体识别任务, 在模型IDCNN-BiLSTM-CRF中使用卷积神经网络和循环神经网络提取特征向量, 并将两个特征向量拼接, 形成既包含全局特征又包含局部特征的向量, 该向量经过映射层后输入到CRF层中, 实验结果表明该模型的整体性能最好. 但是由于医疗领域的特殊性, 仍然需要继续提高医疗实体的识别率, 获取更精确的挖掘结果. 在接下来的工作中, 可以考虑先对医疗文本分词, 然后加入词性或者拼音等特征训练模型, 提高识别率. 此外, 对于医疗文本还要考虑文本中是否含有修饰性实体, 比如表示时间和否定的词汇等, 如“无头痛”, 症状“头痛”前的“无”就是修饰实体. 模型最终结果与参数的调试也有较大的关系, 设置不同的参数, 模型的输出值可能会不同.
| [1] |
Sundheim BM. Named entity task definition, version 2.1. Proceedings of the Sixth Message Understanding Conference. Columbia, MA, USA. 1995. 319–332.
|
| [2] |
Huang ZH, Xu W, Yu K. Bidirectional LSTM-CRF models for sequence tagging. arXiv preprint arXiv: 1508.01991, 2015.
|
| [3] |
苏娅, 刘杰, 黄亚楼. 在线医疗文本中的实体识别研究. 北京大学学报(自然科学版), 2016, 52(1): 1-9. |
| [4] |
张帆, 王敏. 基于深度学习的医疗命名实体识别. 计算技术与自动化, 2017, 36(1): 123-127. DOI:10.3969/j.issn.1003-6199.2017.01.025 |
| [5] |
Li S, Li WQ, Cook C, et al. Independently recurrent neural network (IndRNN): Building a longer and deeper RNN. arXiv preprint arXiv: 1803.04831, 2018.
|
| [6] |
Yu F, Koltun V. Multi-scale context aggregation by dilated convolutions. arXiv preprint arXiv: 1511.07122, 2016.
|
| [7] |
Strubell E, Verga P, Belanger D, et al. Fast and accurate entity recognition with iterated dilated convolutions. Proceedings of 2017 Conference on Empirical Methods in Natural Language Processing. Copenhagen, Denmark. 2017. 2670–2680.
|
| [8] |
杨锦锋, 于秋滨, 关毅, 等. 电子病历命名实体识别和实体关系抽取研究综述. 自动化学报, 2014, 40(8): 1537-1562. |
| [9] |
Mikolov T, Chen K, Corrado G, et al. Efficient estimation of word representations in vector space. ICLR: Proceeding of the International Conference on Learning Representations Workshop Track. AZ, USA. 2013. 1301–3781.
|
| [10] |
Mikolov T, Sutskever I, Chen K, et al. Distributed representations of words and phrases and their compositionality. Proceedings of the 26th International Conference on Neural Information Processing Systems. Lake Tahoe, NV, USA. 2013. 3111–3119.
|
| [11] |
Kenter T, Borisov A, de Rijke M. Siamese CBOW: Optimizing word embeddings for sentence representations. Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Berlin, Germany. 2016. 941–951.
|
 2019, Vol. 28
2019, Vol. 28



