在医学影像领域, 虽然CT技术越来越普及, 但是X光片凭借着放射剂量小, 成本低等优势在学生以及职工体检中仍然占有较大比重. 但是由于X光的2D特性, 导致肋骨与器官影像重叠, 如果疾病位置在这些区域, 会给医生[1]以及针对X光的肺结节检测系统[2]带来干扰.
一直以来, 基于胸部X光图片的肺结节智能检测任务就是智能医疗领域的研究热点, 而肺结节由于其尺寸较小, 位置如果与肋骨区域重合, 会导致结节在图片中难以被发现, 因此肋骨抑制往往是胸部X光图片预处理的必要过程.
X光片中, 双能减影[3](Dual Energy Subtraction, DES)技术, 可以解决数字X光的肋骨影响. 它结合两个不同能量等级的X射线, 然后相减来突出器官组织, 抑制骨骼结构成像. 但是只有较少医院配置了这项技术, 因为它不仅需要专业的设备, 而且与普通的X光相比, DES有着更大的放射剂量, 提高成本的同时还影响患者身体健康. 在图像预处理阶段用软件算法来实现X光图片的肋骨抑制, 可以在无成本增加的情况下辅助医生和肺结节检测系统工作.
如图1(a)所示, 肺结节与肋骨有一定程度的重合, 因此会对检测造成干扰, 而经过肋骨抑制操作之后, 结节特征更为显著.
本文第1部分阐述肋骨抑制的相关研究, 并简单介绍本文所采用的网络模型; 第2部分详细介绍网络结构以及损失函数; 文章第3部分是实验对比与分析, 验证肋骨抑制预处理的有效性; 第4部分对全文进行总结, 并展望下一步工作.
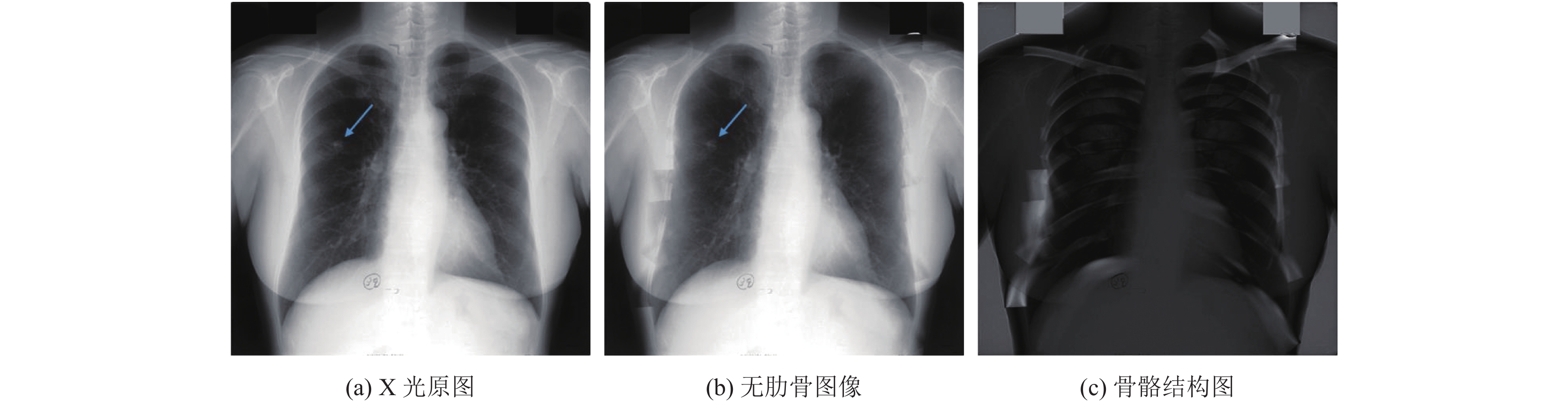
|
图 1 样本示例 |
1 概述 1.1 相关研究
目前胸片中的肋骨影像抑制算法主要分为两类: 基于监督学习的算法和基于无监督学习的算法.
基于监督学习的算法需要DES胸片产生标签, 模型一般为一个回归网络, 通过监督学习, 让模型的输出逼近标签结果. 鉴于肋骨结构较于器官组织更加简单, 如图1(c)所示, 一些基于监督学习的算法会优先选择训练模型学习肋骨结构. 标签通过原图与DES胸片生成, 在测试时, 将原图减去生成的骨骼结构图, 得到最终的无肋骨图像. Chen等人[4]将肺部区域分成多个区域, 分别训练人工神经网络学习对应区域的骨骼结构, 将它们拼接成整幅骨骼图片, 最后将骨骼影像从原图中分离. Yang等人提出CamsNet[5], 使用了多尺度的方法在梯度域上训练原图与肋骨图的映射关系, 然后再重建出肋骨图像, 最后生成无肋骨胸片. 除此之外, Gusarev等人[6]提出将肋骨作为图像噪声, 采用编码器加解码器的网络结构, 将DES图像直接作为训练标签, 能够直接生成无肋骨图像, 将MSE (Mean Squared Error)与MS-SSIM[7](Multi-Scale Structural Similarity Index)共同作为损失函数, 兼顾像素差和结构相似性, 取得了良好的效果.
而基于无监督学习的方法则不需要DES图像作为标签, 但是这些方法都需要分割或者定位肋骨位置, 然后再重建那片区域的像素值. Hogeweg等人[8]提出使用主成分分析(Principle Component Analysis, PCA)的方法来建模肋骨的横截面的形状分布, 然后再将肋骨图像从原图中剔除. Li等人[9]先用传统图像处理结合基于图的方法定位肋骨, 然后同样使用PCA对肋骨进行建模, 最后由原图和肋骨模型重建出无肋骨胸片. 基于无监督学习的方法虽然不需要DES胸片作为标签进行训练, 但是对分割肋骨区域或者定位肋骨边界的精确度有着较高要求.
相较而言, 基于监督学习的方法能够让网络自动学习原图与骨骼图像或者DES胸片直接的映射关系, 在算法设计上更为简单, 而且不依赖肋骨的定位或者分割结果, 缺点是比较难以获得大量的DES胸片数据, 但是数据扩充可以一定程度上解决此问题.
本文采用的是监督学习的方法, 前文提到对肋骨结构建模要易于直接学习无肋骨的胸片图像, 其实在去噪卷积网络中也有这种类似的操作, 如DnCNN[10](Denoising Convolutional Neural Network)中所采用的残差训练策略, 这种方式能够让网络更加容易训练, 加速模型收敛. 与传统去噪任务不同的是, 肋骨成像和常见的高斯噪声区别较大, 具有更多的语义信息. 在医学影像的语义分割任务中, U-net[11]网络结构被广泛使用, 它由语义分割网络FCN[12]改进而来, 采用对称的结构, 将图像到高语义特征的过程看作编码器, 然后将高语义特征到像素级的分类结果视为解码器, 同时在解码器中使用跳跃连接能够将浅层特征与深层特征相结合, 在提取语义信息的同时, 浅层特征还能够保证图像细节. 本文将这两种策略相结合, 通过实验验证了同时采用这两种策略能够使模型取得更优的效果.
1.2 本文工作肋骨抑制的难点在于抑制肋骨影像的同时还要保证原图的细节纹理. 在本文中, 我们采用卷积神经网络作为基础模型, 网络结构类似U-net, 分别验证跳跃连接、残差学习以及增加卷积核数量对模型性能的影响. 除此之外, 依据Zhao等人的实验结果[13], 将MAE (Mean Absolute Error)和MS-SSIM结合共同作为损失函数时能够兼顾像素误差以及图像的结构相似性, 使模型表现更加优异. 本文采用了与其研究结果相同的损失函数以及二者的权重系数.
由于与相关研究的数据集不同, 无法直接通过评估指标进行对比. 因此为了验证模型的有效性, 我们分别将原X光图像与经过肋骨抑制处理后的X光图像分别用目标检测网络RetinaNet[14]进行训练并测试, 实验结果证明使用经过肋骨抑制处理后的胸片进行训练和测试, 能够提升肺结节检测系统的性能表现.
2 算法设计与实现 2.1 神经网络结构由于肋骨抑制操作只是肺结节检测系统的预处理步骤, 而且大多数医院没有GPU集群, 所以我们的目标是让程序能够在单个GPU机器上运行, 为了降低特征层占用的显卡内存, 因此设计了只有6层的CNN模型, 如图2所示, 其中Feature为特征图, 代表网络模型的中间结果, (n1, n2, n3)表示卷积核的数量, 特征图的通道数. 网络结构类似编码器加解码器的框架, 前3层卷积层后都有池化操作进行下采样, 下采样能够提升模型的感受野, 在此过程中特征由图像特征逐渐抽象具有更多的语义信息, 同时降低特征图的尺寸节省显存空间. 虽然空洞卷积操作能够在扩大感受野的同时更多的保留位置细节信息, 但是所有的特征层都是原图的分辨率, 中间结果会占用很大的GPU内存空间, 极有可能导致内存溢出. 后面3层是步长为2的反卷积层, 将特征图解码到原图尺寸.
本文在3层编码器(卷积层+下采样)和3层解码器的基础上, 尝试了2种改进策略:
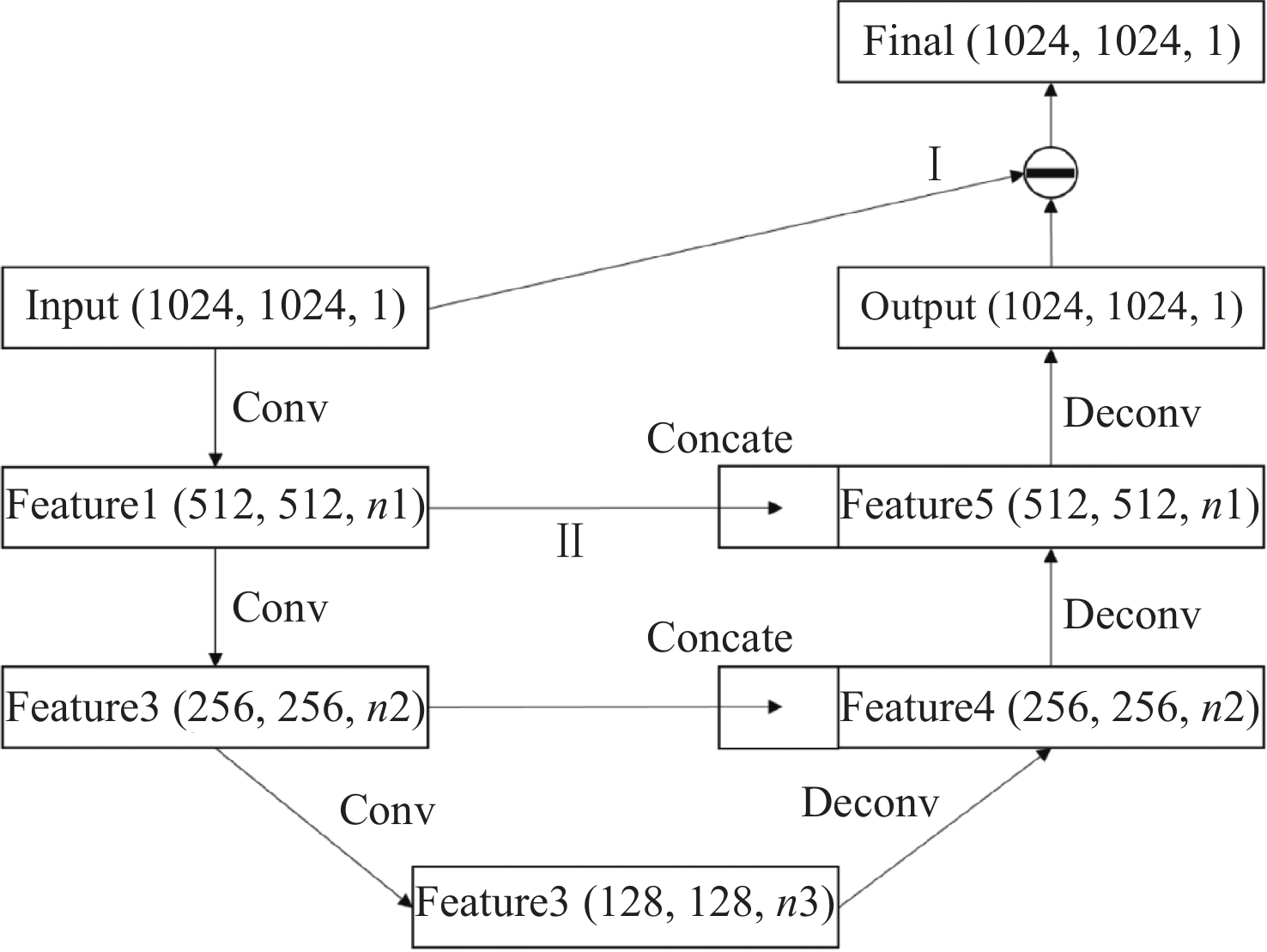
|
图 2 卷积神经网络结构图 |
(1) 残差学习, 如图2中第I部分所示, 其与残差网络[15]的思想相近, 不同的是整个网络变为一个残差块, 输入和理想输出的残差作为模型的预测目标. 前文中提到, 骨骼结构要比无肋骨胸片更加简单, 模型对输入和输出的残差也更容易拟合.
(2) 增加弹跳连接, 如图2中第II部分所示, 将浅层特征图合并到反卷积后的特征图中, 然后再进行下一步反卷积操作. 这种方式一定程度上弥补了下采样带来的空间信息损失, 在不增加额外特征层的基础上, 充分利用了浅层特征, 提高了特征层的利用率.
在后面的实验中, 基础网络结合两个策略, 本文实现了5个网络模型进行性能对比. 分别为基础网络卷积核数量(n1, n2, n3)分别为(16, 32, 64)和(32, 64, 128), 然后在n1=32的基础网络上分别单独加入弹跳连接和残差学习, 以及同时使用. 最后的实验证明了同时使用这两种策略, 能够最大程度提升模型的性能.
2.2 损失函数网络是一个回归模型, 最常用的是MSE和MAE损失函数, MSE由于平方的关系, 对大误差的惩罚力度较大, 但是对小误差比较容忍. 有研究[13]表明, 在图像重建领域MAE比MSE有着更优的性能.
| $ MAE = \frac{1}{N}\mathop \sum \limits_{i = 1}^N \left| {I\left( i \right) - G\left( i \right)} \right| $ | (1) |
| $ MSE = \frac{1}{N}\mathop \sum \limits_{i = 1}^N {\left( {I\left( i \right) - G\left( i \right)} \right)^2} $ | (2) |
式(1) 和式(2) 分别为MAE与MSE的定义, 其中N为一张图像种的像素个数, I为输出图像, G为标签图像. 这两种误差只关注对应像素点之间的误差, 并未考虑到人的视觉特性, 因为我们的最终目的是抑制胸片中的肋骨成像, 同时在肋骨处保持原有的细节, 所以我们并不非常在意比如整体的亮度偏移, 人眼对一个区域的感知容易受到周围区域影响, 因此仅仅关注像素误差可能会造成与视觉感知不一致的效果.
而SSIM (Structural Similarity Index)[16]分别从亮度、对比度和结构3方面度量图像的相似性, 更加符合人眼的视觉特性. SSIM定义如式(3) 所示:
| $ SSIM\left( {I, G} \right) = \frac{{\left( {2{\mu _I}{\mu _G} + {C_1}} \right)}}{{\left( {\mu _I^2 + \mu _G^2 + {C_1}} \right)}}\frac{{\left( {2{\sigma _{IG}} + {C_2}} \right)}}{{\left( {\sigma _I^2 + \sigma _G^2 + {C_2}} \right)}} $ | (3) |
其中,
MS-SSIM在此基础上, 额外考虑了分辨率的因素, 在多个尺度上对图像相似性进行评估. 由于MS-SSIM同样可微, 因此可以将其作为损失函数, 直接用来训练模型. MS-SSIM定义如式(4) 所示:
| $ MsSSIM\left( {I, G} \right) = {l_M}\left( {I, G} \right)\mathop \prod \limits_{j = 1}^M c{s_j}\left( {I, G} \right) $ | (4) |
其中,
| $ Los{s^{MsSSIM}}\left( {I, G} \right) = 1 - MsSSIM\left( {I, G} \right) $ | (5) |
同时使用MAE损失函数和MS-SSIM损失函数能够兼顾像素差异和结构差异, 总的损失函数定义如式(6) 所示:
| $ Loss = \alpha Los{s^{MsSSIM}} + (1 - \alpha) MAE $ | (6) |
其中,
本文的实验数据有240对胸片原图与其对应的DES结果, 我们随机选取220例样本作为训练集, 剩余20例样本作为测试集. 在生成训练样本时, 我们对原数据进行数据扩充, 我们使用了随机平移、裁剪、旋转、水平对称等操作. 这些操作能够一定程度上弥补数据样本量的不足.
此外为了让模型在不同的机器上拍出的胸片都有效, 我们还使用随机伽马变换让训练数据有着不同的对比度, 其中伽马参数范围是[0.5, 2].
我们将原图尺寸限制为1024×1024, 既保证了图像的分辨率, 同时也能让显存能够容纳整个模型以及中间结果.
3.2 评价指标评估模型输出结果与DES标签的相似性, 我们采用了峰值信噪比PSNR(Peak Signal to Noise Ratio)以及SSIM两个评估指标, 其中SSIM如前文中介绍, 更接近人类视觉系统, 定义见式(3). 而PSNR是最广泛使用的评价图像质量的方法, 其定义如式(7):
| $ PSNR = 10*{\log _{10}}\frac{{{{\left( {{2^n} - 1} \right)}^2}}}{{MSE}} $ | (7) |
其中, MSE定义见式(2), 我们使用PSNR来评估两幅图像之间的像素值差距.
3.3 实验结果首先我们将2.1节中提到的5个网络模型用同样的训练数据进行训练, 初始学习率都设置为0.001, 每50个周期学习率衰减为原来的0.2, 总共训练200个周期, 取测试集损失函数最低的模型作为每个网络的最终模型. 然后我们计算每个模型生成的20个结果图像与DES标签的SSIM和PSNR平均值, 具体数据如表1所示.
| 表 1 模型性能对比 |
PSNR和SSIM都是数值越大表明两个图像相似度越高, 实验数据表明, 弹跳连接和残差学习都能够提升模型性能, 如果同时使用, 模型更优. 除此之外, 网络的卷积核数量提升也能提升性能. 在Gusarev等人[6]的实验中也验证了加深模型深度也可以提升模型性能. 增加卷积核数量以及增加模型深度都会增加模型复杂度, 也会增加模型中特征图的内存占用, 因此本文并没有盲目将网络加宽加深.
我们我们从测试集中选取一例样本, 选取最优的模型: (32, 64, 128)的卷积核数目, 同时用跳跃连接和残差学习. 将其输出结果展示, 如图3所示, 能够看出肋骨抑制操作能够一定程度上剔除胸片中的肋骨结构, 使得结节特征更加显著.
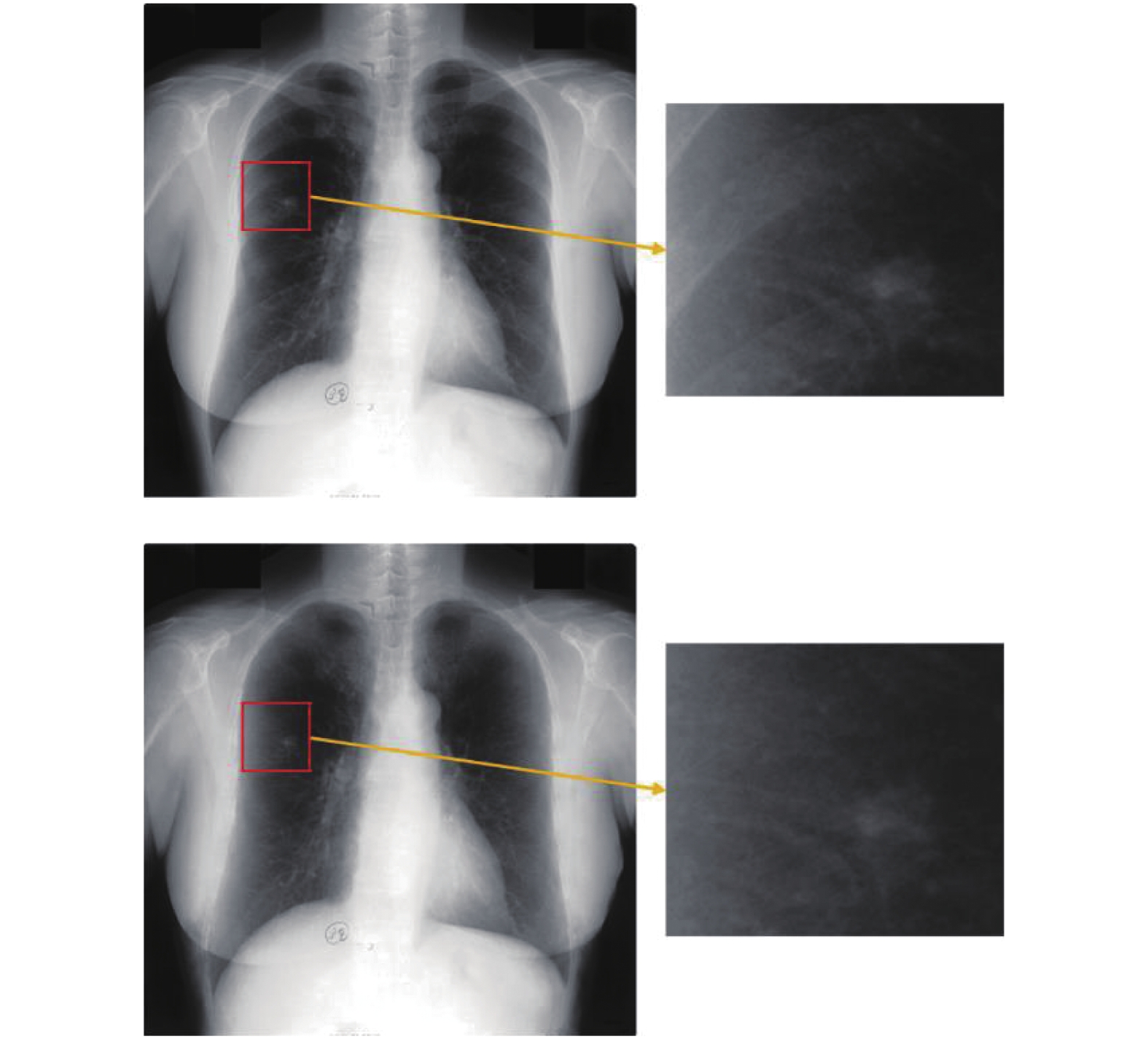
|
图 3 胸片原图与本文模型输出图像 |
由于与先前的研究没有使用同样的数据集, 因此不能直接比较评估指标. 最后为了验证肋骨抑制的有效性, 我们使用目标检测网络RetinaNet[14]作为肺结节检测模型, 在2954张有肺结节的胸片数据集上验证肋骨抑制对肺结节检测的影响. 我们随机选取148张胸片作为测试集, 其余2806张用来训练模型. RetinaNet中的锚点(anchor)长宽比为[0.8, 1, 1.25], 为了使得尺度更加紧凑, 每一个长宽比都有5个不同的尺度分别为[20, 21/5, 22/5, 23/5, 24/5], 以较高的尺度密度, 尽可能地覆盖所有的肺结节. 然后我们使用前面得到的卷积核数目为(32, 64, 128)并且同时有弹跳连接和残差学习的网络模型对胸片数据进行预处理, 数据扩充使用3.1节所述的方法, 用目标检测常用评估指标mAP作对比, 结果如表2所示.
| 表 2 肋骨抑制对检测结果的影响 |
从结果看出, 胸片原图经过肋骨抑制之后, 检测模型更容易取得更优的效果.
4 总结与展望本文基于U-net设计了一个能够抑制胸片中肋骨干扰的卷积神经网络, 通过实验证明了U-net中的弹跳连接, 以及去噪网络中多采用的残差学习策略能够对网络性能有较大的提升. 此外, 经过肋骨抑制处理后的胸片, 作为肺结节检测模型的训练和测试数据, 有助于肺结节检测任务.
下一步的研究工作在肋骨抑制后的胸片数据上, 设计更优的肺结节检测模型, 使其能够在保证检出率的基础上尽可能降低假阳性样本率, 然后构成一个完整的肺结节检测系统, 辅助医生的日常诊断.
| [1] |
Li F, Hara T, Shiraishi J, et al. Improved detection of subtle lung nodules by use of chest radiographs with bone suppression imaging: Receiver operating characteristic analysis with and without localization. American Journal of Roentgenology, 2011, 196(5): W535-W541. DOI:10.2214/AJR.10.4816 |
| [2] |
Chen S, Suzuki K. Computerized detection of lung nodules by means of " virtual dual-energy” radiography. IEEE Transactions on Biomedical Engineering, 2013, 60(2): 369-378. DOI:10.1109/TBME.2012.2226583 |
| [3] |
Vock P, Szucs-Farkas Z. Dual energy subtraction: Principles and clinical applications. European Journal of Radiology, 2009, 72(2): 231-237. DOI:10.1016/j.ejrad.2009.03.046 |
| [4] |
Chen S, Suzuki K. Separation of bones from chest radiographs by means of anatomically specific multiple massive-training ANNs combined with total variation minimization smoothing. IEEE Transactions on Medical Imaging, 2014, 33(2): 246-257. DOI:10.1109/TMI.2013.2284016 |
| [5] |
Yang W, Chen YY, Liu YB, et al. Cascade of multi-scale convolutional neural networks for bone suppression of chest radiographs in gradient domain. Medical Image Analysis, 2017, 35: 421-433. DOI:10.1016/j.media.2016.08.004 |
| [6] |
Gusarev M, Kuleev R, Khan A, et al. Deep learning models for bone suppression in chest radiographs. Proceedings of 2017 IEEE Conference on Computational Intelligence in Bioinformatics and Computational Biology. Manchester, UK. 2017. 1–7.
|
| [7] |
Wang Z, Simoncelli EP, Bovik AC. Multiscale structural similarity for image quality assessment. Proceedings of the 37th Asilomar Conference on Signals, Systems & Computers. Pacific Grove, CA, USA. 2003. 1398–1402.
|
| [8] |
Hogeweg LE, Mol C, de Jong PA, et al. Rib suppression in chest radiographs to improve classification of textural abnormalities. Proceedings of SPIE 7624, Medical Imaging 2010: Computer-aided Diagnosis. San Diego, CA, USA. 2010. 76240Y.
|
| [9] |
Li XC, Luo SH, Hu QM, et al. Rib suppression in chest radiographs for lung nodule enhancement. Proceedings of 2015 IEEE International Conference on Information and Automation. Lijiang, China. 2015. 50–55.
|
| [10] |
Zhang K, Zuo WM, Chen YJ, et al. Beyond a gaussian denoiser: Residual learning of deep CNN for image denoising. IEEE Transactions on Image Processing, 2017, 26(7): 3142-3155. DOI:10.1109/TIP.2017.2662206 |
| [11] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich, Germany. 2015. 234–241.
|
| [12] |
Shelhamer E, Long J, Darrell T. Fully convolutional networks for semantic segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [13] |
Zhao H, Gallo O, Frosio I, et al. Loss functions for image restoration with neural networks. IEEE Transactions on Computational Imaging, 2017, 3(1): 47-57. DOI:10.1109/TCI.2016.2644865 |
| [14] |
Lin TY, Goyal P, Girshick R, et al. Focal loss for dense object detection. Proceedings of 2017 IEEE International Conference on Computer Vision. Venice, Italy. 2017. 2999–3007.
|
| [15] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas, NV, USA. 2016. 770–778.
|
| [16] |
Wang Z, Bovik AC, Sheikh HR, et al. Image quality assessment: From error visibility to structural similarity. IEEE Transactions on Image Processing, 2004, 13(4): 600-612. DOI:10.1109/TIP.2003.819861 |
 2019, Vol. 28
2019, Vol. 28


