卷积神经网络(Convolutional Neural Network, CNN)已经成为图像分类的重要方法[1], 并在医学影像领域得到了快速发展, 具体包括甲状腺癌筛查、乳腺癌诊断、前列腺的Gleason分级、眼底图像的异常筛查等方面. 不断改进和创新的CNN模型, 已然成为提高准确率和效率的关键, 其中池化层是CNN最重要的组成部分之一. 池化层目的在于减少图像尺寸和参数, 提高计算效率, 增大感受野以及提高模型的鲁棒性. 目前最大值池化和平均值池化是在CNN中被广泛使用, 而这两种传统的池化方法虽然处理速度较快, 但会引入边缘光晕、模糊和块效应. 最大值池化是最常用的池化方式, 由于其处理过程过于简单, 使得在激活图上丢失了大量信息, 而平均池化会对某些特征图产生模糊效果[2]. 虽然这些池化方法是高效简单的, 但是阻碍了网络学习最优模型参数的潜力[3].
本文提出了一种基于小波分解卷积神经网络, 利用小波分解中的下采样实现对特征图的池化, 从而减少特征信息的丢失, 并且通过小波分解能够获得特征图的频域信息, 包括低频分量和高频分量[4]. 而传统的卷积神经网络擅长捕获空间域特征[5,6]. 因此, 通过小波分解可将频域信息引入到CNN框架中, 使得模型能够同时学习空域特征和频域特征, 其中小波分解得到的低频信息作为特征图池化的结果, 而高频信息通过捷径连接方式添加到下一层[7]. 可弥补在池化过程中丢失的细节特征. 最后在Camelyon16数据集[8]上与最大值池化和平均值池化进行了对比验证, 从而证明了本文方法的正确性.
目前组织病理学诊断是临床肿瘤诊断的金标准, 但是不断更新的疾病谱系、肿瘤治疗手段, 以及传统玻璃切片易损坏等多重因素导致单纯依靠病理医生已经无法满足临床需求. 因此随着GPU和数字影像设备的快速发展, 全视野数字切片(Whole Slide Image, WSI)在医学影像领域的快速应用, 利用人工智能辅助病理医生对数字病理图像分析, 提高病理诊断的效率和一致性, 已经成为研究热点.
由于CNN在各种任务中出色表现[9], 所以基本上替代了传统手工制作高维特征向量. 从经典的AlexNet, VGG, ResNet到DenseNet, 原始的CNN结构已经扩展到更深的体系结构, 例如ResNet[7]它通过引入捷径连接的方法使得网络模型更深且更易训练. 在受到ResNet的启发, 相继提出了多种改进的CNN网络[10-12]最近的研究工作中证明了CNN与FV-CNN的组合比单独使用CNN可以获得更高的准确率, Lin等通过双线性池化代替全连接层, 在视觉识别方面表现出色[13]. 而CNN中的池化层是其重要的组成部分, 最大值池化和平均值池化是常用的两种池化方法, 虽然它们有效简单, 但是不可避免的丢失了大量的特征信息[3,14].
在频域方面, 利用小波变换处理数字图像已经得到了广泛的研究, 并取得了很大的成果[15,16]. Tian等利用非向下采用小波变换实现了对微观物体的三维形貌重建, 将物体的频域信息与三维空间建立关联, 是小波变换在三维重构领域的突出应用[17]. 小波变换能轻松的获得图像的低频信息和高频信息, 可将频域信息集成到CNN模型中, 使得CNN能够同时学习空域和频域特征. Liu等[18]提出多级小波的U-Net模型, 将小波分解和小波重构嵌入到U-Net架构中, 用于图像复原, 但是它不仅对低频分量进行小波变换, 同时也对高频分量进行小波变换, 因此冗余的小波变换极大的增加了计算负担.
在病理图像的良恶性分类中, Garud等[19]通过175张WSI训练测试CNN模型鉴别乳腺癌良恶性的可靠性, 研究结果显示, 准确率可达到0.89. Lu等[20] 提出基于聚类约束的弱监督病理图像良恶性分类, 通过计算每个图块的注意力得分, 对不同得分的图块进行聚类操作, 最后再训练一个有监督的能够区分正负样本的线性分类器, 以提高整个WSI分类准确率. Mercan等[21]研究证明以计算机为基础的病理诊断方法具有良好的应用前景, 特别是在乳腺癌良恶性分类诊断上. 目前, 传统的CNN网络对病理图像进行良恶性分类, 主要是学习病理图像中空域信息, 鲜有将频域信息引入到CNN网络中, 并且传统的池化层也不可避免的丢失了原特征图中的细节信息.
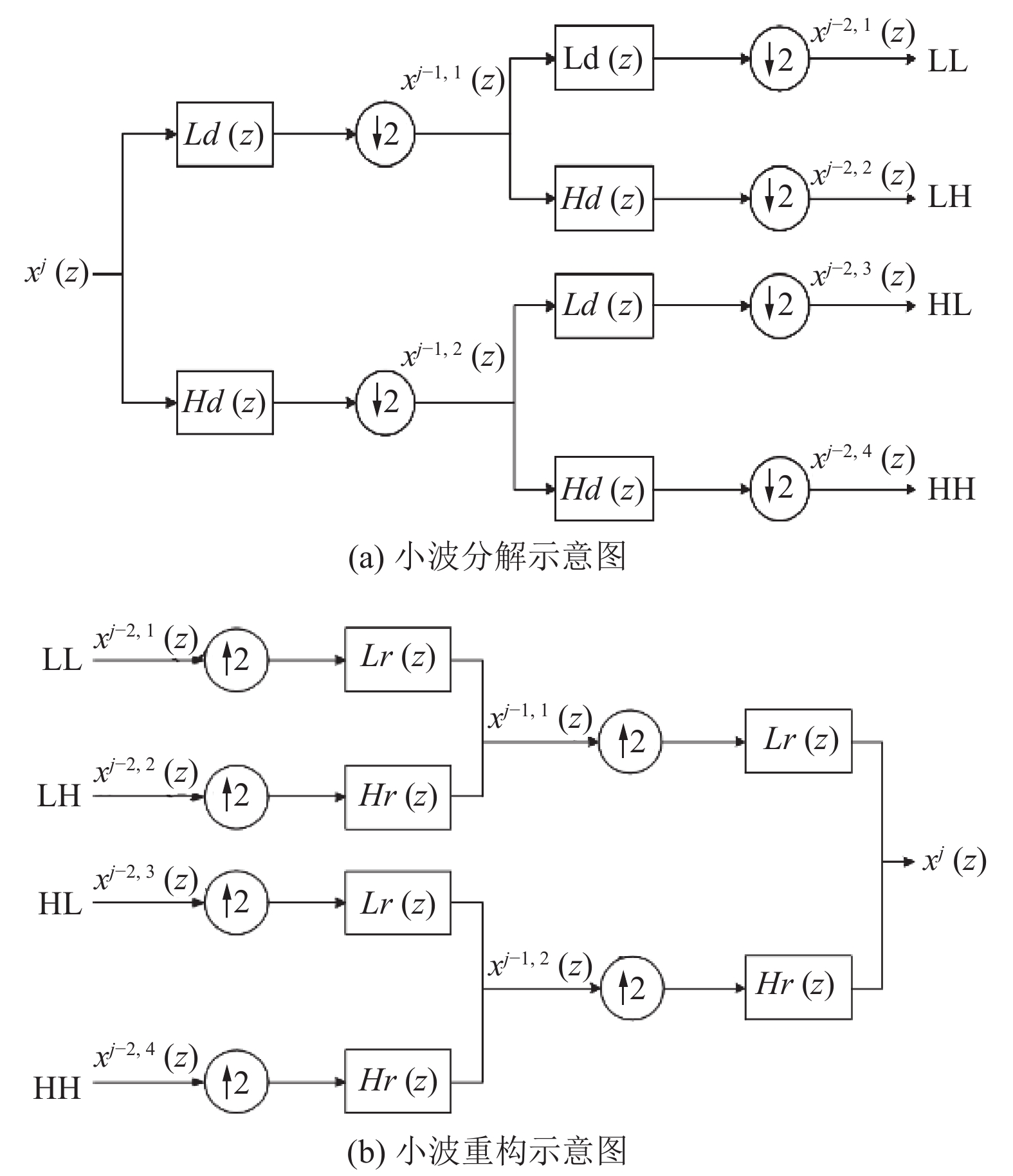
2 方法 2.1 离散小波变换(Discrete Wavelet Transform, DWT)经典快速离散小波变换是二维离散小波变换一种高效实现方法[22], 其分解与重构过程如图1所示, 原始信号
|
图 1 离散小波变换结构框图 Fig. 1 Discrete Wavelet Transform structure block. (a) Wavelet decomposition block diagram, (b) Wavelet reconstruction block diagram |
二维离散小波变换在给定尺度函数
| $\varphi (x,y) = \varphi (x)\varphi (y)$ | (1) |
| ${\psi ^H}(x,y) = \psi (x)\varphi (y)$ | (2) |
| ${\psi ^V}(x,y) = \varphi (x)\psi (y)$ | (3) |
| ${\psi ^D}(x,y) = \psi (x)\psi (y)$ | (4) |
其中, H, V, D分别表示水平高频分量, 垂直高频分量和对角高频分量.
| ${W_\varphi }({j_0},m,n) = \frac{1}{{\sqrt {MN} }}\sum\limits_{x = 0}^{M - 1} {\sum\limits_{y = 0}^{N - 1} {f(x,y){\varphi _{{j_0},m,n}}(x,y)} } $ | (5) |
| $W_\psi ^i(j,m,n) = \frac{1}{{\sqrt {MN} }}\sum\limits_{x = 0}^{M - 1} {\sum\limits_{y = 0}^{N - 1} {f(x,y)\psi _{j,m,n}^i(x,y)} } {\rm{, }}\;i = \{ H,V,D\} $ | (6) |
其中,
| ${\varphi _{j,m,n}}(x,y) = {2^{j/2}}\varphi ({2^j}x - m,{2^j}y - n)$ | (7) |
| $\psi _{j,m.m}^i(x,y) = {2^{j/2}}{\psi ^i}({2^j}x - m,{2^j}y - n),{\rm{ }}i = \{ V,H,D\} $ | (8) |
从式(5)中可以看出, 图像经过小波变换后能够得到图像的近似表达, 其作用与传统的最大值池化和均值池化相同, 在式(6)中小波变换后仍保留了低频分量中丢失的高频分量.
2.2 小波池化与传统池化传统的CNN专注于学习空间域信息, 并且被广泛使用的最大值池化和平均值池化存在丢失大量特征信息的现象, 阻碍了网络学习最优参数的潜力.
从图2中可以看出, 在最大值池化中, 当重要特征幅度低于不重要特征时, 重要特征信息就会丢失. 而在平均值池化中则接收了幅值大的特征和幅值小的特征, 因此会减小幅值大的特征.
在给定具有n个输入分量
| $y = (x * w * P) \downarrow p$ | (9) |
其中,
在平均值池化中, 其本质就是将原图像进行均值滤波后再进行下采样, 由于均值滤波属于低通滤波器并且进行了下采样, 这不可避免的丢失了原图像中的细节信息. 而利用小波分解后得到的高频分量则能够有效弥补这一信息.

|
图 2 最大值池化和平均值池化的缺点 Fig. 2 Shortcomings of Max & Average Pooling |
以最简单的Haar小波为例, 4个滤波器定义为:
| $\begin{split} {f_{\rm LL}} = \;&\left[ {\begin{array}{*{20}{c}} 1&1 \\ 1&1 \end{array}} \right], {f_{\rm LH}} = \left[ {\begin{array}{*{20}{c}} { - 1}&{ - 1} \\ 1&1 \end{array}} \right],\\ {f_{\rm HL}} =\;& \left[ {\begin{array}{*{20}{c}} { - 1}&1 \\ { - 1}&1 \end{array}} \right], {f_{\rm HH}} = \left[ {\begin{array}{*{20}{c}} 1&{ - 1} \\ { - 1}&1 \end{array}} \right]\\[-20pt] \end{split}$ | (10) |
显然,
| ${X_{\rm LL}} = ({f_{\rm LL}} * X) \downarrow 2$ | (11) |
| ${X_{\rm LH}} = ({f_{\rm LH}} * X) \downarrow 2$ | (12) |
| ${X_{\rm HL}} = ({f_{\rm HL}} * X) \downarrow 2$ | (13) |
| ${X_{\rm HH}} = ({f_{\rm HH}} * X) \downarrow 2$ | (14) |
其中,
| $\begin{split} {X_{\rm LL}}(i,j) =\;& (X(2i - 1,2j - 1) + X(2i - 1,2j) \\ &+ X(2i,2j - 1) + X(2i,2j))/4 \end{split}$ | (15) |
| $\begin{split} {X_{\rm LH}}(i,j) =\;& ( - X(2i - 1,2j - 1) - X(2i - 1,2j) \\ & + X(2i,2j - 1) + X(2i,2j))/4 \end{split}$ | (16) |
| $\begin{split} {X_{\rm HL}}(i,j) =\;& ( - X(2i - 1,2j - 1) + X(2i - 1,2j) \\ &- X(2i,2j - 1) + X(2i,2j))/4 \end{split}$ | (17) |
| $\begin{split} {X_{\rm HH}}(i,j) =\;& (X(2i - 1,2j - 1) - X(2i - 1,2j) \\ &- X(2i,2j - 1) + X(2i,2j))/4 \end{split}$ | (18) |
从式(15)中可以看出Harr小波变换后得到的低频分量是与传统CNN中平均池化结果是一致的, 此外还得到了在低频分量中丢失的高频分量. 因此将小波变换的多尺度分析引入到神经网络架构中, 以代替传统的池化方法, 尽管这一想法很简单, 但是其对模型性能的提升是明显的, 正如本文的实验结果所示.
2.3 小波分解卷积神经网络(WDCNN)由3.1和3.2节可知, 二维小波变换会沿行列方向对图像进行下采样, 因此经过一次尺度变换后, 图像尺寸变为原来的四分之一, 实现了与传统池化方法同样的作用. 从式(6)中看出小波变换后保留了低频分量中丢失的高频分量, 能弥补池化过程中丢失的细节特征. 因此本文在此基础上提出一种基于小波分解卷积神经网络(WDCNN), 利用小波分解具有多尺度分析的能力, 实现对图像的池化, 以此代替传统的最大值池化和平均值池化, 并将小波分解后的高频分量引入到CNN中, 使得CNN能够同时学习空域特征和频域特征. 图像经小波变换后分解为4部分: LL表示低频分量, 它是图像的近似表示, HL是图像的高频水平分量, LH是图像的高频垂直分量, HH是图像的高频对角分量, 如图3所示.

|
图 3 小波变换示意图 Fig. 3 Wavelet transform block diagram |
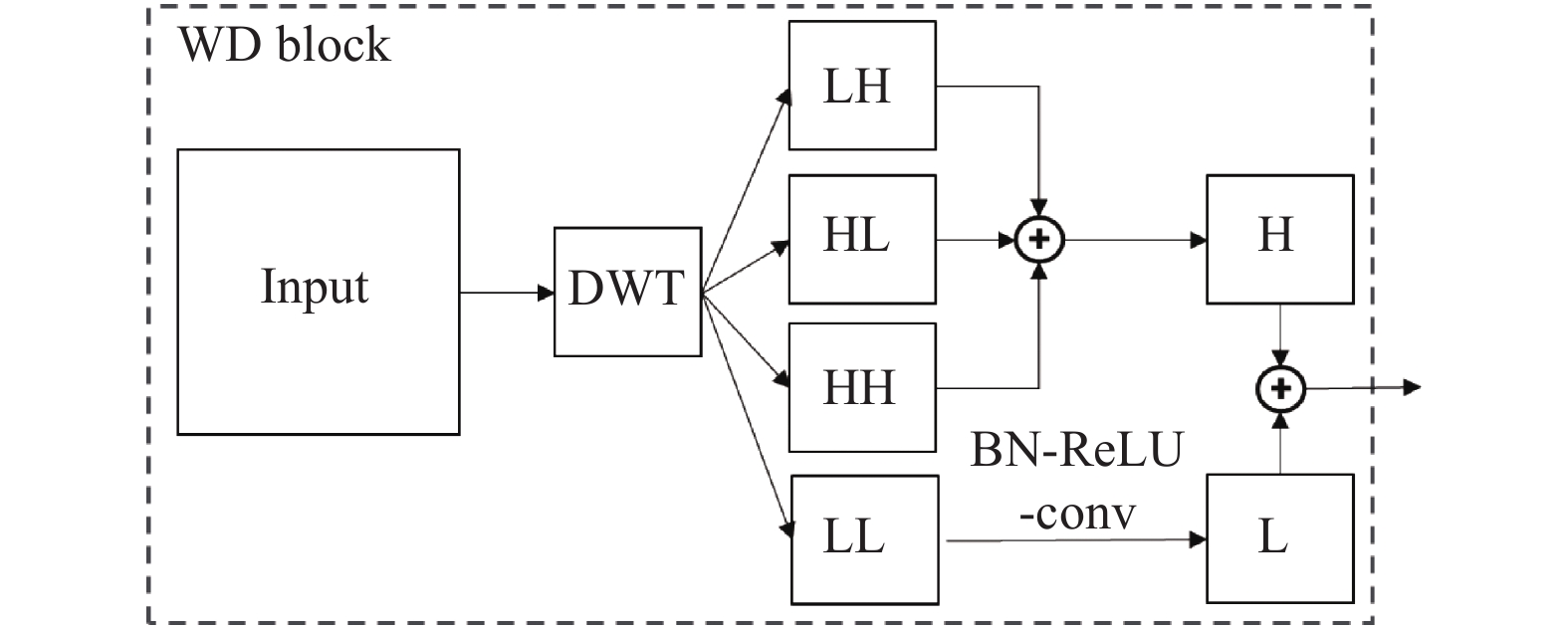
在本文中只对特征图使用一级小波分解, 以获得图像的低频和高频信息, 低频分量作为特征图池化的结果, 而3个高频分量相加后通过捷径连接的方式, 添加到下一层. 在整个过程中不涉及小波重构. 将小波分解引入到CNN网络中, 可构建小波分解模块, 如图4所示.
WD模块的结构简单, 主要可分为两部分: 低频部分和高频部分, 将小波分解后的低频部分作为池化的结果, 然后通过一次常规的BN-ReLU-conv操作, 得到输出结果L; 小波分解后的3个高频分量的值相加, 得到结果H; 最后将L和H的值再次相加, 并且通道数不变, 得到WD模块的输出结果. 将WD模块代替传统的池化层嵌入到CNN中, 即可得到小波分解卷积神经网络(WDCNN), 如图5所示.
|
图 4 小波分解模块示意图 Fig. 4 Wavelet decomposition block diagram |

|
图 5 WDCNN结构示意图 Fig. 5 WDCNN structure block diagram |
图5中BN表示批量标准化, Conv表示卷积核大小为3×3, 步距为1的卷积操作, FC表示全连接层.
从图5中可以看出, 引入WD模块的CNN网络中没有使用传统的池化层来减小特征图的大小, 而是通过WD模块中的小波分解实现对特征图大小的缩减. 此外, 为了充分利用小波分解后得到的图像信息, 本文使用捷径连接[7]的方式, 将小波分解得到的3个高频分量添加到下一层中, 通过这种方式可以弥补低频特征图中丢失的细节信息.
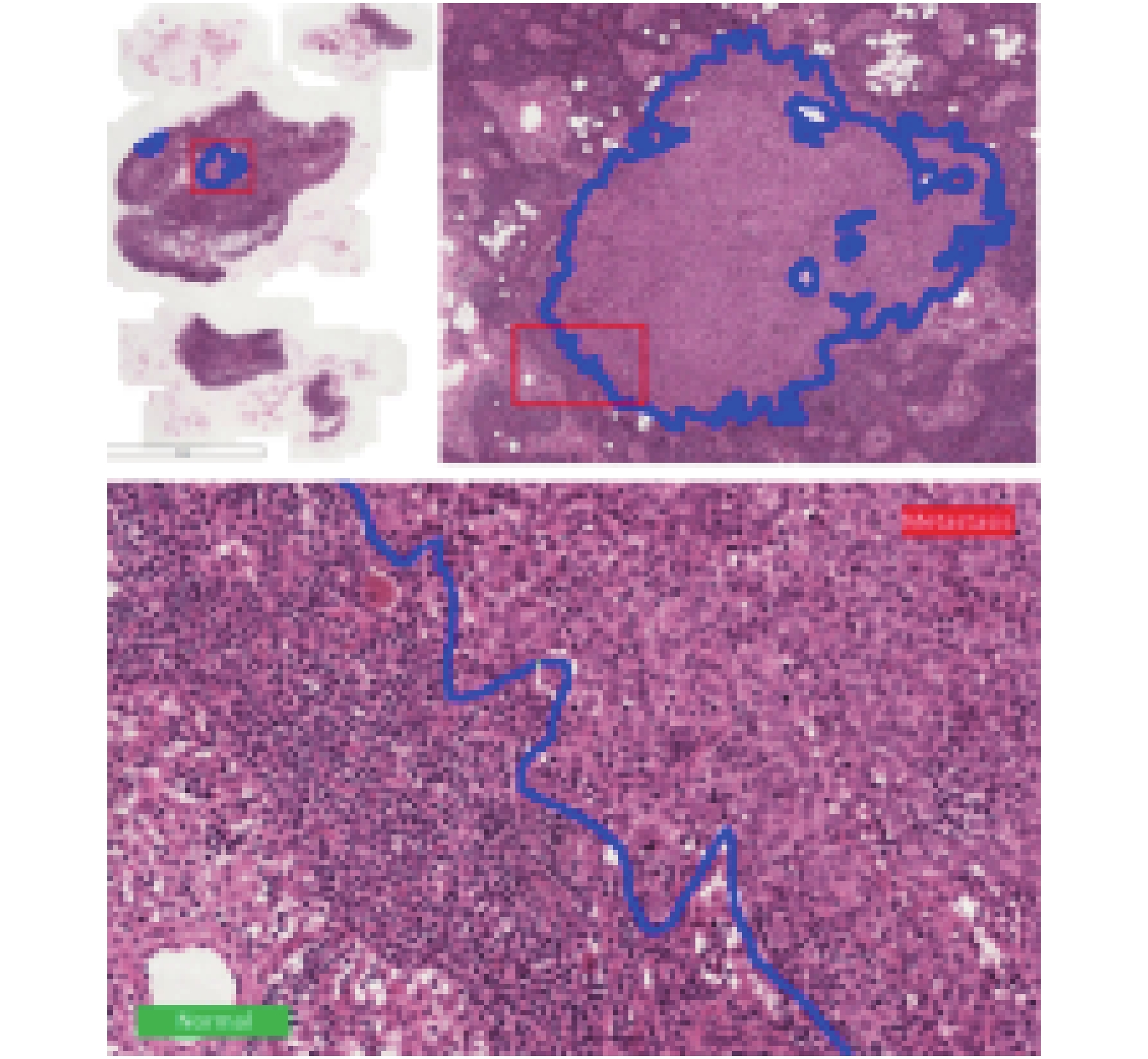
3 实验与讨论 3.1 数据集Camelyon16数据集[8] (
|
图 6 全切片病理图像 Fig. 6 Whole slide pathological image |
图像预处理过程如下:
第1步. 将病理图像从RGB转为HSV (Hue, Saturation, Value)表示色相, 饱和度和亮度.
第2步. 利用阈值分割算法Otsu对病理图像进行背景与组织区域分割, 以获得病理图像中的组织区域. 使用Otsu算法计算出每个通道的最佳分割阈值, 通过实验表明使用H通道和S通道分割出来的组织区域的交集作为最终组织区域.
第3步. 利用病理医生标注的癌变区域的轮廓信息(如图6中的深色曲线)分割出正常组织区域和癌变组织区域.
第4步. 以随即撒点的方式, 在正常区域和癌变区域进行图像块采样, 图像块大小为256×256, 如图7所示.
3.2 评价指标为了全面评价WDCNN模型在病理图像分类任务中的优势, 本文采用准确率(Accuracy)、精确率(Precision)、召回率(Recall)和平衡分数(F1值). 具体公式如下:
| $Accuracy = \frac{{TP + TN}}{{TP + FP + TN + FN}}$ | (19) |
| $Precision = \frac{{TP}}{{TP + FP}}$ | (20) |
| $Recall = \frac{{TP}}{{TP + FN}}$ | (21) |
| $F1 = 2 \times \frac{{Precision \times Recall}}{{Precision + Recall}}$ | (22) |
其中, TP代表正样本被正确识别为正样本的数量; TN代表负样本被正确识别为负样本的数量; FP代表负样本被错误识别为正样本的数量; FN代表正样本被错误识别为负样本的数量.

|
图 7 数据集制作流程 Fig. 7 Datasets production process |
| 表 1 数据集的分布 Table 1 Distribution of datasets |
3.3 实验
为验证WD模块对传统CNN模型在病理图像分类性能上的提升, 本文统一搭建一个五层CNN网络, 如图8所示, 由于在CNN中使用了全局平均池化, 因此要求输入图像的大小一致均为256×256, 为加快网络训练防止梯度爆炸, 在激活函数之前使用了批量归一化[27]. 对于优化函数, 我们使用Adam[28]代替SGD, 并使用ReLU[29]作为激活函数.
图8(b)中可以看出包含5个卷积层以及两个Max/Average池化层. 由于WD模块在池化的同时也做了一次卷积操作, 因此在图8(a)中只显示3个卷积层, 这样做的目的是保证了WDCNN与传统CNN模型的训练参数保持一致, 从而充分验证WD模块对网络性能的提升. 经过计算两个模型的训练参数均为589762.
此外不同的小波基函数在小波分解后会得到不同的低、高频分量, 因此为了选取最优的小波基函数, 在WDCNN中对经典的Haar、Biorthogonal和Daubechies小波基函数也进行了对比验证.
从表2的各项评价指标中可以看出, 相比于最大值池化和平均值池化而言, 在网络中引入WD模块, 能够明显提升CNN模型的分类性能. 从不同小波基函的对比结果来看, 使用Biorthogonal小波基函数对CNN分类性能提升最大.

|
图 8 5层CNN结构框图 Fig. 8 Five-layer CNN structure block diagram, (a) CNN model embedded in WD block, (b) Traditional CNN model |
为了进一步验证WD模块在深度卷积神经网络中的性能, 本文将WD模块引入到经典的VGG16中, 将VGG16中的最大值池化层全部替换为WD模块, 并在Camelyon16数据集中进行对比验证, 证明本文方法的有效性和通用性.
从表3中可以看出, 将传统的最大值池化层替换为WD模块后, WD-VGG16在准确率、召回率和F1值上均优于传统的VGG16. 从而说明了WD模块的引入, 能够显著提升传统CNN在病理图像分类任务中的性能. 图9是WD-VGG16和VGG16的AUC-ROC曲线对比图, 从图中可以看出WD-VGG16具有更高的AUC值, 说明引入WD模块能够提高分类器的稳定性和鲁棒性.
| 表 2 Camelyon16数据集上不同方法的评估 Table 2 Evaluation of different methods in Camelyon16 dataset |
| 表 3 WD-VGG16与VGG16的评估结果 Table 3 Evaluation results of WD-VGG16 and VGG16 |

|
图 9 WD-VGG16和VGG16的ROC曲线 Fig. 9 ROC curve of WD-VGG16 and VGG16 |
4 结束语
本文提出的小波分解卷积神经网络(WDCNN), 利用小波变换多尺度分析的能力, 将特征图分解为低频分量和高频分量, 不仅实现了对特征图的池化, 也将特征图中的细节特征信息通过捷径连接的方式引入到传统CNN中, 使得传统CNN能够同时学习空域信息和频域信息, 提高了模型的泛化能力, 并在病理图像分类任务中表现优异. 最后在Camelyon16数据进行了充分的对比验证, 并选取了最优的小波基函数, 证明了本文方法在病理图像分类任务中的优势. 但是, 相比与最大值池化和平均值池化而言, 引入小波分解模块会增加网络的计算负担, 因此需要优化模型结构, 在不影响网络性能的同时减少网络计算量.
| [1] |
Theodoridis S. Neural networks and deep learning. In: Theodoridis S, ed. Machine Learning. Amsterdam: Academic Press, 2015.
|
| [2] |
Lee CY, Gallagher PW, Tu ZW. Generalizing pooling functions in convolutional neural networks: Mixed, gated, and tree. Proceedings of the 19th International Conference on Artificial Intelligence and Statistics. Cadiz, Spain. 2016. 464–472.
|
| [3] |
Yu DJ, Wang HL, Chen PQ, et al. Mixed pooling for convolutional neural networks. Proceedings of the 9th International Conference on Rough Sets and Knowledge Technology. Shanghai, China. 2014. 364–375.
|
| [4] |
Unser M. Texture classification and segmentation using wavelet frames. IEEE Transactions on Image Processing, 1995, 4(11): 1549-1560. DOI:10.1109/83.469936 |
| [5] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, CA, USA. 2012. 1097–1105.
|
| [6] |
LeCun Y, Boser B, Denker J, et al. Backpropagation applied to handwritten zip code recognition. Neural Computation, 1989, 1(4): 541-551. DOI:10.1162/neco.1989.1.4.541 |
| [7] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas, NV, USA. 2016. 770–778.
|
| [8] |
Litjens G, Bandi P, Ehteshami Bejnordi B, et al. 1399 H & E-stained sentinel lymph node sections of breast cancer patients: The CAMELYON dataset. GigaScience, 2018, 7(6): giy065. |
| [9] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 |
| [10] |
Zagoruyko S, Komodakis N. Wide residual networks. Proceedings of British Machine Vision Conference 2016. York, UK. 2016.
|
| [11] |
Zhang K, Sun M, Han TX, et al. Residual networks of residual networks: Multilevel residual networks. IEEE Transactions on Circuits and Systems for Video Technology, 2018, 28(6): 1303-1314. DOI:10.1109/TCSVT.2017.2654543 |
| [12] |
Huang G, Sun Y, Liu Z, et al. Deep networks with stochastic depth. Proceedings of the 14th European Conference on Computer Vision. Amsterdam, the Netherlands. 2016. 646–661.
|
| [13] |
Lin TY, RoyChowdhury A, Maji S. Bilinear convolutional neural networks for fine-grained visual recognition. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(6): 1309-1322. DOI:10.1109/TPAMI.2017.2723400 |
| [14] |
Zeiler MD, Fergus R. Stochastic pooling for regularization of deep convolutional neural networks. Proceedings of the 1st International Conference on Learning Representations. Scottsdale, AZ, USA. 2013.
|
| [15] |
Kanchana M, Varalakshmi P. Texture classification using discrete shearlet transform. International Journal of Scientific Research, 2013, 2(6): 194-196. |
| [16] |
杨吉雨, 周武能, 段学闯. 基于双正交小波提升机制的图像融合方法. 计算机工程与设计, 2012, 33(1): 290-294. DOI:10.3969/j.issn.1000-7024.2012.01.057 |
| [17] |
Tian YZ, Cui HY, Pan ZY, et al. Improved three-dimensional reconstruction algorithm from a multifocus microscopic image sequence based on a nonsubsampled wavelet transform. Applied Optics, 2018, 57(14): 3864-3872. DOI:10.1364/AO.57.003864 |
| [18] |
Liu PJ, Zhang HZ, Lian W, et al. Multi-level wavelet convolutional neural networks. IEEE Access, 2019, 7: 74973-74985. DOI:10.1109/ACCESS.2019.2921451 |
| [19] |
Garud H, Karri SPK, Sheet D, et al. High-magnification multi-views based classification of breast fine needle aspiration cytology cell samples using fusion of decisions from deep convolutional networks. Proceedings of 2017 IEEE Conference on Computer Vision and Pattern Recognition Workshops. Honolulu, HI, USA. 2017. 828–833.
|
| [20] |
Lu MY, Williamson DFK, Chen TY, et al. Data-efficient and weakly supervised computational pathology on whole-slide images. Nature Biomedical Engineering, 2021.
|
| [21] |
Mercan E, Mehta S, Bartlett J, et al. Assessment of machine learning of breast pathology structures for automated differentiation of breast cancer and high-risk proliferative lesions. JAMA Network Open, 2019, 2(8): e198777. DOI:10.1001/jamanetworkopen.2019.8777 |
| [22] |
Mallat S, Zhong S. Characterization of signals from multiscale edges. IEEE Transactions on Pattern Analysis and Machine Intelligence, 1992, 14(7): 710-732. DOI:10.1109/34.142909 |
| [23] |
Mallat SG. Multifrequency channel decompositions of images and wavelet models. IEEE Transactions on Acoustics, Speech, and Signal Processing, 1989, 37(12): 2091-2110. DOI:10.1109/29.45554 |
| [24] |
尤波, 张宸枫. 结合小波自适应阈值与双边滤波的图像降噪. 计算机工程与设计, 2019, 40(8): 2278-2282. |
| [25] |
Mallat SG. A theory for multiresolution signal decomposition: The wavelet representation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 1989, 11(7): 674-693. DOI:10.1109/34.192463 |
| [26] |
Wang DY, Khosla A, Gargeya R, et al. Deep learning for identifying metastatic breast cancer. arXiv: 1606.05718, 2016.
|
| [27] |
Ioffe S, Szegedy C. Batch normalization: Accelerating deep network training by reducing internal covariate shift. Proceedings of the 32nd International Conference on International Conference on Machine Learning. Lille, France. 2015. 448–456.
|
| [28] |
Kingma DP, Ba J. Adam: A method for stochastic optimization. Proceedings of the 3rd International Conference on Learning Representations. San Diego, CA, USA. 2015.
|
| [29] |
Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks. Proceedings of the 14th International Conference on Artificial Intelligence and Statistics. Fort Lauderdale, FL, USA. 2011. 315–323.
|
 2021, Vol. 30
2021, Vol. 30


