2. 瑞尔森大学 计算机科学系, 多伦多 ON M5B 2K3;
3. 上海智臻智能网络科技股份有限公司 小i机器人研究院, 上海 201803
2. Department of Computer Science, Ryerson University, Toronto ON M5B 2K3, Canada;
3. Shanghai Xiaoi Robot Technology Co. Ltd., Shanghai 201803, China
文本检索会议(TREC)临床决策支持任务(CDS)的目的是构建一个信息检索系统以支持临床决策. 系统接受由医生从电子病历(EMR)中总结的查询, 然后从在线的医学文献集合返回相关文献.
传统的信息检索系统是根据给定查询中关键词的统计量信息计算文章的相关性[1]. 根据文章的相关性得分从高到底进行排序, 相关性最高的作为结果返回. 作为提高信息检索系统性能的有效方法之一, 查询扩展是将包含信息的词加入原始查询, 以便可以使用更多的统计信息来检索相关文章[2–4]. 在TREC CDS任务中, 大部分方法都是使用统计信息, 例如单词出现的次数, TF-IDF分数等来进行扩展单词选择. 这种类型的方法不考虑在语义级别查询和扩展词之间的关系, 并且这类方法在选择扩展词数量的时候都是使用固定的
本文提出的方法不仅考虑了统计量信息, 还考虑了查询和扩展词之间的关系. 考虑到医学文本注释的难度和成本, 我们使用非医疗数据集STS数据集作为训练数据来学习句子和实体之间的关系. 我们选择这些数据的原因是因为我们认为学习句子和实体之间的关系对计算查询和扩展医学实体之间的关系是有用的. 此外, 在测试部分, 我们不仅仅考虑单个医学实体作为扩展的影响, 还考虑了实体组合作为扩展的影响. 所以我们解决了使用固定
在本文, 我们提供了一种查询扩展方式来支持医学信息检索中的临床决策问题. 具体来说, 我们提出了基于注意力机制的神经网络来动态选择扩展医学实体. 我们利用了迁移学习的思想, 将在其他领域学习到的知识应用于医学领域, 以此来解决医学标注成本高, 标注难得问题. 此外, 我们选择最佳的医学实体组合作为扩展来解决固定
本章将介绍在医学领域中基于注意机制网络的查询扩展方法. 该方法利用了迁移学习的思想, 将在其他领域学习到的知识运用到医学领域, 以此来解决医学领域中标注困难和成本高的问题. 由于在模型的训练和测试部分我们使用的是不同的数据, 所以我们将分两部分介绍概念. 在训练过程中,
我们是利用网络资源和MeSH得到扩展候选实体. 我们选择使用网络资源的原因是我们认为搜索引擎返回给我们结果是和原始查询相关的并且结果是按照与查询的相关性从高到低进行排序的. 在之前的工作[12,13], 我们可以发现医学实体对医学查询有正向作用, 所以我们利用MeSH对查询进行映射, 得到扩展医学实体. MeSH[11]是一种广泛使用的医学本体数据库, 由16类医学概念组成. 作为外部知识资源, 如果医学实体可以在前
因为候选实体之间也是存在一定的联系和影响, 所以我们不仅仅考虑单个候选实体的影响, 而且考虑候选实体组合的影响. 考虑到计算的时间复杂性和空间复杂性, 我们只结合任何

|
图 1 查询扩展方法框架 |
1.2 扩展医学实体组合的挑选
和之前只利用统计量信息不同, 我们使用神经网络模型自动的选择最佳扩展医学组合. 考虑到人工标注的困难和开销, 我们选择其他领域的数据集来训练模型. 我们之所以选择其他领域数据集训练模型是因为我们认为实体是句子理解的重要组成部分, 而医学实体是理解医学句子的重要组成部分, 所以学习到的实体和句子之间的知识也是可以用到医学实体和医学句子中. 图2显示了神经网络选择模型的结构, 包括了嵌入层(Embedding Layer), LSTM层(LSTM Layer), 注意力层(Attention Layer)和预测层(Predict Layer). 从图2我们可以看出, 最重要的部分是得到句子和实体组的表示.
嵌入层: 因为我们是学习其他领域的知识并将其运用到医学领域中, 所以只使用词嵌入是不合理的.如果我们只使用了词嵌入, 则在模型训练期间无法训练医学单词, 为了解决这个问题, 我们将词嵌入和字符嵌入结合起来表示句子和实体. 和词嵌入一样, 字符嵌入也是将每个单词映射到高维向量空间, 但是字符嵌入训练的是每个字符的向量. 我们利用卷积神经网络(convolutional neural networks)求得
| $FW({w_i}) = [C({w_i}), W({w_i})]$ | (1) |
其中
LSTM层: 我们使用LSTM生成具有语义构成的句子表征. 众所周知, LSTM是一种典型的递归神经网络变体, 通过引入记忆细胞和门机制, 已被广泛用于长文档建模. 在每个位置

|
图 2 基于注意力机制的网络模型 |
| ${i_t} = \sigma ({W_i}{x_t} + {U_i}{h_{t - 1}})$ | (2) |
| ${f_t} = \sigma ({W_f}{x_t} + {U_f}{h_{t - 1}})$ | (3) |
| ${o_t} = \sigma ({W_o}{x_t} + {U_o}{h_{t - 1}})$ | (4) |
| ${\tilde c_t} = \tanh ({W_c}{x_t} + {U_c}{h_{t - 1}})$ | (5) |
| ${c_t} = {f_t} \otimes {c_{t - 1}} + {i_t} \otimes {\tilde c_t}$ | (6) |
| ${h_t} = {o_t} \otimes \tanh ({c_t})$ | (7) |
其中,
注意力机制层: 我们使用注意力机制来获取实体组中比较重要的实体. 受到神经机器翻译的影响, 注意力机制得到了广泛的使用, 因为它可以帮助模型集中注意力于输入信息中比较重要的部分. 实体组的表征
| $EP = \sum\limits_{i = 1}^m {{\alpha _i}{e_i}} $ | (8) |
其中
| ${\alpha _i} = \frac{{\exp (E({e_i}, FW))}}{{\displaystyle \sum\limits_{K = 1}^m {\exp (E({e_k}, FW))} }}$ | (9) |
| $E({e_i}, FW) = {v^{\rm{T}}}\tanh ({W_h}{e_i} + {W_\alpha }FW + b)$ | (10) |
其中,
预测层: 在得到句子表征和实体组表征之后, 我们将句子表征和实体表征点乘并与句子表征, 实体表征拼接送入二层的深度神经网络, 具体公式如下:
| ${y_1} = ReLU([SP, EP, (SP \otimes EP)] \cdot {W_{v1}} + b)$ | (11) |
| $y = {W_v} \cdot {y_1} + {b_1}$ | (12) |
其中,
这一章主要介绍实验的配置, 包括数据集、查询、参数设置以及评测指标.
数据集: STS数据集包括了从2012年到2017年期间在SemEval中使用过的英文数据集的集合, 一共有8628个句子对. 表1显示的是根据类型和训练-验证-评测的具体划分情况. 在我们的方法中, 我们将STS的训练数据集和验证数据集都作为训练数据集, STS的测试数据集作为验证数据集. 我们在TREC CDS 2014, 2015和2016上检验方法的效果. TREC CDS中的文章都是来自于Open Access Subset of PubMed Central (PMC). PMC是一个免费在线生物医学文章数据库, 所有的文章都是以NXML的形式呈现.
| 表 1 STS数据集结构 |
所有的实体都是用TagMe[14]进行注释并且所有的注释都被保留. 这是ClueWeb上最广泛使用的基于实体的排名方法设置.
查询: 在TREC CDS中, 查询都是由专业查询开发者从患者的实际医疗记录中总结得到的自由文本. 检索到的文章对于回答每个查询的临床问题应该是有用的, 每一个TREC CDS任务都有30个查询.
参数设置: 我们使用5折交叉验证评估该方法. LSTM设置: 隐藏状态设置为100维. 句子部分用ReLU激活函数. Batch大小在{32, 64, 128}中选择, 学习率在{0.0005, 0.001, 0.002}中选择, 迭代次数在{5, 10, 20}中选择, 医疗实体组合的个数从{1, 2, 3, 4, 5}中选择. 另外, 我们为每个查询选择的是谷歌检索的前10个结果.
评测指标:在这次实验中, 我们选择NDCG和P@10作为评测指标. 其中P@10表示前10个检索结果的准确率, NDCG则是衡量前10个检索结果排序的评测指标.
3 实验结果与分析在一章中, 我们主要介绍比较方法和本文提出的方法在2014年, 2015年, 2016年数据集上的实验效果. 因为处理数据和建索引方法的不同, 并且在TREC CDS数据集中未有神经网络方法的实验结果可做对比, 为了验证本文方法的有效性, 我们选择了下面几种对比方法.
BM25: 只用原始query进行检索, 没有任何查询扩展. 检索模型为BM25[15].
WebAssistance: 所有在谷歌检索结果中出现过的医学词都作为查询扩展.
WithoutCombination: 只单单选择前三个分数高的医学实体作为查询扩展. 该方法不考虑实体组合的效果.
LSTM+AdA: 在得到实体表示部分, 使用加权平均的方法获得实体表示, 并没有使用注意力机制的方法.
3.1 与对比方法的比较表2展示了本文方法和对比方法在TREC CDS 2014年, 2015年以及2016年数据上的实验效果. 从表2中可以看出, 和对比方法相比, 本文提出的方法有很大的提升. 本文提出的方法在2014年数据集上, NDCG为0.2521, 和BM25相比, 有5.48%的提升, P@10值为0.3, 和BM25相比, 本文提出的方法有11.11%的提升. 在2016年数据集上, 评测指标NDCG的值为0.2172, P@10的值为0.2733, 同时我们可以看出, 和BM25相比, NDCG有9.42%的提升, P@10有15.46%的提升, 这是很显著的提升. 虽然在2015年数据集上的P@10的效果有所降低, 但是总体来说, 本文提出的方法是对提升检索的效果是有效的. 从表2中我们还可以看出, WebAssistance的实验效果比BM25的效果好, 但是还是没有本文提出的方法效果好, 这是因为WebAssistance将所有的医学实体都作为查询扩展词, 这样会引入噪音, 这也就可以解释在2015年和2016年数据集上效果下降的现象. 同时, 我们也可以从表中看出, 考虑实体组合比只考虑单个实体的效果要好. 注意力机制也被验证是有效的, 因为LSTM+AdA忽略了实体之间的影响.
| 表 2 实验结果 |
3.2 参数学习
| 表 3 各年份查询平均长度 |
3.3 词嵌入和字符嵌入的比较
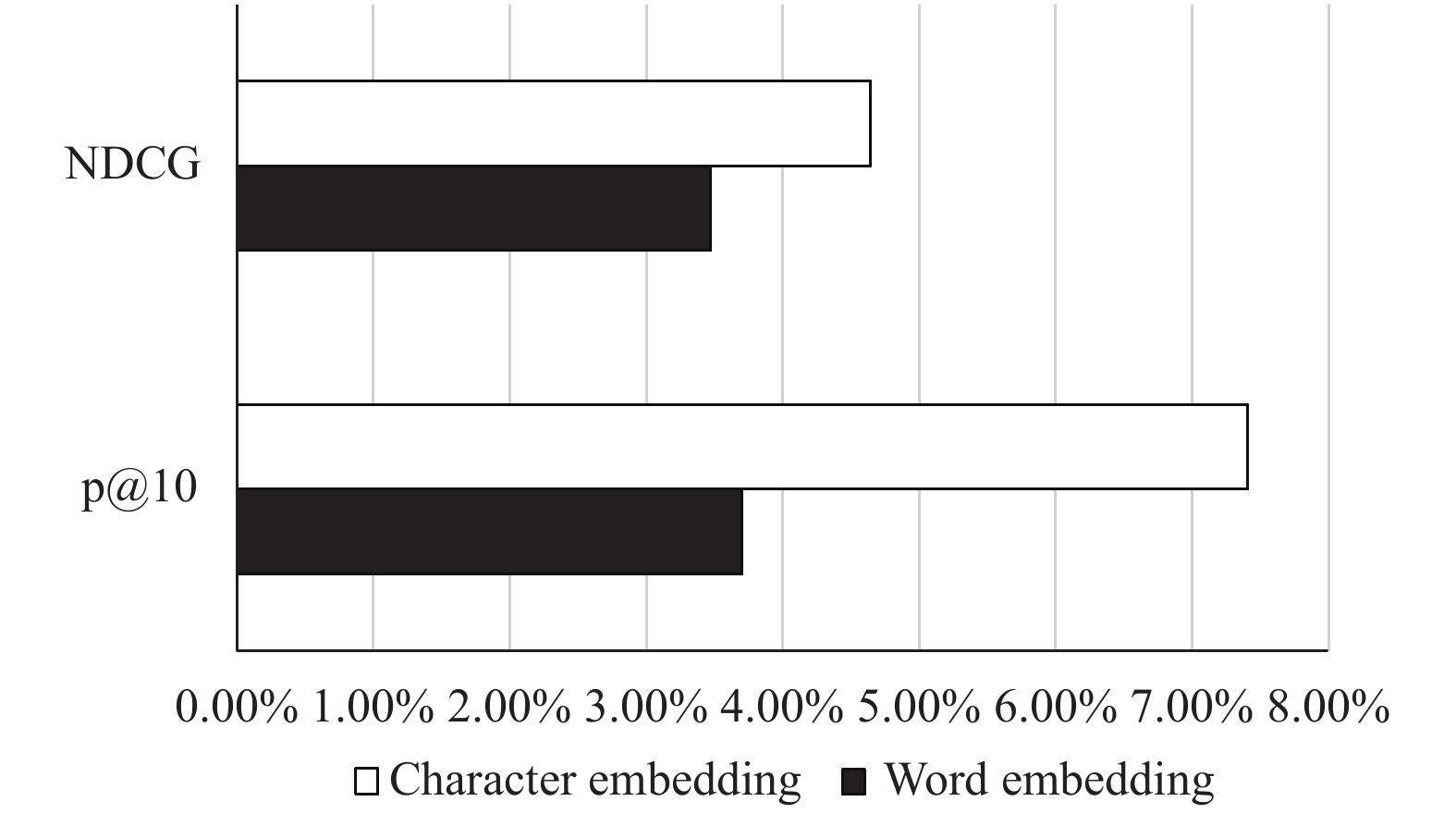
在本文中, 我们同样对词嵌入和字符嵌入对迁移学习的影响做了实验. 在这里, 使用词嵌入和使用字符嵌入的区别是在得到单词表示上. 词嵌入是一种向量训练, 首先通过预训练获得向量, 然后随着模型的训练进一步训练. 字符嵌入的方法是随机初始化字符向量, 然后随着模型的训练进一步训练出字符向量, 最后得到字符向量, 对字符向量的进一步操作才得到单词表示. 从图3, 图4和图5中我们可以发现词嵌入和字符嵌入在一定程度上都是有效的. 在2014年和2016年数据集中, 使用词嵌入的方法要比原始的方法效果好, 使用字符嵌入的效果比使用词嵌入的效果好. 在2014年中, 只使用词嵌入方法的NDCG值为0.2473, P@10为0.28, 从图6, 图7, 图8中我们可以看出和BM25方法相比, NDCG有3.47%的提升, P@10有3.70%的提升. 只使用字符嵌入方法的NDCG值为0.2501, P@10的值为0.29, 和BM25相比, NDCG有4.64%的提升, P@10有7.41%的提升. 在2016年中, 同样可以发现, 使用字符嵌入的效果比使用词嵌入的效果要好. 这表明, 当我们将从其他领域学习到的知识应用于医学领域时, 字符嵌入的性能优于字嵌入. 这个结果是因为我们只使用了医学数据进行测试, 并没有使用医学数据进行训练, 所以在测试时会出现大量未列出的单词, 这也导致了词嵌入性能不显著. 但是无论是哪个领域, 里面的单词都是由字符组成, 并且字符表示会随着模型的优化而更新, 因此我们使用基于字符嵌入能获得更好的单词表示.
4 结论与展望本文主要提出了一个基于知识的神经网络查询扩展模型以提高医疗信息检索效果. 考虑到医学文本注释的难度和成本, 我们将从其他领域学习到的知识应用于医学领域. 与以前的工作不同, 我们解决了固定

|
图 3 2014年不同嵌入效果对比 |

|
图 4 2015年不同嵌入效果对比 |

|
图 5 2016年不同嵌入效果对比 |
|
图 6 2014年不同嵌入效果对比(rate) |

|
图 7 2014年不同嵌入效果对比(rate) |

|
图 8 2014年不同嵌入效果对比(rate) |
| [1] |
Robertson S, Zaragoza H. The probabilistic relevance framework: BM25 and beyond. Foundations and Trends in Information Retrieval, 2009, 3(4): 333-389. |
| [2] |
Salton G, Buckley C. Term-weighting approaches in automatic text retrieval. Information Processing & Management, 1988, 24(5): 513-523. |
| [3] |
Robertson S E. On term selection for query expansion. Journal of Documentation, 1990, 46(4): 359-364. DOI:10.1108/eb026866 |
| [4] |
Xu JX, Croft WB. Query expansion using local and global document analysis. Proceedings of the 19th Annual International ACM SIGIR Conference on Research and Development in Information Retrieval. Zurich, Switzerland. 1996. 4–11.
|
| [5] |
Zhu DQ, Carterette B. Improving health records search using multiple query expansion collections. Proceedings of 2012 IEEE International Conference on Bioinformatics and Biomedicine. Philadelphia, PA, USA. 2012. 1–7.
|
| [6] |
赵静. 大规模汉语语义词典构建[硕士学位论文]. 哈尔滨: 哈尔滨工业大学, 2011.
|
| [7] |
Dong ZD, Dong Q. HowNet–a hybrid language and knowledge resource. Proceedings of International Conference on Natural Language Processing and Knowledge Engineering. Beijing, China. 2003. 820–824.
|
| [8] |
Miller GA, Beckwith R, Fellbaum C, et al. Introduction to WordNet: An on-line lexical database. International Journal of Lexicography, 1990, 3(4): 235-244. DOI:10.1093/ijl/3.4.235 |
| [9] |
Guo YK, Harkema H, Gaizauskas R. Sheffield university and the TREC 2004 Genomics track: Query expansion using synonymous terms. Proceedings of TREC. 2004.
|
| [10] |
Bacchin M, Melucci M. Symbol-based query expansion experiments at TREC 2005 genomics track. Proceedings of TREC. 2005.
|
| [11] |
Medical Library Association. Bulletin of the medical library association. Chicago: Medical Library Association, 1911. |
| [12] |
Hoenkamp E, Bruza P, Song DW, et al. An effective approach to verbose queries using a limited dependencies language model. Proceedings of the 2nd International Conference on the Theory of Information Retrieval: Advances in Information Retrieval Theory. Cambridge, UK. 2009. 116-127.
|
| [13] |
Bendersky M, Croft WB. Discovering key concepts in verbose queries. Proceedings of the 31st Annual International ACM SIGIR Conference on Research and Development in Information Retrieval. Singapore. 2008. 491–498.
|
| [14] |
Ferragina P, Scaiella U. Fast and accurate annotation of short texts with Wikipedia pages. IEEE Software, 2012, 29(1): 70-75. DOI:10.1109/MS.2011.122 |
| [15] |
Manning CD, Raghavan P, Schütze H. An introduction to information retrieval. Cambridge: Cambridge University Press, 2008. 233.
|
 2019, Vol. 28
2019, Vol. 28


